基于ISSR标记的钩栗遗传多样性分析
2016-12-21田艳伶李志辉
田艳伶,李志辉
(1.中南林业科技大学 林学院,湖南 长沙 410004;2.益阳市林业科学研究所,湖南 益阳 413000)
基于ISSR标记的钩栗遗传多样性分析
田艳伶1,2,李志辉1
(1.中南林业科技大学 林学院,湖南 长沙 410004;2.益阳市林业科学研究所,湖南 益阳 413000)
利用 ISSR分子标记对钩栗Castanopsis tibetanaHance 9个野生居群进行了遗传多样性分析。利用100条引物分别对111份钩栗基因组DNA进行PCR扩增,共扩增出158条清晰条带,其中多态性条带141条,多态性条带百分率(P)平均为89.01%。钩栗9个居群的Nei’s基因多样性指数、Shannon’s信息指数分别为0.332 3、0.492 0;总种群基因多样度(Ht)、各种群基因多样度分别为(Hs)0.410 8、0.334 6,表明钩栗的遗传多样性较高。9居群的基因分化系数Gst为 0.185 5,占总遗传多样性的 81.45%。分子方差分析表明,80.21%的遗传差异存在于群体内,19.79%的遗传差异存在于群体间。基因流为2.196 0。用NTSYS 软件计算出9居群111材料的DICE相似系数在0.811 6~0.921 8 之间,遗传亲缘关系较近。各居群间的遗传距离差异较大;其中,邵武与建瓯居群的遗传距离最近,仅为0.081 5; 浏阳和周宁居群的遗传距离最远,为 0.198 4。聚类分析结果表明,采自福建的邵武、建瓯、建阳、屏南和周宁居群聚在一起;恩施和桑植居群聚在一起;永顺和浏阳居群聚为一起,种群遗传变异分布模式基本上与其地理生态格局一致。研究结果表明:供试的钩栗具有较高的遗传多样性,存在着较为频繁的基因交流;基于 ISSR 标记分析能较准确地揭示出钩栗种间的遗传多样性。
钩栗;ISSR标记;遗传多样性;基因分化;聚类分析
钩栗Castanopsis tibetanaHance为壳斗科锥属树种,又名钩锥,钩栲。为常绿阔叶高大乔木,树冠浑厚,叶大荫浓,高达30 m;坚果扁圆锥形,花期4~5月,果次年8~10月成熟[1]。钩栗材质坚硬,耐水腐,为优良的建筑、车船、家具和室内装饰用材[2],兼具用材、绿化、木本粮食等多种功能。其属红锥类,且萌发力强,天然更新良好,生长速度较快,是长江以南常见的主要用材树种,也是具有重要开发前景的珍贵用材树种。
目前,关于钩栗方面的研究还比较少见,仅限于钩栗种群生态及生命表分析[4-5]、种子特性[6-7]与播种育苗[8-9]等方面,对其多样性特别是分子水平上的遗传多样性研究未见报道。且野生钩栗分布广、遗传背景复杂,现存的钩栗常散生于常绿阔叶林中,居群规模小、天然更新长期停留在幼苗阶段,居群处于衰退状态[1,3,6]。
因此,推进钩栗遗传多样性及遗传结构研究,对挖掘利用钩栗野生种质资源和开展种质创新有着重要的意义,可为钩栗珍贵用材林的营建、应用和推广提供必要的理论依据。
简单序列重复间区或微卫星引物PCR(Microsatellite-Primed PCR),也称作锚定简单序列重复(Anchored Simple Sequence Repeat)由加拿大蒙特利尔大学的Zietkiewicz[10]等于1994年提出。ISSR 分子标记技术操作简单,实验过程快速,不需要先知道模板 DNA 的序列,引物设计简单,耗资少,同时多态性高,实验结果重复性好,是物种遗传多态性分析、遗传结构及指纹图谱构建等研究的有效手段[11]。现己广泛应用于种质资源鉴定、系统发育及亲缘关系等方面[12-13],因而是研究植物遗传多样性理想的分子标记技术之一。
本研究首次采用ISSR分子标记技术对钩栗9个种群共111个样本的遗传多样性和遗传结构进行研究,揭示不同地理分布区钩栗种群的遗传关系;探讨钩栗种群的遗传分化趋势,阐明不同生境条件对种群遗传多样性的影响;构建了树状聚类图谱,通过聚类分析方法对钩栗不同居群进行了遗传关系的研究,为良种选育及野生锥栗资源的背景分析、保护及开发利用提为钩栗种质资源有效保护与合理利用以及制定钩栗良种繁育策略提供供理论参考和科学依据。
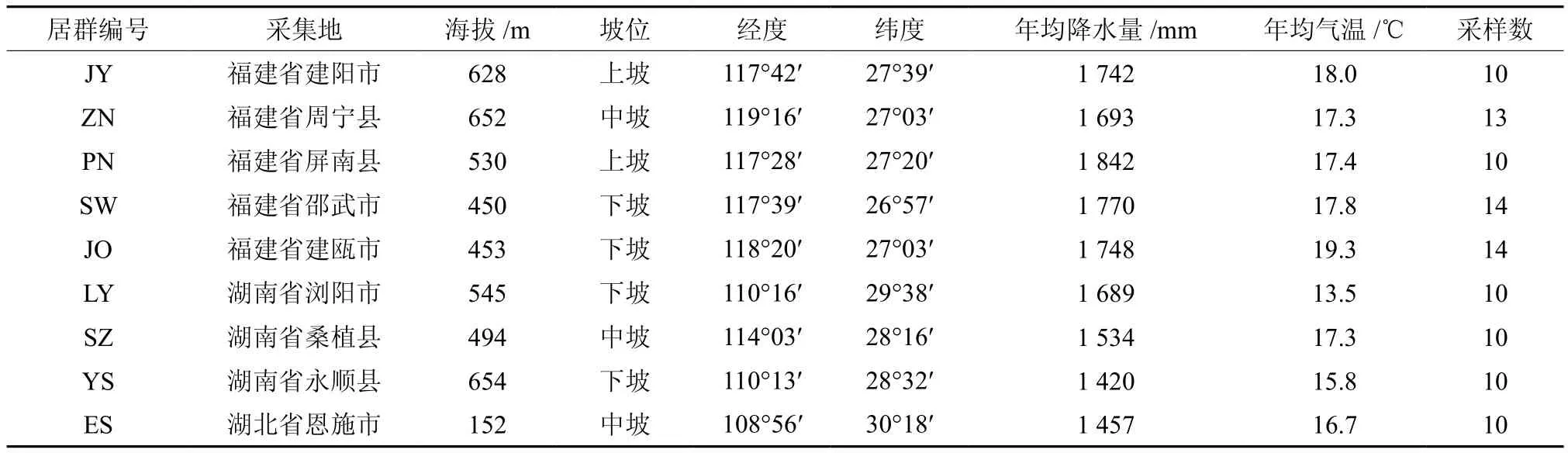
表1 供试材料来源及环境因子Table 1 Tested materials sources and environment factors
1 材料和方法
1.1 实验材料
于2013年在湖南省汨罗市玉池国有林场建立钩栗种质资源收集圃收集到分别来自湖南、湖北、福建等3省区的钩栗种质资源,对要用于种质资源遗传分析的样本,野外采取集幼嫩叶片,装于编号的锡箔纸中,再将锡箔纸包好放于硅胶中干燥封存,运回实验室后放放入-80℃度的超低温冰箱永久保存。各份种质材料的编号和采集地如(表1)所示。
所用仪器和试剂: Eppendorf Centrifuge 5415R冷冻离心机,GeneAmp PCR System 9700扩增仪,电热恒温水浴锅,WH-2微型旋蜗混合仪,DYY-12型稳压稳流电泳仪,Bio-Rad紫外凝胶成像仪;参考加拿大哥伦比亚大学(UBC) 的引物设计ISSR 引物,并由上海英骏生物技术有限公司合成;Marker由广州东盛生物科技有限公司提供;Taq DNA 聚合酶、dNTPs 和 10×PCR buffer 等购自TINGEN公司;CTAB(十六烷基三甲基溴化铵)、0.5 mol/L EDTA (pH8.0)、Tris-base、β-琉基乙醇、1xTBE溶液、琼脂糖溴化乙淀(EB)、Buffer等购自长沙隆和化玻实验用品有限公司。
1.2 实验方法
1.2.1 钩栗总DNA提取
每个钩栗样品秤取30 mg,液氮中充分碾磨,使用TIANGEN公司生产的植物基因组DNA提取试剂盒提取总DNA[14]。且将获得的总DNA产物按一定比例溶解于灭菌双蒸水中,置于-20 ℃的冰箱储存、备用。
1.2.2 ISSR分析
本实验研究通过参照锥栗ISSR遗传分析中引物筛选的方法[15],通过100个ISSR引物对来自不同居群的3个钩栗DNA模板进行PCR扩增,共筛选出10条普带清晰,信号强且,重复性好且扩增结果稳定的引物(见表2),用于钩栗遗传结构分析。
实验采用的最佳反应体系为:20 μL中含30 ng模 板 DNA,3mmol/L Mg2+,0.4 mmol/L dNTPs,0.75 U Taq DNA聚合酶,0.3 μmol/L引物,用纯水补足。钩栗ISSR-PCR反应扩增程序为:94 ℃预变性2 min;然后于94℃变性30 s、50~59.3 ℃退火30 s(根据不同引物的Tm值而设定具体退火温度)、68 ℃延伸150 s,共40个循环;最后于72 ℃延伸7 min保存[14]。用质量体积分数为0.7 %的琼脂糖凝胶检测钩栗DNA的提取浓度及其质量。
1.3 实验数据统计及分析
本实验利用Quantity One软件与人工读取条带相结合,按凝胶同一位置上DNA条带的有无进行统计,有带的记为“1”,无带的记为“0”,建立原始“01”矩阵。按照Nei等[16]的方法,利用 POPFENE Version1.32、NTSYS Version2.10 及AMOVA等软件对9个居群111个钩栗样本的遗传多样性、遗传结构、遗传距离与相似性、居群聚类结果等进行遗传分析。
2 结果与分析
2.1 ISSR-PCR 扩增结果分析
利用优化的反应体系从100 条引物中筛选出10条稳定性强、 重复性好、 多态性丰富的引物,10条引物的序列以及扩增结果见(表2)。 然后利用筛选出的10条引物对111 个供试样品的基因组 DNA 进行 PCR 扩增,PCR扩增产物经电泳得到部分指纹图谱(图1-2)。不同引物组合扩增的多态性条带数均不相同,在250~2 000 bp 内共扩增出158条清晰条带,其中多态性条带141条,多态性条带百分率(P)平均为89.01%。每个引物扩增的条带数目在13~19条之间,平均每条引物扩增出15.8条条带。其中,用引物UBC846 扩增获得的条带数最少,仅13条;用引物UBC880扩增获得的条带数最多,达19条。除引物UBC830之外,其余的 ISSR 引物扩增获得的PPB均大于85.0%,且引物UBC842扩增的PPB均达到 100.0%。每个居群的多态性条带百分率在84.81%~93.67%之间,其中周宁居群最低,永顺居群最高。由此可见,供试的9个钩栗居群具有较丰富的遗传变异和遗传多样性。
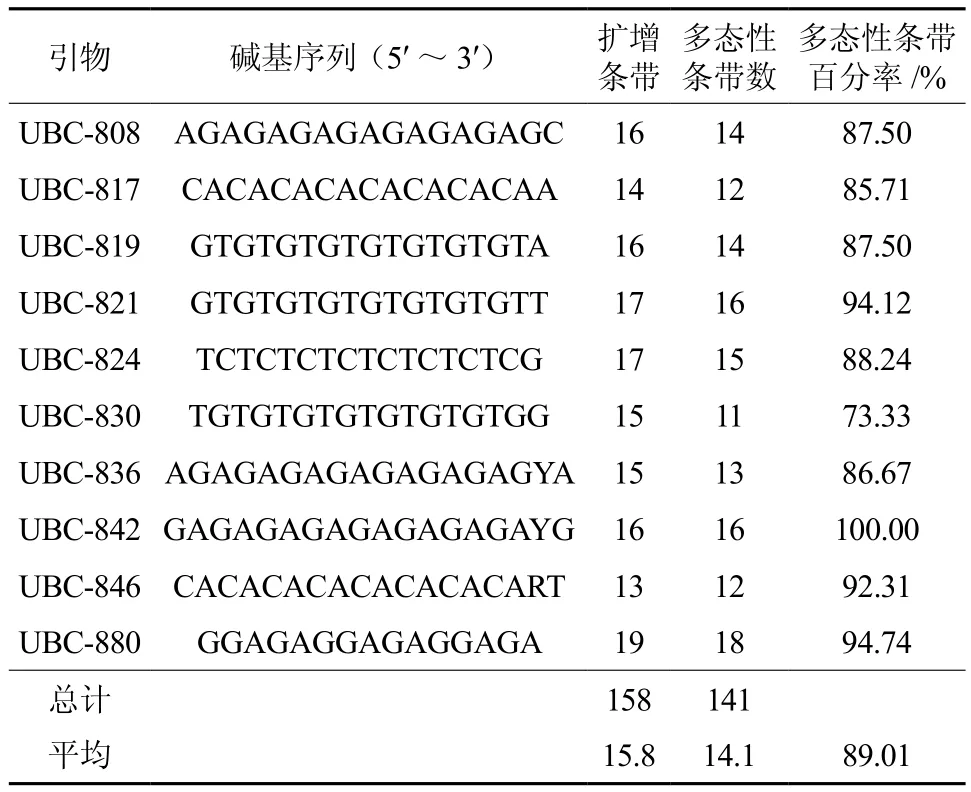
表2 用于钩栗基因组总DNA ISSR -PCR的随机引物碱基序列及扩增结果Table 2 Base sequences of random primers used for ISSRPCR ofgenomic total DNA of Castanopsis tibetana Hance and its amplified results

图1 821号引物在优化后的体系下扩增20个样品的PCR结果Fig.1 The PCR results of 20 specimens with primer 821 and optimized reaction system
图2 880号引物在优化后的体系下扩增23个样品的PCR结果Fig.2 The PCR results of 23 specimens with primer 880 and optimized reaction system
2.2 钩栗居群遗传多样性分析
应用POPGENE32遗传分析软件对得到的111个钩栗样品的“01”矩阵,按9个居群进行群体遗传多样性分析,结果见表3。从表3中可以看出各居群供试材料具有丰富的ISSR标记位点,钩栗居群间基因交流频繁,遗传多样性丰富,是钩栗种质创新的重要来源。其中,永顺与邵武分别为湖南、福建省野生钩栗居群遗传多样性最丰富的区域。
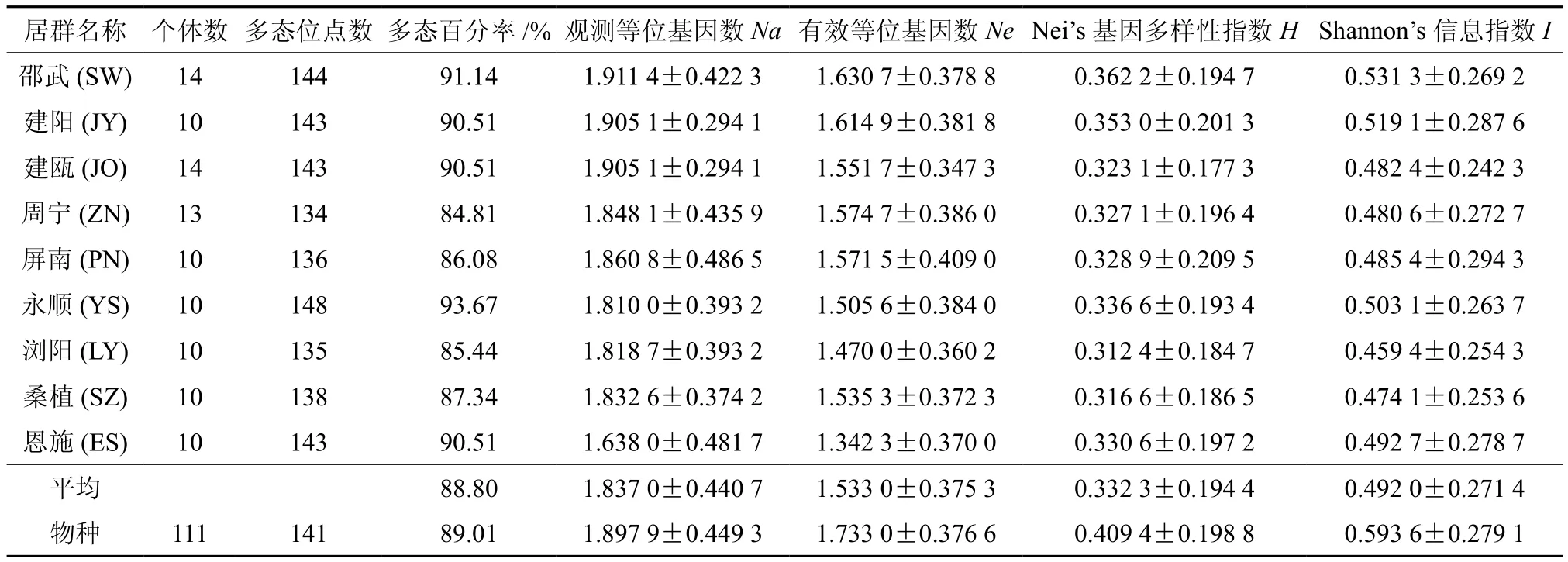
表3 基于ISSR 标记的9个钩栗居群遗传多样性分析结果Table 3 Analysis result of genetic diversity of 9 populations of Castanopsis tibetana Hance based on ISSR marker
由表3可以看出,居群观测等位基因数(Na)为1.897 9,有效等位基因数(Ne)为1.733 0; Nei基因多样性指数(H)值平均值为0.332 3,物种平均值为0.409 4,最大的是邵武(SW)居群为0.362 2,最小的是浏阳(LY)居群仅为0.316 6。9个居群的Nei基因多样性指数(H)值均低于0.4,参照前面提到的研究结果表明,参试的9个钩栗居群的遗传多样性水平较低。钩栗居群的Shannon’s信息指数(I)在0.549 4~0.531 3之间,平均值为0.492 0,物种平均值为0.593 6。从表3中还可以得出,邵武居群的Shannon’s信息指数(I)和Nei基因多样性指数(H)均高于其他六个种群,即在参试的9个钩栗居群中邵武居群的遗传多样性水平最高。
本研究进一步利用 AMOVA软件[17]对钩栗居群内和居群间的分子变异进行了分析。从表4中的结果可以看出:钩栗居群间变异占19.79%,居群内变异占80.21%,居群内和居群间的变异均极显著(p<0.001)。比较发现,AMOVA分析所得结果与Nei’s遗传多样性分析的结果基木一致。
2.3 不同种源间的遗传距离及聚类分析
根据ISSR扩增结果组成二元数据矩阵,用POPGENE 32软件计算9个钩栗居群的遗传相似系数(表5),结果表明:邵武(SW)和建瓯(JO)居群的遗传距离最近,仅为0.081 5,相似系数为0.921 8,表明亲缘关系很近。浏阳(LY)与周宁(ZN)居群的遗传距离最远,为0.198 4,相似系数为0.811 6,表明亲缘关系很远,可能与其分布地距离相对较远、生长环境差异较大有关;参试钩栗居群的遗传距离平均值为0.137 9,平均相似度为0.871 4。根据不同居群亲缘关系的远近,可为今后钩栗育种亲本选择提供理论参考。
进一步计算不同钩栗居群间的遗传距离,依据遗传距离进行UPGMA聚类分析,从而获得9个不同钩栗居群间的ISSR系统聚类图(图3)。从图11可以看出,当λ=0.88时,9个钩栗居群聚为6支,周宁、桑植、恩施、永顺和浏阳各单独聚为一支;而来自福建屏南的居群聚为一支。且屏南居群又分为两个亚支,建阳居群为1个亚支,地理距离最近的邵武和建瓯居群聚为1个亚支。从下面聚类图可以看出,不同的居群的聚类规律符合其在空间上的地理位置,福建省的邵武、建瓯、建阳、屏南和周宁居群聚在了一支;湖北的恩施居群与湖南张家界的桑植居群在地理位置上临近,故也聚为一支;湖南的永顺居群与浏阳居群则独自聚为一支。
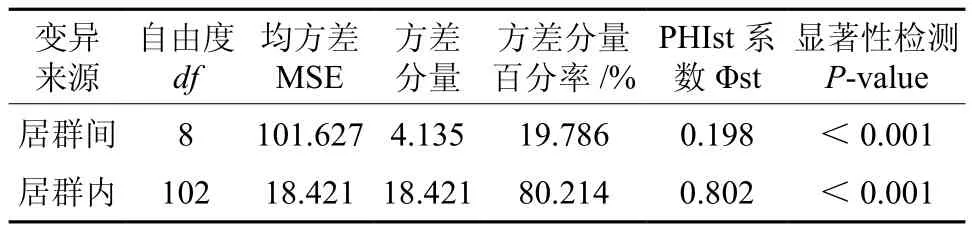
表4 钩栗居群间和居群内分子变异的AMOVA分析结果Table 4 Analysis of moleculuar variance (AMOVA)within/among Castanopsis tibetana Hance populations

表5 遗传一致性(上)和遗传距离表(下)†Table 5 Nei’s unbiased measures of genetic identity (above diagonal) and genetic distance (below diagonal)

图3 基于ISSR 标记的 5 个钩栗居群的 UPGMA 聚类图Fig.3 UPGMA dendrogram of fi ve populations of Castanopsis tibetana Hance based on ISSR marker
2.4 基因分化
通过POPGENE 软件对9个钩锥居群进行遗传分化分析(表6),得出总种群基因多样度(Ht)、各种群基因多样度(Hs)0.410 8、0.334 6,基因流为2.196 0;种群间遗传分化系数(Gst)为(Gst=(Ht-Hs)/Ht)为0.185 5,这表明遗传变异大部分存在于居群内,即居群内的遗传变异占总变异量的81.45%,而只有18.55%的遗传变异存在于居群间。由此可知,参试钩栗的遗传变异以居群内遗传变异为主。

表6 基于ISSR分析的钩栗遗传变异Table 6 Analysis of genetic variation for Castanopsis tibetana Hance by ISSR
3 讨 论
本研究中,用10个ISSR随机引物对钩栗9个居群的基因组DNA进行扩增,得到了158条DNA条带,其中多态性条带有141条,多态性条带百分率达到89.01%,根据条带的差异可以较好地将供试的9个居群区分开来。一般情况下,如果多态性条带百分率超过 50%,就可以认为该物种有较为丰富的遗传多样性[18-19]。Hamrick[20]等也指出:多数多年生木本树种平均多态位点百分数为 65%。由此可知,供试的钩栗有丰富的遗传多态性,利用ISSR分子标记技术能快速准确地检测其遗传变异,也为钩栗提供了分子标记辅助选择与评价的技术方法。总之,从反映物种遗传多样性的多个参数上,包括物种水平的 Nei’s 基因多样度(0.409 4) 和Shannon’s 多样性指数 (0.593 6)等,均表明钩栗种内的遗传多样性十分丰富,对其生存环境有很强的适应能力。其中,Nei’s 基因多样度还高于一般针阔叶树种的估算值(0.206)[21]。无论是在物种水平上,还是在居群水平上,本研究结果均反映出钩栗具有非常丰富的遗传多样性水平和较高的遗传变异度。
由于地理分布隔离的原因,钩栗在长期的自然选择和遗传变异过程中基因频率必然发生变化,某些基因位点可能消失。在多位点检测的基础上通过计算遗传距离并利用聚类分析进行种的识别,具有较高的可靠性[22]。研究结果表明,不同的居群的聚类规律符合其在空间上的地理位置,福建省的邵武、建瓯、建阳、屏南和周宁居群聚在了一支;湖北的恩施居群与湖南张家界的桑植居群在地理位置上临近,故也聚为一支;湖南的永顺居群与浏阳居群则独自聚为一支,说明两者较其他居群的遗传差异相对较大。研究表明:同种居群间的遗传距离一般为 0.03 ~0.20、相似系数一般为 0.80 ~0.97[23]。9个钩栗居群间存在着一定的遗传分化,是因为其受环境差异和地理隔离等因素的影响,然而,根据Thorpe等研究者的标准理论值来看,这9个钩栗居群仍然属于同一地理群体。
从反映物种遗传多样性的多个参数上,包括物种水平的 Nei’s 基因多样度(0.409 4) 、Shannon’s 多样性指数(0.593 6)和多态性条带百分率(89.01%) 等,均表明了钩栗种内的遗传多样性较为丰富,且Nei’s 基因多样度还高于已发表的一般针阔叶树种的估算值(0.206)[21]。
由于本研究的实验材料采样不完整,只取自湖南、湖北、福建3个省份的9个样地,故并不能完全说明钩栗的全部遗传背景。若要全面了解钩栗的遗传背景,仍需扩大钩栗种质资源的收集范围,最好对已发现的具有代表性的省份(如浙江、广西、云南等)进行样品采集,并采用多种方法对钩栗进行遗传多样性和遗传结构的研究分析,为科学地经营、管理、保护和利用钩栗种质基因资源,维持其丰富的遗传育种潜力和遗传多样性奠定基础。
[1]张宏达,任善湘.中国植物志(第22卷)[M].北京:中国科学院中国植物志编辑委员会, 1998: 33-35.
[2]洪 伟,林成来,吴承祯,等,福建建溪流域常绿阔叶防护林物种多样性的研究[J].生物多样性,1999,7(3):208-213.
[3]郑万钧.中国树木志[M].北京:中国林业出版社, 1985:2216-2217.
[4]林 敏,黄宗安.钩栗种群生命表分析[J].福建林业科技,2003, 17(2):9-13.
[5]张嘉生.钩栲群落优势植物种群竞争的研究[J].福建林业科技,2005,13(4):82-85.
[6]王佩兰,许德禄,张 斌,等.钩栗种子形态特征及主要生理指标测定分析[J].种子,2013,11(8):7-10+16.
[7]王佩兰. 不同种源钩栗种子特征及幼苗光合特性比较研究[D].长沙:中南林业科技大学,2015.
[8]陈 养.钩栗人工育苗技术研究[J].林业科技开发,2007,22(03): 89-90.
[9]李纯教.皖南山区钩栲特征特性及播种育苗技术[J].现代农业科技,2012,13(6):185-187.
[10]ZIETKIEWICZ E, RAFALSKI A, Abbuda D. Genome fingerprinting by simple sequence repeat (SSR)-anehored polymerase chain reaction amplifieation[J]. Genomies, 1994,20(2): 176-183.
[11]马克平.试论生物多样性的概念[J].生物多样性,1993,l(l):20-22.
[12]冯夏莲,何承忠,张志毅,等.植物遗传多样性研究方法概述[J].西南林学院学报,2006(1): 51-59.
[13]林乐静,林 立,祝志勇.25份槭属优良种质资源亲缘关系的ISSR分析[J].广西植物, 2015,1(9):9-14+60.
[14]田艳伶,李志辉,杨模华,等.钩栗ISSR-PCR反应体系的建立与优化[J].中南林业科技大学学报, 2015,35(8):32-37.
[15]刘国彬.锥栗自然居群及农家品种遗传多样性的ISSR分析[D].武汉:华中农业大学,2009.
[16]NEI M, LI WH. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proceedings of the National Academy of Sciences of the United States of America, 1979, 76 (10): 5269-5273.
[17]Excoffier L, Smouse P E, Quattro J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondria DNA restriction sites[J].Genetics, 1992, 131(3): 479-491.
[18]李乃伟,贺善安,束晓春,等.基与ISSR标记的南方红豆杉野生种群和迁地保护种群的遗传多样性和遗传结构分析[J].植物资源与环境学报,2011,1(8):25-30.
[19]马丽娟. 东北野生杏居群遗传多样性及遗传结构分析[D].吉林:吉林农业大学,2013.
[20]Hamrick J L, Godt M J W, Sherman-Broyles S L. Factors in fl uencing levels of genetic diversity in woody plant species[J].New For,1992, 6(9): 95-124.
[21]茹考夫斯基.ⅡM.育种的世界植物基因资源.植物育种的遗传学原理[M].北京:科学出版社,1974.
[22]陈大霞,李隆云,钱 敏,等.黄连药材DNA提取及RAPD反应体系的优化[J].中草药,2006,37(8): 1233-1237.
[23]Thorpe J P. The molecular clock hypothesis: biochemical evolution, genetic differentiation and systematics [J]. Annual Review of Ecology and Systematics, 1982, 13(1): 139-168.
Genetic diversity analysis of Castanopsis tibetana Hance based on ISSR marker
TIAN Yan-ling1,2, LI Zhi-hui1
(1. School of Forestry, Central South University of Forestry and Technology, Changsha 410004, Hunan, China;2. Yiyang Forestry Instritue, Yiyang 413000, Hunan, China)
The genetic diversity of nineCastanopsis tibetanaHance populations were analyzed using inter-simple sequence repeat(ISSR) markers. 10 ISSR primers were screened out from l00 primers from UBC for amplifying 111 genomic DNA ofCastanopsis tibetanausing ISSR-PCR. 158 intensive bands were amplified, among which there were 141 polymorphic bands. The polymorphic loci percentage (P) was 89.01%. The Nei’s genetic diversity index, Shannon’s information, number of total genetic diversity index,genetic diversity index are 0.3323, 0.4920, 0.4108, 0.3346. They showed that genetic diversity ofCastanopsis tibetanawas high. Genetic differentiation coef fi cient (Gst) ofCastanopsis tibetanawere 0.1855, it indicated that the variation within population account 81.45%.The results by AMOVA analysis indicated that the variation within population account for 80.21% and variation among populations accounted for 19.79% . As analyzed by NTSYS, the genetic similarity ranged from 0.811 6 to 0.921 8 based on DICE. The genetic relationships among the 111 materials were relatively close. Genetic distance among all populations differs obviously, in which, that between populations of Shaowu and Jianou is the nearest(only 0.0815) while that between populations of Liuyang and Zhouning is the farthest (0.1984). Cluster analysis showed that Shaowu, Jianou, Jianyang, Pingnan and Zhouning populations clustered together. Enshi and Sangzhi population grouped together. Yongshun grouped together with Liuyang population. It was a further indicator that genetic variation of populations and its geographic pattern was basic consistency. It is suggested that populations ofCastanopsis tibetanaHance tested possess higher genetic diversity,and there are frequent gene exchange. The genetic diversity ofCastanopsis tibetanaHance can be accurately revealed by means of ISSR marker analysis.
Castanopsis tibetanaHance; ISSR marker; genetic diversity; gene differentiation; cluster analysis
S718.46
A
1673-923X(2016)12-0129-06
10.14067/j.cnki.1673-923x.2016.12.022
http: //qks.csuft.edu.cn
2014-10-22
国家林业行业公益性项目“珍贵树种钩栗良种选育及栽培关键技术研究”(201204405)
田艳伶,硕士研究生
李志辉,教授,博士生导师,博士;E-mail:lzh1957@126.com
田艳伶,李志辉. 基于ISSR标记的钩栗遗传多样性分析[J].中南林业科技大学学报,2016, 36(12): 129-134, 139.
[本文编校:文凤鸣]
