利用多重数字PCR检测KRAS相关突变
2016-12-20邬升超王蓓丽张春燕楼文晖潘柏申
邬升超 黄 斐 赵 瀛 虞 倩 韩 序 王蓓丽 张春燕 郭 玮 楼文晖 潘柏申△
(1复旦大学附属中山医院检验科,2普外科 上海 200032)
利用多重数字PCR检测KRAS相关突变
邬升超1黄 斐1赵 瀛1虞 倩1韩 序2王蓓丽1张春燕1郭 玮1楼文晖2潘柏申1△
(1复旦大学附属中山医院检验科,2普外科 上海 200032)
目的 建立多重数字PCR体系检测血浆细胞游离DNA(cell-free DNA,cfDNA)中KRAS第2外显子12、13密码子的7种突变。方法 构建7种KRAS突变型质粒用以建立多重数字PCR检测体系,以灵敏度、特异度和动态范围为主要参数评估体系的性能。利用该体系检测15例手术可切除的胰腺导管腺癌(pancreatic ductal adenocarcinoma,PDAC)患者血浆cfDNA中KRAS第2外显子12、13密码子突变情况,并与ARMs的检测结果进行比对。结果 自建的多重数字PCR在0.01%~10%的动态范围内线性良好。两个检测体系的灵敏度分别可达到0.025%和0.043%。15名PDAC患者组织中KRAS突变的阳性率达到100%。数字PCR检测血浆cfDNA所得的结果与组织结果的匹配率达到40%,ARMs检测组织和血浆cfDNA结果的匹配率为20%。15例样本中使用多重数字PCR检测到血浆cfDNA的最低丰度达到0.09%。结论 多重数字PCR具备高灵敏度和特异性,可用于准确定量外周血cfDNA中KRAS相关突变的水平。
多重数字PCR; 血浆游离DNA; KRAS
RAS基因突变被认为与人类肿瘤发生密切相关。RAS家族中的KRAS基因12、13密码子的突变在结直肠癌和胰腺癌中均有较高的发现率,被认为与肿瘤的发生、复发及患者预后直接相关[1-2]。胰腺癌尤其是胰腺导管腺癌(pancreatic ductal adenocarcinoma,PDAC)患者中约有75%~95%携带KRAS突变基因,其与CDKN2A、SMAD4和TP53在基因水平的改变被认为与胰腺癌的发生和发展直接相关[3-4],监测KRAS在体内突变情况的变化有助于理解基因改变与病情发展之间的联系。
外周血细胞游离DNA(cell-free DNA,cfDNA)是来自正常细胞和肿瘤细胞死亡后释放的核酸片段的总称。由于其在体内的半衰期较短且细胞释放的核酸会进入外周血,因此cfDNA较之于组织标本更能实时全面地反映患者体内的肿瘤情况[5-6]。另一方面,患者对非侵入性样本采集方式的接受度更高,cfDNA具有良好的临床应用前景。但是如何准确定量cfDNA一直是个难题:高度片段化、极低的目的片段浓度及复杂的背景噪音干扰,使得传统基于实时荧光定量PCR建立的方法在灵敏度、特异性及重复性上均无法满足要求[7-8]。
液滴式数字PCR作为新一代数字PCR检测系统,得益于无限稀释的概念,具备绝对定量、高灵敏度、高特异性、重复性好以及抗干扰能力强等优点[9]。在肿瘤相关领域,数字PCR已经被用于肺癌患者的液态活检,以辅助指导靶向药物的使用[10];亦可用于结直肠癌患者,高灵敏度的检测系统能够从外周血中发现耐药突变,且外周血与组织的一致性高[11-12]。较之肺癌和结直肠癌,数字PCR在其他癌症尤其是胰腺癌中的相关报道较少。本文旨在自建多重数字PCR体系检测KRAS基因12、13密码子的7个突变,并对该系统进行初步的性能验证。通过对15例PDAC患者的标本进行检测,初步评价该系统的检测能力。
材 料 和 方 法
患者基本信息 选取来自复旦大学附属中山医院普外科病房的15名PDAC患者以及8名来自体检中心的健康人,其中健康人经过影像学确认不存在累及器官的病变,且血清结果显示无异常。15名PDAC患者经影像学发现胰腺占位,考虑为胰腺癌,行胰腺部分或全部切除术,术前获取6 mL全血样本,术中获取组织用以制备4%甲醛溶液固定石蜡包埋切片。所有23名受试者均签署知情通知书。
组织DNA与血浆DNA抽提 从我院病理科获得符合要求的4%甲醛溶液固定的石蜡包埋样本(formalin-fixed and parrffin-embedded,FFPE),每例患者6张,使用QIAamp DNA Mini Kit(德国Qiagen公司)抽提组织DNA,-20 ℃保存备用。术前分别抽取10 mL外周血全血(EDTA抗凝管)和6 mL血清,1 900×g离心后用于CA199和CEA水平的测定。吸取离心后的血浆,16 000×g再次离心,吸取上清,-80 ℃冻存备用。抽提前再次以16 000×g离心,取上清液,使用QIAmp Circulating Nucleic Acid Kit(德国Qiagen公司)抽提cfDNA。使用Nanodrop (美国Thermo Fisher公司)初步定量抽提后的cfDNA浓度,-20 ℃保存。
质粒构建 质粒用于双重和多重数字PCR体系的建立和性能验证。以包含KRAS野生型片段的重组质粒(货号:RC201958,美国Origene公司)为模板定点诱导常见的7种KRAS突变型(G12D、G12V、G12s、G12V、G12A、G12R和G13D)。为确保数字PCR生成的液滴状态稳定,构建好的质粒需使用对应的限制性内切酶切割,使最终用于扩增的包含目的片段的模板长度小于3 000 bp,同时保证质粒处于线性状态。酶切后,使用天根DNA纯化试剂盒(目录号:DP214)纯化质粒,-20 ℃保存备用。为避免质粒污染操作环境及质粒间的交叉污染,实验操作过程中保证仅有一个样本管处于打开的状态,同时实验结束后打开紫外灯照射操作室。
探针和引物的设计 本实验使用RainDrop数字PCR系统,分为收集装置(Source)和读取装置(Sense),分别用于完成液滴的形成和信号采集与分析两个步骤。按照仪器要求,需使用VIC和FAM两种荧光对Taqman探针5’端进行标记,使用MGB淬灭基团对Taqman探针3’端进行标记。为满足多重数字PCR设计时的需要,某种特定的突变形式需要用两条不同荧光标记的Taqman探针,且通过调整探针浓度使得不同突变类型的液滴得以在二维平面中区分[13],具体序列如表1、2所示。

表1 多重数字PCR多重体系1的探针序列和浓度

表2 多重数字PCR多重体系2的探针序列和浓度
多重数字PCR体系构建 多重数字PCR的体系中包含野生型和多种突变型探针。将G12D、G12R、G12V及野生型构成四重数字PCR,命名为体系1(multiplex-1);将G13D、G12A、G12C、G12S以及野生型构成五重数字PCR,命名为体系2(multiplex-2)。体系1和2的体积均为40 μL,包括20 μL Genotyping Master Mix(美国Life Technologies公司)、0.5 μmol/L正向引物(5’-CTGAAAATGACTGAATATAAACTTGTGG-3’)和反向引物(5’-TAGCTGTATCGTCAAGGCAC-TC-3’)、野生型探针和不同类型的突变型探针(表1)、1.6 μL液滴稳定剂(美国RainDance Technologies公司)和不低于50 ng的模板量,剩余用ddH2O补足。整个体系配制完成后,将混合液混匀,瞬时离心,转移至芯片上对应的加样孔中,使用Source将40 μL混合液体制备成10-12级别的均匀液滴。处理完毕的样本使用ABI Veriti96 PCR进行扩增(美国Applied Bio-systems公司),循环条件如下:95 ℃预变性10 min,95 ℃变性15 s、64 ℃退火15 s、60 ℃延伸45 s,共45个循环,98 ℃再延伸10 min后,12 ℃恒温备用。最后将扩增好的液滴转移至Sense中进行读取。
数字PCR数据分析 数据分析使用Raindrop Analyst 10.0.7r2(美国RainDance Technologies公司)完成,具体步骤:(1) 杂音的排除。杂音的存在会严重干扰最后计数的结果,导致假阳性的发生。利用软件中自带的功能“EVENT&对应通道”来判断该坐标轴中散点的性质:如果散点聚成一团出现,则提示为杂音;如果为均匀分布,则提示为阳性液滴,计数有效。(2) 荧光补偿。每个液滴均不可避免存在荧光交叉的情况,具体表现为VIC通道中可以检测到部分FAM荧光信号,反之也是如此。荧光补偿仅能改变“簇”的位置,不改变“簇”的相对位置和其中所含散点的数量。(3) 设门和定值。在多重体系中加入对应质粒获得的散点分布图可用于设定“门”,“门”是一个标准化的概念,其大小和位置是固定的,“门”所包含的散点的集合即为“簇”,散点的个数对应为液滴数,减去空白检测限(limit of blank,LOB)即为目的片段的拷贝数。(4) 结果解读。结合操作者对整个图形的定性分析(噪音点的分布情况,不同突变型之间的距离、总共形成液滴的个数),将结果分为“阴性”、“阳性”和“不置可否”3种情况。对于无法确认的样本使用自建的双重数字PCR进行检测,或增加一倍的模板量后再使用多重数字PCR进行测试,以此为最终可信的结果。
多重数字PCRLOB建立与验证 使用质粒作为样本建立初始LOB-A。利用体系1和2分别对4例KRAS野生型质粒标本(每个体系加入8 μL样本)进行LOB测试,所得结果取均值,根据泊松分布公式计算得到每个基因型对应的LOB-A。再以健康人的混合血浆cfDNA为标本,对LOB-A结果进行验证。同样在每个体系中加入8 μL混合cfDNA,重复8次,计算均值并根据泊松分布公式得到结果LOB-B,将其与LOB-A进行比较。
多重数字PCR动态检测范围测试 使用双重数字PCR定量检测KRAS突变型和野生型的拷贝数,将突变型和野生型质粒(copies/μL)分别按照0.01%、0.1%、1%和10%进行稀释,获得4个浓度梯度的混合样本,供线性验证使用。使用多重数字PCR检测上述样本,以理论拷贝数和实际拷贝数之间的偏移结合复相关系数R2评价多重数字PCR的线性性能。
多重数字PCR的灵敏度测试 两个多重数字PCR体系中分别加入KRAS野生型质粒(约50 000 copy/μL)和任一种突变型质粒(约20 copy/μL),充分混匀后(使用双重数字PCR确认浓度范围)测定混合样本中突变型和野生型的拷贝数,并以“突变型/(突变型+野生型)”的比值MUT/(WT+MUT) %来描述多重数字PCR的灵敏度。
ARMs方法检测组织和外周血KRAS突变 选用商品化试剂盒AmoyDx®KRAS突变检测试剂盒(厦门艾德生物医药科技有限公司)检测抽提后的组织DNA和血浆cfDNA中KRAS基因的突变情况。
结 果
入组患者的基本信息 15例PDAC患者中男性11例,女性4例,平均年龄为(56.21±4.52)岁。根据肿瘤位置不同进行分类:7例在胰头,3例在胰颈,5名在胰尾。根据TNM分期进行分类:7例为ⅠA,8例为ⅡB。血清CA-199的中位水平为141.4 U/mL(0.7~2 010.0 U/mL),血清CEA的中位水平为4.8 U/mL(1.5~54.7 U/mL)。
多重数字PCR体系的建立 通过调整探针的浓度和类型,获得多重数字PCR的两个检测体系(图1)。在Multiplex-1体系中,FAM标记的包含G12D和G12V突变型的液滴分布在靠近横坐标轴的位置,且G12D的荧光强度高于G12V;VIC标记的包含野生型的液滴则分布在靠近纵坐标轴的位置;FAM和VIC标记的包含G12R突变型的液滴位于坐标平面45°角的位置。在体系2中,FAM标记的包含G12C和G12S突变型的液滴位于靠近横坐标轴的位置,且G12S的荧光强度高于G12C;VIC标记的包含野生型和G13D突变型的液滴位于靠近纵坐标轴的位置,FAM和VIC标记的包含G12A突变型的液滴位于坐标平面45°角的位置。所有不包含目的扩增模板的液滴未能进行有效扩增,均位于坐标平面的左下侧。
多重数字PCR LOB的建立 实验组中两个体系分别检测4例野生型质粒标本,拷贝数的取值范围为11 000~14 000 copy/μL,结果取均值,根据泊松分布公式计算得到每个基因型的LOB-A分别为:G12D(9)、G12R(5)、G12V(4)、G13D(8)、G12A(5)、G12C(4)和G12S(7)。在验证组中,两个体系分别检测来自于健康人的混合血浆cfDNA(拷贝数约为180 copy/μL),所得的LOB-B结果分别为:G12D(7)、G12R(4)、G12V(3)、G13D(7)、G12A(5)、G12C(3)和G12S(6)。每个体系使用不同数量级的样本测定LOB,最终得到的结果近似(图2)。
多重数字PCR线性验证 检测MUT/(WT+MUT)%为0.01%、0.1%、1%和10%的样本(图2)。将4个浓度的结果拟合直线,所得的复相关系数R2结果如下:G12V(0.91)、G12D(0.99)、G12R(0.91)、G12A(0.9)、G12S(0.99)、G12C(0.94)、G13D(0.99),提示该多重数字PCR检测系统具有良好的动态检测范围。
多重数字PCR的灵敏度测试 通过对自制的混合样本进行检测,在体系1的47 672个KRAS野生型拷贝中发现21个G12D拷贝,在体系2的48 972个KRAS野生型拷贝中发现25个G12C拷贝,分别减去LOB后实际混合样本中突变型的丰度分别为0.025%和0.043%,即在体系1中每3 972个野生型拷贝中可以检测到1个G12D突变型拷贝,在体系2中每2 332个野生型拷贝中可以检测到1个G12C突变型拷贝(图3)。
LOB-A:LOB calculated according to plasmid; LOB-B:LOB calculated according to cfDNA from pooled plasma cfDNA.
图2 多重数字PCR体系LOB的建立
Fig 2 LOB of multiplex digital PCR
PDAC患者组织和血浆样本的KRAS突变情况使用ARMs方法检测FFPE样本中KRAS突变情况,同时在自建的多重数字PCR平台上检测患者血浆DNA中KRAS的突变情况(表3)。在15例PDAC患者中,组织KRAS突变的阳性率为100%,ARMs检测血浆cfDNA的阳性率为20%,数字PCR的检测结果则为53%。6例患者(40%)的血浆cfDNA结果与组织完全匹配,8例患者未在外周血中发现KRAS相关突变。患者7和患者12的组织结果与血浆cfDNA结果有差异。使用多重数字PCR在患者7的外周血中检测出低丰度的G12S突变型,而ARMs方法无论在组织还是cfDNA中均未检测出该突变。患者12的组织中存在G13D突变,但是在血浆中两种方法均未发现该突变,且ARMs未在cfDNA中检测出低丰度的G12D突变。
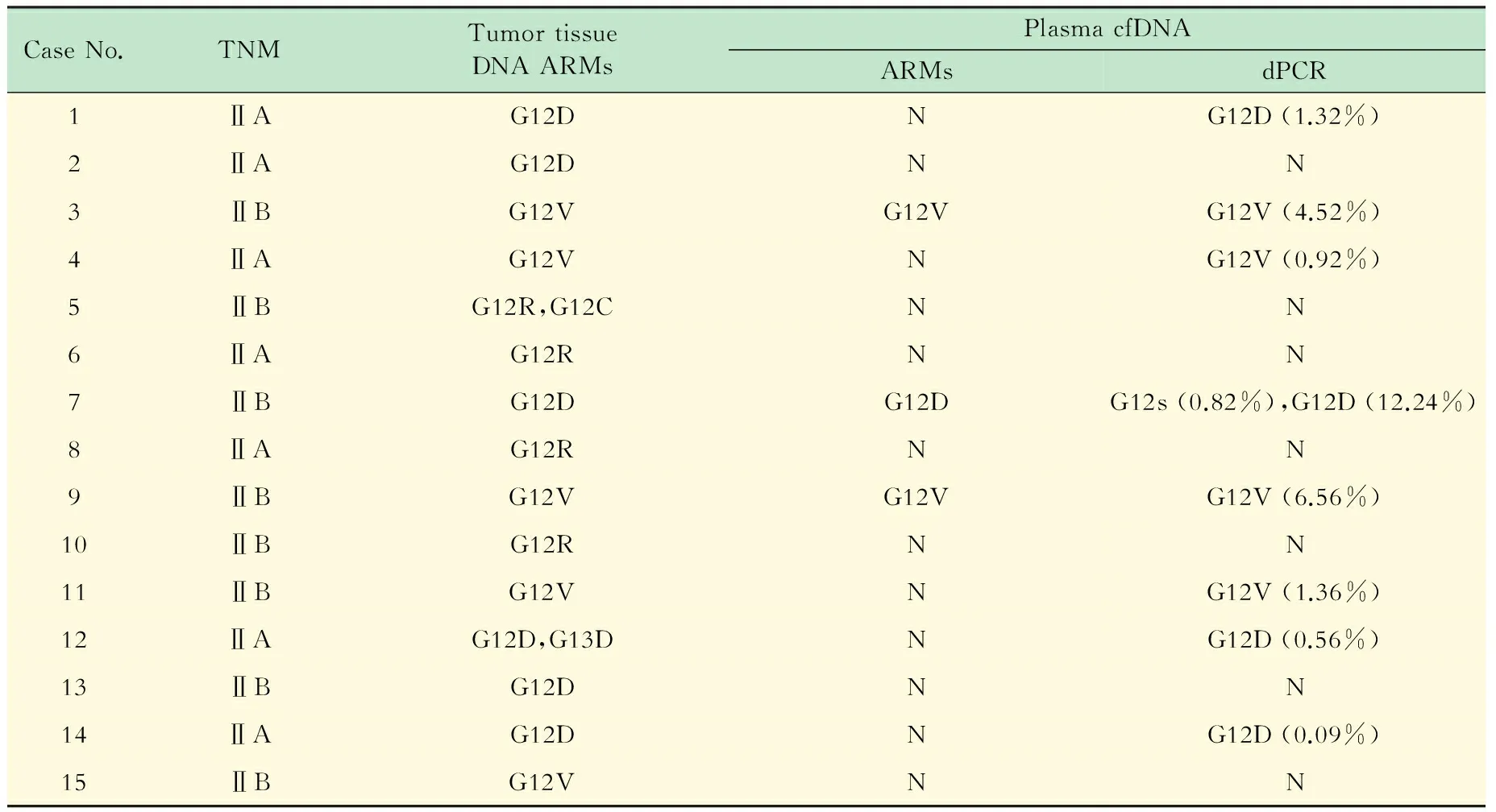
CaseNo.TNMTumortissueDNAARMsPlasmacfDNAARMsdPCR1ⅡAG12DNG12D(1.32%)2ⅡAG12DNN3ⅡBG12VG12VG12V(4.52%)4ⅡAG12VNG12V(0.92%)5ⅡBG12R,G12CNN6ⅡAG12RNN7ⅡBG12DG12DG12s(0.82%),G12D(12.24%)8ⅡAG12RNN9ⅡBG12VG12VG12V(6.56%)10ⅡBG12RNN11ⅡBG12VNG12V(1.36%)12ⅡAG12D,G13DNG12D(0.56%)13ⅡBG12DNN14ⅡAG12DNG12D(0.09%)15ⅡBG12VNN
N:Non-amplified.
讨 论
本实验使用的液滴式数字PCR系统在2个反应孔中实现了对外周血中KRAS基因第2外显子12、13密码子共7个突变位点进行绝对定量分析的目的。经过初步的性能验证结合实际标本的检测,我们认为多重数字PCR具备对多个突变位点在单孔中进行同时分析的能力,特异性、敏感度和重复性均可满足临床需求。
多重数字PCR在双重PCR的基础上通过改变探针的类型和浓度以达到在二维坐标上区分不同基因型的目的。由于数字PCR具备无限稀释分液的能力,确保了单个液滴中仅包含一条模板及反应体系。在单个反应液滴中,如果探针与模板匹配,随着PCR反应循环数的增加可使液滴发出可检测到的荧光,且该荧光强度与探针的浓度成正比。通过FAM和VIC两种荧光标记同一种基因型,可使得液滴具备不同波段的荧光信号,此类液滴通常位于二维坐标轴的右上方(如体系1中的G12R和体系2中的G12A),具体位置随着探针浓度的变化而改变。因此,按照此理论对探针的浓度和标记物类型进行调整,最高可建立十重数字PCR的检测体系。
LOB的确立依赖于大量以野生型为模板进行的空白测试结果的累积。通常可供选择的野生型模板类型包括碎片化的质粒或细胞系、正常人cfDNA以及合成短序列等。实验结果显示,7个突变型均存在一定数量的“假液滴”作为杂音存在于检测体系中。在两个反应体系中发现G12D和G13D的LOB均显著高于所在体系中其他突变类型,可能与加入探针的终浓度相关(G12D∶0.2 μmol/L,G13D:0.25 μmol/L,G12S:0.2 μmol/L)。过高的探针浓度会提高与野生型模板错配的概率,最终表现为LOB升高。相对而言,由于双重PCR仅需添加两种类型的探针,此现象并不常见。本研究对LOB实验和验证的结果提示,无论是样本的浓度还是类型均不会明显改变LOB值,该结论在Valerie等[12]的研究中也被证实。因此,后续类似项目可考虑使用更易获得、制备方便且稳定的质粒作为质控品。
通过多重数字PCR检测15例PDAC患者cfDNA与ARMs方法检测组织标本的结果比对发现,数字PCR在检测血浆cfDNA时优于ARMs方法。以数字PCR的检测结果作为参考,ARMs检测的cfDNA阳性患者的最低检出丰度为0.92%。与之相比,数字PCR的最低检出丰度达到0.09%。以组织结果为金标准,在数字PCR检测出的cfDNA阳性标本中有3例的ARMs检测结果为阴性。因此认为,目前ARMs方法的灵敏度无法完全满足血浆cfDNA的检测需求。
个体间对核酸清除能力的差异、肿瘤负荷、肿瘤分期、肿瘤类型均会影响cfDNA在外周血能够被检出的水平。在本研究的15例Ⅱ期的PDAC患者的cfDNA中,KRAS突变的检出率为53%。患者7出现外周血和组织结果不匹配的情况,通过数字PCR检测出组织中未发现的KRAS G12S突变。对于上述情况可能的解释是单一组织样本不能反映整个肿瘤组织的突变状态,肿瘤亚克隆被认为广泛存在于肿瘤组织中。“液体活检”的优势之一便是当游离核酸释放入血后获取的血液样本囊括了来自于肿瘤组织的大部分信息,这一观点在近期的一些研究中也有类似报道[15-16]。患者12的外周血中并未能检测出组织中发现的G13D突变,经过复检后确认组织和外周血的结果。为确认组织中G12D和G12D的丰度,使用数字PCR检测该样本发现G12D和G13D的丰度分别为12.1%和1.21%。G13D较低的丰度提示肿瘤灶内该类型的突变要远低于G12D,游离到外周血的cfDNA可能因被降解而无法被检测到。因此,外周血cfDNA的结果也并不能够反映肿瘤灶的全部信息。
数字PCR系统具有高灵敏度和特异性,其检测能力已得到广泛认可,同时参照2013年数字PCR的MIQE导则也可逐步规范整个体系的操作步骤[14]。但是,在实际使用中亟需解决以下2个问题:(1) 缺乏对测试结果的标准化解读模式(更多依赖于个人经验);(2) 目前的检测结果与具体病例之间很难建立起有效联系。针对前者可以通过累积大量数据以提升整个系统的检测性能(如LOB、最低检测限、临床决定水平等)。针对后者则需要将“液体活检”理论和数字PCR技术结合,探索其在肿瘤早期诊断、治疗评估、复发监测等领域的应用前景。
[1] RYAN DP,HONG TS,BARDEESY N.Pancreatic adenocarcinoma[J].NEnglJMed, 2014,371(11):1039-1049.
[2] SPINDLER KL,PALLISGAARD N,VOGELIUS I,etal.Quantitative cell-free DNA,kras,and braf mutations in plasma from patients with metastatic colorectal cancer during treatment with cetuximab and irinotecan[J].ClinCancerRes,2012,18(4):1177-1185.
[3] JONES S,ZhHANG X,PARSONS DW,etal.Core signaling pathways in human pancreatic cancers revealed by global genomic analyses[J].Science, 2008,321(5897):1801-1806.
[4] YACHIDA S,WHITE CM,NAITO Y,etal.Clinical significance of the genetic landscape of pancreatic cancer and implications for identification of potential long-term survivors[J].ClinCancerRes, 2012,18(22):6339-6347.
[5] BETTEGOWDA C,SAUSEN M,LEARY RJ,etal.Detection of circulating tumor DNA in early- and late-stage human malignancies[J].SciTranslMed,2014,6(224):224ra224.
[6] SCHWARZENBACH H,HOON DS,PANTEL K.Cell-free nucleic acids as biomarkers in cancer patients[J].NatRevCancer,2011(6),11:426-437.
[7] PEKIN D,SKHIRI Y,BARET JC,etal.Quantitative and sensitive detection of rare mutations using droplet-based microfluidics[J].LabChip,2011,11(13):2156-2166.
[8] CROWLEY E,DI NICOLANTONIO F,LOUPAKIS F,etal.Liquid biopsy:Monitoring cancer-genetics in the blood[J].NatRevClinOncol,2013,10(8):472-484.
[9] HUGGETT JF,COWEN S,FOY CA.Considerations for digital pcr as an accurate molecular diagnostic tool[J].ClinChem,2015,61(1):79-88.
[10] WANG Z,CHEN R,WANG S,etal.Quantification and dynamic monitoring of EGFR T790M in plasma cell-free DNA by digital pcr for prognosis of EGFR-TKI treatment in advanced NSCLC[J].PloSOne,2014,9(11):e110780.
[11] TIE J,KINDE I,WANG Y,etal.Circulating tumor DNA as an early marker of therapeutic response in patients with metastatic colorectal cancer[J].AnnOncol,2015,26(8):1715-1722.
[12] ITALY V,PEKIN D,BENHAIM L,etal.Multiplex picodroplet digital PCR to detect kras mutations in circulating DNA from the plasma of colorectal cancer patients[J].ClinChem,2013(12),59:1722-1731.
[13] WZHONG Q,BHATTACHARYA S,KOTSOPOULOS S,etal.Multiplex digital PCR:Breaking the one target per color barrier of quantitative PCR[J].LabChip,2011,11(13):2167-2174.
[14] HUGGETT JF,FOY CA,BENES V,etal.The digital miqe guidelines:Minimum information for publication of quantitative digital PCR experiments[J].ClinChem, 2013,59(6):892-902.
[15] REINERT T,SCHOLER LV,THOMSEN R,etal.Analysis of circulating tumour DNA to monitor disease burden following colorectal cancer surgery[J].Gut,2015,65(4):625-634.
[16] RUSSO M,SIRAVEGNA G,BLASZKOWSKYl LS,etal.Tumor heterogeneity and lesion-specific response to targeted therapy in colorectal cancer[J].CancerDiscove, 2016,6(2):147-153.
KRAS related mutations detected by multiplex digital PCR
WU Sheng-chao1, HUANG Fei1, ZHAO Ying1, YU Qian1, HAN Xu2, WANG Bei-li1, ZHANG Chun-yan1, GUO Wei1, LOU Wen-hui2, PAN Bai-shen1△
(1DepartmentofClinicalLaboratory,2DepartmentofSurgery,ZhongshanHospital,FudanUniversity,Shanghai200032,China)
Objective To establish amultiplex digital PCR panel detecting 7 most common mutations in codon 12,13 of KRAS exon 2 from plasma cell-free DNA (cfDNA). Methods Plasmids prepared were applied to the establishment of multiplex digital PCR panels.We regarded sensitivity,specificity and dynamic range as main parameters to evaluate the performance.Plasma cfDNA and tissue sample from 15 resectable patients of pancreatic ductal adenocarcinoma (PDAC) were tested with dPCR and ARMs respectively and consistency between these two methods was also evaluated. Results Our self-built multiplex dPCR showed a good linearity performance in the dynamic range of 0.01%-10%.The sensitivity of two panels reached 0.025% and 0.043%,respectively.Among 15 PDAC patients,the consistency between plasma cfDNA given by digital PCR and tissue samples was 40%,while the result was 20% when we used ARMs to detect plasma cfDNA.The lowest abundance of KRAS mutations in plasma cfDNA among 15 PDAC patients was 0.09% according to digital PCR. Conclusions Multiplex digital PCR system shows high sensitivity and specificity in detecting and quantifying mutations of KRAS simultaneously in cfDNA.
multiplex digital PCR; plamsa cell-free DNA; KRAS
国家自然科学基金面上项目(81572064);上海市卫生计生系统重要薄弱学科建设项目(2015ZB0201)
R446.11+9
A
10.3969/j.issn.1672-8467.2016.06.011
2016-01-11;编辑:段佳)
△Corresponding author E-mail:pan.baishen@zs-hospital.sh.cn
*This work was supported by the General Program of National Natural Science Foundation of China (81572064) and the Key Developing Disciplines of Shanghai Municipal Commission of Health and Family Planning (2015ZB0201).
