奶牛乳房炎重要候选疫苗蛋白的抗原表位分析及三联重组表位疫苗的氨基酸序列设计
2016-11-21孔智翔殷书平2殴莎莎刘成艳同韩虎吴三桥陈春琳
刘 祥,孔智翔,殷书平2,殴莎莎,刘成艳,同韩虎,吴三桥,陈 琛,陈春琳
(1.陕西理工大学中德天然产物研究与开发利用联合研究所,陕西 汉中723001;2.陕西医药控股集团生物制品有限公司,陕西 汉中723000)
奶牛乳房炎重要候选疫苗蛋白的抗原表位分析及三联重组表位疫苗的氨基酸序列设计
刘 祥1,孔智翔1,殷书平2,殴莎莎1,刘成艳1,同韩虎1,吴三桥1,陈 琛1,陈春琳1
(1.陕西理工大学中德天然产物研究与开发利用联合研究所,陕西 汉中723001;2.陕西医药控股集团生物制品有限公司,陕西 汉中723000)
为设计奶牛乳房炎三联重组表位多肽疫苗,选取奶牛乳房炎3种主要感染菌的候选疫苗蛋白:金黄色葡萄球菌的Ebps与ClfA,大肠埃希菌的OmpA与OmpC,链球菌的SIP与PGK,通过ABCpred和BepiPred方案,预测获得每种蛋白各有2个优势B细胞表位;利用神经网络与量化矩阵法预测蛋白的CTL表位。结果显示,SIP蛋白无CTL表位,其余蛋白均存在1个CTL细胞表位;采用MHC-Ⅱ类分子结合肽在线程序预测蛋白的Th表位,结果发现每种蛋白各有1个Th细胞表位。使用DNASTAR软件分析6个蛋白的二级结构,结果发现,预测获得的B/T细胞表位大多处于蛋白易于产生表位的暴露表面、无规则卷曲与转角等位置。再通过Protean程序重组拼接获得的B/T细胞抗原表位,最终设计获得抗原性较好的三联表位疫苗氨基酸序列。为新型奶牛乳房炎三联表位疫苗的研制奠定基础。
奶牛乳房炎;表位疫苗;抗原分析
乳房炎是奶牛的多发病,也是造成牛奶产业经济损失的重要原因之一[1],病原菌主要为金黄色葡萄球菌(Staphylococcus aureus)、大肠埃希菌(Escherichia coli)和链球菌(Streptococcus)[2]。目前,奶牛乳房炎疾病的防治措施主要是大量使用抗生素,但抗生素滥用易导致牛奶中的药物残留,以及细菌耐药性的增加。此外,防治奶牛乳房炎疾病的疫苗可分为减毒疫苗、灭活疫苗[3]和蛋白亚单位疫苗[4],但均处于研究的起步阶段,商业化疫苗较少见[5]。而单次免疫可以同时抵御3种主要致病菌感染的蛋白多肽疫苗尚未开发。
表位疫苗是近年发展起来的一种新型疫苗,较传统疫苗,具有安全、稳定、产量高、免疫效果好,以及可人为设计等优点。在肿瘤、肝炎、艾滋病、流感、寄生虫等疾病防治上均有大量研究[6],是疫苗研究的新方向。对奶牛乳房炎研究发现金黄色葡萄球菌的凝集因子A(clumping factor,ClfA)[7]与弹性纤维结合蛋白(elastin binding protein,EbpS)[8];大肠埃希菌的外膜蛋白A(outer membrane protein A,OmpA)[9]与外膜蛋白 C(outermembrane protein C,OmpC)[10];链球菌的膜表面蛋白(surface immunogenic protein,SIP)与磷 酸 甘 油 激 酶 (phosphoglycerate kinase,PGK)[11-13],这些蛋白具有很好的免疫原性,是很好的候选疫苗蛋白。我们期望通过生物信息学方法,分析这些蛋白的B/T细胞抗原表位,并将表位的氨基酸序列进行重组拼接,旨在设计安全、免疫简洁与高效的奶牛乳房炎表位多肽重组疫苗,进而实现奶牛单次免疫就可抵御3种致病菌感染这一目标。
1 材料与方法
1.1 材料
选取金黄色葡萄球菌、大肠埃希菌、链球菌候选疫苗蛋白各2个。依据NCBI网站公布的氨基酸序列:金黄色葡萄球菌ClfA与EbpS蛋白(登录号分别为EFM05894.1和EFM06457.1);大肠埃希菌 OmpC和 OmpA(登录号为AIL15476.1和ELV73478.1);链球菌SIP和PGK(登录号为AEL21614.1和AKU04271.1)。
1.2 方法
1.2.1 B细胞表位预测
采用BepiPred和ABCpred联合预测B细胞表位。BepiPred方法是利用氨基酸的性质和隐形马尔可夫模型在线预测B细胞表位;ABCpred方法采用人工神经网络算法[14]。最终B细胞表位选取两种方案共有的氨基酸序列。
1.2.2 T细胞表位预测
辅助T细胞(Th)抗原表位预测类型选择DRB1-0101、DRB1-0102和DRB1-0301结合肽,预测网站为:http://www.imtech.res.in/raghava/propred/。细胞毒性T细胞(CTL)抗原表位采用量化矩阵与人工神经网络法预测,选择HLA-A2、HLA-A∗0201、HLA-A∗0202、HLA-A∗0203、HLA-A∗0205分子结合肽,通过 CTLpred程序预测[15]。
1.2.3 蛋白二级结构预测
蛋白二级结构采用DNASTAR软件的Protean程序进行预测,获得各个蛋白的抗原表位信息。目的是验证获得的B/T细胞表位是否处于蛋白抗原性较好的肽段位置。
1.2.4 重组表位疫苗的设计
将预测获得的B/T细胞表位进行选择性拼接,每个表位之间采用4个甘氨酸(GGGG)短肽进行分割,以减少各个表位间的相互影响。采用DNASTAR Protean软件分析表位的不同排列方式,以各表位相对独立且具有较好抗原性参数的组合方式,作为重组表位疫苗的氨基酸序列。然后,翻译为核酸序列,最终获得重组表位疫苗基因序列。
2 结果与分析
2.1 B细胞表位预测
采用BepiPred和ABCpred联合预测B细胞表位。BepiPred方法预测蛋白B细胞表位结果如表1所示;ABCpred方法预测结果见表2。综合BepiPred和ABCpred两种方案的共同序列区段,最终获得金黄色葡萄球菌ClfA蛋白B细胞表位区段为:326-330,114-129;Ebps蛋白B细胞表位为:87-102,132-147。大肠埃希菌OmpA蛋白B细胞表位区段为:44-50,126-135;OmpC蛋白 B细胞表位为19-31,129-144。链球菌SIP蛋白B细胞表位区段为:224-238,346-358;PGK蛋白B细胞表位为:64-79,324-329。
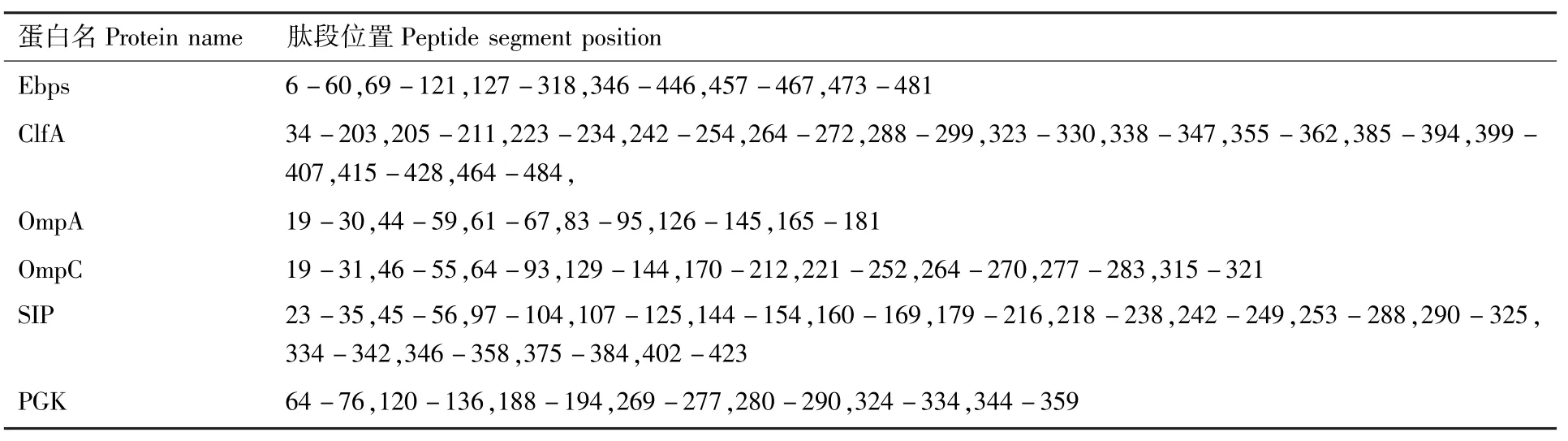
表1 BepiPred方案预测蛋白B细胞表位的肽段位置Table 1 Prediction of B cell epitopes for proteins by BepiPred method
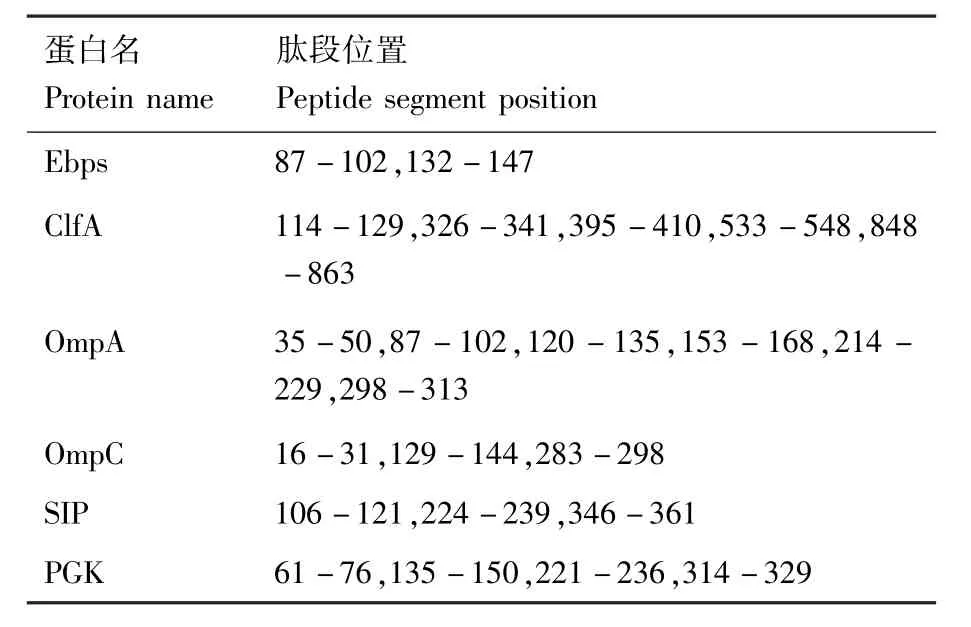
表2 ABCpred方案预测蛋白B细胞表位的肽段位置Table 2 Prediction of B cell epitopes for proteins by ABCpred method
2.2 T细胞表位预测
2.2.1 Th细胞表位预测
采用量化矩阵与人工神经网络法预测Th抗原表位。预测多肽种类选择DRB1∗0101、DRB1∗0102、DRB1∗0301,结果如表3所示。综合多肽共有的区段,获得金黄色葡萄球菌 Ebps与ClfA蛋白Th表位区段分别为:479-487、201-209;大肠埃希杆菌OmpA与OmpC蛋白Th表位区段分别为:284-292、6-12;链球菌SIP与PGK蛋白Th表位区段分别为:240-248、317-325。
2.2.2 CTL细胞表位预测
CTL表位预测采用人工神经网络法结合量化矩阵法,选择HLA-A2、HLA-A∗0201、HLA-A∗0202、HLA-A∗0203、HLA-A∗0205分子结合肽,通过CTLpred程序预测,结果如表4所示。综合多肽共有的区段,获得金黄色葡萄球菌Ebps与ClfA蛋白CTL表位区段分别为:318-326、895-902;大肠埃希菌OmpA与OmpC蛋白CTL表位区段分别为:237-245、295-303;链球菌PGK蛋白CTL表位区段为73-81;链球菌SIP蛋白无优势CTL表位。
2.3 候选疫苗蛋白二级结构预测
为进一步验证预测获得的6种蛋白B/T表位的正确性,利用DNASTAR软件的Protean程序对蛋白二级结构进行预测,检测获得的蛋白B/T细胞表位区段是否处于易于蛋白产生抗体的暴露表面、无规则卷曲与转角等位置。使用Protean程序,从蛋白的亲水性、可柔性、表面可极性与抗原指数方面,预测的6种蛋白抗原性结果如图1至图6所示。结果发现预测的6种蛋白抗原表位均处于抗原性较好的区段,支持预测的B/T细胞表位合理性。

表3 蛋白Th抗原表位预测Table 3 Th epitopes prediction for proteins
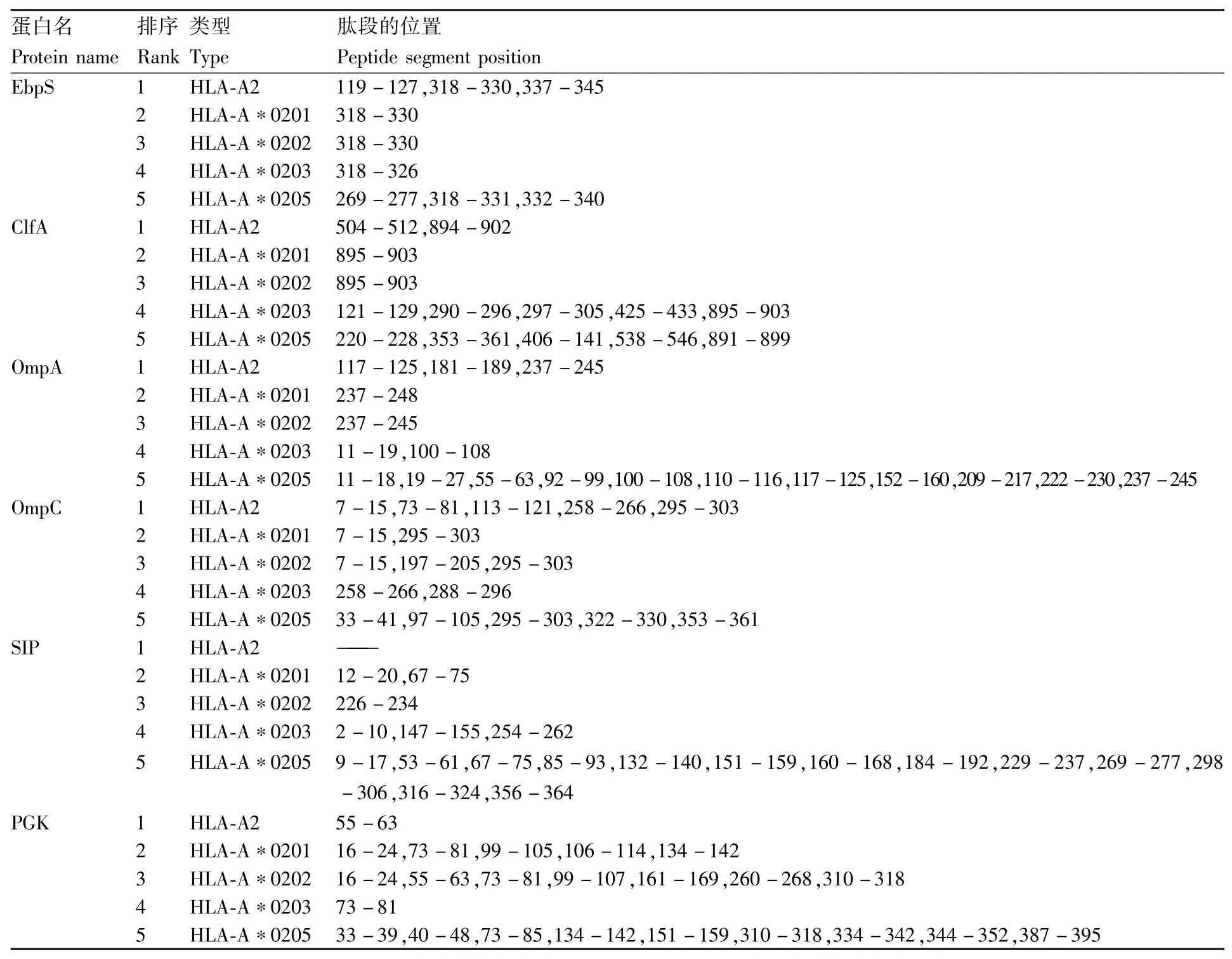
表4 蛋白CTL抗原表位预测Table 4 CTL epitopes prediction for proteins

图1 金黄色葡萄球菌EbpS蛋白表位分子的抗原性分析Fig.1 Antigenicity analysis of S.aureus EbpS protein epitopesmolecular
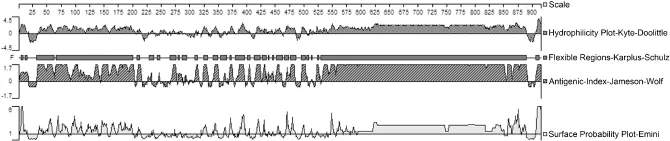
图2 金黄色葡萄球菌ClfA蛋白表位分子的抗原性分析Fig.2 Antigenicity analysis of S.aureus ClfA protein epitopesmolecular

图3 大肠埃希菌OmpA蛋白表位分子的抗原性分析Fig.3 Antigenicity analysis of E.coli OmpA protein epitopesmolecular
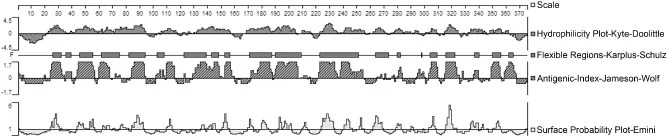
图4 大肠埃希菌OmpC蛋白表位分子的抗原性分析Fig.4 Antigenicity analysis of E.coli OmpC protein epitopesmolecular
2.4 重组串联抗原表位的设计
将预测获得的3种细菌的B/T细胞表位,分别进行拼接组合。每种细菌的2个蛋白表位,按照B细胞表位、CTL表位、Th表位顺序进行编号,再通过Protean程序分析线性表位的各种排列方式,以重组的多表位间相对独立,抗原性参数较好为标准;并且各多肽间接头氨基酸采用4个甘氨酸(GGGG)。结果发现金黄色葡萄球菌Ebps与ClfA蛋白的预测表位排列方式为:epitope 4-epitope 6-epitope 8-epitope 3-epitope 2-epitope1-epitope 5-epitope 7;大肠埃希菌OmpA与OmpC蛋白的预测表位排列方式为:epitope 5-epitope 3-epitope 2-epitope 8-epitope 1-epitope 6-epitope 7-epitope 4;链球菌SIP与PGK蛋白的预测表位排列方式为:epitope 5-epitope3-epitope 2-epitope 6-epitope 4-epitope 1-epitope 7。再将3种菌各自预测完成的串联表位进一步拼合,并采用Protean程序分析发现获得抗原性参数较好的多肽序列(图7)。至此,获得的奶牛乳房炎候选蛋白表位多 肽 序 列 为: IRNGQQIVIGGGG KDDVKGGGGVVNQAVNTSGGGG GMAKVLLPLGGGG PEPIEDNDKHDTIKNAGGGG AGTIDDRQVESSHST EGGGGTGEATTTTTNQANTPAGGGGLLASIGSLGG GG ANAAEVYNKDGNKGGGGALDQLYSQLGGGGD TKSNVYGKNGGGGLSLLVPAGGGGFINNNGPGGGG PEFGGDTYGSDNFMQQGGGGGLRPSVAYLGGGGY LISKGIPAGGGGGVFENPGGGGVRTVAAPRVGGGG TYRAGDPGDHGKGGGGGSLAPVAADLGGGGKEE ADKEGKSLAPVAAGGGGASVAAETPAPVAKVAGG GG VVWNGPMGV(下划线表示接头氨基酸)。翻译为核 酸序 列为:ATTAGAAACGGTCAACAAATCGTTATTGGTGGCGGTGGC AAAGATGATGTAAAAGGTGGCGGTGGC GTAGTTAATCAAGCG GTTAATACAAGTGGTGGCGGTGGC GGCATGGCC AAAGTATTGTTACCATTAGGTGGCGGTGGC CCAG AACCAATCGAAGACAATGATAAACACGATACTA TTAAAAATGCAGGTGGCGGTGGC GCTGGCACAA TAGATGATCGTCAAGTCGAATCATCACACAGTAC TGAAGGTGGCGGTGGC ACTGGTGAAGCTACTACT ACGACAACGAATCAAGCTAATACACCGGCAGGT GGCGGTGGCTTATTAGCATCAATAGGTTCATTA GGTGGCGGTGGC GCAAACGCTGCTGAAGTTTAC AATAAAGACGGCAACAAAGGTGGCGGTGGC GC TCTGGATCAGCTGTACAGCCAGCTGGGTGGCGG TGGC GACACTAAATCCAACGTTTATGGTAAAAA CGGTGGCGGTGGC CTGTCCCTCCTGGTCCCAGC TGGTGGCGGTGGC TTCATCAACAACAATGGCCC GGGTGGCGGTGGC CCAGAATTCGGTGGCGACAC CTACGGTTCTGACAACTTCATGCAGCAGGGTGG CGGTGGC GGTCTGCGTCCGTCTGTAGCATACCT GGGTGGCGGTGGC TACCTGATCTCCAAAGGTAT CCCGGCAGGTGGCGGTGGC GGTGTCTTTGAAAA CCCTGGTGGCGGTGGC GTAAGAACTGTAGCAGC CCCTAGAGTGGGTGGCGGTGGC ACATACCGTGC GGGAGATCCAGGTGATCATGGTAAAGGTGGTGG CGGTGGC TCACTTGCACCGGTAGCTGCTGATTTA GGTGGCGGTGGC AAAGAAGAAGCTGACAAAGA AGGAAAATCACTTGCACCGGTAGCTGCTGGTGGCGGTGGC GCTTCTGTTGCCGCTGAAACACCAGCT CCAGTAGCTAAAGTAGCAGGTGGCGGTGGC GTT GTATGGAACGGACCTATGGGTGTC(下划线表示接头氨基酸的核酸序列)。

图5 链球菌SIP蛋白表位分子的抗原性分析Fig.5 Antigenicity analysis of Streptococcus SIP protein epitopesmolecular
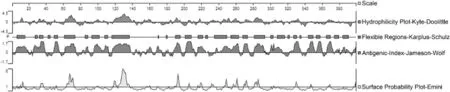
图6 链球菌PGK蛋白表位分子的抗原性分析Fig.6 Antigenicity analysis of Streptococcus PGK protein epitopesmolecular
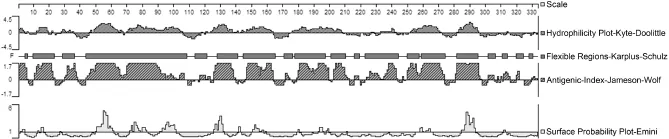
图7 重组多表位分子的抗原性分析Fig.7 Antigenicity analysis of recombinant multi-epitopes molecular
3 讨论
生物信息学具有准确、简便易行等特点,在蛋白结构,尤其在表位疫苗的设计上受到人们重视,可极大地减少试验的盲目性,为人为合成表位多肽疫苗奠定基础[16-17]。奶牛乳房炎感染菌主要为金黄色葡萄球菌、大肠埃希菌和链球菌,这为利用生物信息学合成多联表位疫苗提供可能。通过生物信息学,有实验对金黄色葡萄球菌FnbpA-A基因进行表位设计[18];并合成金黄色葡萄球菌蛋白的串联抗原表位[19-20]。大肠埃希菌表位疫苗主要针对人类的致病大肠埃希菌开展了一些研究[21-22],在奶牛乳房炎研究上尚未展开。链球菌表位疫苗主要进行了GapC蛋白[23]与Sao蛋白[24]的设计合成;在奶牛乳房炎链球菌研究上,布日额等将SIP和PGK的主要抗原区融合表达,获得抗原性较高的融合蛋白[12-13]。因而,现行的试验针对单个病原菌开展了一些表位疫苗的初步研究。本试验利用生物信息学,对奶牛乳房炎三联表位疫苗进行研究,获得了三种致病菌蛋白的B/T细胞优势表位,组装了各自的表位,最终设计获得串联表位多肽,填补三联表位多肽研究的空白,有望进一步提高奶牛乳房炎的防治效果。而该多肽的免疫作用有待试验检测。
B细胞表位预测常见的方法为BepiPred和ABCpred。BepiPred主要利用氨基酸的性质和隐形马尔可夫模型预测线性表位[25];ABCpred基于人工神经网络算法,准确率可高达65.9%[26]。前期,我们通过两种方法的结合,对绿脓杆菌OprF蛋白、溶藻弧菌OmpU蛋白进行了B细胞表位预测[14-15]。本研究采用BepiPred和ABCpred联合预测的方式进行B细胞表位预测。CTL(细胞毒性T淋巴细胞)表位预测采用人工神经网络法结合量化矩阵法[27],在病毒、细菌蛋白CTL表位预测上获得广泛运用[14,28]。Th表位预测方法较为成熟,在病毒蛋白、细菌蛋白均实现很好的Th表位预测[15,27]。本试验借助于这些成熟技术实现CTL、Th细胞表位的预测。
表位间的连接接头分为柔性和刚性两种,实现构建的多肽各表位间相对独立,具有一定的分子刚性[29]。常见的表位接头氨基酸序列有GGGG、AAY、KK与GGGGS[29-31]等。本研究采用GGGG氨基酸接头,通过DANMAN软件对不同表位拼接方式进行优化,最终预测获得抗原性较好的重组多肽的氨基酸序列和翻译的核苷酸序列。为下一步合成表位多肽疫苗的研究奠定了基础。
(
):
[1] 董翔宸,马友记,张勇,等.奶牛乳腺炎主要病原菌的药物敏感性检测[J].中国兽医学报,2015,35(9):1505 -1510.
DONG X C,MA Y J,ZHANG Y,et al.Antimicrobial susceptibility test of main pathogens causing bovine mastitis by mirodilution method[J].Chinese Journal of Veterinary Science,2015,35(9):1505-1510.(in Chinese with English abstract)
[2] 乔富强,乔利敏,姚华.复合生物预混剂对奶牛生产性能和乳品质的影响[J].浙江农业学报,2013,25(4):710 -713.
QIAO FQ,QIAO LM,YAO H.Effects of compound biologicpremix on the performance of dairy cows and milk quality[J]. Acta Agriculturae Zhejiangensis,2013,25(4):710-713.(in Chinese with English abstract)
[3] GURJAR A A,KLAESSIG S,SALMON S A,et al.Evaluation of an alternative dosing regimen of a J-5 mastitis vaccine against intramammary Escherichia coli challenge in nonlactating late-gestation dairy cows[J].Journal of Dairy Science,2013,96(8):5053-5063.
[4] 杨岚,郝永清,何焱,等.金黄色葡萄球菌 CP8-FnBPBClfA偶联蛋白对金黄色葡萄球菌性奶牛乳房炎的免疫效果研究[J].中国预防兽医学报,2015,37(3):229-232.
YANG L,HAO Y Q,HE Y,et al.The study of effect about Staphylococcus aureus CP8-FnBPB-ClfA coupling protein on Staphylococcus aureus mastitis[J].Chinese Journal of Preventive Veterinary Medicine,2015,37(3):229-232.(in Chinese with English abstract)
[5] 丁进东,彭程,贺奋义.奶牛乳房炎金葡菌黏附素亚单位疫苗的基础研究[J].畜牧兽医杂志,2014,33(3):1-4.
DING JD,PENG C,HE F Y.The study of subunit vaccine targeting adhesins of S.aureus induced mastitis in dairy cows[J].Journal of Animal Science and Veterinary Medicine,2014,33(3):1-4.(in Chinese with English abstract)
[6] YUAN X,LIN H,FAN H.Efficacy and immunogenicity of recombinant swinepox virus expressing the A epitope of the TGEVS protein[J].Vaccine,2015,33(32):3900-3906.
[7] SCULLY IL,TIMOFEYEVA Y,KEENEY D,et al.Demonstration of the preclinical correlate of protection for Staphylococcus aureus clumping factor A in amurine model of infection[J].Vaccine,2015,33(41):5452-5457.
[8] 布日额,任晓峰,吴金花,等.牛乳源性金黄色葡萄球菌nEBPS蛋白表达鉴定及多克隆抗体制备[J].中国兽医学报,2013,33(11):1674-1678.
BU R E,REN X F,WU JH,et al.Expression and characterization of N-terminal elastin binding protein of Staphylococcus aureus and preparation of its polyclonal antibody[J].Chinese Journal of Veterinary Science,2013,33(11):1674-1678.(in Chinese with English abstract)
[9] 刘祥,李惠.大肠埃希菌外膜蛋白OmpA表达质粒构建和诱导条件优化[J].生物技术,2013,23(4):14-19.
LIU X,LIH.Construction of E.coli outer protein Omp A expression vector and optimization of inducing conditions[J]. Biotechnology,2013,23(4):14-19.(in Chinese with English abstract)
[10] 俱雄,陈春琳,刘祥,等.大肠埃希菌外膜蛋白OmpC的原核表达与免疫保护作用研究[J].生命科学研究,2015,19(2):131-136.
JU X,CHEN C L,LIU X,et al.Prokaryotic expression and immunoprotection potential of E.coli outer membrane protein OmpC[J].Life Science Research,2015,19(2):131-136.(in Chinese with English abstract)
[11] 胡长敏,徐海洋,陈颖钰,等.SIP亚单位疫苗对无乳链球菌诱导小鼠乳腺炎的保护力评价 [J].中国奶牛,2012(3):1-4.
HU C M,XU H Y,CHEN Y Y,et al.Evaluation of immunization with the SIP against Streptococcus agalactiae induced mousemastitis[J].China Dairy Cattle,2012(3):1-4.(in Chinese with English abstract)
[12] 布日额,吴金花,王金良,等.奶牛乳腺炎无乳链球菌sip与pgk双基因主要抗原区域的融合表达[J].中国兽医学报,2015,35(8):1228-1231.
BU R E,WU JH,WANG J L,et al.Fusion expression of main antigen area of Streptococcus agalactiae sip and pgk double genes[J].Chinese Journal of Veterinary Science,2015,35(8):1228-1231.(in Chinese with English abstract)
[13] 吴金花,布日额,锡林高娃,等.牛乳腺炎无乳链球菌表面免疫相关蛋白及磷酸甘油酸激酶对奶牛的免疫保护作用[J].中国生物制品学杂志,2014,27(8):994-996.
WU JH,BU R E,XIL,etal.Immune protection of surface immunogenic protein of dairy cow mastitis Streptococcus agalactiae and phosphoglycerate kinase to dairy cow[J].Chinese Journal of Biologicals,2014,27(8):994-996.(in Chinese with English abstract)
[14] 刘祥.绿脓杆菌外膜蛋白OprF的生物信息学分析[J].生物技术,2015,25(1):343-348.
LIU X.Bioinformatics analysis of Pseudomonas aeruginos outer membrane protein OprF[J].Biotechnology,2015,25(1):343-348.(in Chinese with English abstract)
[15] 刘祥.溶藻弧菌外膜蛋白OmpU的原核表达、抗原性鉴定与生物信息学分析[J].华北农学报,2015,30(6):37 -43.
LIU X.Prokaryotic expression,antigenicity identification and bioinformatics analysis of Vibrio alginolyticus outer membrane protein OmpU[J].Acta Agriculturae Boreali-Sinica,2015,30(6):37-43.(in Chinese with English abstract)
[16] AMAT-UR-RASOOLH,SAGHIR A,IDREESM.Computational prediction and analysis of envelop glycoprotein epitopes of DENV-2 and DENV-3 pakistani isolates:a first step towards dengue vaccine development[J].PLoSOne,2015,10(3):e0119854.
[17] 胡晓波,朱乃硕.狂犬病毒MHC限制性CTL与Th表位的预测与鉴定[J].中国免疫学杂志,2013,29(7):736 -740.
HU X B,ZHU N S.Prediction and identification of the MHC restricted Th and CTL epitopes of rabies virus[J].Chinese Journal of Immunology,2013,29(7):736-740.(in Chinese with English abstract)
[18] 何焱,郝永清.牛源金黄色葡萄球菌FnbpA-A基因克隆及抗原表位预测[J].中国畜牧兽医,2014,41(5):7 -11.
HE Y,HAO Y Q.Cloning and antigen epitopes prediction of fibronection-binding protein A gene a domain of bovine Staphylococcus aureus[J].China Animal Husbandry&Veterinary Medicine,2014,41(5):7-11.(in Chinese with English abstract)
[19] 高翔,许会会,雷连成.金黄色葡萄球菌FnbpA、ClfA、Ebps抗原表位的串联表达及多克隆抗体的制备[J].中国畜牧兽医,2011,38(5):97-100.
GAO X,XU H H,LEIL C.Tandem expression of FnbpA,ClfA,Ebps antigen epitopes of Staphylococcus aureus and the preparation of the polyclonal antibody[J].China Animal Husbandry&Veterinary Medicine,2011,38(5):97-100.(in Chinese with English abstract)
[20] 刘丽娜,张洁,周静,等.金黄色葡萄球菌疫苗候选分子StbA主要抗原表位区的表达及重组蛋白抗血清的制备[J].中国兽医科学,2015,45(4):375-379.
LIU L N,ZHANG J,ZHOU J,et al.Expression of major antigenic epitope region of candidate protective antigen StbA from Staphylococcus aureus and preparation of antiserum against the recombinant protein[J].Chinese Veterinary Science,2015,45(4):375-379.(in Chinese with English abstract)
[21] KORZENIOWSKA-KOWAL A,KOCHMAN A,GAMIAN E,et al.Antibodies against Escherichia coli O24 and O56 O-specific polysaccharides recognize epitopes in human glandular epithelium and nervous tissue[J].PLoSOne,2015,10(6):e0129492.
[22] GUANW,LIUW,BAO J,et al.Analysis and application of a neutralizing linear epitope on liable toxin B of entero toxin Escherichia coli[J].Applied Microbiology and Biotechnology,2015,99(14):5985-5996.
[23] ZHANG L,ZHOU X,FAN Z,et al.Identification of a conserved B-cell epitope on the GapC protein of Streptococcus dysgalactiae[J].Microbial Pathogenesis,2015,83(4):23 -28.
[24] 王晶,李敏,杜骁杰,等.2型猪链球菌表面蛋白Sao的生物信息学分析及基因工程疫苗的设计 [J].中国病原生物学杂志,2014,9(3):211-215.
WANG J,LIM,DU X J,et al.Bioinformatics analysis of Sao protein and design ofa vaccine against Staphylococcus aureus type 2[J].Journal of Pathogen Biology,2014,9(3):211-215.(in Chinese with English abstract)
[25] CHEN H,YANG HW,WEU JF,et al.In silico prediction of the T-cell and IgE-binding epitopes of Pera 6 and Blag 6 allergens in cockroaches[J].Molecular Medicine Reports,2014,10(4):2130-2136.
[26] SAHA S,RAGHAVA G P.Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J]. Proteins,2006,65(1):40-48.
[27] 王光祥,尚佑军,吕占禄,等.羊口疮病毒F1L蛋白二级结构分析与表位预测[J].中国人兽共患病学报,2012,28(12):1185-1190.
WANG G X,SHANG Y J,LV Z L,et al.Prediction of secondary structures and cell epitopes of F1L protein of ORFV[J].Chinese Journal of Zoonoses,2012,28(12):1185-1190.(in Chinese with English abstract)
[28] LI J,BAI J,GU L,et al.Prediction and identification of HLA-A∗0201 restricted epitopes from leukemia-associated protein MLAA-22 which elicit cytotoxic T lymphocytes[J]. Medical Oncology,2014,31(12):293.
[29] LEROUX L P,DASANAYAKE D,ROMMERRIM L M,et al.Secreted Toxoplasma gondii molecules interfere with expression of MHC-II in interferon gamma-activated macrophages[J].International Journal for Parasitology,2015,45(5):319-332.
[30] 秦培兰,李艳,米荣升,等.微小隐孢子虫三个基因主要抗原表位区的串联表达及其抗原性分析[J].中国动物传染病学报,2012,20(3):36-43.
QIN P L,LIY,MIR S,et al.Tandem expression of major epitope domain of cryptosporidium parvum three antigen genes and its antigenicity analysis[J].Chinese Journal of Veterinary Parasitology,2012,20(3):36-43.(in Chinese with English abstract)
[31] 董林,王艳萍,沈志强,等.猪圆环病毒2型多表位串联体诱导表达及其免疫活性[J].中国兽医学报,2014,11(34):1716-1720.
DONG L,WANG Y P,SHEN Z Q,et al.Induced expression of multi-epitope of porcine circovirus type 2 and its immune activity[J].Chinese Journal of Veterinary Science,2014,11(34):1716-1720.(in Chinese with English abstract)
(责任编辑 卢福庄)
Epitope analysis of cow mastitis important vaccine candidate proteins and design of am ino acid sequence of trip le recombinant epitope vaccine
LIU Xiang1,KONG Zhi-xiang1,YIN Shu-ping2,OU Sha-sha1,LIU Cheng-yan1,TONG Han-hu1,WU San-qiao1,CHEN Chen1,CHEN Chun-lin1
(1.Chinese-German Joint Institute for Natural Product Research,Shaanxi University of Technology,Hanzhong 723001,China;2.Shaanxi Pharmaceutical Holding Group Co.,Ltd,Hanzhong 723000,China)
In order to design cow mastitis triple recombinant epitope polypeptide vaccine,six candidate vaccine proteins were selected,which were from three main pathogens of cowmastitis,concluding Ebps and ClfA of Staphylococcus aureus,OmpA and OmpC of Escherichia coli,SIP and PGK of Streptococcus.Using ABCpred and BepiPred prediction program,each protein had 2 B cell epitopes.CTL cell epitopes were obtained by the prediction method of quantitative matrix and artificial neural network,the results showed that the SIP protein had no CTL cell epitope,and there was 1 CTL cell epitope to other proteins.Th cell epitopes were gained using an online server prediction for MHC class Ⅱ peptide binding affinity,the results showed that each protein had 1 Th cell epitope.DNASTAR software was used to analyze the secondary structure for the six proteins,and the results showed that most of the B/T cellepitopes were located on the exposed surface,the random coil and the corner.The B/T cell epitopes were recomposed by DNASTAR Protean software,and triple epitope vaccine amino acid sequence holding better antigen was designed finally.These laid the foundation for the novel triple epitope vaccine of cow mastitis.
cow mastitis;epitope vaccine;antigenic analysis
S858.23;Q816
A
1004-1524(2016)09-1476-09
10.3969/j.issn.1004-1524.2016.09.04
2015-12-22
国家大学生创新创业训练计划项目(201510720556);陕西省农业科技创新与攻关项目(2016NY-088);陕西理工大学校级科研项目(SLGKY16-13)
刘祥(1982—),男,安徽合肥人,博士,讲师,研究方向为蛋白质组学与免疫学。E-mail:liuxiang888525@163.com
浙江农业学报Acta Agriculturae Zhejiangensis,2016,28(9):1476-1484 http://www.zjnyxb.cn刘祥,孔智翔,殷书平,等.奶牛乳房炎重要候选疫苗蛋白的抗原表位分析及三联重组表位疫苗的氨基酸序列设计[J].浙江农业学报,2016,28(9):1476-1484.
