极度濒危植物巧家五针松基因组微卫星特征分析
2016-11-10阮桢媛王兵益欧阳志勤廖礼彬苏腾伟
阮桢媛 王兵益 欧阳志勤 廖礼彬 苏腾伟 乔 璐
(1.云南林业职业技术学院,昆明 650224; 2.中国林业科学研究院资源昆虫研究所,昆明 650224; 3.云南省环境科学研究院, 昆明 650034)
极度濒危植物巧家五针松基因组微卫星特征分析
阮桢媛1王兵益2*欧阳志勤3廖礼彬2苏腾伟1乔 璐1
(1.云南林业职业技术学院,昆明 650224;2.中国林业科学研究院资源昆虫研究所,昆明 650224;3.云南省环境科学研究院, 昆明 650034)
巧家五针松是世界极度濒危植物,对其SSR引物的开发有助于其遗传学研究以及物种的保护。本研究通过Illumina高通量测序技术获得巧家五针松全基因组序列,并以MISA软件查找得到2 651个微卫星序列,其中单核苷酸重复最多,可能预示了其悠久的进化历史。不同重复类型中,A/T含量显著高于G/C;在不同长度重复单元中,二核苷酸重复微卫星长度变异程度最高;各重复类型微卫星长度与微卫星出现的频率成反比。获得的微卫星序列能够满足巧家五针松的种群遗传学研究,而且反映了该物种的偏好性及对应的潜在功能,并且对该物种的保护提供了资料。
巧家五针松;基因组;微卫星
巧家五针松(PinussquamataX.W.Li)是李乡旺于上世纪90年代发现的仅分布于我国云南省巧家县的珍稀树种[1],天然状态下该种仅存立木31株,其野外种群低于稳定存活界限(野生株数≤5 000株),随时有灭绝的危险,2008年被列入云南省极小种群物种,2010年云南省林业厅、云南省绿色环境发展基金会指导资助开展对其实施野外救护项目,且被列入全球100种最濒危物种名录[2]。针对该物种濒危现状,陆素娟等对五针白皮松濒危原因进行了初步探讨,指出种子产量少、质量低、发芽势弱,气候变化,毁林开荒等因素可能是其处于极度濒危状态的主要原因[3]。为加强五针白皮松的保护工作,进一步分析该种的濒危原因,昆明植物研究所张志勇利用RAPD分子标记对原生地2个亚居群的遗传多样性和遗传结构进行分析,结果显示该物种遗传多样性和遗传分化极低,分析其原因可能是物种演化过程中曾经遭受过严重的灾害,并由于遗传漂变和自交衰退等小种群现象而进一步加剧[4]。
自20世纪30年代,Wright、Fisher和Haldane创建种群遗传学(population genetics)以来[5],DNA分子标记技术因其所需材料少,代表的遗传特征稳定而广泛应用于种群遗传学研究当中。常见的分子标记技术主要有限制性片段长度多态性(restriction fragment length polymorphism,RFLP)、随机扩增多态DNA(random amplified polymorphism DNA,RAPD)、扩增片段长度多态性(amplified fragments length polymorphism,AFLP)、简单重复序列(simple sequence repeats,SSR)、序列特征化扩增区域(sequence characterizedamplified region,SCAR)等,其中RAPD法因其无需知道序列信息,引物具有随机性,在基因组中丰富度高而被大量运用,但是该技术对反应条件敏感,不同杂交之间不能转换等因素在一定程度上限制了它的应用。而SSR技术则因其共显性,涵盖的范围广,揭示多态性高、稳定性高等优点弥补了RAPD技术中的缺陷[6]。
SSR技术广泛应用于种群遗传结构、遗传多样性分析,标记辅助育种,品种亲缘关系分析,估算异交率,品种鉴定和指纹图谱构建和主要性状基因定位等各个领域[7~11],但由于开发SSR引物成本高,很多物种关于SSR信息知之甚少。过去开发SSR引物大多采用磁珠富集法、FIASCO法(fast isolation by AFLP sequences containing repeats)、ISSR巢式PCR法和改进的选择性扩增微卫星(SAM)法等方法,操作复杂,得到SSR数量少。部分借助近缘物种进行的SSR引物开发也因其数量上的局限性和物种之间的差异,限制了引物的使用以及遗传信息的获得。SLAF-seq(Specific-Locus Amplified Fragment Sequencing)是一套简化基因组测序技术,是基于全基因组水平上的高通量测序。用SLAF-seq技术开发的SSR标记具有信息量大、准确率高及成本低廉等特点。目前,将微卫星分子标记应用于巧家五针松的研究还处于空白,将其应用于巧家五针松野生种群与不同栽培种群的进一步研究对于该物种的保护及利用都具有重要的意义。因此,本研究利用巧家五针松的基因组测序,全面了解其中SSR序列特征,为筛选巧家五针松微卫星标记,进而进行遗传多样性和遗传结构分析,以及巧家五针松的保护提供了遗传学资料。同时,微卫星标记由于需要从已知的序列信息中开发合适的多态性信息位点,在资金和时间上都耗费较大,对巧家五针松微卫星的分析与开发利用,有助于将其应用于近缘物种的遗传多样性分析、品种鉴定、遗传图谱构建、主要性状基因定位等一系列生物学研究。
1 材料与方法
1.1 材料
试验材料采自云南省昭通市巧家县药山国家级自然保护区。于2015年4月采集刚萌发的幼嫩针叶,采集后立即置于液氮中带回实验室,置于-80℃冰箱中备用。
1.2 方法
采用CTAB法提取植物DNA,对检测合格的各样品基因组DNA分别用EcoRV+ScaⅠ酶进行酶切。对得到的酶切片段(SLAF标签)进行3′端加A处理、连接Dual-index测序接头、PCR扩增、纯化、混样、切胶选取目的片段,文库质检合格后用Illumina HiSeqTM2500进行测序。测序完成后将得到的序列进行去接头、去低质量阅读框和去污染处理,得到干净序列,并且进行GC含量和Q30分析,以保证测序质量。
利用MISA软件对巧家五针松基因组序列进行所有重复单元的微卫星查找,查找标准为:单核苷酸重复至少为10个重复,二核苷酸重复最少为6个重复,三核苷酸重复、四核苷酸重复、五核苷酸重复和六核苷酸重复均最少为5个重复,混合微卫星,两个SSR距离小于250 bp。
2 结果
2.1 SLAF-seq结果
测序后共得到120 356个SLAF,其平均测序深度为26.97,Q30的比例为87.7%。共开发SSR标记2 651条,平均每45.4个SLAF有1个SSR。
2.2 基因组微卫星的组成和基本特征
对巧家五针松基因组中1~6核苷酸重复完整型和复合型SSR进行查找分析,其部分SSR序列的信息见表1。
表1巧家五针松基因组SSR数据库的部分结果
Table1PartialresultofSSRdataingenomeofP.squamata
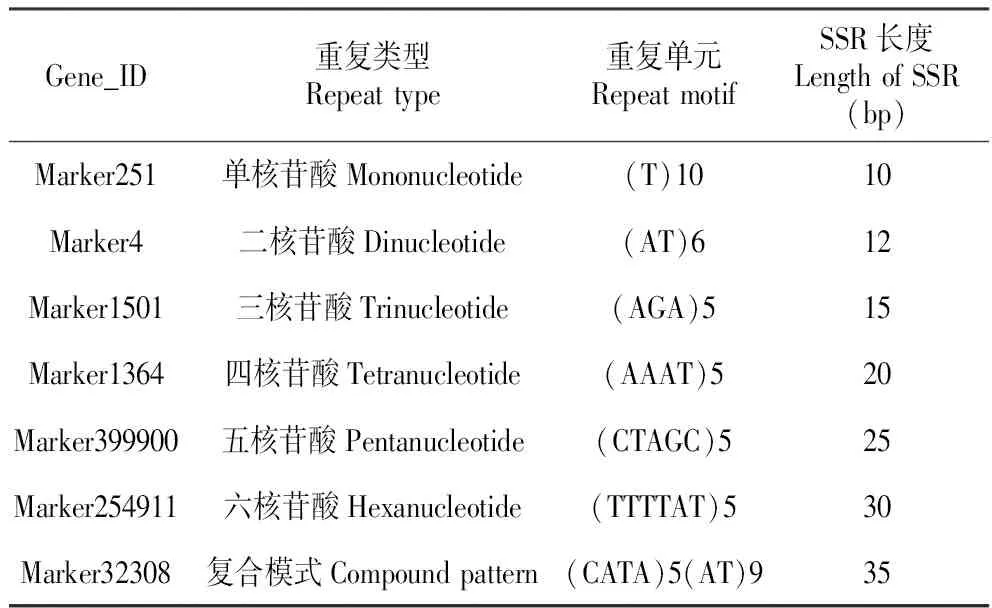
Gene_ID重复类型Repeattype重复单元RepeatmotifSSR长度LengthofSSR(bp)Marker251单核苷酸Mononucleotide(T)1010Marker4二核苷酸Dinucleotide(AT)612Marker1501三核苷酸Trinucleotide(AGA)515Marker1364四核苷酸Tetranucleotide(AAAT)520Marker399900五核苷酸Pentanucleotide(CTAGC)525Marker254911六核苷酸Hexanucleotide(TTTTAT)530Marker32308复合模式Compoundpattern(CATA)5(AT)935
对获得的SSR进行密度分析,不同重复类型的长度不一,所得密度也存在较大差异。在所有的2 651个微卫星中,单核苷酸重复类型最多,达1 603个,占总数的60.47%;其次是二核苷酸重复,共700个,占总数的26.41%;三核苷酸重复、四核苷酸重复、五核苷酸重复和六核苷酸重复所占比例依次降低,各重复单元的微卫星丰度与重复单元的长度呈负相关;非完美型有94个,占总数的3.54%;复合模式仅7个,占总数的0.26%(表2)。
表2巧家五针松基因组中SSR重复单元的分布特征
Table2DistributioncharacteristicsofSSRrepeatmotifingenomeofP.squamata

重复类型Repeattype基序种数NumberofmotifsSSR数量NumberofSSR所占比例Proportion(%)单核苷酸Mononucleotide2160360.47二核苷酸Dinucleotide570026.41三核苷酸Trinucleotide211987.47四核苷酸Tetranucleotide13421.58五核苷酸Pentanucleotide440.15六核苷酸Hexanucleotide330.11非完美型Unperfectpattern94943.55复合模式Compoundpattern770.26合计Total1492651100
表3不同重复基元类型SSR数量、长度及比例
Table3Number,lengthandproportionofSSRswithdifferentrepeatmotifs
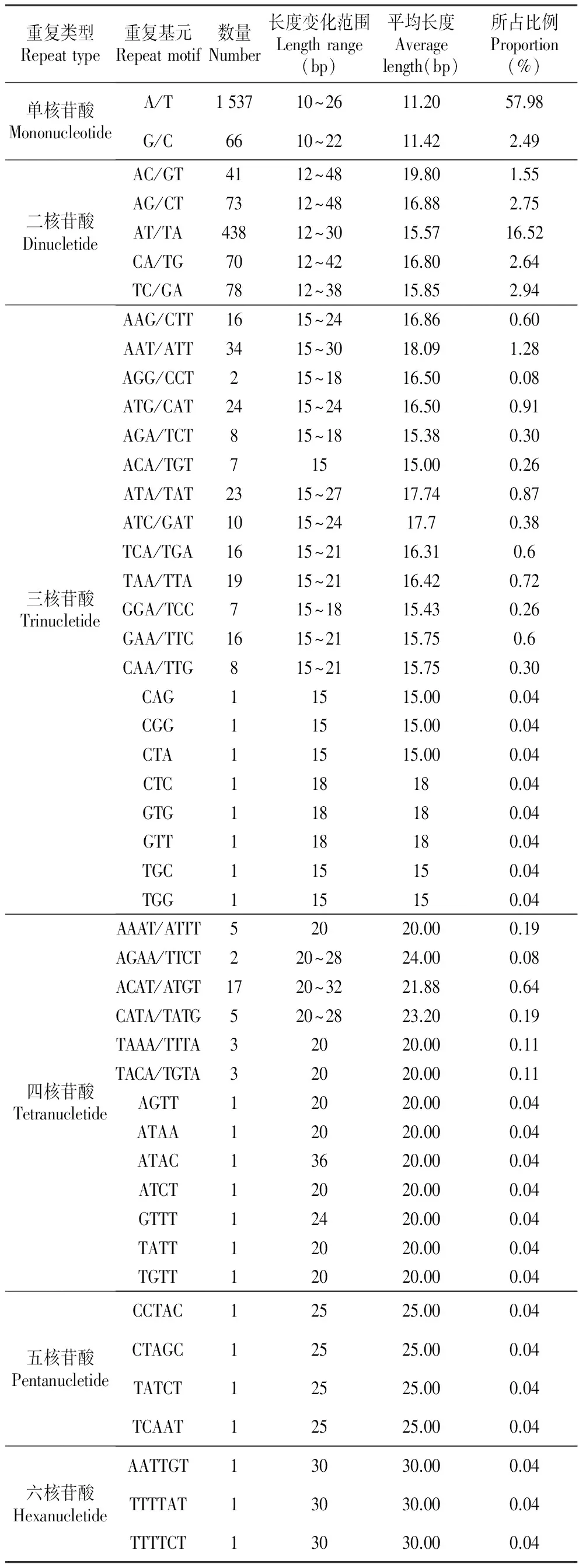
重复类型Repeattype重复基元Repeatmotif数量Number长度变化范围Lengthrange(bp)平均长度Averagelength(bp)所占比例Proportion(%)单核苷酸MononucleotideA/T153710~2611.2057.98G/C6610~2211.422.49二核苷酸DinucletideAC/GT4112~4819.801.55AG/CT7312~4816.882.75AT/TA43812~3015.5716.52CA/TG7012~4216.802.64TC/GA7812~3815.852.94三核苷酸TrinucletideAAG/CTT1615~2416.860.60AAT/ATT3415~3018.091.28AGG/CCT215~1816.500.08ATG/CAT2415~2416.500.91AGA/TCT815~1815.380.30ACA/TGT71515.000.26ATA/TAT2315~2717.740.87ATC/GAT1015~2417.70.38TCA/TGA1615~2116.310.6TAA/TTA1915~2116.420.72GGA/TCC715~1815.430.26GAA/TTC1615~2115.750.6CAA/TTG815~2115.750.30CAG11515.000.04CGG11515.000.04CTA11515.000.04CTC118180.04GTG118180.04GTT118180.04TGC115150.04TGG115150.04四核苷酸TetranucletideAAAT/ATTT52020.000.19AGAA/TTCT220~2824.000.08ACAT/ATGT1720~3221.880.64CATA/TATG520~2823.200.19TAAA/TTTA32020.000.11TACA/TGTA32020.000.11AGTT12020.000.04ATAA12020.000.04ATAC13620.000.04ATCT12020.000.04GTTT12420.000.04TATT12020.000.04TGTT12020.000.04五核苷酸PentanucletideCCTAC12525.000.04CTAGC12525.000.04TATCT12525.000.04TCAAT12525.000.04六核苷酸HexanucletideAATTGT13030.000.04TTTTAT13030.000.04TTTTCT13030.000.04
2.3不同重复基元微卫星序列长度分布及变异情况
巧家五针松SSR序列长度变化范围是10~48 bp,平均长度为14.96 bp,二核苷酸重复长度变化范围最大(12~48 bp),其中以AC/GT和AG/CT长度变化范围最大(12~48 bp);单核苷酸、三核苷酸和四核苷酸重复SSR长度变化范围较小,分别为10~26,15~30和20~36 bp;五核苷酸和六核苷酸长度无变化。在所有重复基元中,所占比例最高的是A/T,达57.68%,其次为AT/TA,占总数的16.52%,再次为TC/GA,占2.94%,可见巧家五针松中所占比例较高的是富含A/T的基元。
对巧家五针松不同长度重复单元微卫星的长度变异情况进行分析,在不同长度重复单元中,二核苷酸重复微卫星长度变异程度最高,有17种不同微卫星变化长度;单核苷酸、三核苷酸、四核苷酸重复微卫星长度变异程度依次降低;五核苷酸和六核苷酸重复微卫星长度无变异(图1)。每种重复类型都随着重复长度的增加,微卫星丰度呈现出递减趋势,即微卫星长度越长,微卫星出现的频率越低。
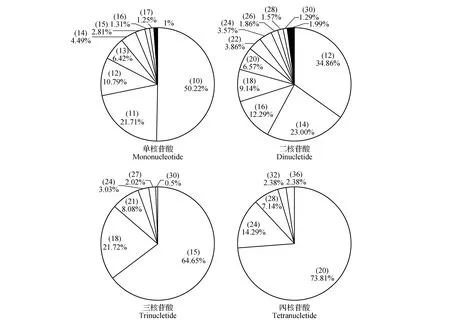
图1 巧家五针松基因组中含不同长度重复单元的微卫星长度变异情况 饼图每一扇区对应不同长度的微卫星标注于所占比例上部括号内,若对应长度微卫星频率≤0.01,则一起合并在黑色扇区内。Fig.1 Length diversification of the microsatellites in genome of P.squamata Microsatellites in different lengths are demonstrated in separate slices. If the corresponding percentage≤0.01,slices were combined for percentages(black slices).
3 讨论
巧家五针松通过全基因组的测序获得大量SSR序列,达到2 651个,而且种类丰富,其中包括了单核苷酸至六核苷酸重复,以及非完美型和复合型重复。传统的利用EST文库、SAM、锚定PCR和微卫星富集等方法开发出SSR标记[12~14],覆盖面较小,无法全面反映各物种微卫星序列分布情况,而巧家五针松在全基因组范围内搜索微卫星序列,有助于今后对其各方面研究的开展。
在获得的巧家五针松基因组所有微卫星中,以单核苷酸重复类型最多,这与许多物种以二核苷酸和三核苷酸重复类型居多不同[15~16]。张晗等在谷子基因组中发现的2 872个SSR中,二核苷酸和三核苷酸重复所占比例达到63.41%和31.86%[17];孔凡明等在一些松属物种中获得的SSR序列也以三核苷酸重复为主[18]。但是在杜仲基因组中,单核苷酸重复所占比例最高(54.34%)[19],小麦中单核苷酸重复所占比例也最多[20],这与巧家五针松的情况一致。通常认为,低级重复单元的大量存在暗示着该物种进化水平较高,而高级重复基元出现频率高的物种具有较短的进化时间或较低的变异频率[21~23]。巧家五针松以单核苷酸重复所占比例最多,可能预示着它具有较高的变异频率或较久的进化历史。已有研究认为巧家五针松与北美西部高山分布的刺果松(Pinusaristata)以及狐尾松(Pinusbalfouriana)有极密切的关系,被列入狐尾松亚组[24],而狐尾松亚组的共同祖先至少在白垩纪末、第三纪初在亚洲和北美洲大陆分离以前就已经存在[25],本研究结果或许在一定程度上证明了巧家五针松具有悠久的进化历史。
微卫星的分布在不同物种之间存在着较大的差异,微卫星的突变率也不相同,并且物种本身碱基组成也是选择的结果[26]。在巧家五针松的单核苷酸重复中以A/T重复基元出现的频率居多,在其它重复类型中A/T出现频率也显著高于G/C,二核苷酸重复以AT/TA为主,三核苷酸重复中AAT/ATT所占比例最高,SSR序列显示出显著的碱基偏好性。在小麦和杨梅中,其偏好性则不同,以G/C为主[27~28];在拟南芥和苹果中呈现出对A/T的偏好[29~30];与巧家五针松亲缘关系较近的马尾松在二碱基重复中也以AT/TA频率最高,达72.92%[31],这或许与不同物种中密码子使用的偏好性有关。巧家五针松各重复基元中G/C的出现,可能会增加某些氨基酸,从而关联于某些特定的功能,如抗逆性、转录调控和信号转导[32~33]。
重复单元的重复次数不同所造成的多态性常表现为复等位性,在不同基因型间存在广泛的多态性,这种多态性或起因于复制过程中的滑动[34]。巧家五针松二核苷酸重复微卫星长度变异程度最高,有17种不同微卫星变化长度,表明其获得或失去重复单元的活跃程度高,具有较丰富的多态性。而该物种不同重复类型微卫星长度越长,微卫星出现的频率越低,这可能与其分布地收缩,现存野生种群数量极少有关。因为微卫星重复序列越长,基因越不稳定,当物种受到强烈趋同选择的压力下,SSR序列富集在较短的序列范围内[12]。
巧家五针松微卫星序列特征的分析反映出获得的微卫星序列能够满足该物种的种群遗传学研究,并可进一步为该物种的保护生物学研究提供资料。该研究获取的巧家五针松微卫星序列完整,由此开发获得的SSR标记对于松属植物及其它亲缘关系较近物种的研究都具有一定的价值。
1.李乡旺.云南松属一新系一新种[J].云南植物研究,1992,14(3):259-260.
Li Xiangwang.A new series and a new species ofPinusfrom Yunnan[J].Acta Botanica Yunnanica,1992,14(3):259-260.
2.李文虎.巧家五针松列入全球最濒危物种名录[J].云南林业,2012,33(6):37.
Li Wenhu.Pinussquamatais listed on the most endangered species in the world[J].Yunnan Forestry,2012,33(6):37.
3.陆素娟,邓莉兰,李乡旺.五针白皮松濒危原因初步研究[J].西北林学院学报,1999,14(1):42-44.
Lu Sujuan,Deng Lilan,Li Xiangwang.A study on endangered causes ofPinussquamata[J].Journal of Northwest Forestry University,1999,14(1):42-44.
4.张志勇,李德铢.极度濒危植物五针白皮松的保护遗传学研究[J].云南植物研究,2003,25(5):544-550.
Zhang Zhiyong,Li Dezhu.Conversation genetics of an extremely endangered pine,Pinussquamata[J].Acta Botanica Yunnanica,2003,25(5):544-550.
5.Wright S.Evolution in populations in approximate equilibrium[J].Journal of Genetics,1935,30:257.
6.刘列钊,林呐.油菜简单重复序列SSR(simple sequence repeat)研究进展[J].生命科学,2004,16(3):173-176.
Liu Liezhao,Lin Na.Research advances of SSR(simple sequence of repeat) in canola[J].Chinese Bulletin of Life Sciences,2004,16(3):173-176.
7.田路明,曹玉芬,董星光,等.SSR分子标记在梨种质资源研究中的应用[J].生物学杂志,2013,30(6):91-94.
Tian Luming,Cao Yufen,Dong Xingguang,et al.Application of SSR molecular marker in pear germplasm resources[J].Journal of Biology,2013,30(6):91-94.
8.杨爱红,张金菊,田华,等.鹅掌楸贵州烂木山居群的微卫星遗传多样性及空间遗传结构[J].生物多样性,2014,22(3):375-384.
Yang Aihong,Zhang Jinju,Tian Hua,et al.Microsatellite genetic diversity and fine-scale spatial genetic structure within a natural stand ofLiruiodendronchinense(Magnoliaceae) in Lanmushan,Duyun City,Guizhou Province[J].Biodiversity Science,2014,22(3):375-384.
9.贺梁琼,熊发前,韩柱强,等.花生种间杂种异源多倍化早期世代性状和SSR变化研究[J].中国油料作物学报,2013,35(5):499-507.
He Liangqiong,Xiong Faqian,Han Zhuqiang,et al.Traits and microsatellites variation of early generations during allopolyploidization ofArachisinterspecific hydridization[J].Chinese Journal of Oil Crop Sciences,2013,35(5):499-507.
10.胡紫璟,毛娟,赵长增,等.利用SSR标记分析27份扁桃种质资源的亲缘关系[J].甘肃农业大学学报,2015,50(4):56-62.
Hu Zijing,Mao Juan,Zhao Changzeng,et al.Idetification of genetic relationship of almond by SSR markers[J].Journal of Gansu Agricultural University,2015,50(4):56-62.
11.李婷,覃道凤,戴璨.利用SSR荧光标记对野慈姑异交率的估测[J].植物科学学报,2015,33(4):554-563.
Li Ting,Qin Daofeng,Dai Can.An estimation of the outcrossing rate inSagitlariatrifoliausing SSR fluorescence markers[J].Plant Science Journal,2015,33(4):554-563.
12.李淑娴,张新叶,王英亚,等.桉树EST序列中微卫星含量及相关特征[J].植物学报,2010,45(3):363-371.
Li Shuxian,Zhang Xinye,Wang Yingya,et al.Content and characteristics of microsatellites detected in expressed sequence tag sequences in Eucalyptus[J].Chinese Bulletin of Botany,2010,45(3):363-371.
13.秦海峰,龙宁,吴建国,等.甜叶菊微卫星富集文库的构建与多态性标记的筛选[J].作物学报,2014,40(3):447-456.
Qin Haifeng,Long Ning,Wu Jianguo.Construction of microsatellite-enriched library and isolation of icrosatellite markers inSteviarebaudiana[J].ACTA Agronomica Science,2014,40(3):447-456.
14.曾庆国,陈艺燕.微卫星位点筛选方法综述[J].生态科学,2005,24(4):368-372.
Zeng Qinguo,Chen Yiyan.The methods of isolating microsatellite loci[J].Ecologic Science,2005,24(4):368-372.
15.袁阳阳,王青锋,陈进明.基于转录组测序信息的水生植物莕菜SSR标记开发[J].植物科学学报,2013,31(5):485-492.
Yuan Yangyang,Wang Qingfeng,Chen Jinming.Development of SSR markers in aquatic plantNymphoidespeltataMenyanthaceae based on information from transcriptom sequencing[J].Plant Science Journal,2013,31(5):485-492.
16.Aggarwal R K,Hendre P S,Varshney R K,et al.Identification,characterization and utilization of EST-derived genic microsatellite markers for genome analyses of coffee and related species[J].Theoretical and Applied Genetics,2007,114(2):359-372.
17.张晗,王雪梅,王东建,等.谷子基因组SSR信息分析和标记开发[J].分子植物育种,2013,11(1):30-36.
Zhang Han,Wang Xuemei,Wang Dongjian,et al.Survey of SSRs in foxtail millet genome and development of SSR markers[J].Molecular Plant Breeding,2013,11(1):30-36.
18.孔凡明,王小龙,陈赢男,等.松属编码区微卫星特征和相应基因功能分析[J].南京林业大学学报:自然科学版,2014,38(2):47-51.
Kong Fanming,Wang Xiaolong,Chen Yingnan,et al.Characterization of microsatellites in coding genes and functional analysis of these genes in pines[J].Journal of Nanjing Forestry University:Natural Sciences Edition,2014,38(2):47-51.
19.吴敏,杜红岩,乌云塔娜,等.杜仲基因组微卫星特征及SSR标记开发[J].林业科学研究,2015,28(3):387-393.
Wu Ming,Du Hongyan,Wuyun Tana,et al.Characterization of genomic microsatellites and development of SSR markers ofEucommiaulmoides[J].Forest Research,2015,28(3):387-393.
20.黄志成,雷树凡,胡景涛,等.利用优化的SSR分子标记技术检测杂交水稻万优481纯度[J].安徽农业科学,2014,42(22):7321-7322.
Huang Zhicheng,Lei Shufan,Hu Jingtao,et al.Identification and purity test of hybrid rice Wanyou 481 with optimized SSR markers[J].Journal of Anhui Agricultural Sciences,2014,42(22):7321-7322.
22.Harr B,Schlötterer C.Long microsatellite alleles in Drosophila melanogaster have a downward mutation bias and short persistence times,which cause their genome-wide underrepresentation[J].Genetics,2000,155(3):1213-1220.
23.高亚梅,韩毅强,汤辉,等.根瘤菌基因组内简单重复序列的分析[J].中国农业科学,2008,41(10):2992-2998.
Gao Yamei,Han Yiqiang,Tang Hui,et al.Anlysis of simple sequence repeats in Rhizobium genomes[J].Scientia Agricultura Sinica,2008,41(10):2992-2998.
24.吴征镒,王荷生.中国自然地理——植物地理[M].北京:科学出版社,1983:1-129.
Wu Zhengyi,Wang Hesheng.Chinese physical geography—Plant geography[M].Beijing:Science Press,1983:1-129.
25.樊国盛,李乡旺,邓莉兰.五针白皮松林区系特点研究[J].中南林学院学报,1996,16(2):23-27,33.
Fan Guosheng,Li Xiangwang,Deng Lilan.A study of floristic geography ofPinussquamataflora[J].Journal of Central-South Forestry University,1996,16(2):23-27.
26.张炜.利用小麦SSR研究其近缘物种的遗传多样性及其微卫星序列的进化[D].成都:四川农业大学,2008.
Zhang Wei.Genetic diversity and evolution of microsatellite segment among triticeae species as revealed by wheat microsatellite markers[D].Chengdu:Sichuan Agricultural University,2008.
27.Song Q J,SHI J R,SINGH S,et al.Development and mapping of microsatellite(SSR) markers in wheat[J].Theoretical and Applied Genetics,2005,110(3):550-560.
28.Jiao Y,Jia H M,Li X W,et al.Development of simple sequence repeat(SSR) markers from a genome survey of Chinese bayberry(Myricarubra)[J].BMC Genomics,2012,13:201.
29.Lawson M J,Zhang L Q.Distinct patterns of SSR distribution in theArabidopsisthalianaand rice genomes[J].Genome Biology,2006,7(2):R14.
30.关玲,章镇,王新卫,等.苹果基因组SSR位点分析与应用[J].中国农业科学,2011,44(21):4415-4428.
Guan Ling,Zhang Zhen,Wang Xinwei,et al.Evaluation and application of SSR loci in apple genome[J].Scientia Agricultura Science,2011,44(21):4415-4428.
31.刘公秉,季孔庶.基于松树EST序列的马尾松SSR引物开发[J].分子植物育种,2009,7(4):833-838.
Liu Gongbing,Ji Kongshu.DesigningPinusmassonianaSSR promers fromPinusEST sequences[J].Molecular Plant Breeding,2009,7(4):833-838.
32.Li S X,Yin T M.Map and analysis of microsatellites in the genome ofPopulus:The first sequenced perennial plant[J].Science in China Series C:Life Science,2007,50(5):690-699.
33.雷淑云,高庆波,付鹏程,等.基于Solexa高通量测序的唐古特红景天(Rhodiolaalgida)微卫星信息分析[J].植物研究,2014,34(6):829-834.
Lei Shuyun,Gao Qingbo,Fu Pengcheng,et al.Analysis on microsatellites inRhodiolaalgidabased on Solexa sequencing[J].Bulletin of Botanical Research,2014,34(6):829-834.
34.Schlötterer C,Tautz D.Slippage synthesis of simple sequence DNA[J].Nucleic Acids Research,1992,20(2):211-215.
Science Research Fund of Yunnan Provincial Education Department(2014Y569);Special Fund for the Protection of Biological Diversity in Yunnan(2013-004);Doctoral Program of Yunnan Forestry Technological College(KY(BS)201402);Science Research Fund of Yunnan Provincial Education Department(2014Y571)
introduction:RUAN Zhen-Yuan(1983—),female,Doctor,lecturer, major is plant physiology and molecular biology.
date:2016-03-16
CharacterizationofMicrosatellitesinGenomeofPinussquamata,aCriticallyEndangeredSpeciesintheWorld
RUAN Zhen-Yuan1WANG Bing-Yi2*OUYANG Zhi-Qin3*LIAO Li-Bin2SU Teng-Wei1QIAO Lu1
(1.Department of Landscape Architecture,Yunnan Forestry Technologicl College,Kunming 650224;2.The Research Institute of Resource Insects,Chinese Academy of Forestry,Kunming 650224;3.Yunnan Institute of Environmental Science,Kunming 650034)
Pinussquamatais a critically endangered species in the world, therefore, developing simple sequence repeat(SSR) primers will facilitate the species genetic research and conservation. The complete genome sequence ofP.squamaiawas determined through Illumina genome analyzer, and 2 651 microsatellites were discovered by MISA software. Single nucleotide repeat number occupied the highest proportion of these microsatellites which may indicate a long evolution history ofP.squamata. Among different repeat types, A/T contents was significantly higher than G/C contents. In terms of distinct length repeat units, the length of dinucleotide repeat microsatellite varies with the highest degree. The length of SSRs with different repeat type was negatively correlated with frequency of occurrences. The available microsatellite sequences can satisfyP.squamaiapopulation genetics, and reveal the species bias and corresponding potential functions.
PinussquamataX.W.Li;genome;microsatellite
云南省教育厅科学研究基金项目(2014Y569);云南省生物多样性保护专项资金资助项目(2013-004);云南林业职业技术学院博士基金项目(KY(BS)201402);云南省教育厅科学研究基金项目(2014Y571)
阮桢媛(1983—),女,博士,讲师,主要从事植物生理与分子生物学研究。
* 通信作者:E-mail:wangbykm@gmail.com
2016-03-16
* Corresponding author:E-mail:wangbykm@gmail.com
S791.24
A
10.7525/j.issn.1673-5102.2016.05.020
