山羊INSIG1基因超表达对乳腺上皮细胞中脂质合成的影响
2016-11-01许会芬朱江江赫秋亚姚大为史怀平
王 苗,罗 军,许会芬,朱江江,赫秋亚,姚大为,史怀平
(西北农林科技大学动物科技学院,陕西省农业分子生物学重点实验室,杨凌 712100)
山羊INSIG1基因超表达对乳腺上皮细胞中脂质合成的影响
王苗,罗军*,许会芬,朱江江,赫秋亚,姚大为,史怀平
(西北农林科技大学动物科技学院,陕西省农业分子生物学重点实验室,杨凌 712100)
本研究旨在通过构建西农萨能奶山羊胰岛素诱导基因1 (INSIG1) 的重组腺病毒(Adenovirus,Ad) 超表达载体,研究该基因超表达后对乳腺上皮细胞中脂质合成的影响,从而为该基因在山羊乳腺脂质合成调控中的功能研究奠定基础。根据GenBank (登录号:JQ665439)中山羊INSIG1基因序列设计引物,PCR扩增得到其CDS区序列。将目的基因连接到穿梭载体pAdTrack-CMV后获得pAdTrack-CMV-INSIG1质粒,将该质粒PmeⅠ线性化后转入大肠杆菌BJ5183感受态细胞进行同源重组,获得pAdEasy-INSIG1重组腺病毒超表达载体。该重组质粒经PacⅠ线性化后转染293A细胞进行病毒的包装及扩繁,利用LaSRT法测定其滴度。将获得的重组腺病毒Ad-INSIG1感染山羊原代乳腺上皮细胞,利用实时荧光定量PCR检测INSIG1及脂质合成相关基因的mRNA表达情况,利用GPO-Trinder酶学反应检测细胞中甘油三酯的含量。结果表明,成功构建了奶山羊INSIG1基因重组腺病毒超表达载体,获得了具有较高滴度 (2×108U·mL-1) 的超表达腺病毒Ad-INSIG1;Ad-INSIG1感染山羊原代乳腺上皮细胞48 h后,与Ad-GFP组相比,INSIG1基因的mRNA表达量上调约500倍,固醇调节元件结合蛋白1 (SREBP1)和SREBP裂解活化蛋白 (SCAP) 的mRNA表达量无明显变化,参与脂肪酸从头合成 (ACCα、FASN) 及去饱和 (SCD1) 基因的mRNA表达量均显著下降 (P<0.05);参与甘油三酯合成的3个关键基因中,GPAM及DGAT2的mRNA表达量显著下降 (P<0.05),AGPAT6的表达无显著变化;同时,催化甘油三酯分解 (ATGL)的基因mRNA表达量也显著下降 (P<0.05);细胞内甘油三酯含量无显著变化。综上所述,在山羊原代乳腺上皮细胞中,INSIG1能够抑制脂质合成相关基因的表达,对山羊乳腺脂质合成具有调控作用。
山羊乳腺上皮细胞;胰岛素诱导基因1 (INSIG1);超表达;脂质合成
胰岛素诱导基因 (Insulin-induced gene,INSIG) 位于内质网膜上,哺乳动物中有INSIG1和INSIG2两个亚型[1-2]。前人研究显示,INSIGs能够与固醇调节元件结合蛋白 (Sterol regulatory element binding protein,SREBPs) 裂解活化蛋白 (SREBP cleavage-activating protein,SCAP) 及3-羟基-3甲基戊二酸单酰辅酶A还原酶 (HMG CoA reductase,HMGR) 结合,从而发挥其生物学功能。INSIGs能使SCAP/SREBP复合体固定在内质网上,阻止其向高尔基体转运活化[3],并且能够促进HMGR的降解[4]。其中SREBP在哺乳动物中是参与胆固醇及脂肪酸合成调节的重要转录因子,HMGR是胆固醇合成的限速酶,因此,INSIGs对脂质及胆固醇代谢有重要的调控作用[5]。但INSIG1和INSIG2转录调节的机制是不同的,前者的表达受核SREBPs的正调节[2],而后者的表达主要受胰岛素的负调节[6];INSIG1与SCAP的亲和力强于INSIG2,在对SREBP的抑制过程中INSIG1蛋白起快速调节作用[7];在肝中,INSIG1表达量占总INSIGs的75%以上[8]。因此,一般认为主要通过INSIG1蛋白实现对SREBPs和HMGR的反馈抑制。另外,研究发现,在牛分娩期前后,乳腺组织中INSIG1的mRNA表达量上调了12倍[9],在对鼠的研究中也有相似的发现[10],提示INSIG1在乳腺组织中可能具有重要作用。然而,INSIG1基因在山羊乳脂生成中的调控功能及分子机制还不清楚。
本研究采用同源重组技术构建INSIG1腺病毒 (Adenovirus,Ad) 超表达载体,利用293A细胞包装、扩增得到高滴度的腺病毒,为后续研究提供试验材料;病毒液感染山羊原代乳腺上皮细胞后,检测脂质合成相关基因表达及细胞中甘油三酯含量的变化,初步探讨INSIG1基因在山羊乳腺上皮细胞中的功能,为明确INSIG1基因在山羊乳腺脂质代谢调控过程中的功能,揭示其对于乳脂合成代谢调控的意义,提供试验依据和理论基础。
1 材料与方法
1.1材料
腺病毒穿梭载体 (pAdTrack-CMV)、含有腺病毒骨架载体(pAdEasy-1)的大肠杆菌(Escherichiacoli,E.coli)BJ5183 菌液、山羊原代乳腺上皮细胞及293A细胞系均由本实验室保存。
试验所用T4 DNA 连接酶、PrimeScript®反转录试剂盒及SYBR®Premix Ex TaqTMⅡ试剂盒、Taq酶、SalⅠ酶及BglⅡ酶等购自TaKaRa大连宝生物工程有限公司 (大连);DNA marker D2000 分子标量、E.coliTOP 10感受态细胞、DNA Marker Ⅲ分子标量、λDNA/Hind Ⅲ marker分子标量、细胞总RNA提取试剂盒、高纯度小提中量试剂盒及琼脂糖凝胶DNA回收试剂盒等购自北京天根生化科技有限公司 (北京);限制性内切酶PmeⅠ、PacⅠ等购自NEB 公司 (美国);FuGENE HD转染试剂购自罗氏 (Roche) 公司 (瑞士);细胞培养板购自NUNC公司 (丹麦);DMEM高糖培养基和DMEM/F-12 培养基均购自HyClone 公司 (北京);胎牛血清购自浙江天杭生物有限公司 (杭州);细胞/组织甘油三酯 (Triacylglycerol,TAG)提取试剂盒购自北京普利莱生物有限公司 (北京)。
1.2方法
1.2.1INSIG1基因的扩增根据西农萨能奶山羊INSIG1基因的序列 (GenBank登录号:JQ665439),设计特异性引物,在上游引物前添加BglII酶切位点 (加粗部分) 及Kozak序列(下划线部分),下游引物添加SalI酶切位点 (阴影部分),并在引物5′端添加保护碱基 (斜体部分)。上下游引物序列:INSIG1-S (5′-3′):GAAGATCTGCCACCATGCCCAGACTGGACGACCACCTCT;INSIG1-A (5′-3′):ACGCGTCGACTCAATCAC-TGTGTGGCTTTTCGG。
以山羊泌乳期乳腺组织cDNA为模板,对INSIG1基因进行PCR扩增。PCR反应体系及反应程序参照王慧等[11]的研究,其中退火温度为63 ℃,延伸时间为1 min。PCR产物采用浓度为1%的琼脂糖凝胶电泳检测,将900 bp左右的目的片段回收纯化后连接载体pMD19-T,连接产物转化感受态细胞E.coliTop 10,提取质粒即pMD19-T-INSIG1,进行SalⅠ/BglⅡ双酶切鉴定,阳性质粒由英潍捷基公司进行测序鉴定。
1.2.2pAdTrack-CMV-INSIG1穿梭载体的构建用SalI和BglII对测序正确的pMD19-T-INSIG1质粒和腺病毒穿梭载体质粒pAdTrack-CMV进行双酶切,电泳检测并回收纯化,用T4连接酶将纯化产物4 ℃连接过夜,连接产物转化到E.coliTop10感受态细胞,涂布于LB固体培养基 (含卡那霉素Kan,100 mg·L-1),37 ℃培养12~14 h。选取较小单克隆菌落培养扩繁后提取质粒,凝胶电泳检测,并利用SalⅠ/BglⅡ进行双酶切鉴定。阳性质粒由英潍捷基公司进行测序鉴定。
1.2.3重组腺病毒载体pAdEasey-INSIG1的构建及鉴定利用氯化钙法,制备含骨架载体pAdEasey-1的感受态细胞E.coliBJ5183[12]。对pAdTrack-CMV-INSIG1阳性质粒进行PmeI酶切线性化,回收产物转化含腺病毒骨架载体pAdEasey-1的E.coliBJ5183感受态细胞中,其中热激时间为55 s,进行同源重组。涂布于LB固体培养基 (Kan,100 mg·L-1),37 ℃培养16~18 h,挑取较小单克隆菌落培养扩繁。提取质粒DNA,凝胶电泳检测,并利用内切酶PacI酶切鉴定。将阳性的重组质粒转化E.coliTOP10感受态细胞中进行大量扩繁。提取质粒送往公司进行测序鉴定。
1.2.4腺病毒Ad-INSIG1的包装、扩增及滴度测定取10 μg重组质粒pAd-INSIG1用PacⅠ酶线性化,通过异丙醇沉淀法纯化回收大片段。按照FuGENE HD Transfection Reagent说明书操作步骤,将回收产物转染6孔细胞培养板中生长融合度为70%~80%的293A细胞中,进行重组腺病毒包装。腺病毒包装过程及扩增方法参考王伟等[13]的研究。利用LaSRT法测定病毒滴度[14]。
1.2.5腺病毒感染乳腺上皮细胞最佳MOI (Multiplicity of infection) 值的测定将生长良好的乳腺上皮细胞接种于24孔细胞培养板内,待生长到汇合度达到70%~80%时,更换培养基,同时分别加入5、10、20和40 μL的腺病毒液,每个处理3个重复。每间隔24 h更换新鲜培养基,72 h后观察细胞中的绿色荧光情况及细胞生长状况,荧光较亮且细胞无明显病变 (细胞变圆且成串分布)的量为在乳腺上皮细胞中最佳的感染量,计算MOI值。
1.2.6Ad-INSIG1腺病毒超表达效果检测将生长状态良好的F12代乳腺上皮细胞接种于24孔细胞培养板中,待细胞汇合度达到80%~90%后更换新鲜培养基,接种腺病毒,每间隔24 h更换一次培养基。设置2个组:对照组 (Ad-GFP)、试验组 (Ad-INSIG1),每个处理设置3个生物学重复。48 h后收集细胞提取总RNA,检测浓度及纯度,将合格的RNA样品反转录为cDNA,以cDNA为模板进行实时荧光定量PCR (Quantitative real-time PCR,qRT-PCR) 分析。反应体系和反应条件均参考孙雨婷等[15]的研究,每个样品重复3次。选择UXT、MRPL39及RSP9为内参基因[16-18]。实时定量引物序列见表1。采用2-ΔΔCt法进行相对定量分析,其中ΔCt=CtINSIG1-Ct内参,ΔΔCt=ΔCtAd-INSIG1-ΔCtAd-GFP,Ct内参为3个内参基因Ct值的几何平均数[19]。
表1实时荧光定量PCR引物
Table1 Primers for qRT-PCR

基因名称Gene引物序列(5'-3')Primersequence产物长度/bpLength扩增效率EfficiencyUXTF:TGTGGCCCTTGGATATGGTTR:GGTTGTCGCTGAGCTCTGTG1012.06MRPL-39F:AGGTTCTCTTTTGTTGGCATCCR:TTGGTCAGAGCCCCAGAAGT1011.94RPS-9F:CCTCGACCAAGAGCTGAAGR:CCTCCAGACCTCACGTTTGTTC642.10INSIG1F:AGCCTCACAAGTTCAAGCGR:ACAGTGCTGCTAATGTCAAGG1322.21SCAPF:CCATGTGCACTTCAAGGAGGAR:TGTCGATCTTGCGTGTGGAG1071.95SREBP1F:CTGCTGACCGACATAGAAGACATR:GTAGGGCGGGTCAAACAGG811.98ACCF:CTCCAACCTCAACCACTACGGR:GGGGAATCACAGAAGCAGCC1712.03FASNF:GGGCTCCACCACCGTGTTCCAR:GCTCTGCTGGGCCTGCAGCTG2261.93SCD1F:CCATCGCCTGTGGAGTCACR:GTCGGATAAATCTAGCGTAGCA2571.92GPAMF:ATTGACCCTTGGCACGATAGR:AACAGCACCTTCCCACAAAG1882.16DGAT2F:CATGTACACATTCTGCACCGATTR:TGACCTCCTGCCACCTTTCT1002.10AGPAT6F:AAGCAAGTTGCCCATCCTCAR:AAACTGTGGCTCCAATTTCGA1002.06ATGLF:GGAGCTTATCCAGGCCAATGR:TGCGGGCAGATGTCACTCT1802.16HSLF:GGGAGCACTACAAACGCAACGR:TGAATGATCCGCTCAAACTCG1361.98
1.2.7INSIG1基因超表达后对脂质合成相关基因表达的影响脂肪酸合酶基因(Fatty acid synthase,FASN)、硬脂酰基辅酶A去饱和酶1基因(Stearoyl-CoA desaturase 1,SCD1) 、乙酰辅酶A羧化酶α基因(Acetyl-CoA carboxylas α,ACCα)、甘油-3-磷酸酰基转移酶基因(Glycerol-3-phosphate acyltransferase,GPAM)、二脂酰甘油酰基转移酶2基因(Diacylgycerol acyltransferase 2,DGAT2)、磷酸甘油酰基转移酶基因6 (1-acyl-sn-glycerol-3-phosphate acyltransferase 6,AGPAT6)、脂肪三酰甘油酯酶 (Adipose triacylglycerol lipase,ATGL)及激素敏感脂肪酶 (Hormone-sensitive lipase,HSL) 被证实为脂质代谢相关基因。利用Primer Premier5.0 软件设计上述基因的qRT-PCR引物(表1)。反应体系、反应条件及数据分析方法同上。
1.2.8INSIG1基因超表达后对乳腺上皮细胞中甘油三酯含量的影响 将生长状态良好的F12代乳腺上皮细胞接种于6 cm细胞培养皿中,待细胞汇合度达到80%~90%后接种腺病毒,每间隔24 h更换一次培养基。病毒感染48 h后弃尽培养基,PBS冲洗2遍,利用试剂盒提取并利用GPO-Trinder酶学反应测量细胞内甘油三酯的含量。每个处理设置3个生物学重复,每个样品测量3次。
2 结 果
2.1INSIG1基因的扩增与鉴定
以泌乳期山羊乳腺组织cDNA为模板,PCR扩增得到山羊INSIG1基因CDS区序列831 bp (图1A)。将PCR产物回收,连接到pMD19-T载体,进行双酶切鉴定 (图1B) 及测序,结果与GenBank中收录的序列一致。表明成功克隆了西农萨能奶山羊INSIG1基因的CDS区序列。
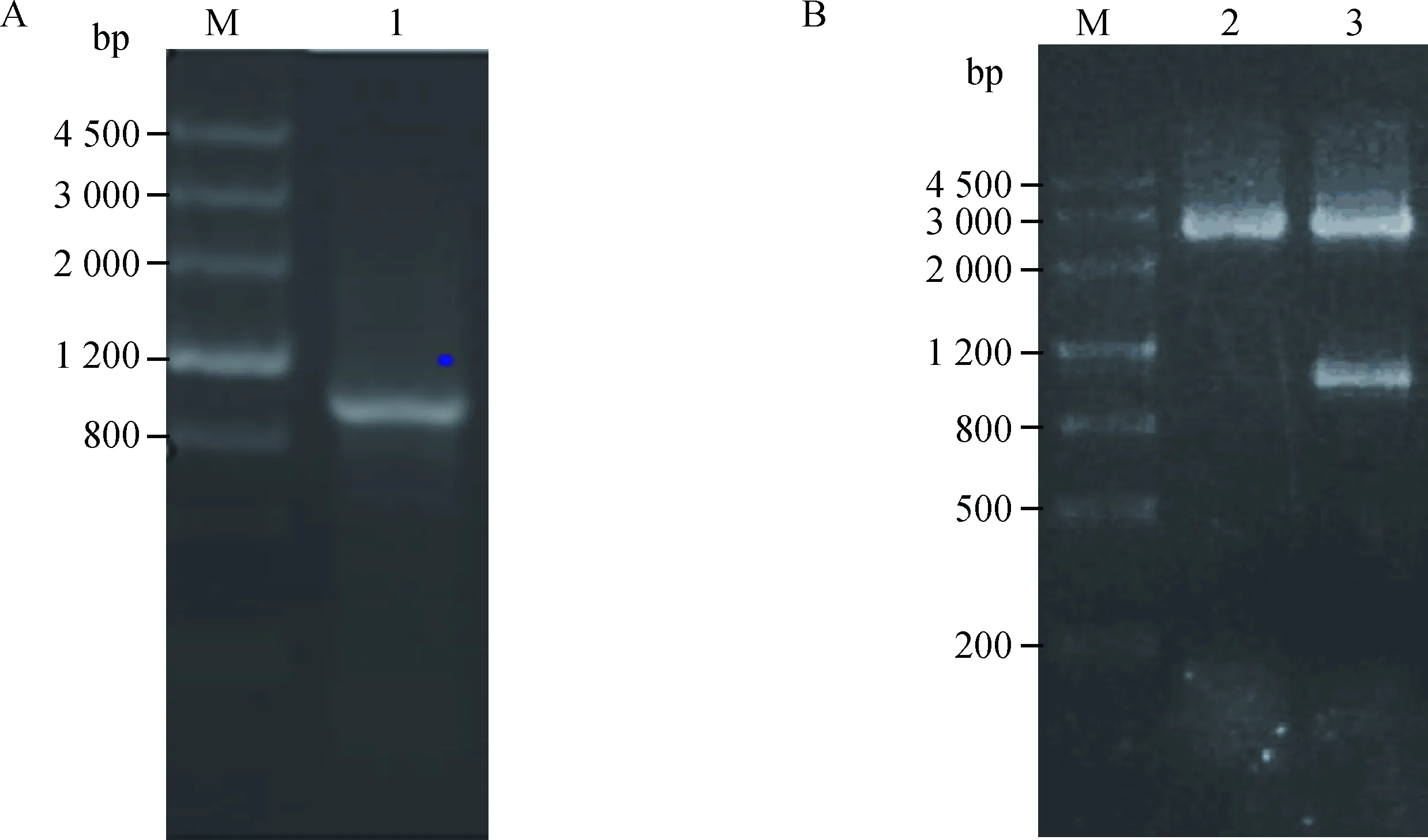
M.DNA相对分子质量标准;1.INSIG1基因CDS区PCR扩增产物;2.pMD19-T-INSIG1质粒;3.pMD19-T-INSIG1经SalⅠ和 Bgl Ⅱ的双酶切产物M.DNA marker Ⅲ;1.Amplification product of INSIG1 CDS region by PCR;2.pMD19-T-INSIG1 vector;3.pMD19-T-INSIG1 vector digested by SalⅠand Bgl Ⅱ图1 INSIG1基因的克隆与鉴定Fig.1 Cloning and identification of INSIG1 gene
2.2腺病毒穿梭载体pAdTrack-CMV-INSIG1的构建与鉴定
用SalⅠ和BglⅡ双酶切pMD19-T-INSIG1及pAdTrack-CMV,回收后连接,获得pAdTrack-CMV-INSIG1质粒,再进行双酶切鉴定,琼脂糖凝胶电泳检测得到约9 kb的质粒条带和831 bp的目的基因条带 (图 2),与预期结果相符。另外,测序结果显示插入的序列与设计序列完全一致,表明腺病毒穿梭载体构建成功。
2.3重组腺病毒载体pAdEasy-INSIG1的构建与鉴定
将PmeⅠ线性化的pAdTrack-CMV-INSIG1转入制备好的E.coliBJ5183感受态细胞中,进行同源重组得到重组质粒pAdEasy-INSIG1,PacⅠ酶切鉴定,琼脂糖凝胶电泳检测得到约30 kb的载体条带和4.5 kb的条带 (图 3),并且测序结果与INSIG1基因克隆的测序结果一致,表明重组腺病毒载体构建成功。
2.4腺病毒的包装、扩增与滴度测定
pAdEasy-INSIG1腺病毒重组质粒经PacⅠ酶切线性化后,转染293A细胞,2 d后荧光显微镜下可观察到部分细胞有绿色荧光蛋白表达,7 d后绿色荧光蛋白表达量增多,同时开始出现噬菌斑,13 d后70%的细胞从培养瓶底脱落时收集病毒,反复感染293A细胞2次后,获得高滴度的腺病毒 (图 4)。经过LaSRT法测定其滴度为2×108U·mL-1。
2.5腺病毒感染乳腺上皮细胞最佳MOI值的确定
将不同剂量的腺病毒感染乳腺上皮细胞72 h后,利用显微镜观察荧光强度及细胞形态,发现24孔板中最适宜的病毒接种量为20 μL,MOI值为200 (图 5)。

M.DNA相对分子质量标准;1.pAdTrack-CMV-INSIG1质粒;2.pAdTrack-CMV-INSIG1质粒的SalⅠ和 Bgl Ⅱ双酶切鉴定M.λ/Hind Ⅲ DNA marker;1.pAdTrack-CMV-INSIG1 vector;2.pAdTrack-CMV-INSIG1 digested by SalⅠ and Bgl Ⅱ图2 穿梭载体pAdTrack-CMV-INSIG1的构建Fig.2 Construction of pAdTrack-CMV-INSIG1 vector

M.DNA相对分子质量标准;1.pAdEasy-INSIG1质粒的Pac Ⅰ酶切鉴定 M.λ/Hind Ⅲ DNA marker;1.pAdEasy-INSIG1 digested by Pac Ⅰ图3 pAdEasy-INSIG1重组质粒的Pac Ⅰ酶切鉴定Fig.3 Digestion identification of pAdEasy-INSIG1 by Pac Ⅰ
2.6Ad-INSIG1腺病毒超表达效果检测
山羊乳腺上皮细胞感染Ad-GFP及Ad-INSIG1 48 h后,收集细胞提取总RNA,qRT-PCR检测INSIG1基因的超表达效果。结果表明,与Ad-GFP感染组相比,Ad-INSIG1感染组中INSIG1基因的mRNA表达量显著上调了500倍左右 (P<0.01) (图 6)。
2.7INSIG1基因超表达后对脂质合成相关基因表达的影响
利用2.6中获得的RNA样品,经qRT-PCR检测脂质合成相关基因的表达情况。结果表明,与对照组相比,超表达INSIG1后,SCAP、SREBP1的mRNA表达量均无显著变化 (图 7A);参与脂肪酸从头合成及去饱和的基因中ACCα、FASN及SCD1的mRNA表达量均显著下降,分别为 32%、21% 和23% (P<0.05) (图 7B);参与甘油三酯合成的3个关键基因中GPAM和DGAT2的mRNA表达量显著下降 (P<0.05),AGPAT6的表达量无明显变化 (图 7C);催化甘油三酯水解的关键酶中ATGL的mRNA表达量显著下降 (P<0.05),HSL的mRNA表达量无明显变化 (图 7D)。
2.8INSIG1基因超表达后对细胞中甘油三酯含量的影响
Ad-GFP、Ad-INSIG1感染乳腺上皮细胞48 h后,收集细胞,利用试剂盒检测细胞中甘油三酯的含量。结果表明,与Ad-GFP组相比,Ad-INSIG1组细胞中甘油三酯的含量无明显变化 (图 8)。
3 讨 论
超表达技术是研究基因功能的重要手段之一,腺病毒超表达系统具有转染效率高、感染细胞范围广、病毒滴度高等优点,现已广泛用于基因表达、疫苗制作和基因治疗等领域[20]。本研究采用的是pAdEasy-1系统,两步法构建重组腺病毒超表达载体,利用293A细胞进行病毒包装与扩增,根据腺病毒载体上GFP荧光情况可方便地观察到基因的表达情况及计算病毒的滴度。该方法操作简便,成功率高。第一代腺病毒的成功包装是获得高滴度病毒液的关键。选择生长状态良好的细胞,当细胞汇合度达到70%左右时,转染腺病毒载体。每天更换培养基以满足病毒包装过程中细胞的高营养需要,观察并记录荧光表达情况及细胞生长状态,一般需要12~14 d[21-22]。试验中在第7 天出现噬菌斑,第13 天发现80%细胞变圆脱壁,及时收毒。经过2次扩增后获得高滴度病毒,滴度为2×108U·mL-1。将该病毒感染乳腺上皮细胞48 h后,发现INSIG1基因mRNA的表达量上升约500倍,超表达效果显著,可作为后续进一步功能研究的试验材料。
INSIG1基因最早由K.L.Mohn等在胰岛素处理鼠的肝癌细胞中发现[23],随后从人体肝细胞中克隆得到该基因,命名为胰岛素诱导基因1 (INSIG1)[1]。人INSIG1基因位于染色体7q36,含5个外显子,编码277个氨基酸,含有6个跨膜区[24]。王慧等[11]和C.F.Wu等[25]分别发现山羊、牛的INSIG1基因CDS区长度均为831 bp,编码276个氨基酸,比人少1个氨基酸,且蛋白跨膜区也只有5个。这一差异提示了在蛋白结构及受调控机制方面,反刍动物INSIG1可能与先前对人INSIG1的研究结果存在差异。近年来对乳腺脂质代谢基因网络的研究发现,INSIG1具有重要的作用。在体外培养的脂肪细胞、牛及山羊乳腺上皮细胞中添加过氧化物酶体增殖物激活受体 (Peroxisome proliferator-activated receptor γ,PPAR) 的激动剂罗格列酮,导致INSIG1基因的表达量显著上升,SREBP靶基因表达受到抑制,脂质合成减少[22,26-27];用t10,c12-CLA处理牛的乳腺上皮细胞后,油红O染色显示细胞内脂质积累增加,而INSIG1基因表达受到抑制,推测INSIG1可能介导了外源多不饱和脂肪酸对脂合成的抑制[27]。此外,在对鼠肝脂质代谢的研究中,发现并验证了INSIG1是miR-24的靶基因之一,miR-24能上调INSIG1基因的表达,脂质合成变化则呈相反趋势[28]。这些研究结果表明,INSIG1可能作为中间媒介,在多种因素对机体脂代谢调控中具有非常重要的作用。但这些因子是直接还是间接作用于INSIG1从而影响脂代谢仍需要进一步验证。

A~C.腺病毒感染293A细胞2、7、13 d后的绿色荧光表达情况;A.2 d后出现少量荧光;B.7 d后出现噬菌斑;C.13 d后细胞脱壁变圆;D.高滴度的腺病毒感染293A细胞24 h后绿色荧光表达情况A-C.Fluorescence of 293A cell after transfection of pAdEasy-INSIG1 for 2,7 and 13 d,respectively;A.Slight green fluorescence was observed after 2 d;B.Plaques were observed after 7 d;C.Desquamated cells were observed after 13 d;D.Fluorescence microscopic image of 293A cell infected by high titer virus for 24 hours图4 腺病毒的包装与扩增 100×Fig.4 Adenovirus packaging and amplification 100×

0 h.感染前空白对照;null.不添加病毒的空白对照;72 h Ad-INSIG1.感染重组腺病毒Ad-INSIG1 72 h 后。上图.白光观察图;下图.荧光观察图0 h.Blank control before infection;null.Blank control without adenovirus infection;72 h Ad-INSIG1.Infected with recombinant adenovirus Ad-INSIG1 for 72 h.Upper panel.White light;Lower panel.Fluorescence图5 感染腺病毒前后的乳腺上皮细胞 100×Fig.5 Goat mammary epithelial cells infected with and without adenovirus 100×

Ad-GFP.对照组;Ad-INSIG1.试验组;**.与对照组相比差异极显著P<0.01;下同Ad-GFP.Control group;Ad-INSIG1.Experiment group;**.P<0.01 compared with control group;The same as below图6 INSIG1超表达效果检测Fig.6 Overexpression analysis of INSIG1
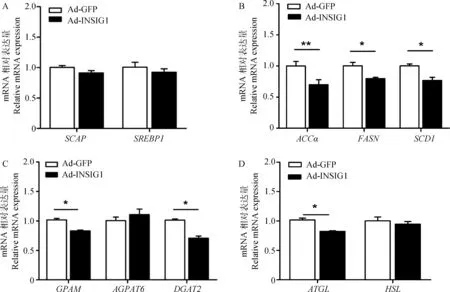
A.SREBP通路;B.脂肪酸从头合成及去饱和;C.甘油三酯合成;D.甘油三酯分解;*.与对照组相比差异显著P<0.05A.SREBP pathway;B.Fatty acid de novo synthesis and desaturation;C.TAG synthesis;D.TAG degradation;*.P<0.05 compared with control group图7 INSIG1基因超表达后对脂质合成相关基因mRNA表达量的影响Fig.7 Effect of INSIG1 gene overexpression on the mRNA expression of lipid synthesis related genes

图8 INSIG1超表达后对乳腺上皮细胞中甘油三酯含量的影响Fig.8 Effect of INSIG1 gene overexpression on the cellular triacylglycerol content
SREBP1是参与脂肪酸代谢调控的重要转录因子,直接调控参与胆固醇、甘油三酯、脂肪酸及磷脂的生物合成及摄取过程中30多个基因的表达[29-31]。SREBP蛋白以非活性的前体形式锚定在内质网上,在SCAP的护送下进入高尔基体,进行蛋白水解活化成核SREBP转入到细胞核内,与靶基因启动子上的固醇调节元件 (SRE) 序列结合而促进其表达[32]。INSIGs能与SCAP结合而抑制SREBP蛋白活化过程,而对其mRNA水平影响不大[3]。本研究发现,在乳腺上皮细胞中INSIG1超表达后,SCAP、SREBP1基因mRNA水平的表达均无显著变化。这一结果与K.Takaishi 等[33]及L.J.Engelking等[34]在活体水平超表达INSIG1试验中的结果一致;同时,在INSIG1基因敲除研究[35]中,也发现SCAP、SREBP1的mRNA表达量无显著变化。另外,ACCα、FASN和SCD1是SREBP的3个重要靶基因,是参与脂肪酸从头合成及去饱和的关键酶。INSIG1超表达后,脂肪酸合成关键基因ACCα、FASN及SCD1的mRNA表达量均显著下降,这与前人研究[33-34]结果一致,表明在乳腺上皮细胞中超表达INSIG1基因能够抑制SREBP蛋白的活化及其靶基因的转录,下调细胞内脂肪酸从头合成及去饱和过程。
甘油三酯是乳脂的主要成分,由长链脂肪酸和甘油形成。在甘油三酯合成的过程中包含3个关键的酶,GPAM参与催化的第一步反应,AGPAT催化甘油三酯 sn-2位置的酯化,DGAT催化二酰甘油加上脂肪酸酰基辅酶A形成三酰甘油,是甘油三酯合成限速酶[36]。细胞中甘油三酯的含量是动态变化的,也可被脂解并用于提供能量和膜脂。ATGL和HSL是催化甘油三酯水解的关键酶。本研究发现,INSIG1超表达后乳腺上皮细胞中GPAM和DGAT2的mRNA表达量显著高于对照组,说明INSIG1对甘油三酯合成具有抑制作用。同时,ATGL基因的mRNA表达量也显著下降。但检测细胞中甘油三酯的含量发现,与Ad-GFP组相比,Ad-INSIG1组中的TAG含量无明显变化,M.R.McFarlane等[35]也发现INSIG1敲除后小鼠血浆及肝中TAG无显著变化。因此,我们推测INSIG1基因超表达后抑制了脂质的合成,同时也导致了细胞中脂动员活动的减弱,最终使细胞内甘油三酯的含量维持动态平衡状态。具体的调控机制还需要进一步的研究探索。
4 结 论
本研究成功构建了奶山羊INSIG1基因重组腺病毒超表达载体,获得了高滴度的重组腺病毒Ad-INSIG1。超表达INSIG1能够下调山羊乳腺上皮细胞脂肪酸及甘油三酯合成相关基因的表达。这些结果将为进一步揭示INSIG1基因在乳脂合成中的功能奠定基础。
[1]PENG Y,SCHWARE E J,LAZAR M A,et al.Cloning,human chromosomal assignment,and adipose and hepatic expression of the CL-6/INSIG1 gene-1[J].Genomics,1997,43(3):278-284.
[2]YABE D,BROWN M S,GOLDSTEIN J L.INSIG2,a second endoplasmic reticulum protein that binds SCAP and blocks export of sterol regulatory element-binding proteins[J].ProcNatlAcadSciUSA,2002,99(20):12753-12758.
[3]YANG T,ESPENSHADE P J,WRIGHT M E,et al.Crucial step in cholesterol homeostasis:sterols promote binding of SCAP to INSIG1,a membrane protein that facilitates retention of SREBPs in ER[J].Cell,2002,110(4):489-500.
[4]SEVER N,YANG T,BROWN M S,et al.Accelerated degradation of HMG CoA reductase mediated by binding of INSIG1 to its sterol-sensing domain[J].MolCell,2003,11(1):25-33.
[5]JO Y,LEE P C,SGUIGNA P V,et al.Sterol-induced degradation of HMG CoA reductase depends on interplay of two Insigs and two ubiquitin ligases,gp78 and Trc8[J].ProcNatlAcadSciUSA,2011,108 (51):20503-20508.
[6]YABE D,KOMURO R,LIANG G,et al.Liver-specific mRNA for Insig-2down-regulated by insulin:implications for fatty acid synthesis[J].ProcNatlAcadSciUSA,2003,100(6):3155-3160.
[7]LEE J N,GONG Y,ZHANG X Y,et al.Proteasemal degradation of ubiquitinated Insig proteins is determined by sefine residues flanking ubiquitinated lysines[J].ProcNatlAcadSciUSA,2006,103(13):4958-4963.
[8]KRAPIVNER S,POPOV S,CHERNOGUBOVA E,et al.Insulin-induced gene 2 involvement in human adipocyte metabolism and body weight regulation[J].JClinEndocrinolMetab,2008,93(5):1995-2001.
[9]BIONAZ M,LOOR J J.Gene networks driving bovine milk fat synthesis during the lactatcycle[J].BMCGenomics,2008,9(366):1-21.
[10]HAN L Q,LI H J,WANG Y Y,et al.mRNA abundance and expression of SLC27A,ACC,SCD,FADS,LPIN,INSIG,and PPARGC1 gene isoforms in mouse mammary glands during the lactation cycle[J].GenetMolRes,2010,9(2):1250-1257.
[11]王慧,罗军,席利萌,等.西农萨能奶山羊INSIG1编码区的克隆序列特征分析和组织表达[J].西北农林科技大学学报,2013,41(8):24-30.
WANG H,LUO J,XI L M,et al.Cloning,sequence characters and tissue expression pattern of INSIG-1 in Saanen Dairy goat[J].JournalofNorthwestA&FUniversity,2013,41(8):24-30.(in Chinese)
[12]朱江江.奶山羊脂肪酸合酶基因乙酰/丙二酸单酰转移酶区域的过表达研究[D].杨凌:西北农林科技大学,2011.
ZHU J J.Overexpression of MAT domain of fatty acid synthase (FASN) gene in dairy goat[D].Yangling:Northwest Agriculture & Forestry University,2011.(in Chinese)
[13]王伟,罗军,赵旺生,等.西农萨能羊FAS基因shRNA序列筛选及其腺病毒载体的构建[J].西北农业学报,2010,19(3):6-12.
WANG W,LUO J,ZHANG W S,et al.Screening of shRNA sequence target Xinong Saanen Goat FAS gene and the construction of recombinant adenovirus vector[J].ActaAgriculturaeBoreali-occidentalisSinica,2010,19(3):6-12.(in Chinese)
[14]江千里,王健民,温丽敏,等.批量快速测定法测定标志基因为GFP的重组病毒滴度[J].第二军医大学学报,2002,23(9):1034-1035.
JIANG Q L,WANG J M,WEN L M,et al.Establishing a realtime large-scale recombinant virus titering method taking GFP as marker gene[J].AcademicJournalofSecondMilitaryMedicalUniversity,2002,23(9):1034-1035.(in Chinese)
[15]孙雨婷,罗军,朱江江,等.奶山羊短链脂肪酸受体GPR41基因的克隆及组织表达分析[J].畜牧兽医学报,2012,43(2):319-323.
SUN Y T,LUO J,ZHU J J,et al.Cloning and analysision tissue expression ofGPR41 gene in dairy goat[J].ActaVeterinariaetZootechnicaSinica,2012,43(2):319-323.(in Chinese)
[16]BIONAZ M,LOOR J J.Identification of reference genes for quantitative real-time PCR in the bovine mammary gland during the lactation cycle[J].PhysiolGenomics,2007,29(3):312-319.
[17]KADEGOWDA A K,BIONAZ M,THERING B,et al.Identification of internal control genes for quantitative polymerase chainreaction in mammary tissue of lactating cows receiving lipid supplements[J].JDairySci,2009,92(5):2007-2019.
[18]BONNET M,BERNARD L,BES S,et al.Selection of reference genes for quantitative real-time PCR normalisation in adipose tissue,muscle,liver and mammary gland from ruminants[J].Animal,2013,7(8):1344-1353.
[19]LI J,JUN L,WANG H,et al.Adipose triglyceride lipase regulates lipid metabolism in dairy goat mammary epithelial cells[J].Gene,2015,554(1):125-130.
[20]LUO J,DENG Z L,LUO X,et a1.Aprotocol for rapid generation of recombinant adenoviruses using the AdEasy system[J].NatProtoc,2007,2(5):1236-1247.
[21]吴敏,罗军,朱江江,等.奶山羊超长链脂肪酸延伸酶6基因ELOVL6的克隆及其对4个脂肪酸代谢基因表达水平的影响[J].农业技术生物学报,2014,22(6):672-680.
WU M,LUO J,ZHU J J,et al.Cloning of elongase of very long chain fatty acids 6 gene(ELOVL 6) and its effect on four genes expression involved in fatty acid metabolism in dairy goat[J].JournalofAgricultureTechnology,2014,22(6):672-680.(in Chinese)
[22]SHI H B,ZHAO W S,LUO J,et al.Peroxisome proliferator-activated receptor γ1 and γ2 isoforms alter lipogenic gene networks in goat mammary epithelial cells to different extents[J].JDairySci,2014,97(9):5437-5447.
[23]MOHN K L,LAZ T M,HSU J C,et al.The immediate-early growth response in regenerating liver and insulin-stimulated H-35 cells:comparison with serum-stimulated 3T3 cells and identification of 41 novel immediate-early genes[J].MolCellBiol,1991,11(1):381-390.
[24]FERAMISCO J D,GOLDSTEIN J L,BROWN M S,et al.Membrane topology of human INSIG-1,a protein regtllslor of lipid synthesis[J].JBidChem,2004,279(9):8487-8496.
[25]WU C F,LIU L X,HUO J L,et al.Isolation,sequence characterization,and tissue transcription profiles of two novel buffalo genes:INSIG1 and INSIG2[J].TropAnimHealthProd,2014,46(1):33-41.
[26]HEIDI R,KAST-WOELBERN,SHARON L,et al.Rosiglitazone induction of INSIG1 in white adipose tissue reveals a novel interplay of peroxisome proliferator-activated receptor γ and sterol regulatory element-binding protein in the regulation of adipogenesis[J].JBiolChem,2004,279(23):23908-23915.
[27]KADEGOWDA A K,BIONAZM,PIPEROVA L S,et al.Peroxisome proliferator-activated receptor-gamma activation and long-chain fatty acids alter lipogenic genenetworks in bovine mammary epithelial cells to various extents[J].JDairySci,2009,92(9):4276-4289.
[28]RAYMOND N G,WU H,XIAO H,et al.Inhibition of microRNA-24 expression in liver prevents hepatic lipid accumulation and hyperlipidemia[J].Hepatology,2014,60(2):554-564.
[29]BROWN M S,GOLDSTEIN J L.A proteolytic pathway that controls the cholesterol content of membranes,cells,and blood[J].ProcNatlAcadSciUSA,1999,96(20):11041-11048.
[30]HORTON J D,GOLDSTEIN J L,BROWN M S.SREBPs:activators of the complete program of cholesterol and fatty acid synthesis in the liver[J].JClinInvest,2002,109(9):25-31.
[31]HORTON J D,SHAH N A,WARRINGTON J A,et a1.Combined analysis of oligo-nucleotide microarray data from transgenie and knockout mice identifies direct SREBP target genes[J].ProcNatlAcadSciUSA,2003,100(21):12027-12032.
[32]NOHTURFFT A,YABE D,GOLDSTEIN,et a1.Regulated step in cholesterol feedback localized to budding of SCAP from ER membranes[J].Cell,2000,102(3):315-323.
[33]TAKAISHI K,DUPLOMB L,WANG M Y,et al.Hepatic insig-1 or-2 overexpress-ion reduces lipogenesis in obese Zucker diabetic fatty rats and in fasted/refed normal rats[J].ProcNatlAcadSciUSA,2004,101(18):7106-7111.
[34]ENGELKING L J,KURIYAMA H,HAMMER R E,et al.Overexpression of Insig-1 in the livers of transgenic mice inhibits SREBP processing and reduces insulin-stimulated lipog-enesis[J].JClinInvest,2004,113(8):1168-1175.
[35]MCFARLANE M R,LIANG G,ENGELKING L J.Insig proteins mediate feedback inhibition of cholesterol synthesis in the intestine[J].JBiolChem,2014,289(4):2148-2156.
[36]ABEN I F,DEGANO L,CALZA F,et al.Milk quality and automatic milking:fat globule size,natural creaming,and lipolysis[J].JDairySci,2005,88(10):3519-3529.
(编辑郭云雁)
Effect ofINSIG1 Overexpression on the Lipid Synthesis in Goat Mammary Gland Epithelial Cells
WANG Miao,LUO Jun*,XU Hui-fen,ZHU Jiang-jiang,HE Qiu-ya,YAO Da-wei,SHI Huai-ping
(ShanxiKeyLaboratoryofMolecularBiologyforAgriculture,CollegeofAnimalScienceandTechnology,NorthwestA&FUniversity,Yangling712100,China)
The aim of this study was to construct an insulin-induced gene-1 (INSIG1) recombinant adenovirus overexpression vector of Xinong Saanen dairy goat,and determine its effect on lipid synthesis in goat mammary epithelial cells (GMEC),to lay the foundation for its functional study in regulating lipid synthesis in goat mammary gland.The primers were designed according to theINSIG1 sequence in GenBank (accession number:JQ665439),then its coding sequences (CDS) was cloned by PCR.The gene fragments were inserted into shuttle vector pAdTrack-CMV to obtain pAdTrack-CMV-INSIG1 vector.After being linearized byPmeⅠ,pAdTrack-CMV-INSIG1 vector was transformed intoEscherichiacoliBJ5183 competent cells to obtain the recombinant adenovirus overexpression vector pAdEasy-INSIG1 by homologous recombination.Next,the pAdEasy-INSIG1 vector,linearized byPacⅠ,was transfected into 293A cells for viral packaging and amplification.LaSRT was used for titer assay.Finally,after infecting the goat mammary gland epithelial cells(GMEC) with Ad-INSIG1 recombinant adenovirus,qRT-PCR was used to measure the mRNA expression ofINSIG1 and genes related to lipid synthesis,and the GPO-Trinder enzyme reaction was used to measure the cellular triacylglycerol (TAG) content.The result showed thatINSIG1 overexpression recombinant adenovirus vector was successfully constructed,and the recombinant adenovirus Ad-INSIG1 with a high titer of 2×108U·mL-1was obtained.Compared with Ad-GFP infected group,the mRNA expression ofINSIG1 increased by about 500-fold after GMEC incubated with Ad-INSIG1 for 48 h.No obvious changes were observed on the mRNA expression of sterol regulatory element binding protein 1 (SREBP1) and SREBP cleavage-activating protein (SCAP),however,there was a significant decrease in the expression genes related to fatty aciddenovosynthesis and desaturasion:acetyl-CoA carboxylas α (ACCα),fatty acid synthase (FASN) and stearoyl-CoA desaturase 1 (SCD1) (P<0.05).Among the 3 key genes involved in TAG synthesis,the transcript abundance of glycerol-3-phosphate acyltransferase (GPAM) and diacylgycerol acyltransferase 2 (DGAT2) were significantly decreased (P<0.05),and 1-acyl-sn-glycerol-3-phosphate acyltransferase 6 (AGPAT6) had no obvious change.Meanwhile,the mRNA expression of adipose triacylglycerol lipase (ATGL),an enzyme catalyzes the initial step of TAG hydrolysis,also decreased significantly (P<0.05).No significant difference was found in the cellular TAG content between 2 groups.In conclusion,INSIG1 can inhibit the expression of genes related to lipid synthesis in GMEC,and may have a regulatory function on lipid synthesis in goat mammary gland.
GMEC;INSIG1;overexpression;lipid synthesis
10.11843/j.issn.0366-6964.2016.09.008
2015-12-21
国家自然科学基金项目(31372281);国家转基因新品种培育重大专项(2014ZX08009-051B)
王苗(1992-),女,湖北汉川人,硕士生,主要从事动物遗传育种与繁殖研究,E-mail:wangmiao9254@sina.com
罗军,教授,博士生导师,主要从事动物遗传育种研究,E-mail:luojun1@yahoo.com
S827;S813.3
A
0366-6964(2016)09-1806-11
