室间隔缺损患者循环miRNA表达差异研究
2016-10-28许美玲吴雪嵩李金栋
许美玲 吴雪嵩 朱 凯 李金栋 王 倩 纪 龙
(1.泰安市泰山区疾病预防控制中心,山东 泰安 271000;2.泰山医学院公共卫生学院,山东 泰安 271016)
室间隔缺损患者循环miRNA表达差异研究
许美玲1吴雪嵩2朱凯2李金栋2王倩2纪龙2
(1.泰安市泰山区疾病预防控制中心,山东 泰安271000;2.泰山医学院公共卫生学院,山东 泰安271016)
目的先天性心脏病(CHD)是环境因素和遗传因素共同作用的结果。室间隔缺损(VSD)是CHD中最常见的一种。研究表明,microRNAs(miRNAs)与CHD的发生发展关系密切,但目前缺乏有关miRNAs与VSD的研究报道。本研究旨在通过血清差异miRNA的表达研究阐明miRNA与VSD发生发展的关系。方法采用miRNA表达谱芯片技术进行VSD患者血清miRNA表达差异分析,并对结果进行实时定量PCR(Real-time PCR)验证,进而对miRNA调控的靶基因进行生物信息学分析。结果通过miRNA芯片在VSD患者和对照血清样本中检测miRNA差异表达,筛选出36个表达差异的miRNA(P<0.05),其中21个miRNA在VSD患者表达显著下调,15个表达显著上调。经过Real-time PCR验证,证实8个miRNAs差异表达有意义。通过生物信息学预测出447个靶基因,经GO分析富集度分值(enrichment score value)最大的生物过程(biological process)为心脏右心室形态发生(cardiac right ventricle morphogenesis),对应的差异表达基因为NOTCH1、HAND1、ZFPM2和GATA3,调控这些基因的miRNA可能包括hsa-let-7e-5p、hsa-miR-222-3p和hsa-miR-433。结论通过微阵列技术分析和RT-PCR验证,筛选出8个差异表达miRNA,为后续实验提供了可供分析的miRNA。利用3个数据库及GO分析, 发现hsa-let-7e-5p、hsa-miR-222-3p和hsa-miR-433的靶基因分别为NOTCH1、HAND1、ZFPM2和GATA3,为进一步在基因水平研究VSD的发病机制提供了新的突破口,为将来可能实现的基因诊断、基因干预治疗以及疾病的预防奠定了一定的实验基础。
室间隔缺损; 微小RNA;NOTCH1;HAND1;ZFPM2;GATA3
先天性心脏病(congenital heart disease,CHD)是胎儿期心脏及大血管发育异常而致的心血管畸形。国外报道先心病发病率为5.4/1000~16.1/1000[1],亚洲国家最高,约为9.3%[2]。我国的监测报告显示CHD的发生率为32.74/10000[3]。CHD是小儿最常见的先天畸形之一,也是导致新生儿及婴儿死亡的最重要的原因[4]。CHD病因复杂,至今尚未完全明确。多数CHD (90%)可能与遗传因素和环境因素有关[5]。常见的CHD主要有房间隔缺损(atrial septal defect, ASD)、室间隔缺损(ventricular septal defect, VSD)、动脉导管未闭(patent ductus arteriosus, PDA)、法洛氏四联症(tetralogy of Fallot, TOF)、大动脉转位(transposition of the great arteries, TGA)、肺动脉闭锁(pulmonary valve atresia, PA)、主动脉缩窄(coarctation of the aorta, COA)和三尖瓣闭锁(tricuspid atresia, TA)等。VSD是较常见的先天性心脏结构畸形之一,约占CHD的20%[6],主要是由于左右心室间隔的缺损导致了左右心室的异常交通。尽管其胚胎学和生理学已经阐明,但其病因与发病机制目前仍不十分清楚[7]。微小RNA( microRNAs,miRNA)是一种转录后调节因子,长度介于18~22个核苷酸的单链、非编码的小分子RNA。可与靶基因mRNA的3'非编码区相互配对结合,在转录后水平负调控靶基因的表达。 miRNA调控细胞的生长、代谢、分化和凋亡,进而参与生物体的生长发育[8]。miRNA除了在细胞内有作用,体液中稳定表达的miRNA在心血管疾病、肿瘤发生等过程中也起了重要作用,循环miRNA可作为潜在的生物标志物用于疾病的诊断[9-10]。近年研究表明,miRNA在心血管疾病的发生与发展方面发挥着重要的功能,参与调控胚胎心脏的发育,在心脏形态发生、心肌细胞生长及分化过程中发挥着极其重要的作用[11]。miRNAs表达谱芯片是筛选与研究疾病发生、发展相关特异miRNAs的重要方法之一,本研究通过采用miRNAs表达谱芯片对VSD患者血浆miRNA表达进行筛选,找出其与正常对照人群差异表达的miRNA,并且运用RT-PCR技术对部分明显差异表达的miRNA进行检测,验证芯片结果的可靠性,运用靶基因预测软件对差异表达的miRNA下游调控的靶基因进行预测,进一步分析其生物学功能,以期为阐明VSD在分子水平上的发病机制提供线索。
1 资料与方法
1.1研究对象
采用以医院为基础的病例—对照研究方法。病例选自2012年8月—2013年6月在济南市儿童医院就诊和治疗的心血管外科患者。分流口直径5~8 mm,超声及术中直视下均明确诊断为CHD,所有患者均为VSD,且不合并其他畸形;对照组为同期上述医院住院治疗的骨外科外伤患者、无CHD遗传家族史并且与病例来自相同地区。按照1∶1匹配收集VSD患者和健康对照各3例进行miRNAs表达谱芯片研究;选取20例VSD患者和15例健康对照人群,以RT-PCR法进行差异miRNA验证。该研究经校伦理委员会批准,所有入选个体均由监护人签署知情同意书,标本用于本实验研究。
1.2样本采集
于入院次日清晨空腹采集研究对象静脉血标本5 ml,EDTA采血管收集,收集的全血静置10 min,1500 r/min离心10 min,上层透明淡黄色液体即为血浆,收集后分装入无RNase的Ep管中,存于-80℃备用。
1.3血浆RNA提取及质量检测
血浆总RNA提取:应用TRIzol and miRNeasy mini kit(美国Qiagen公司)试剂盒提取血浆总RNA。琼脂糖凝胶电泳法检测RNA质量,紫外分光光度仪测定总RNA在260 nm和280 nm处吸收值的比值(OD值=A260/A280)可直接衡量DNA的纯度。OD值在1.8~2.1之间,认为质量合格。实验步骤如下:取250 ml血浆加入750 ml TRI Reagent提取液和20 ml冰醋酸,手动剧烈振荡管体至混匀,室温放置5 min,再加0.2 ml氯仿,强烈震荡15 s,室温放置2~3 min,于4 ℃,12000 r/min离心15 min,将上层水相转入新的离心管内,加入500 ml异丙醇,混匀,15~30 ℃孵育10 min。于4 ℃,12000 r/min离心10 min,弃上清。加1 ml 75%乙醇洗涤沉淀,于4 ℃,7500 r/min离心5 min,自然晾干。
1.4miRNA分离及标记
采用miRCURYTMHy3TM/Hy5TMPower labeling kit (Exiqon, Vedbaek, Denmark)标记试剂盒,用标记酶将Hy3TM荧光基团标记在每份样品中miRNA的3'端,得到用于芯片杂交的荧光探针。分离富集miRNA,分离过程按照试剂盒说明进行。
l)将溶于2 μl水的RNA、1 μl CIP buffer及CIP混合,置于37 ℃孵育30 min,于95 ℃孵育5 min以终止反应;
2)将3 μl标记缓冲液、l.5 μl荧光标记物(Hy3TM)、2 μl DMSO,2 μl标记酶加入上述步骤的混合液中。置于16 ℃孵育1 h进行标记反应,然后于65 ℃孵育15 min以终止反应。
1.5miRNA芯片杂交
选择miRCURYTMLNA Array (v.18.0) (Exiqon) 表达谱芯片,总的25 μl被标记Hy3TM的样品混合液与25 μl杂交缓冲液混合,置于95 ℃变性2 min,冰上孵育2 min,然后使用phalanTM热收缩杂交袋将标记好的探针与miRCURYTM芯片于56 ℃条件下杂交16~20 h。
1.6芯片扫描及图像数据处理
采用GenePix 4000B microarray scanner (Axon Instruments, Foster City,CA)扫描仪进行芯片扫描,采集荧光强度。采用Genepix Pro 6.0 software (Axon)图像分析软件对采集的杂交图像进行数字化分析,用每个探针的原始信号值减去该点的背景值得到每个探针的修正值;用同一批次实验中在每张芯片上修正值≥50的非对照探针做标准化,以这部分探针中值作为标准化因子对整张芯片的点做标准化处理。经过标准后处理后,分别用VSD组/对照组计算相应miRNAs分子的差异倍数,挑选差异大于1.5或小于0.67的miRNA作为有表达差异的miRNAs。
1.7实时荧光定量PCR验证部分差异表达
随机挑选表达差异的8个miRNAs进行验证,以has-miR-93为内参照,引物参照GenBank数据库基因序列进行设计。PCR 反应条件:取100 ng总miRNAs加入20 μl逆转录体系,16 ℃30 min,42 ℃40 min,85 ℃5 min,反应结束后,将其放在冰上待用。再取2 μl cDNA加入8 μl实时PCR体系,扩增反应程序为:95 ℃预变性10 min;95 ℃10 s,60 ℃60 s,95 ℃15 s,测定荧光强度,重复40个循环;从60 ℃缓慢加热到99 ℃,绘制溶解曲线;数据采用2-△△CT法进行分析。
1.8miRNAs靶基因预测
采用targetscan (http://www.microrna.org/microrna/home.do),mirbase(http://pictar.mdc-berlin.de),和Miranda (http://www.targetscan.org/vert_60/)3个数据库,输入VSD组和正常对照组表达差异的miRNAs,预测得到差异miRNA的靶基因,然后取3个数据库预测共有的靶基因(交集部分)。
1.9miRNA与靶基因的相关性分析
利用Gene Ontology数据库对上述差异miRNA调控的靶基因进行注释,得到靶基因的所有功能注释,采用Fisher精确检验计算每个功能的显著性水平(P-value),根据显著性水平进行筛选,得到靶基因的显著性功能及功能所属的基因。筛选标准:P≤0.05,P值越低,GO越显著。
1.10统计学方法

2 结 果
2.1人群特征
进行miRNAs表达谱芯片研究的3例VSD患者和3例健康对照组人群按照性别、年龄、出生地、出生方式、母亲孕期情况、遗传家族史等方面1∶1配对。另外20例VSD患者和15例健康对照人群按照年龄(P=0.354)、性别(P=0.573)进行匹配,基本情况见表1。

表1 病例组和对照组基本情况
2.2血清miRNA微阵列芯片表达谱分析结果
利用miRCURY LNA Array (version 11.0) miRNA芯片技术检测了3例VSD患者和3例健康对照血浆中的miRNA表达特征,初步确定了VSD差异miRNA表达谱。微阵列芯片杂交结果显示:与对照组相比,有36个表达差异的miRNA(P﹤0.05),其中21个miRNA在VSD患者表达显著下调,表达显著上调的miRNA有15个,见表2、图1。

表2 VSD患者表达差异的miRNAs

图1microRNA芯片筛选VSD血浆microRNA差异表达热图
2.3差异表达miRNA的RT-PCR定量检测分析
结合生物信息学分析,随机选择8个差异表达的miRNA(其中表达下调的miRNA共7个,分别为hsa-let-7e-5p、hsa-miR-155-5p、hsa-miR-222-3p、hsa-miR-379-5p、hsa-miR-409-3p、hsa-miR-433、hsa-miR-487b;表达上调miRNA共1个,为hsa-miR-498)运用RT-PCR技术进一步进行检测,结果与miRNA芯片结果基本相符合,提示miRNA芯片结果能正确反映VSD患者和对照组人群血浆中miRNA的表达差异。同时,更精确的测定了上述miRNA表达水平的变化。即hsa-let-7e-5p表达下调0.27倍(P=0.024),hsa-miR-155-5p表达下调0.45倍(P=0.033),hsa-miR-222-3p表达下调0.51倍(P﹤0.001),hsa-miR-379-5p表达下调0.18倍(P=0.013),hsa-miR-409-3p表达下调0.56倍(P﹤0.001),hsa-miR-433表达下调0.10倍(P﹤0.001),hsa-miR-487b表达下调0.24倍(P=0.025),hsa-miR-498表达上调4.33倍(P﹤0.001),图2。
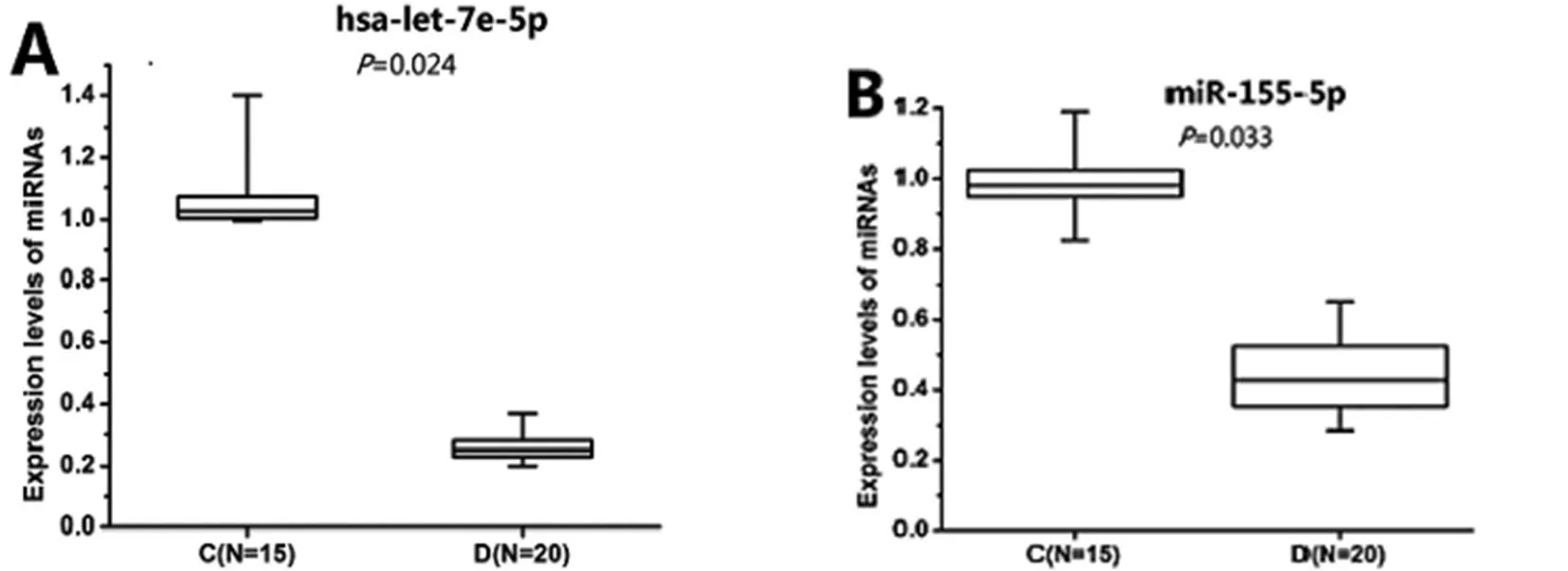


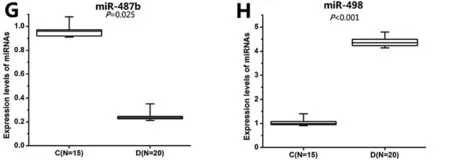
图2 20例VSD与15例对照8种血浆microRNAs相对表达量
2.4差异miRNAs靶基因预测
对VSD组和正常对照组表达差异的36个miRNAs靶基因进行预测。研究发现与VSD的发生可能有关的15个差异表达miRNAs的447个靶基因,并且大部分差异miRNAs的靶基因中含有与心脏发育直接相关的关键基因(HAND1、NKX2-4、TBX-1、MAP2K4、GATA4、NOTCH1、ZFPM2等)。15个差异表达miRNAs的代表性靶基因,见表3。

表3 VSD患者36个差异表达miRNAs的靶基因预测
2.5靶基因GO分析
筛选出的447个可能参与VSD发病机制的潜在功能靶基因,通过GO分析生物学过程后揭示了这些差异表达的miRNA在cardiac right ventricle morphogenesis等过程中富集,排名前十的生物学过程,见表4,图3。富集度最高的GO相关靶基因为NOTCH1、HAND1、ZFPM2、GATA3与cardiac right ventricle morphogenesis发育有关,并且调控这4个基因的miRNA分别为hsa-let-7e-5p、hsa-miR-222-3p和hsa-miR-433,在VSD患者血浆中表达均下调(P<0.05),见表3。
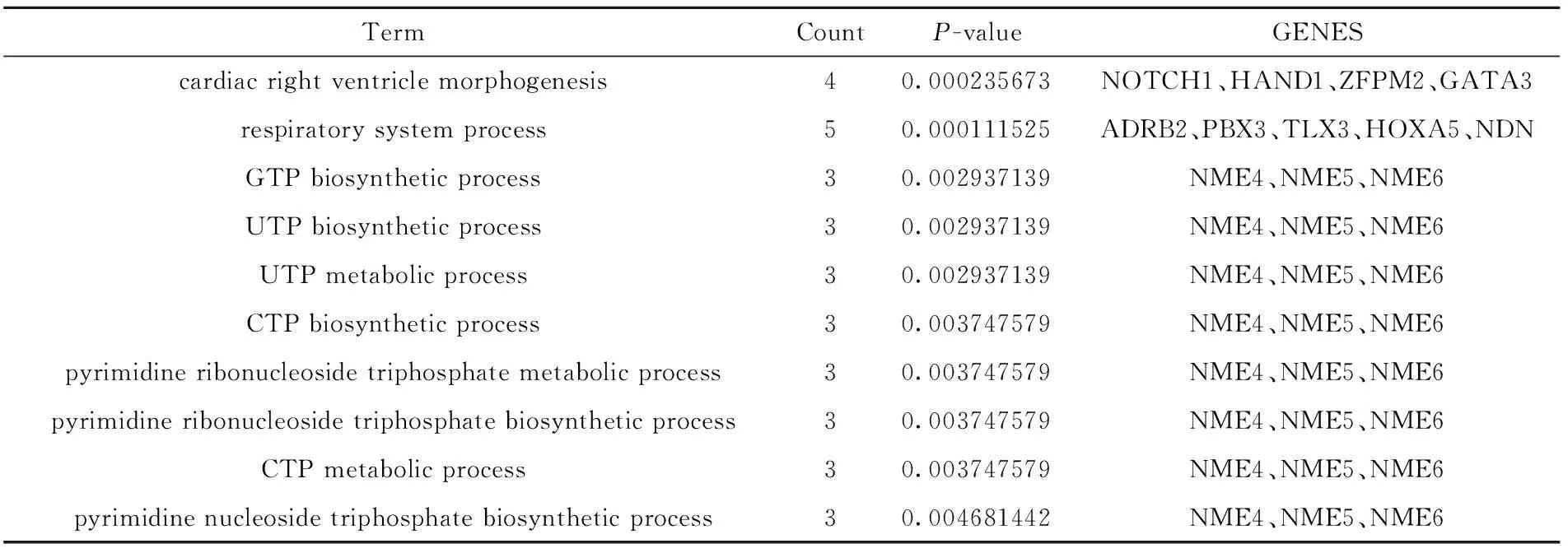
表4 miRNA的预测靶基因Gene Ontology分析*
*表中仅列出功能显著性排名前10的GO terms。

图3 预测靶基因富集度居前的生物学过程条目(部分)
2.6构建miRNA与靶基因的调控网络
对靶基因通过显著性功能分析,构建miRNA与靶基因的调控网络。取差异miRNA与显著性功能所属的靶基因,构建差异表达的miRNA与调控VSD有关基因的调控网络,见图4。得到网络核心调控的关键miRNA(15个)和被调控的核心靶基因,并提示这些miRNA可能与VSD相关。

图4microRNA-Gene-Network调控网络示意图
3 讨 论
miRNA在很多生物的生理和病理过程中发挥重要作用,miRNA基因数目只为蛋白质编码基因数目的1%,但是通过复杂的调控网络可以调控大约30%的人类基因,影响细胞发育、增殖和凋亡,并与许多疾病相关。miRNA与许多先CHD的发病机制有关[12],并且在心脏相关疾病的诊断和治疗中发挥越来越重要的作用[13-14]。有研究表明,miR-1和miR-133控制心肌及骨骼的发育[15]。Huang等[16]研究发现miRNAs在颅面部和心血管系统发生中起核心作用,如发生变异,将导致神经-颅面部-心脏先天性缺陷。VSD是先天性心脏病中最常见的一种类型,虽已查明多个可能与VSD有关的miRNA调控的靶基因,如在敲除小鼠Dicer基因后,小鼠出现VSD表型,发现miRNA-21和miRNA-181a在VSD的发生、发展中起着重要作用[17],但与VSD 发生、发展相关的miRNAs数量繁多,对其表达调控机制仍不十分清楚,因此仍需进一步的深入研究和发掘。最新研究发现,循环miRNA在血清中非常稳定,不能被血液中降解其他RNA分子的酶降解,可以作为先天性心脏病敏感和特异的生物学标记[18- 19]。miRNAs表达谱芯片是筛选与研究疾病发生、发展相关特异miRNAs的重要方法之一,本研究采用miRNAs表达谱芯片筛选VSD患者和正常对照人群中表达差异的miRNAs,并初步确定了这些miRNA调控的靶基因,为阐明VSD在分子水平上的发病机制提供可靠线索。
本研究通过检测3例VSD患者血浆中差异表达的miRNA,发现与正常对照人群(3例)相比存在36个差异表达的miRNA,其中21个表达下调,15个表达上调,见表1。采用RT-PCR技术对其中8个miRNA进行验证,结果表明,8个miRNA与芯片结果基本相符。预测出的447个靶基因中,大部分靶基因是与心脏发育有关。进一步对447个靶基因进行功能显著性分析。富集度最高的GO相关靶基因功能表现为调控右心室形态发育,其关键基因有包括NOTCH1、HAND1、ZFPM2和GATA3等4个。调控这些基因的3个miRNA分别为hsa-let-7e-5p、hsa-miR-222-3p和hsa-miR-433,在VSD患者血浆中表达均下调(P﹤0.05),见表3。
分析结果提示,hsa-miR-433可能调控的靶基因为NOTCH1和GATA3。已有研究表明,NOTCH1与心室发育密切相关,心室发育过程中心肌前体细胞分化为外部致密层和内部肌小梁层,在胚胎发育第9.5天发现NOTCH1于小梁层底部的心内膜表达,而敲除NOTCH1大鼠胚胎肌小梁形成缺陷,且失去特征标志物表达能力[20]。余章斌等[21]通过芯片筛选和定量PCR表明VSD胎儿心肌组织NOTCH1表达下调,从而影响下游的效应基因Hes1,其表达也下调,表明NOTCH信号通路在胎儿VSD中起重要作用。
GATA家族目前有六个基因(GATA1-6)。 GATA4是一个在心脏发生调控网络中起调控者作用的转录因子,参与心脏的正常发育及功能基因的表达[22]。GATA4 基因正常水平的表达对心脏的环化、心房、心室发育、房室瓣形成及动脉干分隔都起着关键作用,是心脏发育成熟并发挥其正常生理功能所必需的[23]。对斑马鱼的研究表明,GATA4不仅参与心脏的形成,而且对心肌受损后的修复与重建也起着重要作用[24]。GATA3是该家族中另一重要的转录因子,特异性表达于Th2细胞中,在Th2细胞的增殖、分化和发育中起关键作用[25],有关GATA3与VSD关系的研究极少,金立臣等[26]研究发现,在VSD合并肺动脉高压(pulmonary arterial hypertension, PAH)组患儿中GATA3基因表达比单纯PAH患儿显著降低(P﹤0.05),提示GATA3基因的低表达可能与VSD的发生有关。
hsa-miR-222-3p参与调控的靶基因ZFPM2,又名FOG2(transeription factor GATA4, FOG2),同属GATA转录因子家族,在心脏发育过程中必不可少。FOG2基因与GATA4基因发生特异性相互作用,同时抑制GATA4依赖性转录过程[27]。FOG2基因敲出小鼠于胚胎期 E13.5天左右因心脏缺陷导致胚胎期致死,其心脏发育缺陷主要包括心肌发育不良心室壁变薄、大的室间隔缺损、严重的房室心内膜垫缺失 、主动脉骑跨和严重的冠状动脉血管丛发育不全[28],可能与VSD的发生有关。
在预测的4个靶基因中,HAND1是HAND家族的重要成员之一,和HAND2一起作为参与心脏发育调控的转录因子,与心室肌细胞分化成形、传导束、心脏流出道、心外膜及心内膜的发育密切相关[29]。HAND1在调控过程中通过启动子区域的四个结合位点与GATA4相结合,共同发挥调控作用[30]。Cheng等在中国人群VSD患者中也发现HAND1基因存在突变,提示HAND1可能参与房室间隔缺损的发生[31]。Zhao等[32]研究发现,miR-1可以在恰当的时候阻止HAND2蛋白的合成,使心脏得以正常发育。但目前尚无有关调控HAND1的miRNA与VSD的研究报道。我们在实验中发现,hsa-let-7e-5p在VSD发生过程中显著下调,对芯片结果进行分析发现:与对照组相比,hsa-let-7e-5p表达水平下降了0.27倍(P=0.024),图3。提示hsa-let-7e-5p很有可能参与VSD进程且发挥了重要的调控作用。推测这些表达差异的miRNAs与右心室形态发育特异的转录因子结合,从而引起VSD发生,但是这些信息学分析需要进一步的荧光素酶报告基因结合实验进行鉴定。
多数CHD属于多基因遗传病,其发生涉及多个miRNA调控的多种基因的表达,这些基因相互影响形成调控网络[33],从而影响心脏的发育。本研究筛选到VSD患者和正常对照人群血浆差异表达miRNAs,首次报道hsa-let-7e-5p、hsa-miR-222-3p和hsa-miR-433可能通过与心脏特异的靶基因结合,从而引起心脏发育异常,这些靶基因如NOTCH1、HAND1、ZFPM2和GATA3等可能在右心室形态发育中相互影响,从而导致VSD发生(见图5)。随着特异性心脏发育相关miRNAs的进一步深入研究,结合靶基因的功能性研究,以及差异miRNA作为无创性诊断标志物,为进一步研究VSD的病因、以及早期诊断和治疗提供了新的思路,具有重大的临床意义。
[1] Arias López I,Martínez Tallo E, Campo Sanpedro F, et al.Incidence and clinical characteristics of congenital heart disease in Badajoz Province, Span[J].An Pediatr (Barc), 2008, 69(1): 23-27.
[2]Van der Linde D, Konings EE, Slager MA, et al. Birth prevalence of congenital heart disease worldwide: a systematic review and meta-analysis[J]. J Am Coll Cardiol, 2011, 58(21): 2241-2247.
[3]Ministry of Health of the People's Republic of China. The maternal and child health monitoring and annals communication[R]. Beijing: Maternal and Child Health and Community Health Division of the Ministry of Health, 2011, (3): 4-9.
[4]Van der Bom T, Zwinderman AH, Meijboom FJ, et al. The changing epidemiology of congenital heart disease[J]. Nat Rev Cardiol,2011,8: 50-60.
[5]Arnold C, Christopher PH, Bemadette M. March of Dimes: Global Report on Birth Defects, the Hidden Toll of Dying and Disabled Children[M]. New York: White Plains, 2006.
[6]Gao Y, Huang GY. Advances in the etiology of congenital heart disease and epidemiological studies[J].Chin J Evid Based Pediatr, 2008, 3(3): 213-222.
[7]Penny DJ,Vick GW 3rd.Ventricular septal defect[J]. Lancet, 2011, 377( 9771): 1103 -1112.
[8]BartelD P. MicroRNAs: genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281-297.
[9]Cheng Y, Tan N, Yang J, et al. A translational study of circulating cell-free microRNA-1 in acute myocardial infarction[J]. Clin Sci (Lond), 2010, 119(2): 87-95.
[10]Turchinovich A, Weiz L, Langheinz A, et al. Characterization of extracellular circulating microRNA[J]. Nucleic Acids Res, 2011, 39: 7223-7233.
[11]LI FQ, Sun TT. Research Advances of MicroRNA in Cardiovascular Disease and Application of the Relevant Treatment[J]. Medical Recapitulate, Jan, 2013, 19(1): 43-45.
[12]Yu ZB, Han SP, Guo XR. Research on miRNA related to heart development[J]. J Med Mol Biol, 2010, 7(1): 66-69.
[13]Yu Z, Han S, Hu P, et al.Potential role of maternal serum microRNAs as a biomarker for fetal congenital heart defects[J].Med Hypotheses, 2011, 76(3): 424-426.
[14]Huang ZP, Neppl RL Jr, Wang DZ.Application of microRNA in cardiac and skeletal muscle disease gene therapy[J].Methods Mol Biol, 2011, 709: 197-210.
[15]Zhao Y, Ransom JF, Li A, et al. Dysregulation of cardiogenesis, cardiac conduction, and cell cycle in mice lacking miRNA-1-2[J]. Cell, 2007, 129(2): 303-17.
[16]Zhan-Peng Huang, Jian-Fu Chen, Jenna N Regan, et al. Loss of miRNAs in neural crest leads to cardiovascular syndromes resembling humancongenital heart defects[J]. Arterioscler Thromb Vasc Biol, 2010, 30(12): 2575-2586.
[17]Huang ZP, Chen JF, Regan JN, et al.Loss of microRNAs in neural crest leads to cardiovascular syndromes resembling human congenital heart defects[J].Arterioscler Thromb Vasc Biol, 2010, 30(12): 2575-2586.
[18]Ai J, Zhang R, Li Y, et al. Circulating microRNA-1 as a potential novel biomarker for acute myocardial infarction[J]. Biochem Biophys Res Commun, 2010, 391(1):73-77.
[19]Wang G K, Zhu J Q, Zhang J T, et al. Circulating microRNA: a novel potential biomarker for early diagnosis of acute myocardial infarction in humans[J]. Eur Heart J, 2010, 31(6):659-666.
[20]Kratsios P, Catela C, Salimova E, et al.Distinct roles for cell-autonomous Notch signaling in cardiomyocytes of the embryonic and adult heart[J]. Circ Res, 2010,106(3): 559-572.
[21]Yu Z B, Han S P, Zhu C, et al. Screening and Identification of Genes in Myocardial Tissue of Fetus with Ventricular Septal Defect by Using Human Genome Microarray[J]. J Appl Clin Pediatr, 2011, 26(8):1425-1428.
[22]Gruber PJ. Cardiac Development: New Concepts[J]. Clinics in Perinatology, 2005, 32( 4): 845-856.
[23]Burch JBE. Regulation of GATA gene expression during vertebrate development[J]. Semin Cell Dev Biol, 2005, 16(1): 71-81.
[24]Kikuchi K, Hoidway JE, Werdich AA, et al. Primary contribution to zebrafish heart regeneration by gata4 +cardiomyocytes[J]. Nature, 2010, 464: 601-605.
[25]Sara A, Miller Amy S. Common themes emerge in the transcriptional control of T helper and developmental cell fate decisions regulated by the T-box, GATA and ROR families [J]. Immunology, 2009, 216: 306-315.
[26]Jin LC. The Expression of T-bet and GATA-3 mRNA in Peripheral Blood Mononuclear Cells of Youg children suffered from VSD combined with different level of PAH[D].Master's degree thesis of Hebei Medical University, 2012.
[27]Lu JR, McKinsey TA, Xu H, et al. FOG-2, a heart- and brain-enriched cofactor for GATA transcription factors[J]. Mol Cell Biol,1999, 19: 4495-4502.
[28]Zhou B, Ma Q, Kong SW, et al. Fog2 is critical for cardiac function and maintenance of coronary vasculature in the adult mouse heart[J]. J Clin Invest,2009, 119: 1462-1476.
[29]Vincentz J W, Barnes R M, Firulli A B. Hand factors as regulators of cardiac morphogenesis and implications for congenital heart defects [J]. Birth Defects Res A Clin Mol Teratol, 2011, 91(6): 485-94.
[30]Zhang H,Lu ZH. Bioinformatic Analysis of Cardiac Developmental GRNs in Mouse[J]. Journal of Biomedical Engineering Research, 2011, 30(2): 95-100.
[31]Cheng Z, Li B L, Li Z, et al. Two novel HAND1 mutations in Chinese patients with ventricular septal defect[J]. Clin Chim Acta, 2012, 413: 675-677.
[32]Zhao Y, Samal E, Srivastava D, et al. Serum response factor regulates a muscle-specific microRNA that targets Hand during cardio-genesis[J]. Nature, 2005, 436(7048): 214-220.
[33]Wessels MW, Willems PJ. Genetic factors in non-syndromic congenitalheart malformations[J]. Clin Genet, 2010, 78(2):103-123.
Characterization of circulating microRNA expression in patients with a ventricular septal defect
XU Mei-ling1WU Xue-song2ZHU Kai2LI Jin-dong2WANG Qian2JI Long2
(1.Taishan District Center for Disease Control and Prevention, Taian 271000,China;2. School of Public Hhealth, Taishan Medical University, Taian 271016,China)
Objective: To characterize the expression of miRNAs that might be involved in the development or reflect the consequences of VSD. Methods: MiRNA microarray analysis and reverse transcription-polymerase chain reaction (RT-PCR) were employed to determine the miRNA expression profile from 3 patients with VSD and 3 VSD-free controls. The expression of eight selected miRNAs (hsa-let-7e-5p, hsa-miR-155-5p, hsa-miR-222-3p, hsa-miR-379-5p, hsa-miR-409-3p, hsa-miR-433, hsa-miR-487b and hsa-miR-498) were then independently validated by RT-PCR of plasma samples from 20 VSD patients and 15 VSD-free controls. A total of 446 target genes were predicted by bioinformatic methods. Results: Thirty-six differentially expressed miRNAs were found in the patients with VSD and the VSD-free controls. Compared with VSD-free controls, expression of 15 miRNAs were up-regulated and 21 miRNAs were down-regulated in the VSD group. The results of the RT-PCR were consistent with those of the microarray analysis. Gene ontology analysis indicated that the top target genes were mainly related to cardiac right ventricle morphogenesis. Additionally, we predicted NOTCH1, HAND1, ZFPM2, and GATA3 as targets of hsa-let-7e-5p, hsa-miR-222-3p and hsa-miR-433. Conclusion: The study has reported for the first time three circulating miRNA profiles for patients with VSD and VSD-free controls and validated these results in a larger group of subjects. These findings may reveal important insights into the pathogenesis of VSD.
VSD; microRNAs; NOTCH1; HAND1; ZFPM2; GATA3
山东省医药卫生科技发展计划项目(2013WS0320)。
许美玲(1972—),女,山东宁阳人,主管技师,主要从事专业卫生检验工作。
纪龙,男,讲师。
R541.1
A
1004-7115(2016)09-0961-07
10.3969/j.issn.1004-7115.2016.09.001
2016-05-10)
