汉语阅读障碍儿童S100B基因多态性研究*
2016-09-20莫胜男邵珊珊刘玲飞张晓慧宋然然
罗 秀, 莫胜男, 孔 锐, 邵珊珊, 刘玲飞, 王 佳, 张晓慧, 宋然然△
1华中科技大学同济医学院公共卫生学院儿少卫生与妇幼保健学系,武汉 4300302武汉大学中南医院设备处,武汉 430071
汉语阅读障碍儿童S100B基因多态性研究*
罗秀1,莫胜男2,孔锐1,邵珊珊1,刘玲飞1,王佳1,张晓慧1,宋然然1△
1华中科技大学同济医学院公共卫生学院儿少卫生与妇幼保健学系,武汉4300302武汉大学中南医院设备处,武汉430071
目的研究S100B基因与汉语儿童阅读障碍的关联性。方法采用病例-对照的研究方法,选取阅读障碍儿童409名,非阅读障碍对照儿童(对照组)410名。口腔拭子收集口腔上皮细胞以提取基因组DNA,采用Sequenom Mass Array platform分型技术对819个样本S100B基因的2个SNP位点rs3788266和rs9722进行基因多态性研究。统计分析采用t检验和χ2检验以及非条件Logistic回归等方法。结果①根据S100B基因易感位点的筛选条件和基因型分型结果,仅rs9722位点纳入下一步分析;②S100B基因rs9722单核苷酸位点基因型及等位基因分布在阅读障碍儿童组与对照组之间差异无统计学意义(均P>0.05);③基因型、加性模型、隐性模型、显性模型显示S100B基因的rs9722位点多态性与阅读障碍的发生均无统计学差异(Padjusted>0.05)。结论S100B基因的rs9722位点的多态性可能不是影响汉语阅读障碍发生的遗传易感因素。
S100B基因;阅读障碍;病例对照研究;关联性研究
发展性阅读障碍(developmental dyslexia)是指在智力正常、拥有完整的感知觉能力,接受了适当的社会文化教育的情况下,仍不能进行精确或流利的文字认知以及在拼写、编码等方面存在缺陷的神经发育紊乱疾病[1-2]。在中国学龄儿童中阅读障碍的发病率为3.9%~12.6%,男女比例为1.6~3.0[3-4]。家系研究和双生子研究表明阅读障碍有很强的遗传性[5-7],大量的遗传关联性的研究认为9个基因座DYX1~DYX9与阅读障碍的发生有关[8]。
S100B基因位于21q22.3,主要编码钙连接蛋白,在大脑的海马区有较高含量的表达,该蛋白主要是由星形胶质细胞产生,并通过旁分泌和自分泌来影响神经元和胶质细胞[9]。S100B蛋白广泛分布于细胞质和细胞核中,并参与细胞周期调控和分化等过程。在部分脑组织损伤的疾病中,该蛋白被公认为是脑组织活跃度和受损伤的良好标记物,例如在创伤性脑损伤、局部缺血、神经的退行性病变和精神类疾病中[10]。S100B蛋白与RAGE(晚期糖基化蛋白受体)结合,从而激活信号通路促使神经元轴突的延长与增殖以及星形细胞的增多[11]。Nishiyama等[12]认为S100B蛋白具有调节突触的可塑性进而影响学习和记忆的功能。在学习和记忆加工的过程中突触的可塑性是非常重要的,但Lee Swanson[13]发现阅读障碍儿童在这方面是存在缺陷的。国外2项家系研究发现S100B与阅读障碍的发生有关联性[14-15]。S100B基因-阅读障碍的关联性研究主要是在国外的家系研究中,在人群中的研究尤其是汉语人群中的研究并不多见。本研究采用病例对照研究方法,在中国人群中进行候选基因S100B的易感位点的遗传多态性研究,以期发现该基因的变异与阅读障碍之间的关联性,为从分子水平揭示阅读障碍的发生机制提供参考依据。
1 资料与方法
1.1研究对象
本研究对象来源于“同济阅读环境与阅读障碍研究”(READ)[4,16-18]中的部分数据,共有409名汉语阅读障碍儿童和410名对照组儿童纳入本次研究。阅读障碍诊断标准是根据国际疾病分类诊断第10版(ICD-10)规定的汉语阅读障碍的定义和诊断标准:①学习成绩位于同班成绩的后10%的学生;②《儿童汉语阅读障碍量表》[19-20]得分高于同年级学生2个标准差以上;③《儿童学习障碍筛查量表》[21]总分低于65分;④无视听觉障碍及器质性脑病及智力正常。同时满足以上5个条件即诊断为阅读障碍。另外在阅读障碍儿童同小学、同年级、同班级按照1∶1比例,选择与阅读障碍儿童年龄及性别匹配的阅读能力正常、智商正常及排除视听觉障碍及器质性脑病的儿童作为对照组。所有研究对象参加本研究均取得本人及父母或其他监护人的知情同意。实验方案被华中科技大学同济医学院伦理委员会批准。
1.2研究方法
1.2.1DNA提取采用口腔拭子收集口腔上皮细胞以提取基因组DNA。使用QIAamp DNA Investigator试剂盒DP56504(凯捷公司,北京)提取口腔上皮细胞中的DNA,并通过NanoDrop 1000分光光度计检测DNA浓度和纯度。
1.2.2S100B功能性常见变异SNPs的选取与基因分型步骤:①根据F-SNP database(http://compbio.cs.queensu.ca/F-SNP)筛选出具有编码蛋白,剪切调控以及转录调控等功能性SNP位点;②基于HapMap database(http://hapmap.ncbi.nlm.nih.gov/),在以上SNP位点中筛选出在中国人群中最小等位频率大于5%的SNP位点;③对满足以上条件的SNP位点进行连锁不平衡分析http://www.broadinstitute.org/mpg/snap/ldsearchpw.php。r2≥0.80(即存在高度连锁)的一对SNP位点中,只保留其中之一。经筛选,只有rs3788266和rs9722符合以上条件被选取。采用Sequenom Mass Array platform分型技术对以上SNP位点进行基因分型。
1.3统计学方法
通过拟合优度χ2检验判断本研究对照组中位点基因型频率分布是否符合Hardy-Weinberg平衡。使用Pearson χ2检验或t检验比较病例和对照组的性别、年龄以及SNPs的基因型频率和等位基因频率的差异。SNP位点多态性与阅读障碍发病风险的关联性研究采用非条件Logistic回归分析,得到经性别和年龄校正后的优势比(OR)值及95%的置信区间(95%CI)。统计分析采用SPSS 13.0软件包实现且采用双侧检验,检验水准α=0.05。
2 结果
2.1SNP位点的分型结果
13个DNA样本未能成功分型(其中包括6个病例,7个对照),故最终得到403名阅读障碍儿童组和403名对照组儿童的DNA样本基因分型结果,两组儿童性别、年龄差异无统计学意义(均P>0.05),其基本的人口学特征见表1。
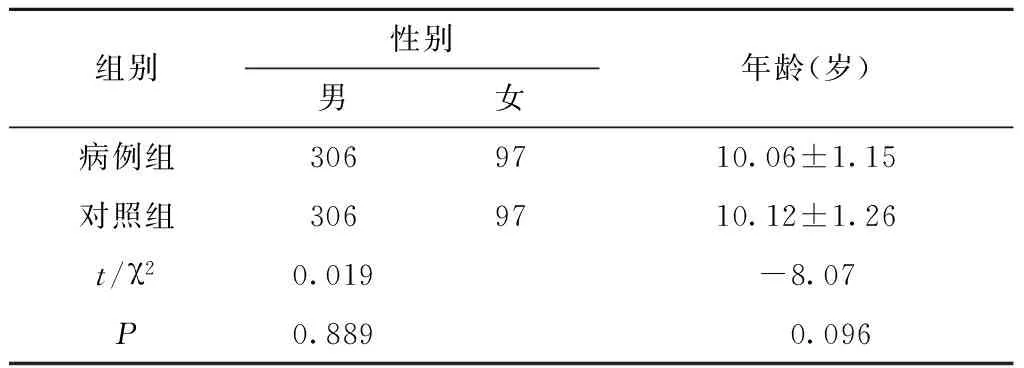
表1 病例组与对照组人口学基本特征
由于rs3788266位点不符合Hardy-Weinberg平衡检验(P<0.05),故这个位点需排除,最终只有rs9722符合要求。
2.2S100B各位点基因多态性情况
S100B基因的rs9722位点上等位基因和各基因型频率在阅读障碍组和对照组之间的分布情况见图1,rs9722位点上等位基因频率和基因型频率差异无统计学意义(均P>0.05)。
2.3S100B基因多态性与阅读障碍的相关性分析
以年龄、性别为协变量进行校正,通过非条件Logistic回归模型对rs9722位点4种遗传模型(即基因型、加性模型、显性模型及隐性模型)进行分析,计算出OR值和95%置信区间,结果如表2所示。
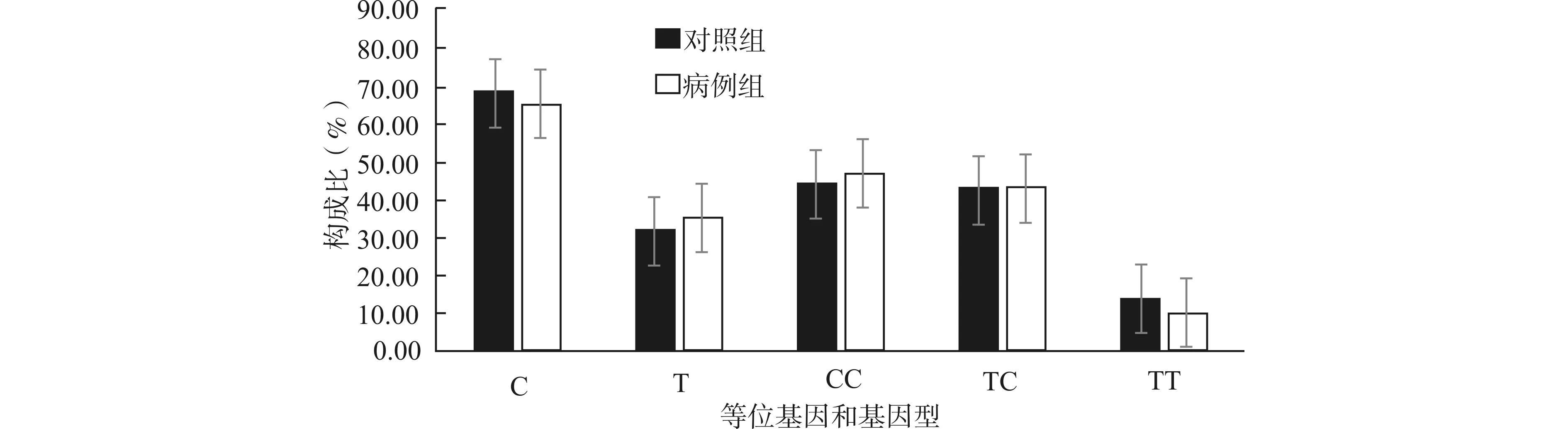
图1 rs9722位点等位基因和各基因型频率在阅读障碍病例组和对照组间分布情况Fig.1 Distribution of the frequencies of alleles and genotypes of rs9722 in case group and control group
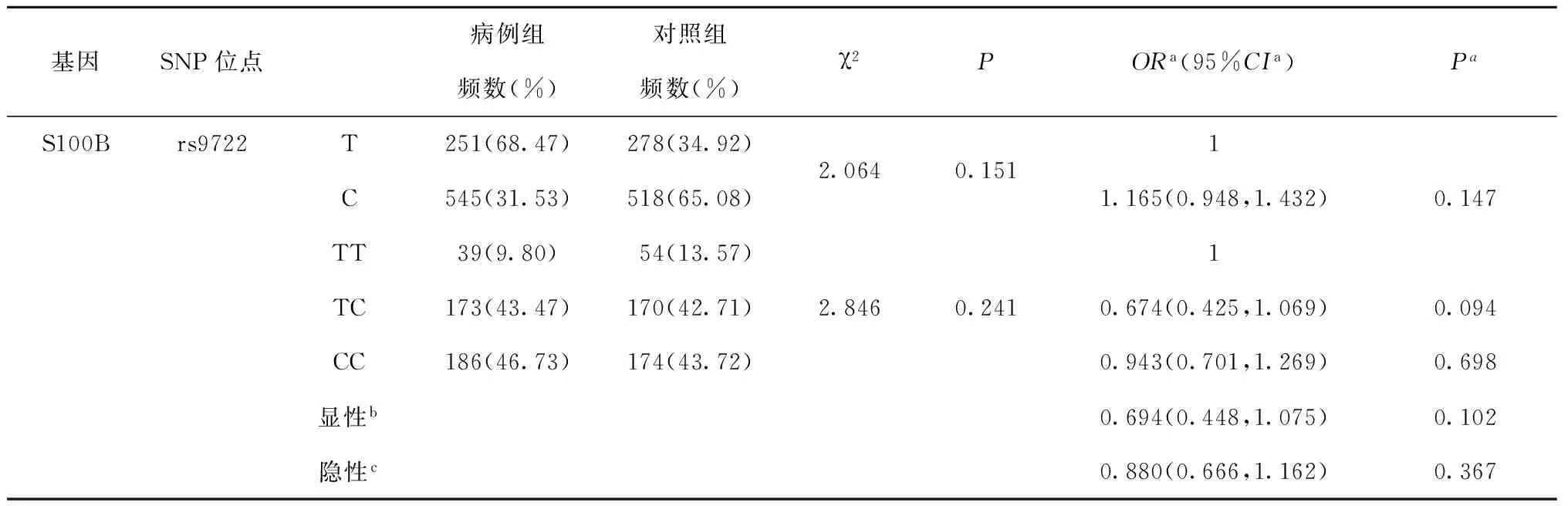
基因SNP位点病例组对照组频数(%)频数(%)χ2PORa(95%CIa)PaS100Brs9722T251(68.47)278(34.92)C545(31.53)518(65.08)2.0640.15111.165(0.948,1.432)0.147TT39(9.80)54(13.57)TC173(43.47)170(42.71)CC186(46.73)174(43.72)2.8460.24110.674(0.425,1.069)0.0940.943(0.701,1.269)0.698显性b0.694(0.448,1.075)0.102隐性c0.880(0.666,1.162)0.367
a经过性别、年龄校正之后的Logistic回归模型计算得到的OR值、95%CI值和P值,b显性代表显性模型,c隐性代表隐性模型
S100B基因的rs9722位点上,基因型CC和等位基因C分别相对于基因型TT和等位基因T;显性模型中,携带C基因型(CC+TC)的个体与携带TT基因型的个体相比;隐性模型中,携带CC基因型个体与携带TT+TC基因型的个体相比结果可知,4种遗传模型均显示S100B基因的rs9722位点的多态性阅读障碍组与对照组相比均无统计学差异(Padjusted>0.05)。
3 讨论
本研究在403例阅读障碍和403例非阅读障碍的中国汉语儿童中进行S100B基因多态性研究,通过统计分析发现S100B基因的rs9722位点的遗传变异与阅读障碍相关性无统计学意义。然而这与荷兰[14]、芬兰和德国[15]的家系研究报道中称S100B基因与阅读障碍的发生有关联性并不一致。
阅读作为一项高级的认知活动,包括编码、语音加工等过程,其发生机制极其复杂[22-23]。在语言的加工过程中欧美和中国人群的大脑通路不同,接收神经信息的大脑额下回区域有差异,以英语为母语的人群位于颞叶皮层的左下部分,而以中文为母语的人群则为颞叶皮层的双侧前面部分[24]。同样,阅读障碍的生物学病理生理机制因文化的不同而不一致,西方人群阅读障碍是脑部左颞顶区域功能的紊乱[25],而在中国人群中阅读障碍与脑部左额中脑回灰质体积相关[26]。S100B基因与阅读障碍在人群中的关联性研究在国内外开展都较少,而阅读障碍作为一种神经发育障碍类的疾病,其病理机制也是非常复杂的,关于基因和基因组学方面的研究其结果具有很大的差异性[27]。例如KIAA0319在巴西人[28]、英格兰人[29]的研究中发现该基因的变异与阅读障碍有相关性,而在中国人[30]、印度人[31]以及德国人[32]的研究中则发现其与阅读障碍并无相关性。此外,在欧洲的8个国家都发现DYX1C1、DCDC2、KIAA0319基因与阅读障碍有关,但是在这8个国家中进行的队列研究则发现这些基因与阅读障碍都没有相关性[33]。
S100B蛋白在神经元发育与再生过程中可能是一种营养因子,大脑中S100B蛋白的含量发生变化会引起动物行为紊乱和认知缺陷[9]。通过在小鼠胚胎干细胞的同源重组,生成无S100B基因的小鼠。将野生型和突变型的小鼠进行组织学分析和水迷宫实验,结果发现突变型小鼠的脑组织在解剖学上没有异常,海马区长时程增强(LTP)增加,海马依赖的学习和空间记忆能力会增强。研究者通过钙处理能力实验和不同水平的长时程增强的处理等,结果证实突变小鼠的突触可塑性增加与外源性S100B蛋白通过细胞膜传导,参与随后神经元信号级联反应有关。此外,也有研究者认为S100B蛋白就像一个“紧急阀”,在神经活动过频时可能有负性调节神经活性作用,避免脑组织受损[12]。
本次研究可能是首次报道S100B基因多态性与汉语阅读障碍发生的关联性,此次结果只能说明S100B基因的rs9722位点没有关联性,并不能说明S100B基因与阅读障碍无关。期望在未来的研究中能够尽可能多地选择SNP数量和扩大样本量以了解该基因与阅读障碍的关联性。
[1]Lyon G R.A definition of dyslexia[J].Ann Dyslexia,2003,53(1):1-14.
[2]Peterson R L,Pennington B F.Developmental dyslexia[J].Lancet,2012,379(9830):1997-2007.
[3]Chan D W,Ho C S,Tsang S,et al.Prevalence,gender ratio and gender differences in reading-related cognitive abilities among Chinese children with dyslexia in Hong Kong[J].Educ Stud,2007,33(2):249-265.
[4]Sun Z,Zou L,Zhang J,et al.Prevalence and associated risk factors of dyslexic children in a middle-sized city of China:a cross-sectional study[J].PLoS One,2013,8(2):e56688.
[5]Luciano M,Evans D M,Hansell N K,et al.A genome-wide association study for reading and language abilities in two population cohorts[J].Genes Brain Behav,2013,12(6):645-652.
[6]de Kovel C G F.Genomewide scan identifies susceptibility locus for dyslexia on Xq27 in an extended Dutch family[J].J Med Genet,2004,41(9):652-657.
[7]Swagerman S C,van Bergen E,Dolan C,et al.Genetic transmission of reading ability[J].Brain Lang,2015.Doi:10.1016/j.bandl.2015.07.008.
[8]Peterson R L,Pennington B F.Developmental dyslexia[J].Annu Rev Clin Psychol,2015,11:283-307.
[9]Rothermundt M,Peters M,Prehn J H M,et al.S100B in brain damage and neurodegeneration[J].Microsc Res Tech,2003,60(6):614-632.
[10]Goncalves C A,Leite M C,Nardin P.Biological and methodological features of the measurement of S100B,a putative marker of brain injury[J].Clin Biochem,2008,41(10/11):755-763.
[11]Huttunen H J,Kuja-Panula J,Sorci G,et al.Coregulation of neurite outgrowth and cell survival by amphoterin and S100 proteins through receptor for advanced glycation end products(RAGE)activation[J].J Biol Chem,2000,275(51):40096-40105.
[12]Nishiyama H,Knopfel T,Endo S,et al.Glial protein S100B modulates long-term neuronal synaptic plasticity[J].Proc Natl Acad Sci U S A,2002,99(6):4037-4042.
[13]Lee Swanson H.Dynamic testing,working memory,and reading comprehension growth in children with reading disabilities[J].J Learn Disabil,2011,44(4):358-371.
[14]Poelmans G,Engelen J J M,van Lent-Albrechts J,et al.Identification of novel dyslexia candidate genes through the analysis of a chromosomal deletion[J].Am J Med Genet B Neuropsychiatr Genet,2009,150B(1):140-147.
[15]Matsson H,Huss M,Persson H,et al.Polymorphisms in DCDC2 and S100B associate with developmental dyslexia[J].J Hum Genet,2015,60(7):399-401.
[16]He Z,Shao S,Zhou J,et al.Does long time spending on the electronic devices affect the reading abilities? A cross-sectional study among Chinese school-aged children[J].Res Dev Disabil,2014,35(12):3645-3654.
[17]Shao S,Kong R,Zou L,et al.The roles of genes in the neuronal migration and neurite outgrowth network in developmental dyslexia:single-and multiple-risk genetic variants[J].Mol Neurobiol,2015.Doi:10.1007/s12035-015-9334-8.
[18]Kong R,Shao S,Wang J,et al.Genetic variant in DIP2 A gene is associated with developmental dyslexia in Chinese population[J].Am J Med Genet B Neuropsychiatr Genet,2016,171(2):203-208.
[19]吴汉荣,宋然然,姚彬.儿童汉语阅读障碍量表的初步编制[J].中国学校卫生,2006,27(3):189-190.
[20]吴汉荣,宋然然,姚彬.儿童汉语阅读障碍量表的信度效度分析[J].中国学校卫生,2006,27(6):468-469,471.
[21]静进,余淼,邓桂芬.学习能力障碍筛查量表的修订和在小学生中的试用[J].心理发展与教育,1995,1(2):24-29.
[22]Nicolson R I,Fawcett A J,Dean P.Developmental dyslexia:the cerebellar deficit hypothesis[J].Trends Neurosci,2001,24(9):508-511.
[23]Vandermosten M,Boets B,Poelmans H,et al.A tractography study in dyslexia:neuroanatomic correlates of orthographic,phonological and speech processing[J].Brain,2012,135(3):935-948.
[24]Ge J,Peng G,Lyu B,et al.Cross-language differences in the brain network subserving intelligible speech[J].Proc Natl Acad Sci U S A,2015,112(10):2972-2977.
[25]Seo H C,Edvardsen R B,Maeland A D,et al.Hox cluster disintegration with persistent anteroposterior order of expression in Oikopleuradioica[J].Nature,2004,431(7004):67-71.
[26]Siok W T,Niu Z,Jin Z,et al.A structural-functional basis for dyslexia in the cortex of Chinese readers[J].Proc Natl Acad Sci U S A,2008,105(14):5561-5566.
[27]Geschwind D H,Flint J.Genetics and genomics of psychiatric disease[J].Science,2015,349(6255):1489-1494.
[28]Svidnicki M C,Salgado C A,Lima R F,et al.Study of candidate genes for dyslexia in Brazilian individuals[J].Genet Mol Res,2013,12(4):5356-5364.
[29]Scerri T S,Morris A P,Buckingham L,et al.DCDC2,KIAA0319 and CMIP are associated with reading-related traits[J].Biol Psychiatry,2011,70(3):237-245.
[30]Sun Y,Gao Y,Zhou Y,et al.Association study of developmental dyslexia candidate genes DCDC2 and KIAA0319 in Chinese population[J].Am J Med Genet B Neuropsychiatr Genet,2014,165(8):627-634.
[31]Venkatesh S K,Siddaiah A,Padakannaya P,et al.Analysis of genetic variants of dyslexia candidate genes KIAA0319 and DCDC2 in Indian population[J].J Hum Genet,2013,58(8):531-538.
[32]Kirsten H,Wilcke A,Ligges C,et al.Association study of a functional genetic variant in KIAA0319 in German dyslexics[J].Psychiatr Genet,2012,22(4):216-217.
[33]Becker J,Czamara D,Scerri T S,et al.Genetic analysis of dyslexia candidate genes in the European cross-linguistic NeuroDys cohort[J].Eur J Hum Genet,2014,22(5):675-680.
(2016-01-26收稿)
Polymorphisms of S100B Gene in Children with Developmental Dyslexia in China
Luo Xiu1,Mo Shengnan2,Kong Rui1etal
1DepartmentofMaternalandChildHealth,SchoolofPublicHealth,TongjiMedicalCollege,HuazhongUniversityofScienceandTechnology,Wuhan430030,China2DepartmentofEquipment,ZhongnanHospitalofWuhanUniversity,Wuhan430071,China
ObjectiveTo study the association between S100B polymorphisms and developmental dyslexia(DD) among children in China.MethodsThe case-control method was used in the study.There were 409 dyslexia children and 410 healthy controls.Genomic DNA was extracted from oral swab samples.Gene polymorphisms were detected in two SNP sites rs378826 and rs9722 in 819 samples by using the Sequenom Mass Array platform.Thettest,χ2test and unconditional logistic regression were used for statistical analysis.Results①Only rs9722 entered the following analysis based on the screening of the susceptibility site of S100B and the results of genotyping.②The differences in the distribution of allele frequencies and genotypes of rs9722 between two groups were not statistically significant(P>0.05).③Genotype,additive,dominant and recessive models showed that rs9722 polymorphisms were not associated with development dyslexia(Padjusted>0.05).ConclusionThe genetic variants of rs9722 in S100B gene may not be a genetic susceptibility factor of developmental dyslexia in China.
S100B;developmental dyslexia;case control study;association study
,Corresponding author,E-mail:songranran@hust.edu.cn
R179
10.3870/j.issn.1672-0741.2016.04.008
*国家自然科学基金资助项目(No.81273092),湖北省卫生计生委西医类一般项目(No.WJ2015MB019),湖北省自然科学基金资助项目(No.2014CKB491)
罗秀,女,1991年生,硕士研究生,E-mail:luoxiu@hust.edu.cn
