鲍曼不动杆菌耐氨苄西林-舒巴坦与青霉素结合蛋白基因变异的相关性
2016-09-20肖淑珍褚海青张静波郭庆兰
肖淑珍, 褚海青, 赵 兰, 张静波, 李 冰, 郭庆兰
鲍曼不动杆菌耐氨苄西林-舒巴坦与青霉素结合蛋白基因变异的相关性
肖淑珍1, 褚海青2, 赵 兰2, 张静波2, 李 冰2, 郭庆兰3
目的 测定鲍曼不动杆菌临床分离株对常用抗菌药物的敏感性;分析青霉素结合蛋白(PBP)基因的变异,探索其与鲍曼不动杆菌对氨苄西林-舒巴坦耐药的相关性。方法 收集上海市4所教学医院临床分离的鲍曼不动杆菌67株,采用琼脂稀释法测定其对常用抗菌药物的最低抑菌浓度。扩增所有临床株的7种PBP基因并测序。比较该菌氨苄西林-舒巴坦敏感组与耐药组之间核苷酸序列差异。结果 临床分离鲍曼不动杆菌对常用抗菌药物耐药率高,对氨苄西林-舒巴坦耐药率为67.6%。测序结果显示敏感株及耐药株间未发现PBP的氨基酸变异。结论 临床分离鲍曼不动杆菌对氨苄西林-舒巴坦耐药率较高。其耐药机制与PBP变异无相关性。
鲍曼不动杆菌; 青霉素结合蛋白; 氨苄西林-舒巴坦
鲍曼不动杆菌属不发酵糖革兰阴性杆菌,根据CHINET细菌耐药监测资料显示,自2010年起鲍曼不动杆菌在医院感染中占第3位,成为仅次于大肠埃希菌和肺炎克雷伯菌的重要革兰阴性杆菌[1-3]。近年来,鲍曼不动杆菌耐药趋势日益严重,部分菌株仅对β内酰胺酶抑制剂复方制剂、黏菌素、替加环素敏感。
舒巴坦是对鲍曼不动杆菌具有抗菌活性的β内酰胺酶抑制剂,当多重耐药鲍曼不动杆菌感染时,舒巴坦是一种合适的选择[4-6]。含舒巴坦复方制剂是国内治疗鲍曼不动杆菌感染常用的抗菌药物。近年国内外研究显示舒巴坦复方制剂(氨苄西林-舒巴坦及头孢哌酮-舒巴坦)对鲍曼不动杆菌抗菌活性明显下降[1-3]。
本研究旨在测定鲍曼不动杆菌临床分离株对常用抗菌药物的敏感性,分析青霉素结合蛋白(PBP)基因的变异,初步探索与氨苄西林-舒巴坦耐药的相关性。
1 材料与方法
1.1材料
1.1.1菌株来源 收集2011年7月—2012年5月上海市4所教学医院临床分离的鲍曼不动杆菌67株,剔除同一患者重复菌株,其中包括仁济医院5株,第六人民医院7株,华山医院27株,瑞金医院28株。所有菌株经检测blaoxa-51-like及16S-23S rRNA基因[7-8]测序鉴定证实。质控菌株:大肠埃希菌ATCC 25922,铜绿假单胞菌ATCC 27853。
1.1.2主要药物和试剂 亚胺培南、美罗培南、阿米卡星、哌拉西林、头孢他啶、米诺环素、环丙沙星、氨苄西林-舒巴坦、舒巴坦、头孢哌酮-舒巴坦、哌拉西林-他唑巴坦、黏菌素、替加环素、甲氧苄啶-磺胺甲唑等抗菌药物(上海科佳药检器材公司);dNTP、Taq DNA酶(TaKaRa,大连)。
1.1.3主要仪器 PCR扩增仪ABI 2700及RTPCR扩增仪ABI PRISM 7500( ABI,美国);电泳仪、凝胶成像仪及紫外线投射凝胶成像系统(Bio-Rad,美国)。
1.2方法
1.2.1抗菌药物敏感性试验 采用琼脂稀释法检测67株鲍曼不动杆菌对15种抗菌药物的最低抑菌浓度(MIC),计算MIC范围、MIC50、MIC90。结果判定参照2012年美国临床和实验室标准化协会(CLSI)标准[9]。多重耐药(MDR)是指对下列5类抗菌药物中至少3类耐药的菌株,包括:头孢菌素、碳青霉烯类抗生素、含有β内酰胺酶抑制剂的复合制剂(包括哌拉西林-他唑巴坦、头孢哌酮-舒巴坦、氨苄西林-舒巴坦)、氟喹诺酮类抗菌药物、氨基糖苷类抗生素。
1.2.2PCR检测基因 从平皿上挑取3~4个菌落置于300 μL ddH2O中,100 ℃煮沸15 min后12 000 g离心15 min,取上清液即为DNA模板。PCR引物由上海生工生物工程技术服务有限公司合成,引物序列及反应体系见参考文献[10]。PCR产物送上海生工生物工程技术服务有限公司测序,以鲍曼不动杆菌标准菌株ATCC17978作为参照菌株进行碱基序列比对。
2 结果
2.1药敏结果
67株鲍曼不动杆菌对常用抗菌药物的药敏试验结果见表1。参考2012年CLSI标准,鲍曼不动杆菌对常用抗菌药物的耐药率较高,对头孢菌素类如头孢他啶的耐药率为82.0%;对碳青霉烯类如亚胺培南、美罗培南的耐药率分别为65.6%、41.8%;对氨基糖苷类如阿米卡星耐药率67.1%;对酶抑制剂复方制剂氨苄西林-舒巴坦和哌拉西林-他唑巴坦的耐药率分别为67.6%和73.1%。根据氨苄西林-舒巴坦的药敏结果将细菌分组,包括耐药组(46株)、中介组(6株)和敏感组(15株)。耐药组细菌对其他抗菌药物也普遍表现为耐药(除黏菌素外),MDR率达69.6%;而敏感组细菌耐药谱则相对较窄,未发现MDR菌株。
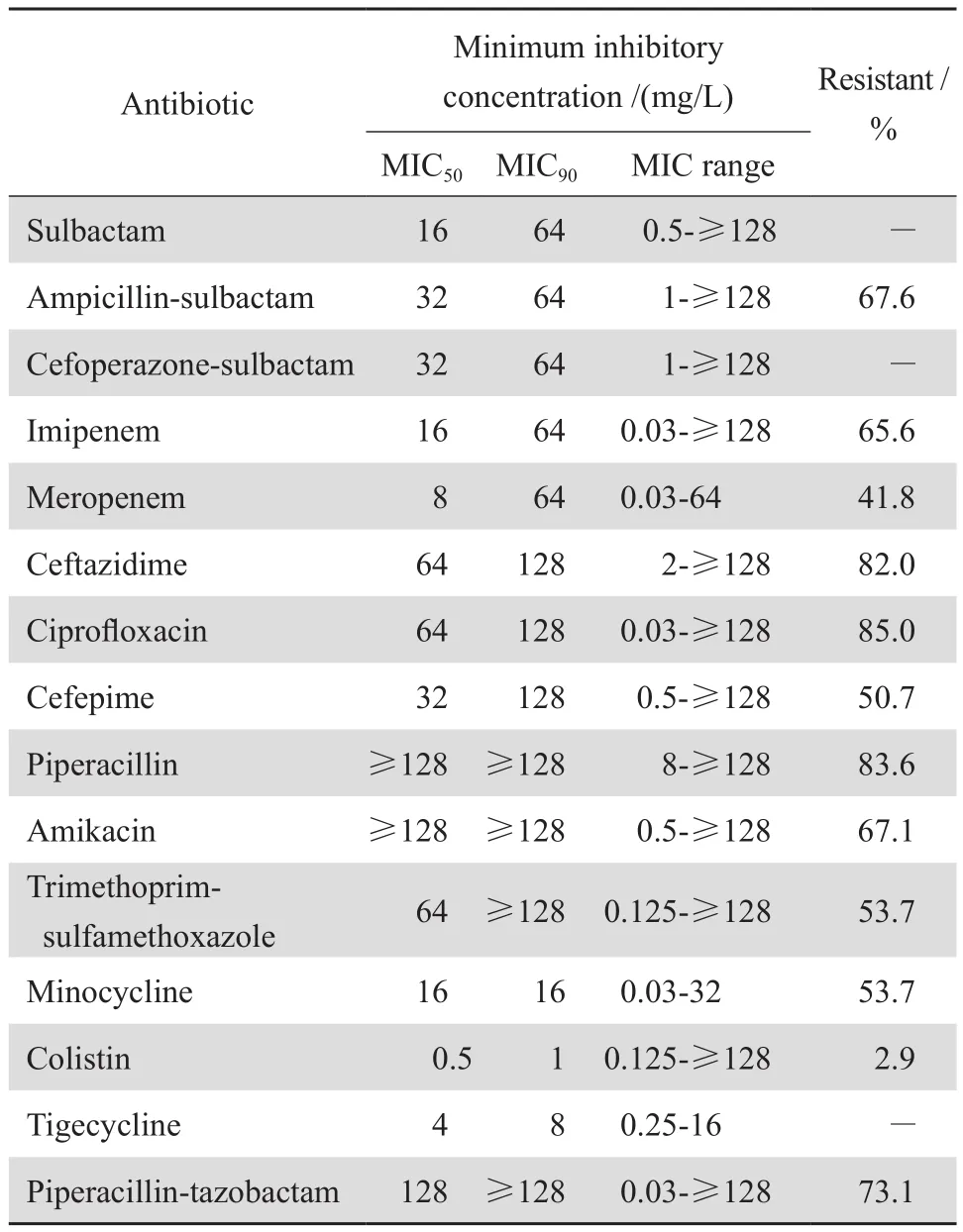
表1 常用抗菌药物对67株鲍曼不动杆菌抗菌活性Table 1 Activity of antimicrobial agents against 67 strains of Acinetobacter baumannii
2.2PBP基因扩增及测序
67株鲍曼不动杆菌均成功扩增到7种PBP基因并进行测序。与标准菌株比对,敏感组与耐药组间存在核苷酸序列差异,但两者未发现氨基酸序列的差异,见表2。7种基因氨基酸序列见图1。
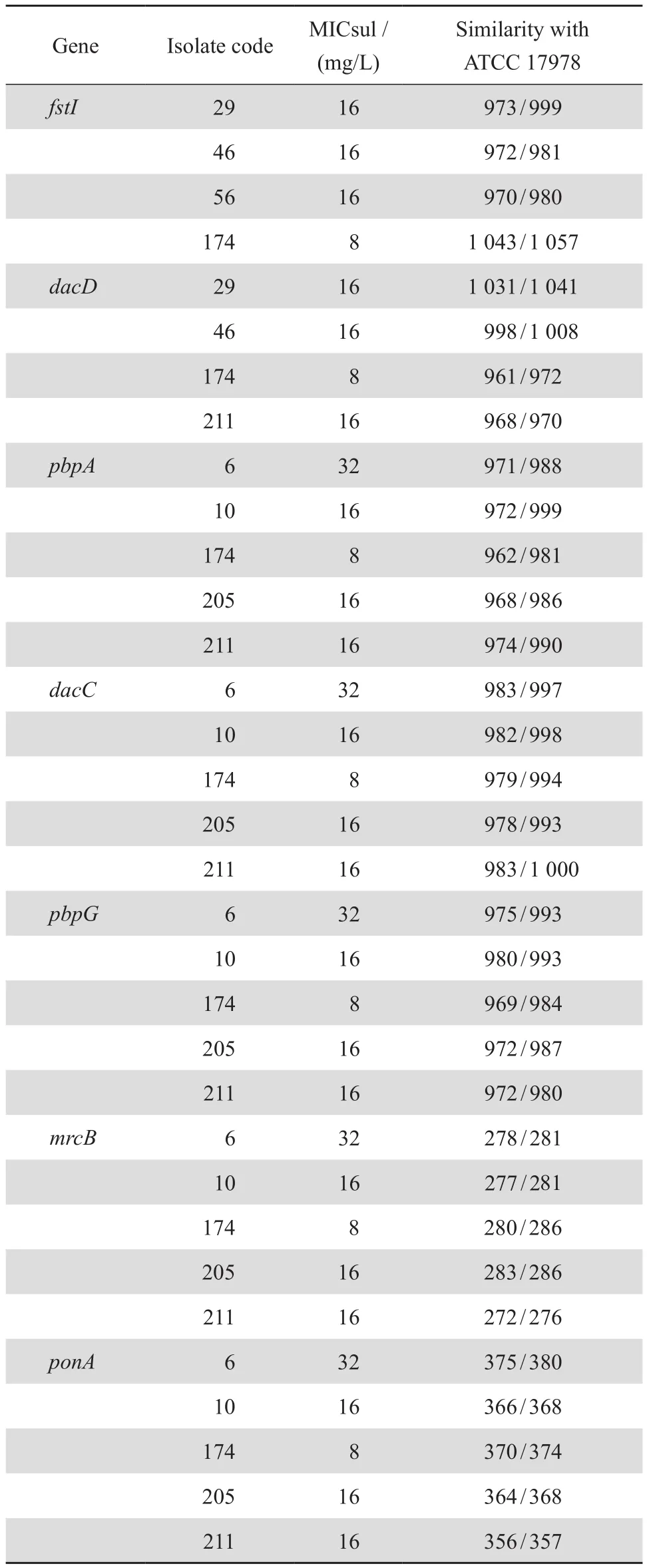
表2 不同菌株中碱基序列变异分析Table 2 Analysis of gene mutations in different strains


图1 临床菌株与标准菌株氨基酸序列对比Figure 1 Deduced amino acid sequences of several genes compared between clinical strains and reference strains
3 讨论
近年来,鲍曼不动杆菌对舒巴坦复方制剂的敏感率逐年下降。CHINET细菌耐药监测数据显示2007—2009年,鲍曼不动杆菌对氨苄西林-舒巴坦的敏感率分别是 39.6%、37.6%和37.1%[11-13]。国外文献也报道,含舒巴坦复方制剂对鲍曼不动杆菌抗菌活性明显下降[1-3]。不同地区不同医院鲍曼不动杆菌的耐药率也有不同,2010年安徽、北京、重庆、昆明等地报道的耐药率分别是58.0%、60.6%、67.1%及71.7%[1]。本研究中,鲍曼不动杆菌对氨苄西林-舒巴坦的耐药率为67.6%,提示上海地区耐药水平较高。
PBP是细菌细胞膜的重要组成成分,主要参与细菌细胞壁的合成[10]。不同细菌中PBP的种类及含量均不相同,如粪肠球菌中有5种PBP,肺炎链球菌中有分子量43~100 ku的7种PBP,在鲍曼不动杆菌中存在7种PBP[14-15]。有研究表明PBP在某些细菌耐药中起着重要作用,其基因突变或者与抗菌药物亲和力下降将导致细菌对某些药物耐药[16-18]。
GEHRLEIN等[19]发现在1株亚胺培南耐药的鲍曼不动杆菌中,外膜蛋白没有异常,也未发现产β内酰胺酶,作者推测PBP的变异直接导致了细菌的耐药。近期有国外学者对临床分离鲍曼不动杆菌中PBP进行分析,在26株菌株中均检测到7种PBP基因,与标准菌株相比较后发现临床菌株中存在碱基高突变区域,但氨基酸序列与标准菌株则保持高度的一致性[19]。本研究提示氨苄西林-舒巴坦耐药与敏感组菌株中均未发现PBP存在氨基酸有义突变,因此推测氨苄西林-舒巴坦耐药与PBP变异没有相关性。
本研究显示,临床分离鲍曼不动杆菌对氨苄西林-舒巴坦耐药率较高,其耐药机制与PBP变异无相关性。
[1]朱德妹,汪复,胡付品,等. 2010年中国CHINET细菌耐药性检测[J].中国感染与化疗杂志,2011,11(5):321-329.
[2]汪复,朱德妹,胡付品,等. 2012年中国CHINET细菌耐药性检测[J].中国感染与化疗杂志,2013,13(5):321-330.
[3]朱德妹,杨洋,蒋晓飞,等. 2011年中国CHINET细菌耐药性检测[J].中国感染与化疗杂志,2012,12(6):401-411.
[4]BRICEÑO DF,QUINN JP,VILLEGAS MV. Treatment options for multidrug-resistant nonfermenters [J]. Anti Infect Ther, 2012,8(3):303-315.
[5]BASSETTI M,RIGHI E,ESPOSITO S, et al. Drug treatment for multidrug-resistant Acinetobacter baumannii infections [J]. Future Microbiol,2008,3(6):649-660.
[6]RAFAILIDIS PI, IOANNIDOU EN, FALAGAS ME. Ampicillin/sulbactam: current status in severe bacterial infections [J]. Drugs, 2007,67(13):1829-1849.
[7]YIN D, DU J, DUAN J. 16S rRNA methylase and genetic relatedness of multidrug-resistant Acinetobacter spp [J]. Chin J Lab Med, 2009, 32: 673-677.
[8]CHANG HC, WEI YF, DIJKSHOORN L, et al, Species-Level identification of isolates of the Acinetobacter calcoaceticus-Acinetobacter baumannii complex sequence analysis of the 16S-23S rRNA gene spacer region [J]. J Clin Microbiol, 2005, 43(4):1632-1735.
[9]Clinical and Laboratory Standards Institute. Performance Standards for Antimicrobial Susceptibility Testing[S]. Twenty-Second Informational Supplement. 2012,M100-S22.
[10]CAYÔ R, RODRíGUEZ MC, ESPINAL P, et al. Analysis of genes encoding penicillin-binding proteins in clinical isolates of Acinetobacter baumannii [J]. Antimicrob Agents Chemother,2011, 55(12):5907-5913.
[11]汪复,朱德妹,胡付品,等. 2007年中国CHINET细菌耐药性检测[J].中国感染与化疗杂志,2008,8(5):325-333.
[12]汪复,朱德妹,胡付品,等. 2008年中国CHINET细菌耐药性检测[J].中国感染与化疗杂志,2009,9(5):321-329.
[13]汪复,朱德妹,胡付品,等. 2009年中国CHINET细菌耐药性检测[J].中国感染与化疗杂志,2010,10(5):325-334.
[14]MOISAN H, PRUNEAU M, MALOUIN F. Binding of ceftaroline to penicillin-binding proteins of Staphylococcus aureus and Streptococcus pneumoniae [J]. J Antimicrob Chemother, 2010, 65(4):713-716.
[15]ARBLOA A, SEGAL H, HUGONNET JE, et al. Role of class A penicillin-binding proteins in PBP5-mediated beta-lactam resistance in Enterococcus faecalisc[J]. J Bacteriol, 2004, 186 (5):1221-1228.
[16]GISKE CG, BUARØ L, SUNDSFJORD A, et al. Alterations of porin, pumps, and penicillin-binding proteins in carbapenem resistant clinical isolates of Pseudomonas aeruginosa[J]. Microb Drug Resist, 2008, 14(1):23-30.
[17]CHIU CH, SU LH, HUANG YC, et al,. Increasing ceftriaxone resistance and multiple alteration of penicillin-binding proteins among penicillin-resistant Streptococcus pneumoniae isolates in Taiwan [J]. Antimicrob Agents Chemother, 2007, 51(9):3404-3406.
[18]NAGAI K, DAVIES TA, JACOBS MR, et al. Effects of amino acid alterations in penicillin-binding proteins( PBPs) 1a, 2b, and 2x on PBP affinities of penicillin, ampicillin, amoxicillin, cefditoren,cefuroxime, cefprozil, and cefaclor in 18 clinical isolates of penicillin-susceptible, -intermediate, and -resistant pneumococci [J]. Antimicrob Agents Chemother, 2002, 46(5):1273-1280.
[19]GEHRLEIN M, LEYING H, CULLMANN W, et al. Imipenem resistance in Acinetobacter baumannii is due to altered penicillinbinding proteins [J]. Chemotherapy, 1991, 37(6):405-412.
Correlation between ampicillin-sulbactam resistance and penicillin-binding protein gene variation in Acinetobacter baumannii
XIAO Shuzhen, CHU Haiqing, ZHAO Lan, ZHANG Jingbo, LI Bing, GUO Qinglan. (Department of Clinical Microbiology, Ruijin Hospital, Shanghai Jiao Tong University School of Medicine, Shanghai 200025, China)
Objective To determine the susceptibility of Acinetobacter baumannii isolates to commonly used antibiotics,characterize mutation status of penicillin-binding protein (pbp) gene and analyze the association between pbp gene and ampicillinsulbactam resistance. Methods A total of 67 A. baumannii strains were collected from four teaching hospitals in Shanghai. The minimum inhibitory concentrations of antibiotics were determined for A. baumannii by agar dilution method. PCR and gene sequencing were conducted to analyze 7 pbp genes in all stains. The nucleotide sequence was compared between the strains susceptible to ampicillin-sulbactam and the strains resistant to ampicillin-sulbactam. Results The A. baumannii isolated from patients showed high resistance rate to common antibiotics, 67.6% resistant to ampicillin-sulbactam. Sequencing analysis showed that no amino acid variation was found for all 7 pbp genes whether the strains were susceptible or resistant. Conclusions Clinical isolates of A. baumannii are highly resistant to ampicillin-sulbactam, while pbp gene is not involved in the mechanism of ampicillinsulbactam resistance.
Acinetobacter baumannii; penicillin-binding protein; ampicillin-sulbactam
·论著·
R378
A
1009-7708(2016)01-0057-04
10.16718/j.1009-7708.2016.01.013
上海市科学技术委员会自然科学基金项目(12ZR1426200),上海市科学技术委员会医学引导项目(14411962900)。
1. 上海交通大学医学院附属瑞金医院检验科,上海 200025;
2. 上海同济大学附属上海市肺科医院呼吸科;
3. 复旦大学附属华山医院抗生素研究所。
肖淑珍(1988—),女,硕士研究生,主要从事微生物耐药机制研究。
褚海青,E-mail: chu_haiqing@126.com。
2015-06-11
2015-07-09
