三重PCR检测黄瓜靶斑病菌、炭疽病菌和细菌性角斑病菌
2016-09-09高士刚徐丽慧罗金燕戴富明
高士刚, 曾 蓉, 徐丽慧, 罗金燕, 陈 磊, 戴富明
(1上海市农业科学院生态环境保护研究所,上海 201403;2上海市设施园艺重点实验室,上海 201403;3上海市农业技术推广服务中心,上海 201103)
三重PCR检测黄瓜靶斑病菌、炭疽病菌和细菌性角斑病菌
高士刚1,2, 曾 蓉1,2, 徐丽慧1,2, 罗金燕3, 陈 磊3, 戴富明1,2
(1上海市农业科学院生态环境保护研究所,上海 201403;2上海市设施园艺重点实验室,上海 201403;3上海市农业技术推广服务中心,上海 201103)
【目的】开发一种可同时检测黄瓜靶斑病菌(Corynespora cassiicola)、黄瓜炭疽病菌(Colletotrichum orbiculare)和黄瓜细菌性角斑病菌(Pseudomonas syringae pv. lachrymans)的三重PCR快速检测方法。【方法】根据ITS或16S rDNA序列分别设计黄瓜靶斑病原菌、黄瓜炭疽病菌和黄瓜细菌性角斑病菌特异性检测引物;通过鉴定引物的特异性,筛选可在目标菌株中扩增出特异片段的特异引物;选择可以组合的3种病原菌特异性引物进行三重PCR,对引物浓度、退火温度、延伸时间和循环数分别进行优化,优化三重PCR体系。【结果】针对黄瓜靶斑病菌、黄瓜炭疽病菌和黄瓜细菌性角斑病菌分别设计出5对、7对和6对特异性引物,其中引物CC4F/CC4R 和CC5F/CC5R可特异扩增黄瓜靶斑病菌ITS,引物CL1F/CL1R、CL2F/CL2R、CL3F/CL3R、CL3F/CL4R、CL3F/CL5R、CL3F/CL6R和CL3F/CL7R可特异扩增黄瓜炭疽病菌ITS,引物PS3F/PS4R和PS4F/PS4R可特异扩增黄瓜细菌性角斑病菌16S rDNA。引物CC5F/CC5R、CL3F/5R和PS3F/4R扩增片段长度分别为370、275和698 bp,其混合扩增产物可在3%琼脂糖凝胶上得到充分分离,这3对引物选择作为三重PCR引物。在三重PCR反应体系中,当引物CC5F/CC5R、CL3F/5R和PS3F/4R的终浓度分别为0.16、0.4和0.16 μmol·L-1时,3个目的片段能同时得到有效扩增;当退火温度大于65℃时,部分目的片段不能有效扩增。最终建立并验证了适合上述3种黄瓜主要病原菌的三重PCR检测体系,即25 μL PCR反应体系中含有12.5 μL 2×HiffTMPCR Master Mix(With Dye)、0.16 μmol·L-1CC5F/CC5R、0.4 μmol·L-1CL3F/CL5R、0.16 μmol·L-1PS3F/PS4R。反应程序:95℃预变性3 min;95℃变性30 s,65℃退火30 s,72℃延伸2 min,35个循环;最后72℃延伸10 min。【结论】建立的三重PCR方法能够快速检测田间采集的黄瓜发病叶片中的黄瓜靶斑病菌、黄瓜炭疽病菌和黄瓜细菌性角斑病菌,灵敏度均可达到0.4 pg·μL-1。
黄瓜靶斑病菌;黄瓜炭疽病菌;黄瓜细菌性角斑病菌;分子检测;三重PCR
0 引言
【研究意义】黄瓜靶斑病菌多主棒孢(Corynespora cassiicola)是半知菌亚门棒孢属(Corynespora)的植物病原真菌,其寄主范围十分广泛,可危害黄瓜、茄子、番茄、烟草、橡胶等多种蔬菜和经济作物。该病菌危害黄瓜叶片形成靶斑病,严重时也可侵染叶柄和茎蔓,已成为影响黄瓜生产的重要病害之一[1-2]。黄瓜炭疽病菌(Colletotrichum orbiculare)和丁香假单胞杆菌流泪病致病变种(Pseudomonas syringae pv. lachrymans)也是黄瓜上另外两个重要的病原菌,分别引起黄瓜炭疽病[3-4]和黄瓜细菌性角斑病[5]。黄瓜炭疽病从幼苗到成株期均可发病,严重时叶片干枯,茎蔓和叶柄染病;黄瓜细菌性角斑病主要危害叶片和瓜条。黄瓜靶斑病、炭疽病和细菌性角斑病早期在黄瓜叶片上常常会形成相似症状而难于区分,如黄瓜靶斑病在不同黄瓜品种上的早期症状表现为黄褐色小斑点,周围有时会有黄色晕圈,易与细菌性角斑病、炭疽病混淆[6],因此,生产上容易误将防治炭疽病或细菌性角斑病的农药用于靶斑病的防治,导致防治效果不佳,也耽误了正确用药的最佳时机。病害的早期防治对病害的发生及控制非常关键,因此在病害的发病初期进行准确快速的诊断具有重要意义。【前人研究进展】传统的病害诊断方法主要是根据植株的发病症状、病原的形态特征和柯赫氏法则[7-10],但是操作复杂、耗时,往往耽误病害防治的关键时期。近年来,PCR分子检测技术广泛应用于病害的诊断,使得病害诊断更加准确、快速、便捷[7],目前已经有利用多种PCR技术如常规PCR、实时荧光定量PCR和三重PCR快速分子检测黄瓜病原菌的报道。陈璐等[6]利用常规PCR特异扩增actin基因片段来检测黄瓜靶斑病菌;王伟青等[11]根据ITS序列开发出黄瓜靶斑病种子带菌的特异性 PCR分子检测方法,可特异检测出带菌种子;高苇等[12]利用实时荧光定量PCR技术,建立了快速、准确、特异检测土壤中黄瓜靶斑病菌的方法;KUAN等[13-14]分别利用real-time PCR技术建立了特异检测炭疽病菌的方法;王哲等[15-16]分别利用PCR技术建立了快速检测黄瓜细菌性角斑病菌的方法;王楠等[17]利用三重 PCR技术同时特异检测黄瓜炭疽病菌、菌核病菌和细菌性萎蔫病菌,提高了检测效率。【本研究切入点】上述研究主要是针对黄瓜靶斑病菌、炭疽病菌和细菌性角斑病菌中的单一病菌进行 PCR分子检测,因此开发同时检测黄瓜靶斑病菌、炭疽病菌和细菌性角斑病菌的三重 PCR特异检验方法可以提高黄瓜靶斑病、炭疽病和细菌性角斑病的早期诊断效率。【拟解决的关键问题】分别设计黄瓜靶斑病菌、炭疽病菌和细菌性角斑病菌的特异性引物,建立三重PCR检测方法并对其反应体系及反应条件进行优化,开发一种快速、灵敏的检测方法,为这3种病害的早期防控提供技术支持。
1 材料与方法
试验于2014年9月至2015年12月在上海市农业科学院生态环境保护研究所完成。
1.1供试菌株
供试菌株的种名、来源及数量见表 1,其中黄瓜靶斑病菌、炭疽病菌和细菌性角斑病菌为检测病菌。
1.2 基因组DNA提取
1.2.1 病原真菌基因组 DNA的提取 从上海田间新采集的黄瓜白粉病和黄瓜霜霉病发病叶片表面分别刮取专性寄生真菌白粉菌和古巴假霜霉菌孢子,参照HARJU等[18]的方法提取其基因组DNA。灰葡萄孢、链格孢、尖镰孢黄瓜专化型、新月弯孢霉、拟盘多毛孢、辣椒疫霉、串珠镰孢和胶孢炭疽等兼性寄生真菌分别在PDA平板上28℃黑暗培养7 d后,用接种针挑取适量菌丝,利用CTAB法[19]提取兼性寄生真菌基因组DNA。本文提取的所有基因组DNA均采用Infinite M200 PRO NanoQuant(TECAN,瑞士)进行浓度测定。
1.2.2 病原细菌基因组 DNA的提取 分别挑取黄瓜细菌性角斑病菌丁香假单胞杆菌流泪致病变种和瓜类细菌性果斑病菌嗜酸菌西瓜亚种单菌落接种于 5 mL TSB培养基中,37℃振荡(200 r/min)培养至稳定期,参照SUO等[20]的方法提取其基因组DNA。
1.2.3 发病组织DNA的快速提取 黄瓜在温室大棚生长至16—18叶期时,分别接种黄瓜炭疽病菌和细菌性角斑病菌。采集发病早期即1级(病斑面积占整个叶面积的5%以下)或3级(病斑面积占整个叶面积的5%—10%)黄瓜炭疽病和细菌性角斑病的发病叶片,同时采集自然发病的1级或3级黄瓜靶斑病叶片和未发病的健康叶片。黄瓜靶斑病和炭疽病叶片病害分级标准参考国标 GB/T 17980.112—2004,黄瓜细菌性角斑病叶片病害分级标准参考国标GB/T 17980.110—2004。从新鲜发病叶片和健康叶片上分别剪取等质量的发病组织和健康叶片,根据 WANG等[21]的方法快速提取发病组织和健康叶片基因组DNA。
1.3 特异性引物设计
分别以多主棒孢、黄瓜炭疽病菌、灰葡萄孢、古巴假霜霉菌、白粉菌和链格孢等基因组DNA为PCR模板,用真菌ITS通用引物ITS1和ITS4[22]扩增它们的ITS序列。PCR反应体系为25.0 μL:2×HiffTMPCR plus Master Mix(With Dye)(上海翊圣生物科技有限公司)12.5 μL,ITS1/ITS4(10 μmol·L-1)各1.0 μL,DNA模板2.0 μL,ddH2O 8.5 μL。PCR反应程序:95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸45 s,35个循环;最后72℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测后送至生工生物工程(上海)股份有限公司进行序列测定。利用MEGA 4.0软件对上述各真菌ITS序列进行序列比对,再利用引物设计软件Primer Premier 5.0分别设计多主棒孢和黄瓜炭疽病菌的特异性引物(图1)。

表1 供试菌株Table 1 Fungal and bacterial strains used in this study
分别以丁香假单胞杆菌流泪致病变种及其近缘物种嗜酸菌西瓜亚种基因组DNA为PCR模板,用细菌16S rDNA通用引物F27/R1492[23]扩增其16S rDNA,PCR反应体系及条件同上。利用上述同样方法设计丁香假单胞杆菌流泪致病变种的特异性引物(图 2)。本文所用引物序列如表2所示。
1.4 特异性引物筛选

图1 黄瓜靶斑病菌和炭疽病菌的特异引物设计Fig. 1 Design of specific primers for C. cassiicola and C. orbiculare

图2 黄瓜细菌性角斑病菌的特异引物设计Fig. 2 Design of specific primers for P. syringae pv. lachrymans
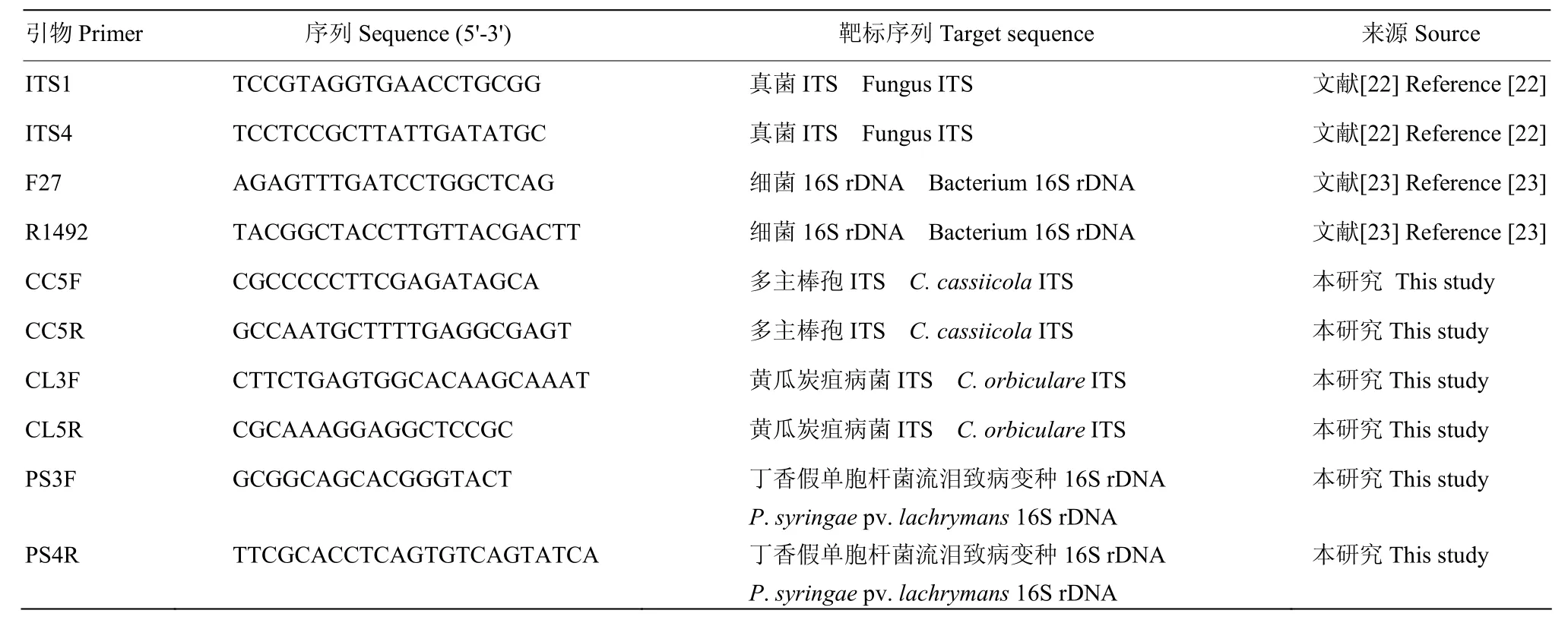
表2 引物及其序列Table 2 Primers used in this study
分别以各病菌基因组DNA做PCR模板,筛选针对多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种单一菌株的特异性引物。引物组合 CC1F/ CC1R、CC2F/CC2R、CC3F/CC3R、CC4F/CC4R、CC5F/ CC5R分别扩增多主棒孢ITS;CL1F/CL1R、CL2F/CL2R、 CL3F/CL3R、CL3F/CL4R、CL3F/CL5R、CL3F/CL6R、CL3F/CL7R分别扩增黄瓜炭疽病菌ITS;PS1F/PS1R、PS2F/PS2R、PS3F/PS3R、PS3F/PS4R、PS4F/PS3R、PS4F/PS4R分别扩增丁香假单胞杆菌流泪致病变种16S rDNA。PCR反应体系为25.0 μL:2×HiffTMPCR plus Master Mix(With Dye)12.5 μL,正向/反向引物(10 μmol·L-1)各1.0 μL,DNA模板(2 ng·μL-1)2.0 μL,ddH2O 8.5 μL。PCR反应程序:95℃预变性3 min;95℃变性30 s,65℃退火30 s,72℃延伸45 s,35个循环;最后72℃延伸10 min。PCR反应结束后,取6 μL PCR产物进行1%琼脂糖凝胶电泳检测。最终筛选出引物组合CC4F/CC4R和CC5F/CC5R可特异扩增多主棒孢ITS;CL1F/CL1R、CL2F/CL2R、CL3F/CL3R、CL3F/CL4R、CL3F/CL5R、CL3F/CL6R和CL3F/CL7R可特异扩增黄瓜炭疽病菌 ITS;PS3F/PS4R和PS4F/PS4R可特异扩增丁香假单胞杆菌流泪致病变种16S rDNA。
1.5 三重PCR反应体系建立及优化
1.5.1 引物组合的选择 引物组合 CC5F/CC5R、CL3F/CL5R和 PS3F/PS4R扩增片段的长度分别为370、275和698 bp,其混合产物可在3%琼脂糖凝胶上被有效分离,因此选择作为三重PCR引物。
1.5.2 引物浓度优化 为了在同一三重PCR反应中使 3个目的片段均得到有效扩增,需要对 CC5F/ CC5R、CL3F/CL5R和PS3F/PS4R的浓度进行优化。在25 μL PCR反应体系中,设定CC5F/CC5R的3个浓度梯度为0.08、0.16和0.24 μmol·L-1,CL3F/CL5R 的 3个浓度梯度为 0.24、0.32和 0.40 μmol·L-1,PS3F/PS4R的3个梯度为0.16、0.24和0.32 μmol·L-1。根据3对引物3个不同浓度梯度,设置18个引物用量组合(表3)。三重PCR反应体系为25.0 μL:2×HiffTMPCR plus Master Mix(With Dye)12.5 μL,CC5F/CC5R (10 μmol·L-1) 0.2/0.4/0.6 μL,CL3F/CL5R(10 μmol·L-1)0.6/0.8/1.0 μL,PS3F/PS4R(10 μmol·L-1)0.4/0.6/0.8 μL,多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种基因组DNA(2 ng·μL-1)各2.0 μL,补加ddH2O至25.0 μL。三重PCR反应程序:95℃预变性3 min;95℃变性30 s,65℃退火30 s,72℃延伸2 min,35个循环;最后72℃延伸10 min。PCR反应结束后,取6 μL PCR产物进行3%琼脂糖凝胶电泳检测。
1.5.3 退火温度优化 设定 65、66、67和 68℃ 4个退火温度梯度。三重PCR反应体系为25.0 μL:2× HiffTMPCR plus Master Mix(With Dye)12.5 μL,CC5F/CC5R(10 μmol·L-1)0.4 μL,CL3F/CL5R(10 μmol·L-1)1.0 μL,PS3F/PS4R(10 μmol·L-1)0.6 μL,多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种基因组DNA(2 ng·μL-1)各2.0 μL,补加ddH2O 至25.0 μL。三重PCR反应程序:95℃预变性3 min;95℃变性30 s,65/66/67/68℃退火30 s,72℃延伸2 min,35个循环;最后72℃延伸10 min。PCR反应结束后,取6 μL PCR产物进行3%琼脂糖凝胶电泳检测。1.5.4 延伸时间和循环次数优化 设定 0.5、1、1.5 和2 min 4个延伸时间;25、30、35、40和45次5个循环次数。最终确定最佳延伸时间为 2 min,最佳循环次数为35次。

表3 三重PCR反应体系中引物CC5F/CC5R、PS3F/PS4R和CL3F/CL5R的浓度Table 3 Concentration of CC5F/CC5R, PS3F/PS4R and CL3F/ CL5R used in triplex PCR
1.6 三重PCR特异性检测
根据多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种基因组DNA的浓度,将3种病菌基因组DNA等量混匀。分别以多主棒孢、灰葡萄孢、古巴假霜霉菌、白粉菌、黄瓜炭疽病菌、丁香假单胞杆菌流泪致病变种和链格孢基因组DNA以及多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种混合基因组DNA做模板,利用优化好的三重PCR反应体系和条件进行三重PCR,验证该三重PCR体系的特异性。
1.7 三重PCR灵敏度分析
在 25 μL反应体系中,将 3种目标病菌基因组DNA模板浓度 10倍梯度稀释至 0.4 ng·μL-1、0.04 ng·μL-1、4 pg·μL-1、0.4 pg·μL-1、0.04 pg·μL-1、4 fg·μL-1、0.4 fg·μL-1和0.04 fg·μL-1。利用优化好的三重PCR体系和条件进行扩增,根据扩增产物的 3%琼脂糖凝胶电泳结果确定三重PCR反应的灵敏度。
1.8 黄瓜发病叶片DNA的特异检测
根据黄瓜靶斑病、黄瓜炭疽病和黄瓜细菌性角斑病发病叶片的基因组DNA浓度,将3种发病叶片基因组DNA等量混匀以及两两混匀,分别作为三重PCR特异检测体系的DNA模板,同时以健康叶片基因组DNA作为对照。利用优化好的三重PCR体系进行扩增,特异检测发病叶片中的黄瓜靶斑病菌、黄瓜炭疽病菌和黄瓜细菌性角斑病菌,验证该三重PCR特异检测体系。
2 结果
2.1 引物组合CC5F/CC5R、CL3F/CL5R和PS3F/PS4R
的特异性检测
CC5F/CC5R、CL3F/CL5R和PS3F/PS4R分别在多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种基因组DNA中扩增出370、275和 698 bp的特异性目的片段,而在灰葡萄孢、古巴假霜霉菌、白粉菌、链格孢、嗜酸菌西瓜亚种、尖镰孢黄瓜专化型、新月弯孢、拟盘多毛孢、辣椒疫霉、串珠镰孢、胶孢炭疽等非目标病菌基因组DNA和阴性对照中没有扩增出条带(图3)。
2.2 三重PCR反应体系及条件优化
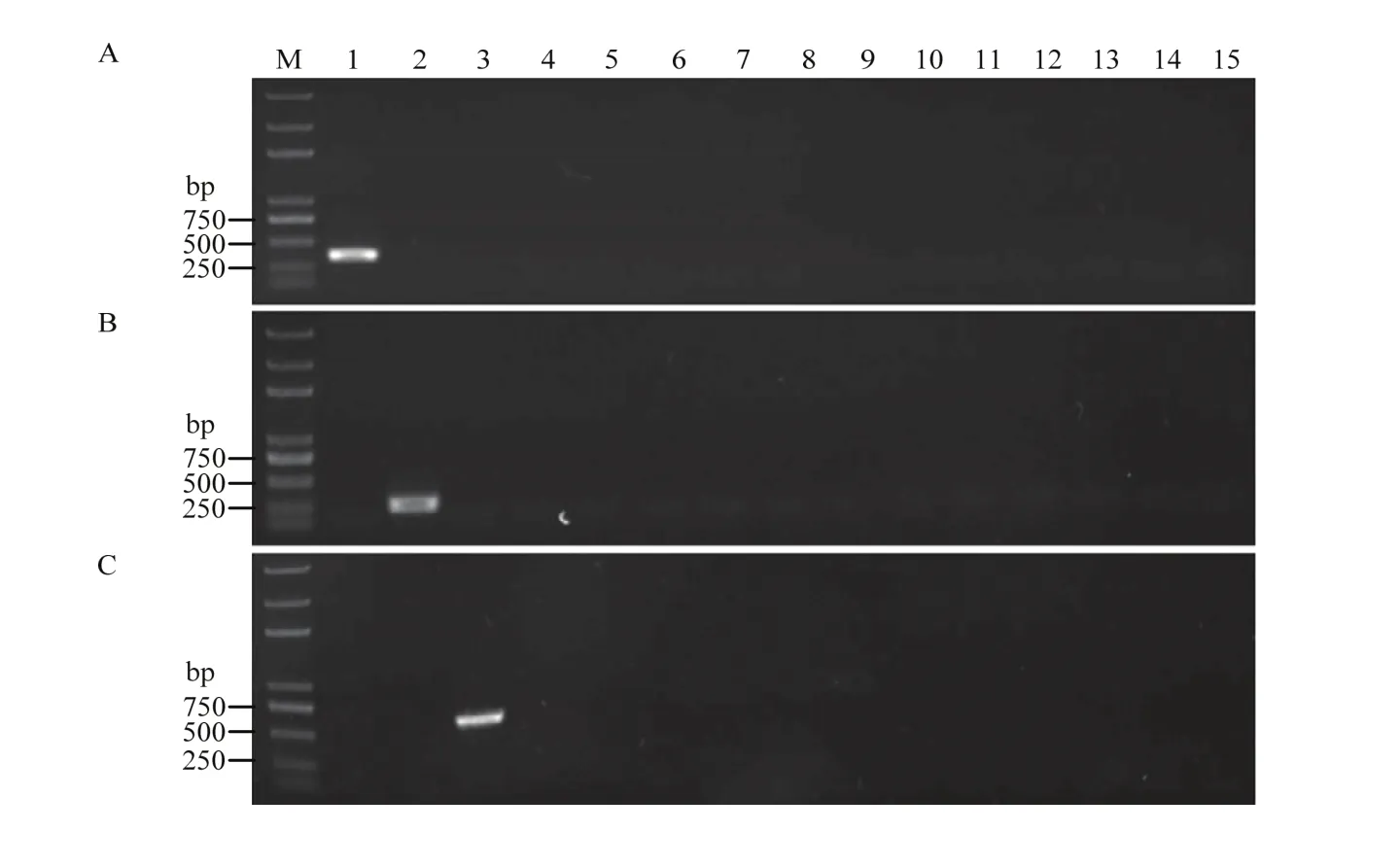
图3 引物CC5F/CC5R、CL3F/CL5R和PS3F/PS4R的特异性检测Fig. 3 Specificity test of CC5F/CC5R, CL3F/CL5R and PS3F/PS4R primer pairs
通过对三重PCR反应体系中引物CC5F/CC5R、CL3F/CL5R和PS3F/PS4R的浓度进行优化,发现25 μL 反应体系中 CC5F/CC5R、CL3F/CL5R 和PS3F/PS4R的终浓度分别为0.16、0.4和0.16 μmol·L-1时能同时有效扩增3个目的片段(图4)。当三重PCR退火温度大于 65℃时,部分目的片段不能有效扩增(图5)。最终优化的三重PCR反应体系:2×HiffTMPCR Plus Master Mix(With Dye)12.5 μL,CC5F/CC5R、CL3F/CL5R和PS3F/PS4R的终浓度分别是0.16、0.4 和0.16 μmol·L-1,3种目标病菌基因组DNA模板(2 ng·μL-1)各2.0 μL,补加ddH2O至25.0 μL;优化的三重PCR反应条件:95℃预变性3 min;95℃变性30 s,65℃退火30 s,72℃延伸2 min,35个循环;最后72℃延伸10 min。
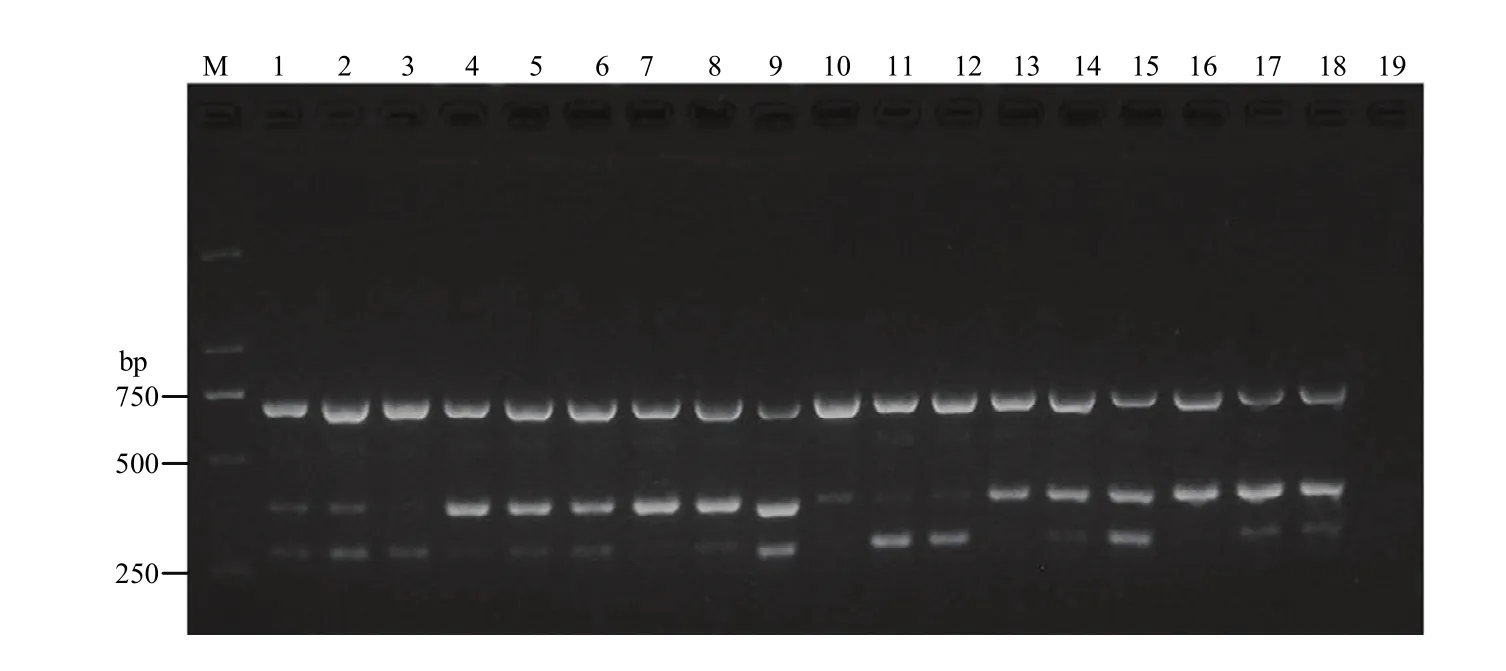
图4 三重PCR引物浓度优化Fig. 4 Optimization for the concentration of primers used in triplex PCR
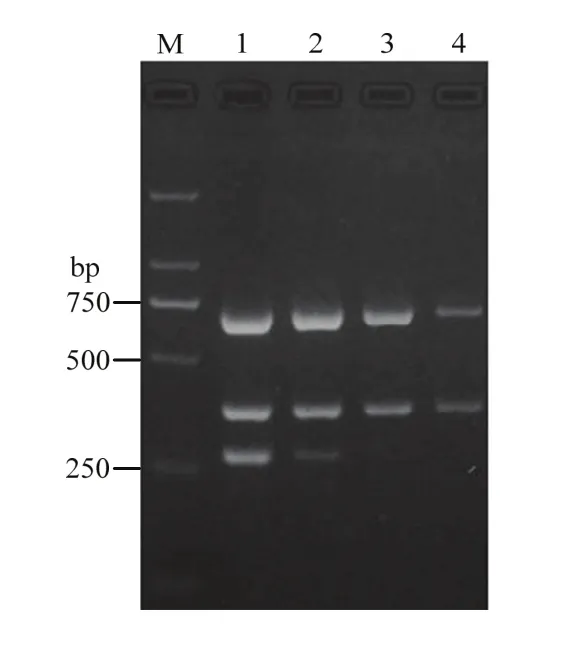
图5 三重PCR退火温度优化Fig. 5 Optimization for the annealing temperature of triplex PCR
2.3 三重PCR特异性检测
利用优化的三重PCR,同时扩增目标和非目标病菌基因组DNA,以检测该三重PCR扩增目标病菌基因组DNA的特异性。结果表明,该三重PCR可在目标菌株的混合或非混合基因组DNA中扩增出清晰的特异性目的条带,即在多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种基因组DNA中分别扩增出长度为370、275和698 bp的特异条带,而在灰葡萄孢、古巴假霜霉菌、白粉菌、链格孢、嗜酸菌西瓜亚种、尖镰孢黄瓜专化型、新月弯孢、拟盘多毛孢、辣椒疫霉、串珠镰孢、胶孢炭疽等非目标病菌基因组DNA和阴性对照中没有扩增出条带(图6)。
2.4 三重PCR检测体系灵敏度分析
为了确定优化后三重PCR的灵敏度,将25 μL反应体系中多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种基因组DNA的浓度从0.4 ng·μL-1依次10倍梯度稀释至0.04 fg·μL-1,并用优化的反应体系及条件进行扩增。结果表明,三重PCR同时扩增3个模板时,能够检测到0.4 pg·μL-1的基因组DNA(图7)。
2.5 黄瓜发病叶片的三重PCR快速检测
在25 μL PCR反应体系中,分别以发病黄瓜叶片基因组DNA做模板,同时以多主棒孢、黄瓜炭疽病菌和丁香假单胞杆菌流泪致病变种等量混合基因组DNA做阳性对照,利用三重PCR特异检测这3种病菌。结果显示,在黄瓜靶斑病、黄瓜炭疽和黄瓜细菌性角斑病的黄瓜发病叶片基因组DNA中分别能扩增出长度为370、275和698 bp的特异片段,其长度与阳性对照一致,而在健康黄瓜叶片DNA和阴性对照中没有扩增出条带(图8),说明该三重PCR检测体系可以对发病叶片中的目标病菌进行特异检测,可以同时对黄瓜靶斑病、黄瓜炭疽病和黄瓜细菌性角斑病进行早期快速分子诊断。

图6 三重PCR特异检测病菌Fig. 6 Specific detection of triplex PCR for pathogens

图7 三重PCR灵敏度分析Fig. 7 Sensitivity assay of triplex PCR
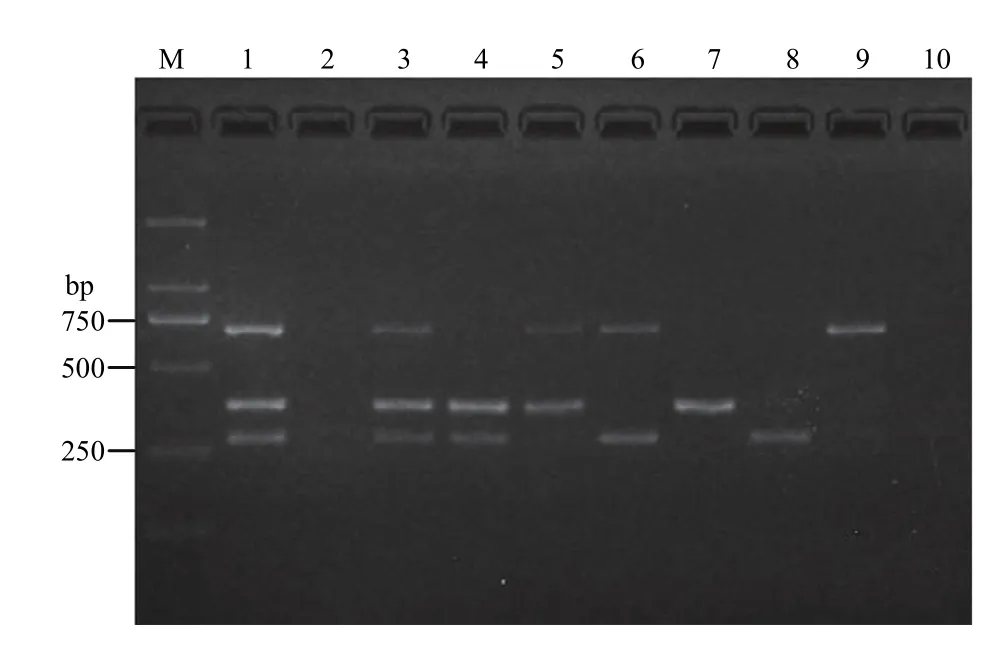
图8 三重PCR特异扩增病菌侵染的黄瓜叶片DNAFig. 8 Specific amplification for the mix of pathogen-infected cucumber tissues DNA by triplex PCR
3 讨论
多重 PCR主要应用于多种病原微生物的同时检测,具有省时、成本低、效率高的优点[24]。黄瓜靶斑病、炭疽病和细菌性角斑病在症状上存在相似之处,在病害诊断上容易将这3种病害混淆。此外,针对单一病害的分子检测不能快速、准确对病害进行甄别,往往错用防治药剂或耽搁病害的最佳防治时期。因此,亟需开发一种同时检测这3种病害的多重PCR检测技术。
目前,针对瓜类黑星病菌、枯萎病菌和蔓枯病菌以及黄瓜炭疽病菌、菌核病菌和细菌性萎蔫病菌的三重PCR检测已经有报道[17,25],但针对黄瓜靶斑病菌、炭疽病菌和细菌性角斑病菌的三重 PCR检测方法未见相关报道,只是对单一病菌进行特异分子检测[6,16]。多重PCR体系中存在两对以上引物,因此必须保证多对引物之间不形成引物二聚体;同时又要确保扩增产物的片段长度不能太接近,否则难以在琼脂糖凝胶上得到充分的分离;由于在扩增时不同引物对之间竞争其他反应组分,必须对反应体系中不同引物对的浓度进行优化[26]。除了引物之外,Taq DNA聚合酶、dNTP、MgCl2、退火温度、延伸时间和循环次数等对多重PCR结果也均有影响[24,26]。为了简化多重PCR操作过程,本试验选用2×HiffTMPCR Plus Master Mix来替代Taq DNA聚合酶、dNTP和MgCl2,不需要对Taq DNA聚合酶、dNTP和MgCl2进行优化。因此本试验对引物浓度、退火温度、延伸时间和循环次数进行了优化,摸索出可同时检测黄瓜靶斑病菌、黄瓜炭疽病菌和黄瓜细菌性角斑病菌最优的三重PCR反应体系及条件。
该三重PCR检测体系的灵敏度可达0.4 pg·μL-1,与高永洋等(1 pg·μL-1)[25]和王楠等(0.4 pg·μL-1)[17,27]开发的三重PCR检测方法接近,处于同一数量级上;比陈璐等(4 pg·μL-1)[6]的黄瓜靶斑病菌PCR检测方法高1个数量级,但比王伟青等(50—100 fg·μL-1)[11]的黄瓜靶斑病种子带菌PCR分子检测低约1个数量级,可能是由于引物扩增效率和引物扩增序列的拷贝数不同导致这种差异[28]。
4 结论
三重PCR反应体系25 μL:12.5 μL 2×HiffTMPCR Plus Master Mix(With Dye),0.16 μmol·L-1CC5F/ CC5R、0.4 μmol·L-1CL3F/CL5R和0.16 μmol·L-1PS3F/ PS4R;三重 PCR反应程序:95℃预变性 3 min,35个循环(95℃变性30 s,65℃退火30 s,72℃延伸2 min),最后72℃延伸10 min。该检测体系可在4 h内从田间病株叶片中检测出黄瓜靶斑病菌、炭疽病菌和细菌性角斑病菌,其灵敏度可达0.4 pg·μL-1。
References
[1] 曾蓉, 陆金萍, 戴富明. 上海地区黄瓜靶斑病病原鉴定及ITS的分析. 上海交通大学学报 (农业科学版), 2011, 29(4): 13-16. ZENG R, LU J P, DAI F M. Pathogen identification of cucumber target spot diseae in Shanghai and its sequence analysis. Journal of Shanghai Jiaotong University (Agricultural Science), 2011, 29(4):13-16. (in Chinese)
[2] SHIMOMOTO Y, SATO T, HOJO H, MORITA Y, TAKEUCHI S,
MIZUMOTO H, KIBA A, HIKICHI Y. Pathogenic and genetic variation among isolates of Corynespora cassiicola in Japan. Plant Pathology, 2011, 60(2): 253-260.
[3] IRIEDA H, TAKANO Y. Identification and characterization of virulence-related effectors in the cucumber anthracnose fungus Colletotrichum orbiculare. Physiological and Molecular Plant Pathology, 2016, 95: 87-92.
[4] FUKADA F, KUBO Y. Colletotrichum orbiculare regulates cell cycle G1/S progression via a two-component GAP and a GTPase to establish plant infection. The Plant Cell, 2015, 27(9): 2530-2544.
[5] CHOJAK J, KUŹNIAK E, ŚWIERCZ U, SEKULSKA- NALEWAJKO J,GOCŁAWSKI J. Interaction between salt stress and angular leaf spot (Pseudomonas syringae pv. lachrymans) in cucumber. Vegetable Crops Research Bulletin, 2012, 77: 5-16.
[6] 陈璐, 石延霞, 谢学文, 柴阿丽, 李宝聚. 黄瓜棒孢叶斑病菌 PCR检测方法的建立. 园艺学报, 2014, 41(3): 585-592. CHEN L, SHI Y X, XIE X W, CHAI A L, LI B J. PCR assay for detection of Corynespora cassiicola, the causal agent of Corynespora leaf spot of cucumber. Acta Horticulturae Sinica, 2014, 41(3):585-592. (in Chinese)
[7] MA Z H, MICHAILIDES T J. Approaches for eliminating PCR inhibitors and designing PCR primers for the detection of phytopathogenic fungi. Crop Protection, 2007, 26(2): 145-161.
[8] LIEVENS B, THOMMA B P. Recent developments in pathogen detection arrays: implications for fungal plant pathogens and use in practice. Phytopathology, 2005, 95(12): 1374-1380.
[9] ALVAREZ A M. Integrated approaches for detection of plant pathogenic bacteria and diagnosis of bacterial diseases. Annual Review of Phytopathology, 2004, 42: 339-366.
[10] 刘志恒, 郑川, 黄欣阳, 唐爽爽, 李健冰, 焦俊. 茄子绒菌斑病病原菌鉴定及生物学特性研究. 植物保护, 2014, 40(3): 58-64. LIU Z H, ZHENG C, HUANG X Y, TANG S S, LI J B, JIAO J. Pathogen identification and biological characteristics of eggplant leaf mold. Plant Protection, 2014, 40(3): 58-64. (in Chinese)
[11] 王伟青, 岳瑾, 董杰. 黄瓜棒孢叶斑病种子带菌PCR分子检测. 中国植保导刊, 2013, 33(8): 13-17. WANG W Q, YUE J, DONG J. Molecular detection of Corynespora cassiicola in cucumber seed by PCR. China Plant Protection, 2013,33(8): 13-17. (in Chinese)
[12] 高苇, 李宝聚, 王万立, 郝永娟, 石延霞. 土壤中黄瓜棒孢叶斑病病原菌实时荧光定量PCR检测技术研究. 华北农学报, 2014, 29(2):71-74. GAO W, LI B J, WANG W L, HAO Y J, SHI Y X. Detection of Corynespora cassiicola in soil with real-time quantitative PCR. Acta Agriculturae Boreali-Sinica, 2014, 29(2): 71-74. (in Chinese)
[13] KUAN C P, WU M T, HUANG H C, CHANG H. Rapid detection of Colletotrichum lagenarium, causal agent of anthracnose of cucurbitaceous crops, by PCR and real-time PCR. Journal of Phytopathology, 2011,159(4): 276-282.
[14] YANG H C, HAUDENSHIELD J, HARTMAN G. Multiplex real-time PCR detection and differentiation of Colletotrichum species infecting soybean. Plant Disease, 2015, 99(11): 1559-1568.
[15] 王哲, 陈青, 田茜, 李志红, 朱水芳, 赵文军. 应用 PCR方法快速检测黄瓜细菌性角斑病菌. 植物检疫, 2011, 25(6): 29-32. WANG Z, CHEN Q, TIAN Q, LI Z H, ZHU S F, ZHAO W J. Using PCR for rapid detection of Pseudomonas syringae pv. lachrymans. Plant Quarantine, 2011, 25(6): 29-32. (in Chinese)
[16] 王平, 樊金娟, 刘长远, 赵奎华, 梁春浩. 黄瓜细菌性角斑病的分子检测. 中国农学通报, 2012, 28(25): 150-153. WANG P, FAN J J, LIU C Y, ZHAO K H, LIANG C H. The detection of Pseudomonas syringae pv. based on PCR. Chinese Agricultural Science Bulletin, 2012, 28(25): 150-153. (in Chinese)
[17] 王楠, 王伟. 三重 PCR检测黄瓜炭疽病菌、菌核病菌和细菌性萎蔫病菌. 植物病理学报, 2014, 44(2): 129-138. WANG N, WANG W. Triplex PCR detection of Colletotrichum orbiculare, Sclerotinia sclerotiorum and Erwinia tracheiphila in infected cucumber tissues. Acta Phytopathologica Sinica, 2014, 44(2):129-138. (in Chinese)
[18] HARJU S, FEDOSYUK H, PETERSON K R. Rapid isolation of yeast genomic DNA: Bust n' Grab. BMC Biotechnology, 2004, 4: 8.
[19] GAO S G, ZHOU F H, LIU T, LI Y Y, CHEN J. A MAP kinase gene,Clk1, is required for conidiation and pathogenicity in the phytopathogenic fungus Curvularia lunata. Journal of Basic Microbiology, 2013, 53:214-223.
[20] SUO Y J, HUANG Y Y, LIU Y H, SHI C L, SHI X M. The expression of superoxide dismutase (SOD) and a putative ABC transporter permease is inversely correlated during biofilm formation in Listeria monocytogenes 4b G. PLoS ONE, 2012, 7(10): e48467.
[21] WANG H, QI M, CUTLER A J. A simple method of preparing plant samples for PCR. Nucleic Acids Research, 1993, 21(17): 4153-4154.
[22] WHITE T J, BRUNS T, LEE S, TAYLOR J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics//INNIS M A, GELFAND D H, SNINSKY J J, WHITE T J. PCR Protocols: A Guide to Methods and Applications. London, USA: Academic Press,1990: 315-322.
[23] LANE D J. 16S/23S rRNA sequencing//STACKEBRANDT E,GOODFELLOW M. Nucleic Acid Techniques in Bacterial Systematics. NY, USA: John Wiley & Sons New York, 1991: 115-175.
[24] 赵红庆, 苑锡铜, 黄留玉. 多重PCR技术在病原检测中的应用. 生物技术通讯, 2007, 18(5): 863-865. ZHAO H Q, YUAN X T, HUANG L Y. Application of multiplex PCR in detection of pathogens. Letters in Biotechnology, 2007, 18(5):863-865. (in Chinese)
[25] 高永洋, 王楠, 高观朋, 王伟. 瓜黑星病菌、枯萎病菌和蔓枯病菌的三重PCR检测. 植物病理学报, 2010, 40(4): 343-350. GAO Y Y, WANG N, GAO G P, WANG W. Triplex PCR detection of Cladosporium cucumerinum, Fusarium oxysporum f. sp. niveum and Mycosphaerella melonis in infected plant tissues. Acta Phytopathologica Sinica, 2010, 40(4): 343-350. (in Chinese)
[26] MARKOULATOS P, SIAFAKAS N, MONCANY M. Multiplex polymerase chain reaction: a practical approach. Journal of Clinical Laboratory Analysis, 2002, 16(1): 47-51.
[27] 王楠, 王剑, 尹丹韩, 高观朋, 王伟. 三重 PCR检测草莓灰霉病菌、炭疽病菌和黄萎病菌. 中国农业科学, 2010, 43(21): 4392-4400. WANG N, WANG J, YIN D H, GAO G P, WANG W. Triplex PCR detection of Botrytis cinerea, Colletotrichum gloeosporioides and Verticillium dahliae in infected strawberry plant tissues. Scientia Agricultura Sinica, 2010, 43(21): 4392-4400. (in Chinese)
[28] EDWARDS M C, GIBBS R A. Multiplex PCR: advantages development, and application. PCR Methods and Applications, 1994,3(4): 65-75.
(责任编辑 岳梅)
Triplex PCR Detection for Corynespora cassiicola, Colletotrichum orbiculare and Pseudomonas syringae pv. lachrymans
GAO Shi-gang1,2, ZENG Rong1,2, XU Li-hui1,2, LUO Jin-yan3, CHEN Lei3, DAI Fu-ming1,2
(1Institute of ECO-Environment and Plant Protection, Shanghai Academy of Agricultural Sciences, Shanghai 201403;2Key Laboratory of Horticultural Facilities in Shanghai, Shanghai 201403;3Shanghai City Agricultural Technology Extension and Service Center, Shanghai 201103)
【Objective】The objective of this study is to develop a rapid triplex PCR detection method of Corynespora cassiicola, Colletotrichum orbiculare and Pseudomonas syringae pv. lachrymans. 【Method】 Specific primers for the three pathogens were designed based on ITS sequences of C. cassiicola and C. orbiculare and 16S rDNA sequence of P. syringae pv. lachrymans, which were confirmed by amplifying specific fragments. Three pairs of primers specific to C. cassiicola, C. orbiculare or P. syringae pv. lachrymans were suitable for triplex PCR and selected for triplex PCR. Primer concentration, annealing temperature, extension time and cycle number were optimized to develop the triplex PCR detection system. 【Result】Five, seven andsix pairs of primers were designed specific to C. cassiicola, C. orbiculare and P. syringae pv. lachrymans, respectively. CC4F/CC4R and CC5F/CC5R primers could specifically amplify ITS sequence of C. cassiicola; CL1F/CL1R, CL2F/CL2R, CL3F/CL3R,CL3F/CL4R, CL3F/CL5R, CL3F/CL6R and CL3F/CL7R could specifically amplify ITS sequence of C. orbiculare; PS3F/PS4R and PS4F/PS4R primers could specifically amplify 16S rDNA sequence of P. syringae pv. lachrymans. Fragments in lengths of 370, 275 and 698 bp were amplified by CC5F/CC5R, CL3F/5R and PS3F/4R, respectively, which were separated fully by 3% agarose gel electrophoresis, and the three pairs of primers were used for triplex PCR. Three target fragments were amplified effectively with 0.16 μmol·L-1of CC5F/CC5R, 0.4 μmol·L-1of CL3F/5R and 0.16 μmol·L-1of PS3F/4R in 25 μL of triplex PCR system. Part target fragments were not amplified effectively at >65℃ of annealing temperature. Finally, the triplex PCR system suitable for the detection of C. cassiicola, C. orbiculare and P. syringae pv. lachrymans were developed and confirmed. This PCR system contained 12.5 μL of 2×HiffTMPCR Master Mix (With Dye), 0.16 μmol·L-1of CC5F/CC5R, 0.4 μmol·L-1of CL3F/CL5R and 0.16 μmol·L-1of PS3F/PS4R. The PCR program was as follows: an initial denaturation at 95℃ for 3 min followed by 35 cycles of denaturation (95℃for 30 s), annealing (65℃ for 30 s) and extension (72℃ for 2 min), and a final extension at 72℃ for 10 min. 【Conclusion】The detection method could rapidly detect C. cassiicola, C. orbiculare and P. syringae pv. lachrymans from leaves infected by the three pathogens, the sensitivity of which was 0.4 pg·μL-1.
Corynespora cassiicola; Colletotrichum orbiculare; Pseudomonas syringae pv. lachrymans; molecular detection;triplex PCR
2016-01-28;接受日期:2016-02-24
上海市科技兴农重点攻关项目[沪农科攻字(2012)第2-10号,沪农科攻字(2013)第5-3号]、上海市科技兴农重大项目[沪农科重字(2010)第4-1号]
联系方式:高士刚,E-mail:shggao@163.com。通信作者戴富明,Tel:021-62202956;E-mail:fumingdai@163.com
