基因工程中的单酶切与双酶切
2016-08-20舒端阳
舒端阳
(广东省揭阳华侨高级中学 522000)
1 单酶切
由于高中生普遍没有接触过分子生物学,概念上比较模糊,所以高中生物学教材只介绍相对简单的单酶切,有利于学生的理解和接受。但单酶切在实验室操作的时候,容易出现反连甚至是载体之间的自连现象,因此单酶切的方法并非适应基因工程的所有项目。
例如,2012年高考江苏卷第32题就涉及此问题,相关信息如下:图1 表示含有目的基因D的DNA 片段长度(bp即碱基对)和部分碱基序列,图2表示一种质粒的结构和部分碱基序列。现有MspI、BamHI、MboI、SmaI 4 种限制性核酸内切酶,它们识别的碱基序列和酶切位点分别为C↓CGG、G↓GATCC、↓GATC、CCC↓GGG。提出的问题是:若将图2中质粒和目的基因D通过同种限制酶处理后进行连接,形成重组质粒。但经检测,部分含有重组质粒的大肠杆菌菌株中目的基因D不能正确表达,其最可能的原因是什么?
分析:根据题意,应采用BamHI进行单酶切。所得到的目的基因D与质粒两头的黏性末端都是相同的。由于在进行连接反应时,目的基因和载体是随机碰撞的,且两者的两端黏性末端都相同。所以,基因D既可以与质粒正向连接(正连),即目的基因顺时针地按其上游到下游的方向与载体相连(与启动子的顺时针方向一致),这样,目的基因就能从上游到下游正常转录,表达产生所需的蛋白质,产生预期的性状;目的基因也可以与质粒反向连接(反连),即目的基因的上下游方向颠倒地与载体相连,结果目的基因的转录从下游到上游,导致密码子全部改变,目的基因不能正常表达。为了防止出现反连现象,实验中常用双酶切的方法。
2 双酶切
双酶切,即采用两种不同的限制酶同时处理目的基因和质粒。因为限制酶具有特异性,不同的酶作用于不同的碱基序列,所以得到的黏性末端也是不同的。当应用DNA连接酶进行连接时,就只能存在一种正连情况,而不会出现反连的问题。正因为可以有效地防止错误连接的现象出现,双酶切的做法被广泛应用于基因工程中表达载体的构建,也可以用于制备体外转录的模板。
RNA干扰(RNAi)是由双链RNA(dsRNA)引起的同源mRNA特异性降解的现象,可以定向地关闭某个基因的表达,因而被广泛应用于生物学的众多领域。要对某个基因进行有效的沉默,就需要得到与这个基因具有相同序列的dsRNA,而制备dsRNA的过程需要应用双酶切和单酶切的处理方法。
如何制备dsRNA呢?通常要选择一种叫做pLITMUS28i的质粒,该质粒最大的特点是带有两个T7启动子,且转录方向是相反的,在两个启动子之间有一些常用的酶切位点。
首先,用双酶切(BamHI和EcoRI)同时处理目的基因和质粒pLITMUS28i;连接后获得重组质粒;将重组质粒平均分成两份,分别被BamHI和EcoRI进行单酶切,获得转录用的模板;加入RNA聚合酶和4种核糖核苷酸(NTP)后,在室温下进行体外转录,得到单链RNA。因为这些单链RNA本身是互补的,所以低温下退火即可制备得到dsRNA,通过脂质体转入相应受体细胞就可用于进行RNAi的相关实验。
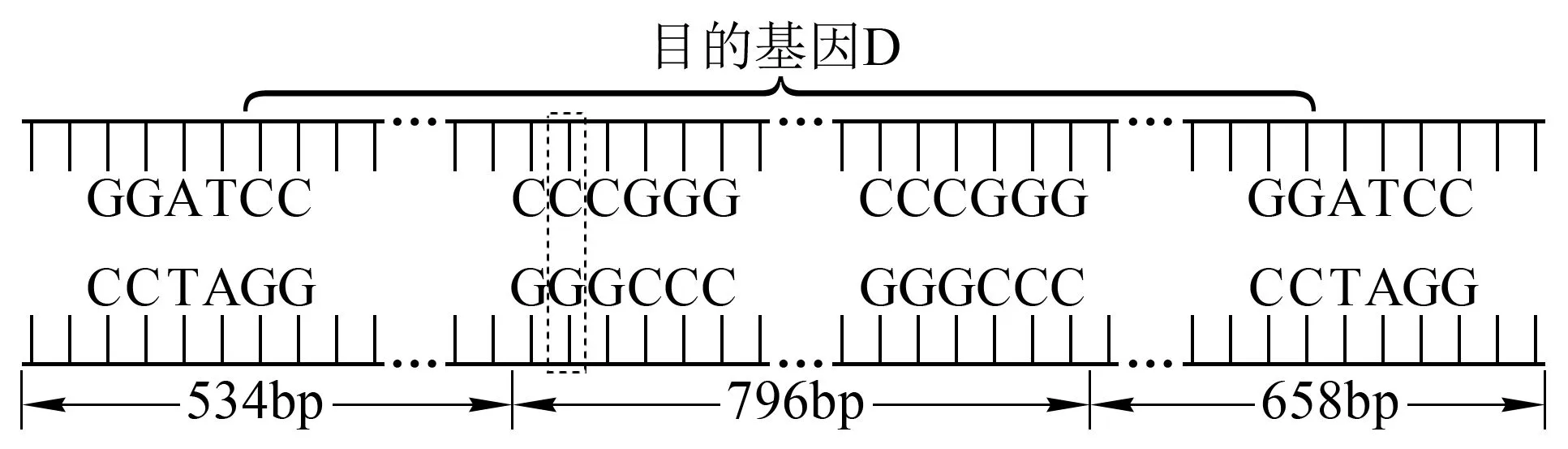
图1 含有目的基因D的DNA片段长度和部分碱基序列
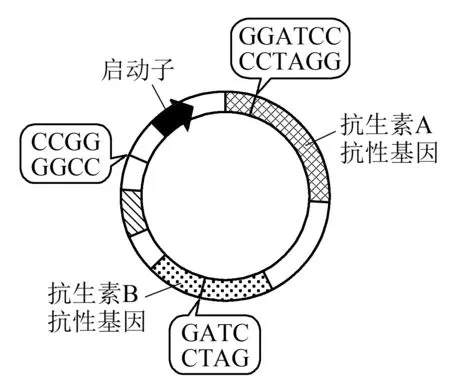
图2 一种质粒的结构和部分碱基序列
