类酵母共生菌中两个组氨酸合成基因在褐飞虱发育中的作用
2016-08-09唐耀华万品俊郝培应傅强俞晓平
唐耀华 万品俊 郝培应 傅强,* 俞晓平,*
(1中国计量大学 生命科学学院 浙江省生物计量与检验检疫技术重点实验室, 杭州 310018;2中国水稻研究所 水稻生物学国家重点实验室, 杭州 310006; *通讯联系人, E-mail: fuqiang@caas.cn; yxp@cjlu.edu.cn)
类酵母共生菌中两个组氨酸合成基因在褐飞虱发育中的作用
唐耀华1,2万品俊2郝培应1傅强2,*俞晓平1,*
(1中国计量大学 生命科学学院 浙江省生物计量与检验检疫技术重点实验室, 杭州 310018;2中国水稻研究所 水稻生物学国家重点实验室, 杭州 310006;*通讯联系人,E-mail:fuqiang@caas.cn;yxp@cjlu.edu.cn)
TANGYaohua,WANPinjun,HAOPeiying,etal.Rolesoftwogenesinvolvedinhistidinebiosyntheticpathwayinyeast-likesymbiontindevelopmentofNilaparvata lugens (Stål).ChinJRiceSci, 2016, 30(4): 406-416.
唐耀华, 万品俊, 郝培应, 等. 类酵母共生菌中两个组氨酸合成基因在褐飞虱发育中的作用. 中国水稻科学, 2016, 30(4): 406-416.
摘要:褐飞虱[Nilaparvata lugens (Stål)]是我国水稻上的主要害虫,专一性吸食必需氨基酸缺乏的水稻筛管液。利用基因组和转录组数据,人们构建了褐飞虱及其体内类酵母共生菌(YLS)参与组氨酸等必需氨基酸的合成途径。在此基础上,本研究旨在通过基因克隆和RNA干扰研究YLS的His2和His6(分别命名为EdeHis2和EdeHis6)在褐飞虱生长、发育和存活中的作用。同源搜索和系统发育分析表明EdeHis2和EdeHis6均源于YLS基因组,与绿僵菌His2和His6高度同源并在系统发育树中形成一个簇,但在褐飞虱基因组中无同源基因。基因的时空表达分析表明,His2和His6在褐飞虱的各个龄期均表达且呈现一定的波动性,在褐飞虱脂肪体中的表达量均显著高于头、足、体壁和中肠。此外,在褐飞虱头和翅的基因组中未能扩增到目的片段,而在腹部能扩增出目的片段。分别注射外源双链RNA(dsEdeHis2或dsEdeHis6)后的第2、4、6天,EdeHis2的表达量分别显著下调45%~60%,EdeHis6下调27%~55%。dsEdeHis2和dsEdeHis6分别使褐飞虱的死亡率提高了8.3%和9.2%,雌、雄虫的若虫期历期延长了0.43 d、0.33 d和0.65 d、0.36 d,但均未达到显著水平。另外,注射dsEdeHis6的褐飞虱雌雄成虫翅畸形率分别为11%和13%,显著高于对照组。综上所述,源于YLS的EdeHis2和EdeHis6参与褐飞虱组氨酸的合成,与褐飞虱的存活、发育和翅发育相关。
关键词:褐飞虱; 组氨酸; 类酵母共生菌; His2; His6; RNA干扰
褐飞虱[Nilaparvatalugens (Stål)]是主要的水稻害虫,专一性吸食水稻筛管液,严重威胁我国乃至许多亚洲国家的水稻生产[1, 2]。褐飞虱能够利用氨基酸不平衡的水稻汁液,一般认为其体内的共生菌具有营养补偿作用[3, 4]。人为减除褐飞虱类酵母共生菌(YLS)之后,褐飞虱表现出发育迟缓,存活率降低;对组氨酸、赖氨酸、亮氨酸、异亮氨酸、苏氨酸、缬氨酸和苯丙氨酸等必需氨基酸的需求量增加[3-5]。
组氨酸对多数昆虫来说是必需氨基酸,一般需要从食物中摄取,而褐飞虱等半翅目昆虫,其所吸食水稻汁液中的组氨酸等必需氨基酸却相对匮乏[6]。利用褐飞虱、YLS的基因组[7]和转录组[8]的研究结果表明,YLS基因组中含有参与编码组氨酸合成相关酶的所有基因,而在褐飞虱基因组中未发现。此外,也有研究表明,缺失组氨酸的人工饲料持续饲养褐飞虱后,能显著影响褐飞虱的羽化率[9]。由此推测,YLS合成的组氨酸对褐飞虱的生长发育具有重要的作用。然而,截至目前,尚未见关于褐飞虱中编码组氨酸合成相关酶的基因克隆和功能分析,特别是缺乏这些基因对褐飞虱发育作用的生物学证据。
为研究褐飞虱中YLS合成的组氨酸对褐飞虱生长的作用,本研究利用在YLS基因组中检索到的两个组氨酸合成关键基因分别能编码组氨醇磷酸磷酸化酶(His2)和磷酸核糖亚氨甲基-5-氨基咪唑-甲酰胺核糖核苷酸异构酶(His6)。对其进行基因克隆、序列特征鉴定和系统发育分析,利用荧光定量PCR技术检测其在褐飞虱中的时空表达,进一步通过RNA干扰技术研究该基因对褐飞虱生长发育及存活的影响,以期揭示YLS参与组氨酸合成及其对褐飞虱的重要性。
1材料与方法
1.1供试昆虫
实验所用虫源为中国水稻研究所培育的褐飞虱TN1种群,饲养环境温度(27±1)℃,相对湿度为(80±10)%,光周期16h光照/8h黑暗。
1.2总RNA的提取和cDNA第一链合成
1.2.1样品准备
取20头产卵盛期的褐飞虱雌成虫接到TN1水稻苗上,待产卵1d后剪下茎秆并在解剖镜下剥离卵块(50粒),放入1.5mL的eppendorf管中。采用类似的方法收集1~5龄若虫及初羽化雌雄成虫(1日龄)各40头。另外,选取80头初羽化雌成虫并在解剖镜下分离头(Head)、足(Leg)、体壁(Integument)、中肠(Midgut)、卵巢(Ovary)和脂肪体(Fatbody)置于1.5mLeppendorf管中。以上每处理设置3个生物学重复。所有样品用液氮速冻后置于-80℃冰箱保存备用。
1.2.2总RNA提取
褐飞虱总RNA的提取采用Qiagen公司生产的总RNA提取试剂盒(RNeasyMiniKit,Qiagen,Valencia,CA),提取方法参照试剂盒说明书。利用1.2%琼脂糖凝胶电泳和分光光度计(Nano-Drop1000spectrophotometer,ThermoFisherScientific,Rockford,USA)检测RNA的完整性和浓度,用于cDNA第一链的合成。
1.2.3cDNA第一链合成
cDNA第一链的合成采用Toyobo公司的反转录试剂盒(ReverTraAceqPCRRTkit,ToyoboCo.LTD,Osaka,Japan),操作参照试剂盒说明书进行,模板浓度为1μg,以OligdT为引物,合成cDNA第一链。
1.3基因检索、克隆与序列分析
分别检索褐飞虱、YLS(Fan等[10]将其命名为Entomomyces delphacidicolastr.NLU)和杀雄菌(Arsenophonus nilaparvatae)的基因组[7],仅在YLS基因组中找到两条单拷贝基因,分别能编码组氨醇磷酸磷酸化酶(His2)和磷酸核糖亚氨甲基-5-氨基咪唑-甲酰胺核糖核苷酸异构酶(His6)。利用PrimerPremier5.0设计引物(表1)对其进行RT-PCR克隆。以混合褐飞虱各龄期cDNA的模板进行PCR扩增,反应体系如下: 10×ExTaq 缓冲液 2.500μL,dNTPs(2.5mmol/L)2.000μL,正、反向引物(10mmol/L)各1.000μL,ExTaq酶0.125μL,cDNA模板1.000μL,双蒸水17.375μL。反应条件如下:94℃下预变性5min,94℃下变性30s,58℃下退火45s,72℃下延伸1min,38个循环,72℃下延伸10min。扩增产物用1.2%琼脂糖凝胶电泳检测,切胶回收后连于TOPO2.1载体(Invitrogen,Carlsbad,CA)上,转化至DH5α感受态细胞(Novagen,Darmstadt,Germany)中,随机挑选10个单克隆在ABI3730测序仪上进行双端测序。
通过EXPASY的ComputepI/Mw工具(http://web.expasy.org/compute_pi/)预测蛋白的分子量和等电点。利用ClustalW软件[11]进行多序列比对,MEGA6软件[12]绘制进化树,均采用默认参数。
1.4基因组DNA提取及PCR扩增
在解剖镜下分离出褐飞虱(羽化后1d)雌成虫的头(Head)、翅(Wing)和腹(Abdomen),置于1.5mL的eppendorf管中,参照王渭霞[13]等的方法提取基因组DNA。用分光光度计测量DNA浓度,设计PCR引物(跨内含子)进行PCR扩增,扩增体系及反应条件同1.3。每个处理分别设置3个技术重复和3个生物学重复。
1.5dsRNA的合成与生物测定
采用Ambion试剂盒(MEGAscriptT7HighYieldTranscriptionkit,Austin,USA)进行dsRNA体外转录。按照试剂盒说明书,取8μL纯化后的DNA模板,加入10×T7 缓冲液2μL,T7酶预混液2μL,ATP、GTP、CTP、UTP各2μL,37℃下温浴5h,随后置于72℃下处理5min。冷却至常温后加入21μL无核酸酶的水,5μL10×消化缓冲液,2μL脱氧核糖核酸酶和2μL核糖核酸酶,在37℃下温浴45min以去除剩余DNA模板及单链RNA,用醋酸钠和乙醇纯化dsRNA。在分光光度计下测量dsRNA的浓度,根据浓度加入不同体积的无核糖核酸酶的水,使dsRNA浓度为1.5μg/μL。
注射dsRNA参照本实验室的方法[14, 15]。选取褐飞虱3龄1日若虫(150头),通过显微注射仪将目的基因dsRNA或dsGFP注射至试虫体内(每头50nLdsRNA)。注射1d后,移除死亡的褐飞虱,随后逐日观察试虫的存活数、若虫发育情况及表型。此外,以150头不做任何注射的试虫为空白对照组(CK)。在处理后的第2、4、6天分别取10头试虫用于总RNA提取,用于实时荧光定量PCR(RT-qPCR)。每处理分别设置3个技术重复和生物学重复。
1.6RT-qPCR
RT-qPCR分析采用Toyobo公司生产的SYBRGreenMix试剂盒,参考操作说明,反应体系共20.0μL,包括6.4μLddH2O,10μLTaq酶预混液,10.0μmol/L上下游引物各0.8μL,2.0μL模板。基因的相对表达量采用2-△△CT法[16]计算,内参选用NlRPS11、NlRPS15[16,17]。
1.7数据分析
数据分析采用DPS数据处理系统[18],采用单因素方差分析(One-wayANOVA)或双因素方差分析(Two-wayANOVA),并采用Duncan新复极差法进行多重比较;百分数用反正弦平方根转换。文中所用数据为平均值±标准误(SE)。
2 结果与分析
2.1EdeHis2和EdeHis6基因的克隆与序列分析
本研究克隆得到了YLS基因组中两个参与组氨酸合成的基因,分别命名为EdeHis2和EdeHis6
表1RT-PCR、dsRNA合成和qPCR引物
Table1.PrimersusedforRT-PCR,dsRNAsynthesisandqPCR.

引物名称Primername正向引物(5'-3')Forwardsequence(5'-3')反向引物(5'-3')Reversesequence(5'-3')扩增长度Ampliconsize/bp基因克隆Geneclone EdeHis2CAAAAGCTTGACCCTCCCCTCTACAGGTCGCGCTCCTCATA378 EdeHis6GCTCTCCCTCGGAATGACGTGACCTAGGAGCTGCTTTTCC830基因组PCRGenomicPCR gEdeHis2GAGCCTTTAGAAACGCCAGTCCGAGTGAGAGTGCATAGTGA125 gEdeHis6AGACAGTGACGGACATGGAGACCAAGTCCTCGTCGATACC124 gNlRPS11CCGATCGTGTGGCGTTGAAGGGATGGCCGACATTCTTCCAGGTCC159双链RNA合成dsRNAsynthesis dsGFPT7-CCTGAAGTTCATCTGCACCACT7-TGATGCCGTTCTTCTGCTTGT355 dsEdeHis2T7-TTCGGTGTCCTCGTTCCTTCT7-CAGGTCGCGCTCCTCATATC272 dsEdeHis6T7-TCAGACCAACTACGTGTCGCT7-CGGAACAGTACGGCTCAAGT440定量PCRReal-timequantitativePCR qEdeHis2GAGCCTTTAGAAACGCCAGTCCGAGTGAGAGTGCATAGTGA125 qEdeHis6AGACAGTGACGGACATGGAGACCAAGTCCTCGTCGATACC124 qNlRPS11CCGATCGTGTGGCGTTGAAGGGATGGCCGACATTCTTCCAGGTCC159 qNlRPS15TAAAAATGGCAGACGAAGAGCCCAATTCCACGGTTGAAACGTCTGCG150
(GenBank登录号分别为:KU664333和KU664334)。EdeHis2开放阅读框(openreadingframe,ORF)全长858bp,编码285个氨基酸,预测蛋白分子量为32.1kD,等电点为5.91。EdeHis6的ORF全长813bp,编码270个氨基酸,预测蛋白分子量为29.6kD,等电点为6.09。
EdeHis2编码的推导蛋白质包含14个α螺旋和11个β折叠(图1-A),其中α1~α14和β1~β10组成组氨醇磷酸聚合酶结构域(Polymeraseandhistidinolphosphatase,PHP),这一结构是典型的组氨酸合成酶PHP的结构域。多序列比对表明EdeHis2与其同源基因的相似性达到48%~75%,其中与绿僵菌(Metarhizium guizhouense)His2的相似性最高(75%),其次是明尼苏达被毛孢(Hirsutella minnesotensis)、偏侧蛇虫草菌(Ophiocordyceps unilateralis)和葡萄穗霉菌(Stachybotrys chlorohalonata)(均为72%)。此外,基于邻接法(neighbor-joining,NJ)对10种真菌His2和EdeHis2系统进化进行了分析,发现EdeHis2与绿僵菌His2聚为一支,并与尖孢镰刀菌、白僵菌His2构成肉座菌目(图2-A)。
在EdeHis6方面,其推导的编码产物包含11个α螺旋和15个β折叠(图1-B),其中α1~α10和β1~β14组成His_biosynth结构域。多序列比对表明,EdeHis6与绿僵菌His6等序列相似性达57~87%,其中与绿僵菌、明尼苏达被毛孢(Hirsutellan minnesotensis)His6的相似性最高(均为87%)。系统进化树表明,整个系统进化树被分成了两个簇,一个由真菌His6组成,另一个由细菌His6组成,其中EdeHis6与绿僵菌His6在进化关系上最为接近(图2-B)。
2.2EdeHis2和EdeHis6的表达分析
单因素方差分析结果表明,EdeHis2在褐飞虱不同器官或组织中的表达差异显著(P=0.014),其中,脂肪体中表达量最高,显著高于中肠、头、足和表皮,与卵巢中的表达量无显著差异(图3-A)。EdeHis6的表达量在褐飞虱卵巢中最高,其次是脂肪体,均显著高于其在中肠、头、足和表皮的表达量(P=0)(图3-B)。EdeHis2和EdeHis6在褐飞虱的各个龄期均表达,EdeHis2的表达量在卵期最高,随着龄期逐渐降低,至成虫期最低(P=0)(图3-C);EdeHis6在一龄期表达最高,三龄和五龄次之,在卵和成虫中表达量最低(P=0)(图3-D)。基因组PCR结果表明,EdeHis2和EdeHis6分别能在腹部基因组DNA中扩增出目的条带,而在头基因组DNA和翅基因组DNA不能扩增出目的条带(图3-E)。
2.3dsRNA对EdeHis2和EdeHis6表达的影响
二因素方差分析结果表明,注射dsEdeHis2与对照相比,EdeHis2的表达量有显著差异(P=0),注射后2、4、6d之间没有显著差异(P=0.16)。荧光定量PCR检测结果表明,与空白对照组相比,注射dsEdeHis2后的第2、4、6天,EdeHis2表达量分别显著下调45%、60%和49%,而注射dsGFP的试虫与空白对照组无差异(图4-A)。与之类似,二因素方差分析结果表明注射dsEdeHis6后EdeHis6的mRNA水平较CK相比存在显著差异(P=0),但注射后的第2、4、6天对EdeHis6无显著影响(P=0.90)。注射dsEdeHis6后的第4、6天,EdeHis6的mRNA水平较CK相比显著下调47%、55%(图4-B),而EdeHis6表达量在注射后的第2天仅下调27%,与CK没有显著差异。与CK相比,注射dsGFP后第2、4、6天试虫的EdeHis6表达量均没有显著差异。
2.4dsRNA对褐飞虱生长、发育的影响
单因素方差分析结果表明,注射dsEdeHis2或dsEdeHis6对褐飞虱的死亡率无显著影响(P=0.065),对雌雄虫的若虫发育历期均无显著影响(P=0.76,P=0.18)。分别注射dsEdeHis2或dsEdeHis6之后,褐飞虱的死亡率分别为(10.00±2.00)%和(10.89±2.89)%,与空白对照(1.67±1.67)%相比显著高了8.3%(图5-A)和9.2%(图5-B)。另外,注射dsGFP褐飞虱的死亡率(4.57±2.52)%与CK相比无显著差异。分别注射dsEdeHis2或dsEdeHis6之后,褐飞虱雄虫的若虫发育历期分别为(7.98±0.10)d和(8.21±0.32)d,与空白对照组(7.56±0.15)d相比,延长0.43d和0.65d;雌虫的若虫发育历期分别为(8.66±0.05)d和(8.69±0.56)d,与对照(8.33±0.24)d相比,延长0.33d和0.36d,但均未达到显著差异水平(图5-C~D)。
注射dsEdeHis6的褐飞虱羽化至成虫时,雌虫的翅发育不完全(11%),雄虫的翅呈现弯折(13%),雌雄虫的旧表皮均不能完全褪下(图6-A~B)。注射dsGFP褐飞虱雌雄虫的翅发育正常(图6-C~D)。此外,注射EdeHis2褐飞虱成虫未发现明显异常表型。
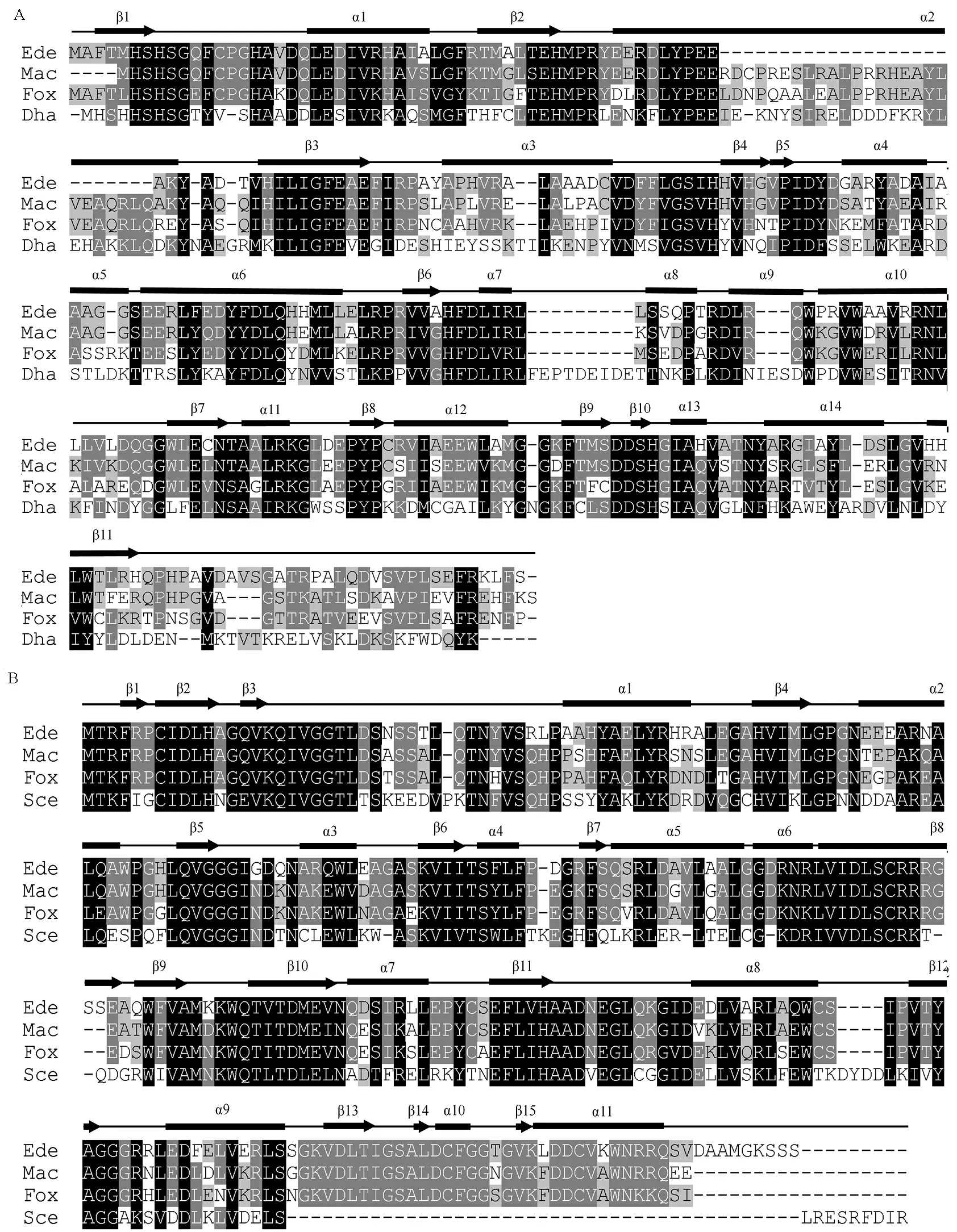
His2同源物种的简写及orthoDB登录号:Ede, 褐飞虱类酵母共生菌;Mac, 绿僵菌,E9DRU4;Fox, 尖孢镰刀菌,J9N254;Dha, 德巴利氏酵母,B5RTB4。His6同源物种的简写及登录号:Ede, 褐飞虱类酵母共生菌;Mac, 绿僵菌,E9DSK3;Fox, 尖孢镰刀菌,F9G3J5;Sce, 酿酒酵母,A6ZVP7。
OriginspeciesofHis2andtheirorthoDBaccessionnumbers:Ede, Entomomyces delphacidicola;Mac, Metarhizium acridumCQMa102,E9DRU4;Fox, Fusarium oxysporum,J9N254;Dha, Debaryomyce shansenii,B5RTB4.OriginspeciesofHis6andtheirorthoDBaccessionnumbers:Ede,Entomomyces delphacidicola;Mac, Metarhizium acridum,E9DSK3;Fox, Fusarium oxysporum,F9G3J5;Sce, Saccharomyces cerevisiae,A6ZVP7.
图1His2(A)和His6(B)氨基酸序列的多序列比对
Fig. 1.AminoacidsequencealignmentsofHis2 (A)andHis6(B)withtheirhomologues.
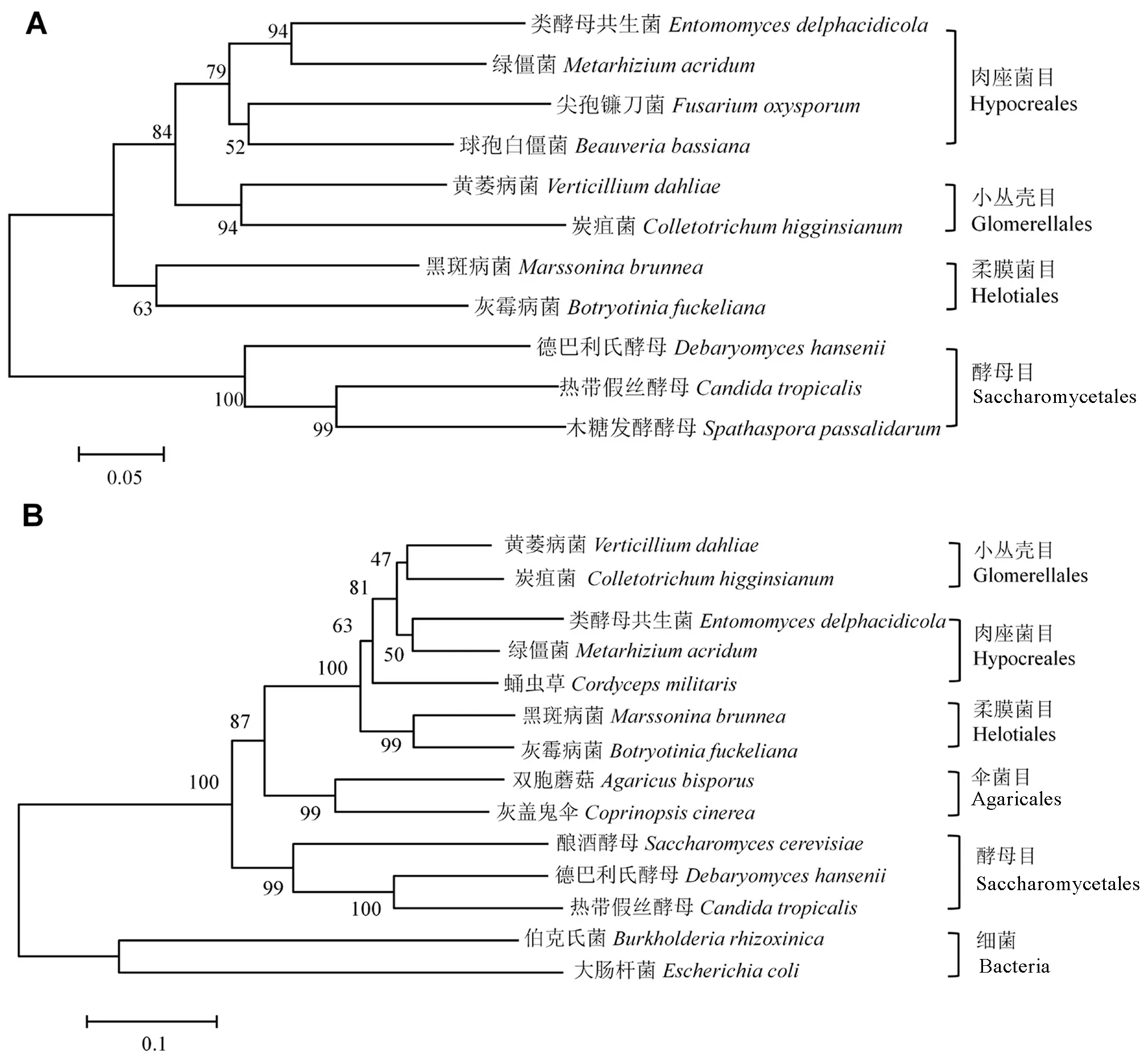
His2来源及orthoDB登录号: 绿僵菌,E9DRU4; 尖孢镰刀菌,J9N254; 德巴利氏酵母,B5RTB4; 热带假丝酵母,C5MGD3; 木糖发酵酵母,G3AV99; 黄萎病菌,G2XJV7; 炭疽菌,H1W439; 白僵菌,J4VYI6; 黑斑病菌,K1WGV8; 灰霉病菌,M7U2Q0。His6来源及登录号: 绿僵菌,E9DSK3; 蛹虫草,G3J516; 酿酒酵母,A6ZVP7; 热带假丝酵母,C5MDV0; 德巴利氏酵母,Q6BUV9; 木糖发酵酵母,G3AV99; 黄萎病菌,G2X7U1; 炭疽菌,H1V6J4; 白僵菌,J4VYI6; 黑斑病菌,K1WJH6; 灰霉病菌,M7U4B5; 双胞蘑菇,K5XY72; 灰盖鬼伞,A8NXH0; 伯克氏菌,CBW74635.1; 大肠杆菌,CCQ29416.2。
OriginofHis2andtheiraccessionnumbers: Metarhizium acridumCQMa,E9DRU4; Fusarium oxysporum,J9N254; Debaryomyces hansenii,B5RTB4; Candida tropicalisMYA-3404,C5MGD3; Spathaspora passalidarum,G3AV99; Verticillium dahlia,G2XJV7; Colletotrichum higginsianum,H1W439;Beauveria bassianaARSEF2860,J4VYI6; Marssonina brunneaf.sp. ,K1WGV8; Botryotinia fuckeliana,M7U2Q0.OriginofHis6andtheiraccessionnumbers: Metarhizium acridumCQMa,E9DSK3;Cordyceps militarisCM01,G3J516; Saccharomyces cerevisiaeYJM789,A6ZVP7; Candida tropicalisMYA-3404,C5MDV0; Debaryomyces hansenii,Q6BUV9; Spathaspora passalidarum,G3AV99;Verticillium dahlia,G2X7U1; Colletotrichum higginsianum,H1V6J4; Beauveria bassianaARSEF2860,J4VYI6; Marssonina brunneaf.sp.,K1WJH6; Botryotinia fuckeliana,M7U4B5; Agaricus bisporus,K5XY72; Coprinopsis cinerea,A8NXH0; Burkholderiar hizoxinica,CBW74635.1; Escherichia coli,CCQ29416.2.
图2His2(A)和His6(B)氨基酸序列的系统进化分析(邻接法)
Fig. 2.PhylogeneticanalysisofEdeHis2(A)andEdeHis6 (B)bytheneighbor-joiningmethod.
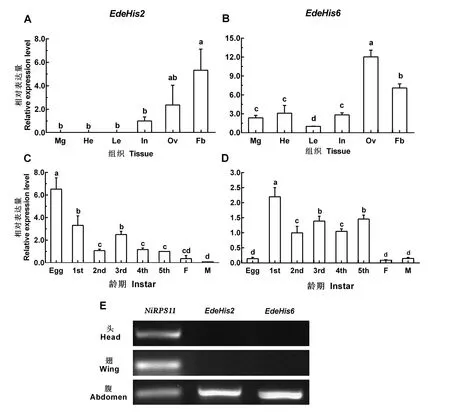
Mg-中肠;He-头;Le-足;In-体壁;Ov-卵巢;Fb-脂肪体;Egg-卵;1st、2nd、3rd、4th、5th:1-5龄褐飞虱;F-雌成虫;M-雄成虫. 图中数据(平均值±标准误;n=2)上不同小写字母者表示差异达5%显著水平(Duncan新复极差法)。下同。
Mg,Midgut;He,Head;Le,Leg;In,Integument;Ov,Ovary;Fb,Fatbody. 1stto5thindicate1st- 5thinstarnymph,respectively;F,Femaleadult;M,Maleadult.Values(mean±SE, n=2)markedbydifferentlowercaselettersaresignificantlydifferentatthe5%levelbytheDuncan’stest.Thesameasinfiguresbelow.
图3EdeHis2和EdeHis6的组织(A、B、E)和龄期(C、D)表达
Fig.3.Tissuespecific(A,BandE)andtemporal(CandD)expressionpatternsofEdeHis2andEdeHis6.
3讨论
2014年公布的褐飞虱基因组、YLS基因组和杀雄菌(Ars)基因组[7]揭示了褐飞虱体内氨基酸合成的代谢途径及其基因的来源。本研究克隆的EdeHis2、EdeHis6均源于YLS基因组,而在褐飞虱基因组、Ars基因组中均未检索到编码His2和His6的基因或同源序列。组氨酸是褐飞虱的必需氨基酸,其参与合成的基因在褐飞虱基因组及Ars基因组中均缺失。然而,YLS中EdeHis2、EdeHis6分别与真菌的His2或His6高度同源,且具有典型α螺旋、β折叠和结构域,这表明EdeHis2和EdeHis6可能作为功能蛋白参与YLS合成组氨酸的过程,而褐飞虱则不参与这一过程。此外,系统进化树分析结果表明,EdeHis2、EdeHis6均与肉座菌目中绿僵菌同源基因在进化关系上接近,这和Xue等基于基因组的研究结果一致[7]。与此同时,不同组织或器官表达谱结果表明EdeHis2、EdeHis6在褐飞虱脂肪体中的表达量显著高于中肠、头、足和表皮的表达量。前人研究表明YLS主要存在于褐飞虱腹部脂肪体中,不存在于头、足、表皮等组织[19, 20],本研究结果与之较为类似。另外,基因组PCR结果表明,EdeHis2和EdeHis6能够在腹部基因组中扩增出目的条带,而在不存在YLS的头和翅中未扩增出相应的条带。由此可见,本研究克隆的EdeHis2、EdeHis6源于YLS。
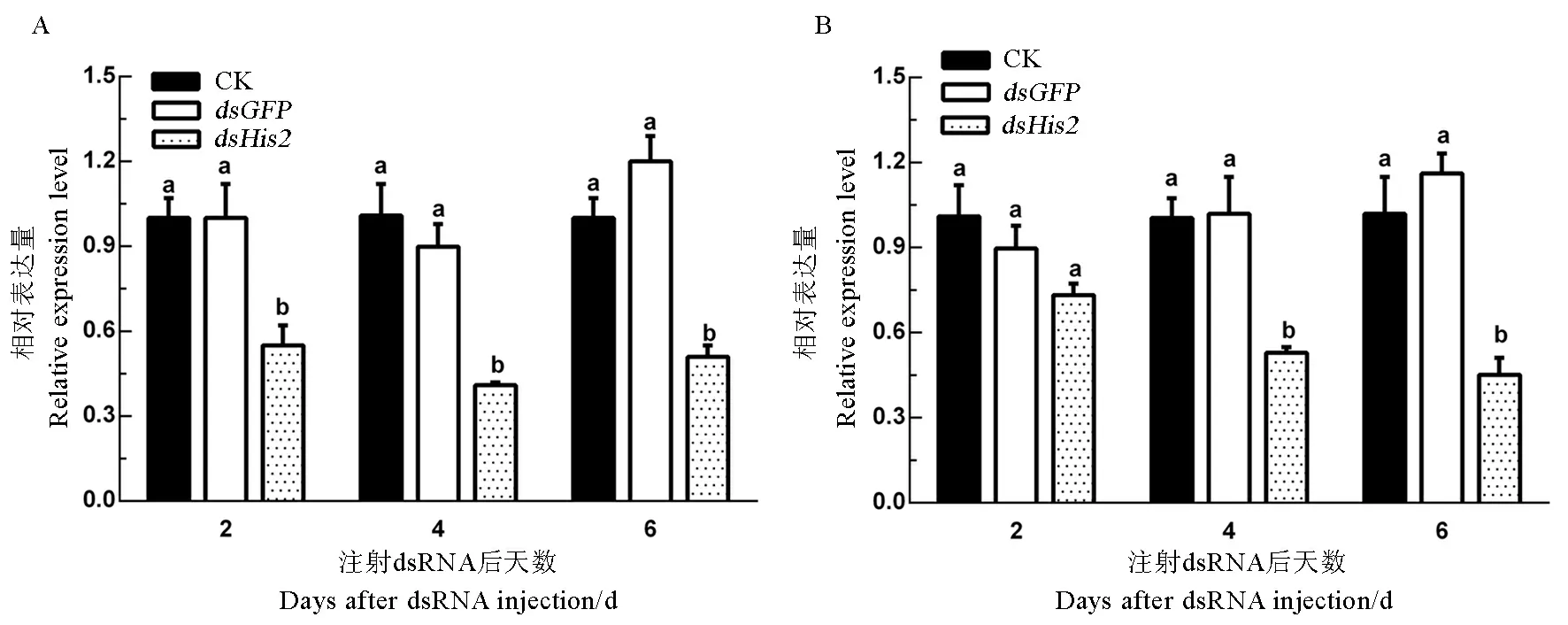
图4注射dsRNA后第2、4、6天EdeHis2(A)和EdeHis6(B)的表达量
Fig. 4.EffectsofinjectionofdsRNAonthemRNAlevelofEdeHis2 (A)andEdeHis6 (B).
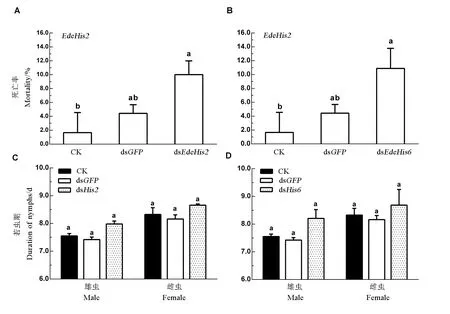
图5注射dsRNA对褐飞虱死亡率(A、B)和若虫发育历期(C、D)的影响
Fig. 5.Mortality(A,B)andnymphduration(C,D)ofN. lugensasaffectedbydsRNAinjection.
YLS向褐飞虱提供氨基酸营养,当人为移除褐飞虱体内的YLS后,褐飞虱表现为生长缓慢[4, 21, 22]。分别注射dsEdeHis2或dsEdeHis6后,褐飞虱的死亡率显著升高、若虫发育历期略微延长,这表明干扰EdeHis2或EdeHis6对褐飞虱生长、发育、存活有一定的负面作用。这与Wan等[6]的研究结果类似,干扰源于YLS的Lys合成基因SDH,试虫的死亡率和发育历期均显著升高。此外,本研究干扰EdeHis6后的褐飞虱翅表现为畸形,这一表型变化与干扰Lys代谢相关基因引发的畸形较为相似[6, 23]。由此可见,EdeHis6在褐飞虱的发育和翅型发育中具有重要的作用。这一调节过程可能是氨基酸不同代谢途径交叉调节的现象导致[24]。组氨酸代谢与嘌呤、叶酸、色氨酸和甲硫氨酸等有着重要的关系[25-27]。EdeHis6基因编码的酶ProFAR-I催化合成的PRFAR可以用于合成嘌呤、硫胺素焦磷酸和甲硫氨酸[28]。EdeHis6基因的沉默会导致PRFAR不能合成,而干扰EdeHis2不会影响PRFAR的合成,但会导致PRFAR的积累。注射EdeHis6的褐飞虱出现了翅畸形,而注射EdeHis2的褐飞虱未出现,表明PRFAR合成的缺失可能导致嘌呤、硫胺素焦磷酸和甲硫氨酸等的合成减少,进而影响翅的发育。

图6注射dsEdeHis6后羽化的雄雌虫(A、B)和注射dsGFP后羽化的雌雄虫(C、D)
Fig. 6.EffectsofdsEdeHis6 (A,B)anddsGFP(C,D)onthewingsdevelopmentofN.lugens.
综上所述,来源于YLS的EdeHis2和EdeHis6参与褐飞虱组氨酸的合成,与褐飞虱的死亡率、发育历期和翅型的发育相关。
参考文献:
[1]WangY,ChenJ,ZhuYC,etal.Susceptibilitytoneonicotinoidsandriskofresistancedevelopmentinthebrownplanthopper, Nilaparvata lugens (Stål) (Homoptera:Delphacidae). Pest Manag Sci, 2008, 64(12): 1278-1284.
[2]BottrellDG,SchoenlyKG.Resurrectingtheghostofgreenrevolutionspast:Thebrownplanthopperasarecurringthreattohigh-yieldingriceproductionintropicalAsia. J Asia-Pacif Entomol, 2012, 15(1): 122-140.
[3]傅强,张志涛,胡萃,等. 高温处理后褐飞虱体内共生酵母菌和氨基酸需求的变化. 昆虫学报,2001,44(4):534-540.
FuQ,ZhangZT,HuC,etal.Theeffectsofhightemperatureonbothyeast-likesymbiontsandaminoacidrequirementsofNilaparvata lugens. Acta Entomol Sin, 2001, 44(4): 534-540.(inChinesewithEnglishabstract)
[4]ChenYH,BernalCC,TanJ,etal.Planthopper"adaptation"toresistantricevarieties:Changesinaminoacidcompositionovertime. J Insect Physiol, 2011, 57(10): 1375-1384.
[5]王国超,傅强,赖凤香,等. 褐飞虱体内类酵母共生菌与氨基酸营养的关系. 昆虫学报,2005,48(4): 483-490.
WangGC,FuQ,LaiFX,etal.Relationshipbetweenyeast-likesymbiotesandaminoacidrequirementsinthericebrownplanthopper, Nilaparvata lugens (Stål) (Homoptera:Delphacidae).Acta Entomol Sin, 2005, 48(4): 483-490.(inChinesewithEnglishabstract)
[6]WanPJ,YangL,YuanSY,etal.RNAinterference-aidedknockdownofaputativesaccharopinedehydrogenaseleadstoabnormalecdysisinthebrownplanthopper, Nilaparvata lugens (Stål) (Hemiptera:Delphacidae). Bull Entomol Res, 2015, 105(4): 390-398.
[7]XueJ,ZhouX,ZhangC-X,etal.Genomesofthericepestbrownplanthopperanditsendosymbiontsrevealcomplexcomplementarycontributionsforhostadaptation. Genom Biol, 2014, 15(521): 1-19.
[8]WanPJ,YangL,WangWX,etal.Constructingthemajorbiosynthesispathwaysforaminoacidsinthebrownplanthopper, Nilaparvata lugensStål(Hemiptera:Delphacidae),basedonthetranscriptomedata. Insect Mol Biol, 2014, 23(2): 152-164.
[9]傅强. 褐飞虱全纯人工饲料继代饲养技术及营养生理学研究.杭州:浙江大学,1999.
FuQ.Continousrearingandnutitionalphysiologyofthebrownplanthopper, Nilaparvata lugens (Stål)onchemicallydefineddiets.Hangzhou:ZhejiangUniversity, 1999.(inChinesewithEnglishabstract)
[10]FanHW,NodaH,XieHQ,etal.GenomicanalysisofanAscomycetefungusfromthericeplanthopperrevealshowitadaptstoanendosymbioticlifestyle. Genome Biol Evol, 2015, 7(9): 13-34.
[11]LarkinMA,BlackshieldsG,BrownNP,etal.ClustalWandClustalXversion2.0. Bioinformatics, 2007, 23(21): 2947-2958.
[12]TamuraK,PetersonD,PetersonN,etal.MEGA5:Molecularevolutionarygeneticsanalysisusingmaximumlikelihood,evolutionarydistance,andmaximumparsimonymethods. Mol Biol Evol, 2011, 28(10): 2731-2739.
[13]王渭霞,罗举,赖凤香,等. 水稻褐飞虱内生共生细菌Arsenophonus的鉴定和系统分析. 昆虫学报,2010,53(6): 647-654.
WangWX,LuoJ,LaiFX,etal.IdentificationandphylogeneticanalysisofsymbioticbacteriaArsenophonusfromthericebrownplanthopper, Nilaparvata lugens (Stål) (Homoptera:Delphacidae).Acta Entomol Sin, 2010, 53(6): 647-654.(inChinesewithEnglishabstract)
[14]WangWX,LiKL,ChenY,etal.IdentificationandfunctionanalysisofenolasegeneNlEno1fromNilaparvata lugens (Stål) (Hemiptera:Delphacidae). J Insect Sci, 2015, 15(1): 1-9.
[15]LiKL,WanPJ,WangWX,etal.RaninvolvedinthedevelopmentandreproductionisapotentialtargetforRNA-interference-basedpestmanagementinNilaparvata lugens. PLoS ONE, 2015, 10(11):e0142142.
[16]LivakKJ,SchmittgenTD.Analysisofrelativegeneexpressiondatausingreal-timequantitativePCRandthe2(T)(-DeltaDeltaC)method. Methods, 2001, 25(4): 402-408.
[17]YuanM,LuY,ZhuX,etal.Selectionandevaluationofpotentialreferencegenesforgeneexpressionanalysisinthebrownplanthopper, Nilaparvata lugens (Hemiptera:Delphacidae)usingreverse-transcriptionquantitativePCR. PLoS ONE, 2014, 9(1):e86503.
[18]TangQY,ZhangCX.DataProcessingSystem(DPS)softwarewithexperimentaldesign,statisticalanalysisanddataminingdevelopedforuseinentomologicalresearch. Insect Sci, 2013, 20(2): 254-260.
[19]ChengDJ,HouRF.Histologicalobservationsontransovarialtransmissionofayeast-likesymbioteinNilaparvata lugensStål(Homoptera,Delphacidae). Tissue Cell, 2001, 33(3): 273-279.
[20]DongS,PangK,BaiX,etal.Identificationoftwospeciesofyeast-likesymbiotesinthebrownplanthopper, Nilaparvata lugens. Curr Microbiol, 2011, 62(4): 1133-1138.
[21]FuQ,ZhangZ,HuC,etal.Theeffectsofhightemperatureonbothyeast-likesymbiontsandaminoacidrequirementsofNilaparvata lugens. Acta Entomol Sin, 2001, 44(4): 534-540.
[22]WilkinsonTL,IshikawaH.OnthefunctionalsignificanceofsymbioticmicroorganismsintheHomoptera:AcomparativestudyofAcyrthosiphon pisumandNilaparvata lugens. Physiol Entomol, 2001, 26(1): 86-93.
[23]WanPJ,YuanSY,TangYH,etal.PathwaysofaminoaciddegradationinNilaparvata lugens (Stål)withspecialreferencetolysine-ketoglutaratereductase/saccharopinedehydrogenase(LKR/SDH). PLoS ONE, 2015, 10(5):e0127789.
[24]HinnebuschAG.MechanismsofgeneregulationinthegeneralcontrolofaminoacidbiosynthesisinSaccharomyces cerevisiae. Microbiol Revi, 1988, 52(2): 248-273.
[25]KoslowskyS,RieglerH,BergmullerE,etal.HigherbiomassaccumulationbyincreasingphosphoribosylpyrophosphatesynthetaseactivityinArabidopsis thalianaandNicotiana tabacum. Plant Biotechnol J, 2008, 6(3): 281-294.
[26]ZhangY,MorarM,EalickSE.Structuralbiologyofthepurinebiosyntheticpathway. Cellul Mol Life Sci, 2008, 65(23): 3699-3724.
[27]IngleRA.Histidinebiosynthesis.TheArabidopsisbook/AmericanSocietyofPlantBiologists, 2011, 1-15.
[28]StepanskyA,LeustekT.Histidinebiosynthesisinplants. Amino Acids, 2006, 30(2): 127-142.
收稿日期:2016-02-22; 修改稿收到日期: 2016-03-24。
基金项目:国家自然科学基金资助项目(31371939); 国家科技支撑计划资助项目(2012BAD19B03)。
中图分类号:S435.112+.3; S476.12
文献标识码:A
文章编号:1001-7216(2016)04-0406-11
RolesofTwoGenesInvolvedinHistidineBiosyntheticPathwayinYeast-LikeSymbiontinDevelopmentofNilaparvata lugens (Stål)
TANGYao-hua1,2,WANPin-jun2,HAOPei-ying1,FUQiang2, *,YUXiao-ping1, *
(1ZhejiangProvincalKeyLaboratoryofBiometrologyandInspectionandQuarantine,CollegeofLifeScience,ChinaJiliangUniversity,Hangzhou310018,China;2StateKeyLaboratoryofRiceBiology,ChinaRiceResearchInstitute,Hangzhou310006,China;*Correspondingauthor,E-mail:fuqiang@caas.cn;yxp@cjlu.edu.cn)
Abstract:Nilaparvata lugens is a serious phloem-feeding pest of rice in China. Based on genome and transcriptome data of N.lugens and yeast-like symbiont (YLS, also named Entomomyces delphacidicola str. NLU), the major biosynthesis pathways for amino acids in N.lugens were constructed. In this study, we cloned two genes, EdeHis2 and EdeHis6, which catalyze critical steps in histidine biosynthesis pathway, and revealed the negative effects of double-stranded RNA (dsRNA) on the growth, development and survival rate of N.lugens. Homology searches and phylogenetic analysis showed that EdeHis2 and EdeHis6 origin from YLS genome, share high similarities with that of Metarhizium acridum and formed a clad in the phylogenetic tree, whereas no His2- or His6-like genes was found in N.lugens genome. Temporal expression profiles of EdeHis2 and EdeHis6 showed that both genes were ubiquitously but unevenly expressed among the different life stages, and the spatial expression pattern showed they have higher expression levels in the fat body rather than head, leg, integument and midgut. Furthermore, no target products were amplified in head and wing genomic DNA of N. lugens, rather than that in abdomen genomic DNA. At two, four and six days after dsEdeHis2 or dsEdeHis6 injection, the mRNA abundance of target genes was decreased by 45%-60% (EdeHis2)or 27%-55% (EdeHis6), comparing with blank control.Down-regulation of EdeHis2 or dsEdeHis6 slightly increased the mortality by 8.3% or 9.2%, and delayed the nymphal duration of male and female by 0.43 and 0.65 day and 0.33 and 0.36 day, respectively. Moreover, both male (11%) and female adult (13%) showed wing deformation after injection of dsEdeHis6, higher than that in the blank control. In conclusion, EdeHis2 and EdeHis6 that origin from YLS were involved in histidine biosynthesis pathway, contributed to the survivor, development and wing formation of N. lugens.
Key words:Nilaparvata lugens; yeast-like symbiont; histidine; His2; His6; RNAi
