凤丹牡丹ANS(PoANS)基因克隆、特性及表达1)
2016-08-06李果李厚华张延龙辛转霞魏新翠唐豆豆
李果 李厚华 张延龙 辛转霞 魏新翠 唐豆豆
(西北农林科技大学,杨凌,712100) (济南市学大教育培训学校) (西北农林科技大学)
凤丹牡丹ANS(PoANS)基因克隆、特性及表达1)
李果李厚华张延龙辛转霞魏新翠唐豆豆
(西北农林科技大学,杨凌,712100)(济南市学大教育培训学校)(西北农林科技大学)
摘要通过RT-PCR技术,从凤丹牡丹花瓣中克隆出编码ANS基因的cDNA序列,并对其进行生物信息学分析。对不同开花阶段的凤丹花瓣进行实时荧光定量及总花青素质量分数测定,构建原核表达载体PET-28a-ANS,并转化大肠杆菌BL21感受态,经IPTG诱导进行体外表达。结果表明:该序列编码区域共1 065 bp,编码354个氨基酸,其蛋白相对分子质量为40 475.5 ,亚细胞定位为细胞质的可能性最大;表达分析显示,ANS基因表达水平与不同开花时期的总花青素质量分数及花色相关,原核表达得到相对分子质量约为44 000的ANS-His-tag融合蛋白,与预测蛋白大小相一致。
关键词凤丹牡丹;ANS基因;表达分析;原核表达
凤丹牡丹(Paeonia ostii‘feng dan’)是中国传统的药用植物和新兴的油料作物[1],具有很高的经济价值,同时也是现代观赏牡丹的主要原始亲本之一[2-3]。凤丹花色主要为白色或浅粉色,单瓣花,在栽培演化进程中受其他种或品种群的影响较小[2]。
决定植物组织颜色的花青素(anthocyanidins)是类黄酮代谢途径中的重要产物[4]。花青素合成酶基因(Anthocyanidin synthase)是花青素合成途径中的一个重要基因,它能够催化无色花青素合成有色花青素[5],花青素能够使植物呈现蓝、紫、粉、红等颜色[4]。ANS基因最初通过转座子标签技术从玉米中克隆出来[6],已有研究发现,ANS基因在绿色紫苏中不表达,在红色紫苏中表达较高,证明了其与花青素的积累有密切关系[7],同时对蛇莓着色的研究也表明,ANS基因表达量与颜色积累有关[8]。目前,ANS基因的研究多集中于模式植物以及果蔬类植物,对于观赏类特别是在开花过程中花瓣颜色变化程度较高的植物中研究较少,且多集中在表达分析层面,对于基因本身以及编码蛋白的研究很少[9]。
凤丹直接从野生原种驯化而来,本身花色较为单一,但其开花过程中,花色变化较为明显,证明其具有很高的观赏潜力[2]。本研究在凤丹花瓣中克隆出ANS基因及其序列,并进行了生物信息学分析,通过实时荧光定量并结合花青素积累情况分析ANS基因表达与花色变化的关系,以期为凤丹花色形成机理及表达调控提供理论基础。通过原核表达获得ANS基因编码蛋白,为体外验证花青素合成酶功能奠定基础。为后期进行凤丹花色调控,提高观赏性奠定理论基础。
1材料与方法
植物材料取自西北农林科技大学野生牡丹资源圃保存的凤丹不同开花时期,参考前人对花期的划分[10],将‘凤丹’牡丹分为花蕾期、未展期、初展期、盛花期和末花期五个时期(图1)的花瓣,分别采集多株花型、花色较为接近的花瓣,用于总花青素质量分数测定,用液氮速冻后储存于-80 ℃备用。
植物RNA提取试剂盒购自OMEGA公司,Taq酶、高保真酶、限制性内切酶、连接酶、克隆载体DNA Marker购自TaKaRa公司,琼脂糖胶回收试剂盒购自于北京康为世纪生物科技有限公司,质粒提取试剂盒购自BIOMIGA公司,无缝克隆试剂盒购自近岸蛋白公司,氨苄青霉素(Ampicillin)、卡那霉素(Kanamycin)、X-gal、IPTG等试剂均为进口分析纯。引物合成、测序等均为北京奥科公司完成。大肠杆菌(E.coli)TOP10、BL21、原核表达载体pET-28a均由本实验室保存。文中未注明的其他试剂均为国产或进口分析纯。

图1 不同发育时期的凤丹花
RNA提取与cDNA合成:RNA提取采用OMEGA公司的植物总RNA提取试剂盒按照说明进行,提取的RNA用1%琼脂糖凝胶电泳检测,并利用紫外分光光度计进行质量浓度及质量检测。
以每个时期总RNA为模板,使用PrimeScriptTMRT reagent Kit with gDNA Eraser,先消除基因组DNA后,反转录合成cDNA第一链,置于-80 ℃备用。
基因克隆:根据NCBI网站上已经报道的其他植物的ANS基因序列,设计特异性引物(表1),以反转录合成的cDNA为模板,扩增凤丹ANS基因cDNA序列。扩增条件为94 ℃预变性10 min;94 ℃变性30 S,58 ℃退火30 S,72 ℃延伸50 S(共30个循环);72 ℃延伸10 min,4 ℃保存反应产物。PCR产物使用1%琼脂糖凝胶电泳检测,纯化回收目的条带,与PGEM-T载体进行连接,连接产物采用热激法转化大肠杆菌TOP-10感受态细胞,经蓝白斑筛选,以及菌落PCR验证后,将阳性产物于Amp终质量浓度为100 μg·L-1的LB液体培养基过夜培养后,送北京奥科鼎盛公司测序。将验证正确的重组质粒命名为pGEM-T-ANS。
生物信息学分析:使用NCBI BLAST进行氨基酸序列同源分析,利用ExPASyProtParam Tool(http://web.expasy.org/protparam/),HNN SECONDARY STRUCTURE PREDICTION METHOD,Swiss-Model Workplace(http://swissmodel.expasy.org/)和TMGMM 2.0等软件进行蛋白质各级结构进行预测。编码蛋白翻译后的修饰位点检测在KinasePhos(http://kinasephos.mbc.nctu.edu.tw/)中完成。
原核表达分析:首先将PET-28a载体利用EcoRI,HindIII双酶切进行线性化处理,再根据载体切口两端的15 bp同源序列设计特异性引物nrANSf和nrANSr(表1),以重组pGEM-T-ANS载体质粒为模板,克隆两端带有PET-28a同源序列的PoANS基因。凝胶电泳分别回收线性化载体以及PCR产物,在NovoRec®重组酶的作用下进行连接。连接产物首先采用热激法转化大肠杆菌TOP-10感受态细胞,经kana抗性筛选,挑取单一菌落过夜摇菌后提取质粒进行PCR验证及双酶切验证,将阳性重组子于kana终质量浓度为50 μg·L-1的LB液体培养基过夜培养后,送北京奥科鼎盛公司测序。将测序正确的重组子命名为PET-28a-ANS。
重组载体PET-28a-ANS验证正确后转入大肠杆菌BL21感受态细胞,加入kana终质量浓度为50 μg·L-1的LB培养基过夜培养。用LB液体培养基按V(过夜培养菌液)∶V(液体LB培养基)=1∶50稀释过夜培养的菌液,培养一段时间后至OD600为0.6左右,加入终浓度为1 mmol·L-1的IPTG溶液,分别取空载体菌液,重组载体0、2、4、6、8 h的诱导菌液,加入上样缓冲液,煮沸10 min后离心,吸取上清液进行SDS-PAGE电泳。经考马斯亮蓝染色,脱色后,照相保存[11]。
不同开花时期的表达分析:选择PoANS基因靠近5’端的一段序列设计实时定量引物rtANSf和rtANSr(表1),所得扩增片段长度应为234 bp,选用牡丹β-微管蛋白基因(β-Tubulin,登录号:EF608942)作为内参基因[12],设计特异性引物Tubf和Tubr(表1),对应的扩增片段长度为182 bp。使用IQTM5多重实时荧光定量PCR仪(Bio-Rad),以五个不同花期cDNA为模板,参照TaKaRa公司的SYBR@PremixExTaqTMⅡ说明书,进行实时荧光定量分析,反应程序如下,95 ℃预变性1 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s(共40个循环);95 ℃ 1 min,65 ℃ 1 min,95 ℃ 20 s(步进0.5 ℃·s-1)。每个样品做4次重复反应,以牡丹β-Tubulin基因为内参,使用2-ΔΔCT法进行数据分析[13]。
不同开花时期花瓣总花青素质量分数的测定:将不同时期花瓣液氮速冻后,取0.3 g样品用1%甲醇盐酸4 ℃提取24 h,12 000 r·min-1离心15 min,取上清液定容至10 mL。将样品分别用pH值1.0的25 mmol·L-1的氯化钾溶液和pH值4.5的400 mmol·L-1的醋酸钠溶液按一定的稀释倍数稀释后利用紫外分光光度计测定530 nm和700 nm处的吸光值,同一时期的花瓣每组做3次重复。然后按照以下公式计算(总花青素质量分数以矢车菊素-3-葡萄糖苷的质量分数计算)[14]:
A=A1-A2;
总花青素质量分数=(A×MW×DF×V×1 000)/(m×ε)。式中:A为吸光度;A1为氯化钾稀释样品液530 nm和700 nm处的吸光值之差;A2为醋酸钠稀释样品液530 nm和700 nm处的吸光值之差;MW为相对分子质量;DF为稀释倍数;V是溶液定容体积;m为称取的花瓣质量;ε为矢车菊素-3-葡萄糖苷的摩尔消光系数[15])。

表1 所用引物序列
2结果与分析
2.1凤丹花青素合成酶(PoANS)基因的克隆与生物信息学分析
以凤丹花cDNA为模板,使用上述引物以及高保真酶扩增出一段大约1 000 bp的片段(图2)。将该片段连接PGEM-T载体,转化大肠杆菌后,经测序获得该片段完整序列,命名为PoANS。
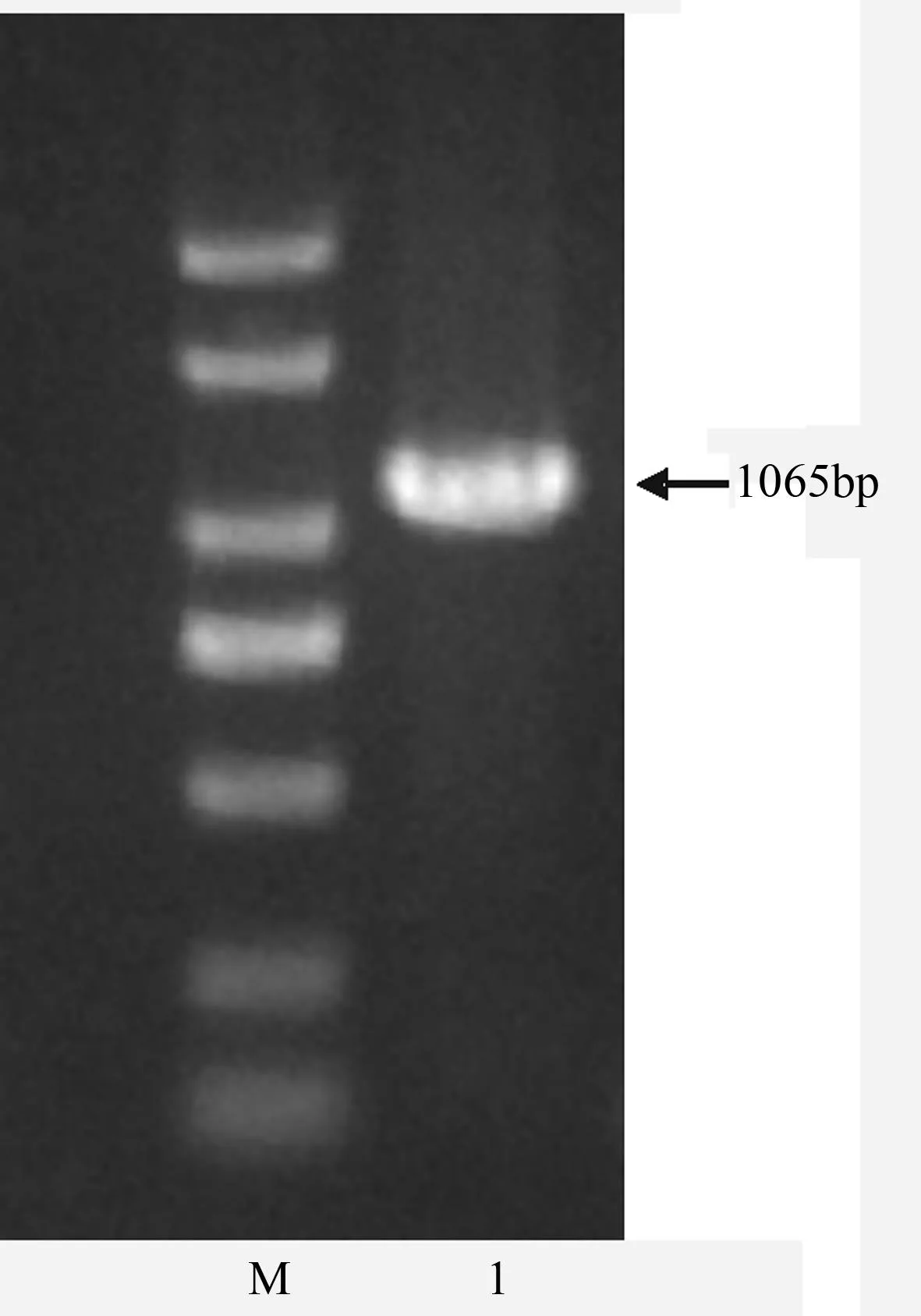
M为2 000 bpMarker;1为凤丹ANS基因序列。
序列分析结果显示,‘凤丹’牡丹ANS基因编码区共1 065 bp,编码354个氨基酸(图3),其蛋白相对分子质量为40 475.5,氨基酸种类及所占比例见表2。

图3 PoANS基因cDNA编码区基因序列及其所对应的氨基酸序列
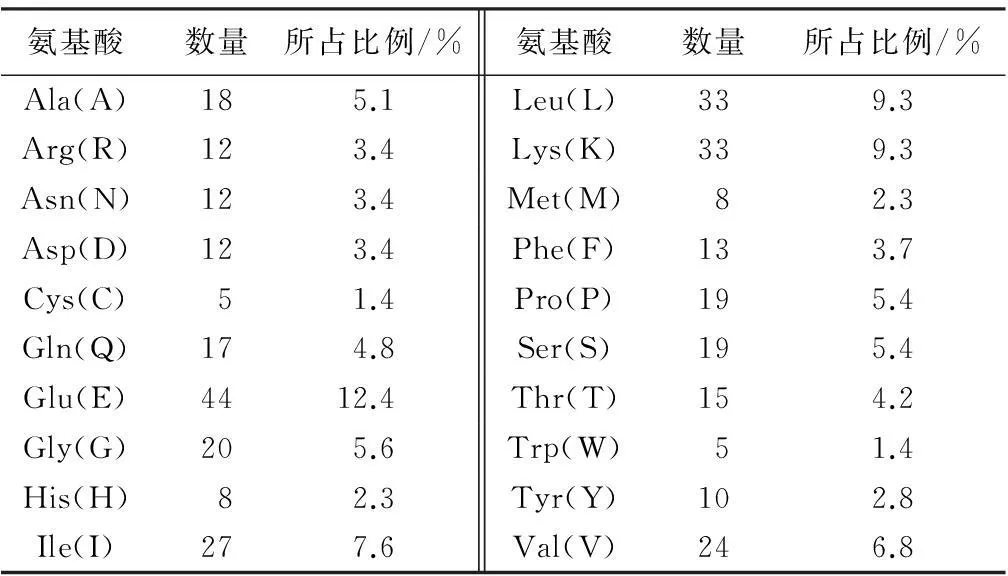
氨基酸数量所占比例/%氨基酸数量所占比例/%Ala(A)185.1Leu(L)339.3Arg(R)123.4Lys(K)339.3Asn(N)123.4Met(M)82.3Asp(D)123.4Phe(F)133.7Cys(C)51.4Pro(P)195.4Gln(Q)174.8Ser(S)195.4Glu(E)4412.4Thr(T)154.2Gly(G)205.6Trp(W)51.4His(H)82.3Tyr(Y)102.8Ile(I)277.6Val(V)246.8
PoANS蛋白质二级结构预测结果表明,其在175~195和230~257有两个跨膜区;二级结构由α螺旋(占41.53%)、随机卷曲(占27.68%)、延伸链(占19.21%)和β旋转(占11.58%)组成。对PoANS蛋白质的理化性质分析结果表明,PoANS蛋白理论等电点为5.42,半衰期较长,为30 h,不稳定系数为53.08,属于比较稳定的蛋白质,脂溶系数为90.85,总平均疏水性为-0.405。NCBI CDD软件在线预测结果表明,PoANS蛋白中含有PLN03178功能域(图4),Swiss-Model Workplace在线预测蛋白质三级结构(图5)。

图4 PoANS蛋白保守结构域预测结果

图5 PoANS蛋白三级结构预测结果
PoANS蛋白在细胞质的可能性最大,可信度为4.723(CELLO v.2.5预测结果);PoANS蛋白可能含有2个丝氨酸磷酸化位点,分别位于第33个氨基酸和302个氨基酸;2个络氨酸磷化位点,分别位于第115个氨基酸和167个氨基酸;1个苏氨酸磷酸化位点,位于第261个氨基酸(图6)。
2.2PoANS基因的原核表达
从抗性筛选呈阳性的大肠杆菌TOP10中提取质粒,进行PCR验证(图7)和双酶切验证(图8),使用BamHI和NotI双酶切,因PoANS序列中含有EcoRI和HindIII酶切位点。结果显示,PCR扩增得到1 000 bp左右条带,双酶切得到5 000 bp和1 000 bp左右的两条带,证明原核表达重组载体构建成功。测序结果表明,无碱基位点发生突变,可以进行诱导表达。
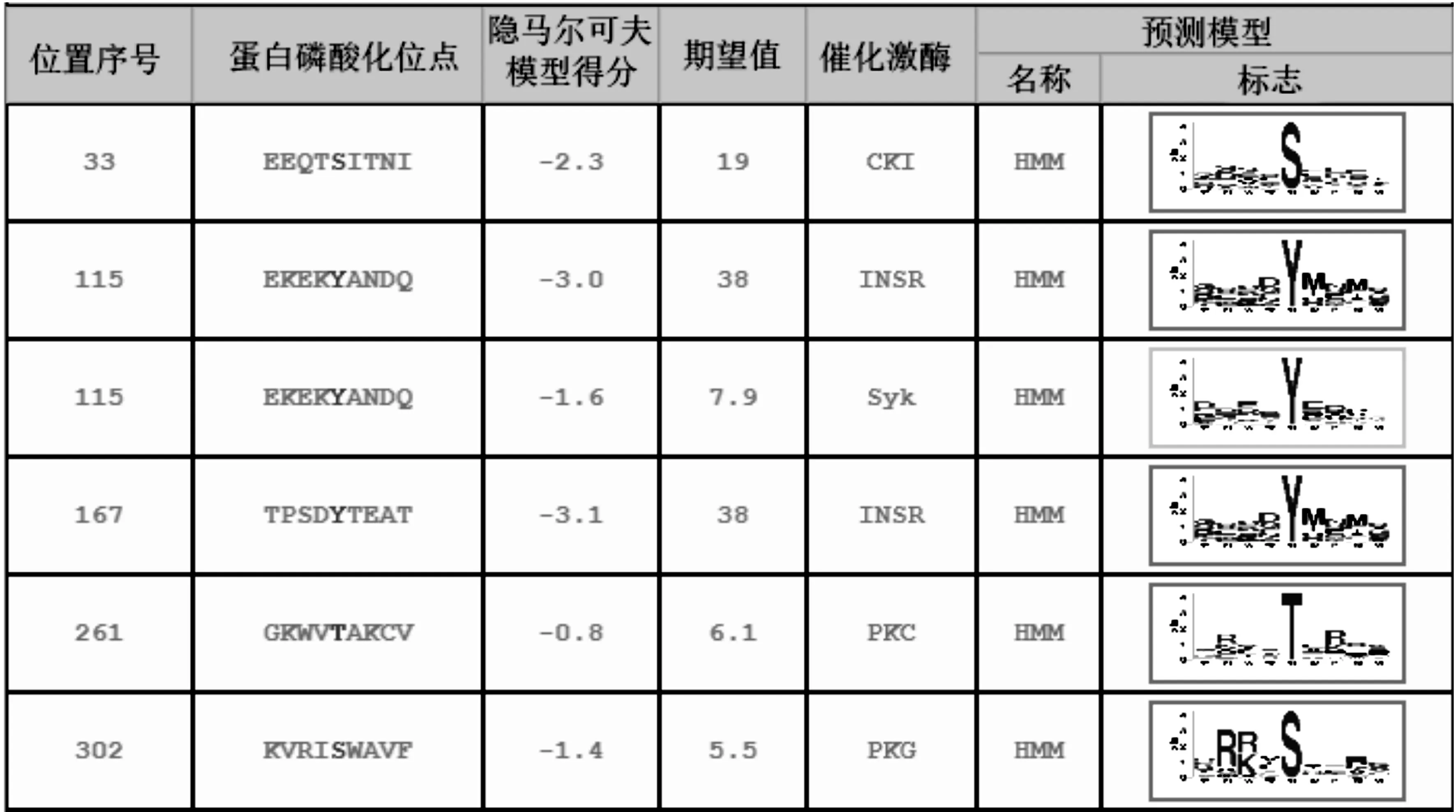
图6 PoANS蛋白磷酸化位点预测结果

图7 重组质粒PCR验证
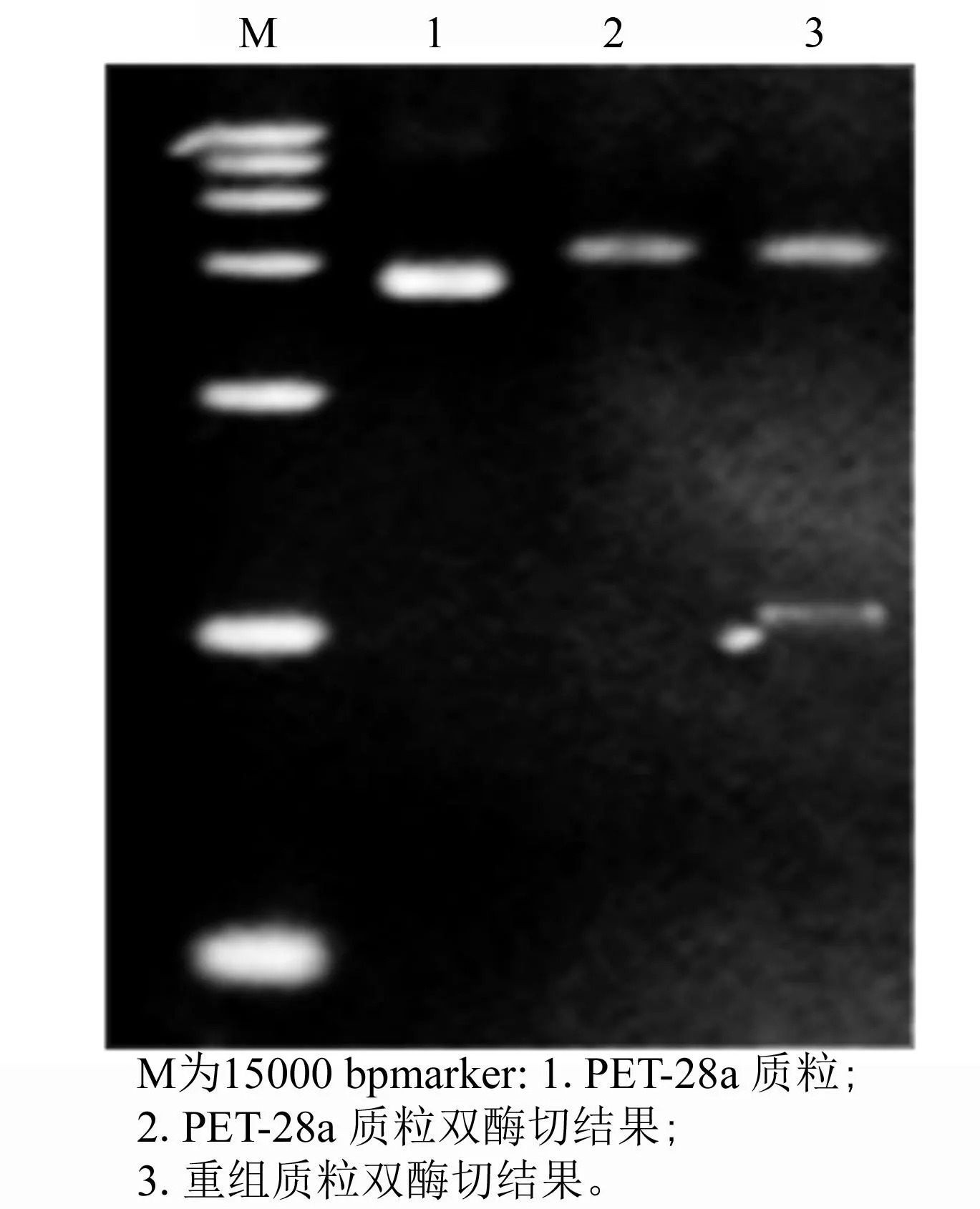
图8 重组质粒双酶切验证
SDS-PAGE电泳结果显示,空载体和0 h左右的菌液表达均不明显,而随着诱导时间的增加蛋白表达逐渐增加,可以看到,在相对分子质量为43 000左右的位置有明显异于空载体的蛋白表达。PoANS蛋白相对分子质量为40 000左右,原蛋白加上His-tag标签大概为43 000,由此表明,有融合蛋白成功表达(图9)。
2.3PoANS基因表达水平分析
ANS基因的表达受到植物的整体代谢控制,同时花青素的积累也受植物体内各种因素如底物质量浓度、其他类黄酮途径合成酶,以及外界环境等影响。为了研究PoANS基因在凤丹开花过程中对于花色的影响,故对5个不同开花时期的花瓣进行荧光定量PCR分析。以5个不同时期的花瓣总RNA反转录合成的cDNA为模板,以β-Tubulin为内参基因得到PoANS基因在‘凤丹’牡丹5个时期花瓣的相对表达量(表3,图10),PoANS基因的表达水平呈现出先下降再上升,最后再下降的趋势,在盛花期达到最高,相对表达量为14.68,花色也最鲜艳,而到末花期,PoANS基因相对表达水平最低,而花瓣也几乎为纯白色。
经过紫外分光光度计测量及后期数据处理分析,可发现花瓣中总花青素的质量分数呈先下降后上升的趋势,在盛花期达到最高,而在末花期,未检测到花青素(表3)。
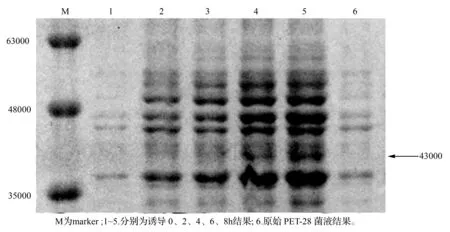
图9 SDS-PAGE电泳结果

发育时期PoANS基因相对表达量花青素积累量/mg·kg-1花蕾期8.21±0.23137.33±5.67未展期4.18±0.1866.65±6.12初展期5.37±0.2083.92±5.49盛花期14.68±0.25152.45±5.16末花期1.00±0.130

图10 PoANS基因定量产物电泳图
3结论与讨论
本研究利用RT-PCR技术成功从凤丹牡丹的花瓣中克隆出花青素合成酶基因(PoANS),并对该基因进行了生物信息学分析、原核表达分析以及表达分析。结果表明,PoANS开放阅读框共有1 065个碱基,共编码354个氨基酸,相对分子质量为40 475.5,蛋白消光系数为42 650,其不稳定性系数为53.08,说明较稳定,在细胞质的可能性最大。通过构建原核表达载体,经过IPTG诱导后,在不同时间收集菌液,经过SDS-PAGE电泳发现,带有His-tag标签的融合蛋白大小预期基本一致,且融合蛋白随着时间的增长,表达质量浓度有明显的增加,但到8 h左右,增加的幅度已经不是很明显,故诱导的最佳时间为6 h左右。原核表达的成功为下一步进行体外酶促反应,进一步验证PoANS基因的功能奠定了基础。
通过对花不同发育时期PoANS基因的表达分析发现,在未开放的花蕾中PoANS表达量较高,而此时的花瓣颜色也呈浅粉色,花青素质量分数也比较高,随着花逐渐开放,基因表达量先下降再上升,在盛花期达到最高,而此时的花青素积累量也达到最高值,花色也最为鲜艳,证明了ANS基因的不同表达水平对花色的形成具有重要影响,而Zhao et al.[16]的研究中也发现,ANS的高表达能够促进花青素的积累,结合本试验的结果,均证实了ANS基因与花青素质量分数的直接联系。本试验在末花期中并没有检测到花青素的存在,而此时也有PoANS少量表达,Zhao et al.[17]的研究中也有类似发现,可能原因是花青素作为底物被下游合成途径的酶所消耗,导致未检测到花青素成分。文中研究结果为下一步利用基因工程手段提高ANS表达水平,以及通过RNAi及病毒干扰技术干扰花青素合成途径下游基因、调控花青素合成与积累、培育兼具经济价值与观赏价值的凤丹新品种奠定了一定的理论基础。
参考文献
[1]李育材.中国油用牡丹工程的战略思考[J].中国工程科学,2014,10(16):58-63.
[2]李嘉珏.中国牡丹起源的研究[J].北京林业大学学报,1998,20(2):22-26.
[3]洪涛,张家勋,李嘉珏,等.中国野生牡丹研究(一):芍药属牡丹组新分类[J].植物研究,1992,12(3):223-234.
[4]SCHAEFER H M, WILKINSON D M.Red leaves, insects and coevolution: a red herring?[J].Trends in Ecology & Evolution,2004,19(12):616-618.
[5]TURNBULL J J, SOBEY W J, APLIN R T, et al.Are anthocyanidins the immediate products of anthocyanidin synthase?[J].Chemical Communications,2000,32(24):2473-2474.
[6]ADRIANE M, SUSANNE H, WILLIAM M, et al .The En/Spm transposable element of Zea mays contains splice sites at the termini generating a novel intron from a dSpm element in the A2 gene[J].Embo Journal,1990,9(10):3051-3057.
[7]GONG Z Z, YAMAZAKI M, SUGIYAMA M, et al.Cloning and molecular analysis of structural genes involved in anthocyanin biosynthesis and expressed in a forma-specific manner in Perilla frutescens[J].Plant Molecular Biology,1997,35(6):915-927.
[8]DEBES M A, ARIAS M E, GRELLET-BOURNONVILLE C F, et al.White fruited Duchesnea indica (Rosaceae) is impaired in ans gene expression[J].American Journal of Botany,2011,98(12):2077-2083.
[9]张云洁,潘怡辰, 王汝茜,等.植物花青素生物合成途径相关基因的研究进展[J].安徽农业科学,2014,42(34):12014-12016,12080.
[10]ZHANG C, WANG W N, WANG Y J, et al.Anthocyanin biosynthesis and accumulation in developing flowers of tree peony (Paeonia suffruticosa) ‘Luoyang Hong’[J].Postharvest Biology and Technology,2014,97:11-22.
[11]王亚杰.水母雪莲黄酮合成酶FNSⅡ基因克隆和功能分析[D].杨凌:西北农林科技大学,2014.
[12]王彦杰,董丽, 张超,等.牡丹实时定量PCR分析中内参基因的选择[J].农业生物技术学报,2012,20(5):521-528.
[13]王亚杰,李厚华,付婉艺,等.水母雪莲红色细胞系类黄酮含量和相关基因表达[J].生物工程学报, 2014,30(8):1225-1234.
[14]LI Q, WANG J, SUN H Y, et al.Flower color patterning in pansy (Viola×wittrockiana Gams.) is caused by the differential expression of three genes from the anthocyanin pathway in acyanic and cyanic flower areas[J].Plant Physiol Biochem,2014,84:134-141.
[15]杨兆艳.pH示差法测定桑椹红色素中花青素含量的研究[J].食品科技,2007,32(4):201-203.
[16]ZHAO D Q, TAO J, HAN C X, et al.Flower color diversity revealed by differential expression of flavonoid biosynthetic genes and flavonoid accumulation in herbaceous peony (Paeonia lactiflora Pall.)[J].Molecular Biology Reports,2012,39(12):11263-11275.
[17]ZHAO D Q, TANG W H, HAO Z J, et al.Identification of flavonoids and expression of flavonoid biosynthetic genes in two coloured tree peony flowers[J].Biochem Biophys Res Commun,2015,459(3):450-456.
第一作者简介:李果,男,1991年10月生,西北农林科技大学风景园林艺术学院,硕士研究生。E-mail:547569415@qq.com。 通信作者:李厚华,西北农林科技大学风景园林艺术学院,副教授。E-mail:lihouhua73@163.com。
收稿日期:2016年1月24日。
分类号Q785;Q786
Isolation, Characterization and Expression Analysis of Anthocyanidin Synthase (ANS) Gene in Paeonia ostii‘feng dan’//
Li Guo, Li Houhua, Zhang Yanlong, Xin Zhuanxia(Northwest A&F University, Yangling 712100, P.R.China); Wei Xincui(Jinan Xueda School); Tang Doudou(Northwest A&F University)//
Journal of Northeast Forestry University,2016,44(7):64-69.
We isolated a completed CDS sequence encoding ANS gene, designated as PoANS, from Paeonia ostii ‘feng dan’ by RT-PCR.The opening reading frame (ORF) of PoANS is 1 065 bp that encoded 354 amino acid, relative molecular mass is 40 475.5, the subcellular localization most likely be cytoplasm.By real-time quantitative polymerase chain reaction analysis and ultraviolet photometric analysis, the transcript level of PoANS was related with the total anthocyanidins.Prokaryotic expression confirmed that its protein molecular weight agreed with expectations.
KeywordsPaeonia ostii‘feng dan’; Anthocyanin synthase; Expression analysis; Prokaryotic expression
1)林业公益性行业科研重大专项(201404701)、国家自然科学基金项目(31570697)。
责任编辑:任俐。
