额尔古纳自然保护区6种林型土壤真菌群落结构的多样性
2016-08-06赵文静赵宝军崔岱宗赵敏
赵文静 赵宝军 崔岱宗 赵敏
(东北林业大学,哈尔滨,150040) (中国内蒙古森工集团) (东北林业大学)
额尔古纳自然保护区6种林型土壤真菌群落结构的多样性
赵文静赵宝军崔岱宗赵敏
(东北林业大学,哈尔滨,150040)(中国内蒙古森工集团)(东北林业大学)
摘要采用PCR-DGGE技术,研究了额尔古纳国家级自然保护区黑桦(Betula davurica)林、山杨(Populus davidiana)林、落叶松(Larix gmelinii)林、白桦(Betula platyphylla)林、樟子松(Pinus sylvestris)落叶松混交林、落叶松白桦混交林土壤中真菌群落结构的异同。结果表明:土壤真菌种类与森林类型密切相关,每个林型还存在着其特有的菌种。各林型土壤真菌多样性高低顺序依次为落叶松白桦混交林、落叶松林、樟子松落叶松混交林、山杨林、白桦林和黑桦林,同时各个林型土壤中优势真菌种类是不相同的;林型相似,土壤中真菌种类越相近,相反,林型差异越大,土壤中真菌种类差异就越大。
关键词额尔古纳国家级自然保护区;土壤真菌;18S rDNA;PCR-DGGE;真菌多样性
额尔古纳国家级自然保护区具有独特的林型和土壤类型,植物类型比较丰富和复杂。虽然森林土壤中蕴育着丰富的未开发土壤真菌,但是土壤中可培养的微生物在自然界中只占0.1%~10.0%[1-2],很难客观、全面地认识土壤中微生物的真实情况。目前,利用分子生物学技术研究生物生态学已经比较广泛,这为认识环境中的微生物开辟了新途径。在土壤微生物多样性的诸多研究方法中,PCR-DGGE技术因稳定性高、操作简便等优点已经被越来越多地运用于土壤微生物多样性的研究中。本研究通过PCR-DGGE技术对额尔古纳国家级自然保护区黑桦(Betula davurica)林、山杨(Populus davidiana)林、落叶松(Larix gmelinii)林、白桦(Betula platyphylla)林、樟子松(Pinus sylvestris)落叶松混交林、落叶松白桦混交林中土壤真菌的多样性进行分析,对6种林型土壤中真菌数量、种类进行了相似性分析,明确了土壤真菌分布规律、数量及种类,为额尔古纳自然保护区土壤真菌的开发利用提供理论依据。
1材料与方法
1.1材料
2012年8月20日选择额尔古纳国家级自然保护区黑桦林、山杨林、樟子松落叶松混交林、落叶松白桦混交林、落叶松林、白桦林作为研究样地。用5点法采集各林型土壤样品,并将其装入无菌聚乙烯袋中,编号后带回实验室,4 ℃保存[3]。
1.2土壤总DNA的提取和纯化
土壤总DNA的提取方法参照文献[4]、[5]。土壤总DNA的纯化方法参照文献[6]。提取后的DNA粗提液用琼脂糖凝胶回收试剂盒进行纯化,纯化后的DNA用于PCR扩增。
1.3PCR扩增
以纯化后的土壤总DNA为模板,选择真菌18 S rDNA通用引物NS1与GCFung[7]对土壤总DNA进行扩增。引物序列为:NS1-5′-GTA GTC ATA TGC TTG TCT C-3′;GCFung-5′-CGC CCG CCG CGC CCC GCG CCC GGC CCG CCG CCC CCG CCC CAT TCC CCG TTA CCC GTT G-3′。PCR产物片段大小和扩增效果用1%的琼脂糖凝胶进行检测。
PCR反应体系:引物各4 μL(2.5 μmol·L-1),10×PCR Buffer(含Mg2+)2 μL,Taq DNA聚合酶(2.5 U·μL-1)0.4 μL,dNTP(10 mmol·L-1)0.4 μL,DNA模板0.5 μL,去离子水8.7 μL,总体积20 μL。
PCR反应条件:94 ℃预变性5 min,94 ℃变性1 min,54 ℃退火1 min,72 ℃延伸1 min,35个循环,最后72 ℃延伸10 min,4 ℃保存。
1.4DGGE凝胶的制备及染色
选用15%~45%(100%的变性剂是7 mol·L-1尿素与40%去离子甲酰胺的混合物)的变性梯度凝胶。因为银染法有易于操作和灵敏度高(为EB染色200倍)等优点[8-9],所以选择银染法对DGGE凝胶进行染色。
1.4.1DGGE凝胶的制备
(1)将玻璃板倒上洗涤剂,自来水冲洗干净后,用去离子水润洗,自然风干,将部件正确组装,在组装过程中旋拧螺母时用力要均一,避免玻璃板受力不均而碎裂。
(2)调节灌胶器齿轮到起始位置,用体积调节螺母设定所需要的凝胶体积。
(3)分别吸取高低变性胶16 mL于2个50 mL的三角瓶中,向每个三角瓶中分别加入70 μL 10% APS和15 μL TEMED,摇动三角瓶,充分混匀。
(4)分别吸取15 mL高浓度和低浓度胶溶液到标有“高”与“低”的注射器中,排空注射器中的空气后,将注射器固定在制胶器上。
(5)将“Y”型接头两端连接到2个注射器上,“Y”型接头另一端与一个19号平头针头相连,将平头针头固定在玻璃板中间后,准备灌胶。
(6)缓慢转动灌胶器齿轮,慢慢的将变性胶灌到玻璃板中,待胶溶液到玻璃板顶部时,灌胶停止,将梳子插入凝胶中。
(7)清洗灌胶系统,避免堵塞。
(8)凝胶室温放置2 h后即可凝固,待凝胶聚合后小心拔出梳子,凝胶制备完成。
1.4.2DGGE凝胶的染色
(1)电泳完成后,将玻璃板小心从胶板盒上取下,待玻璃板完全冷却后,小心将玻璃板分开。
(2)在装有去离子水的塑料托盘中,小心将凝胶从玻璃板上取下,去离子水洗涤2次,每次洗涤后尽量控尽凝胶上的去离子水。
(3)在装有凝胶的塑料托盘中倒入固定液,使凝胶完全浸没在固定液中,摇床振荡15 min。
(4)倒掉固定液,凝胶用去离子水洗涤3次,每次洗涤时间为3 min。将染色液倒入装有凝胶的托盘中,置于摇床上避光振荡20 min。
(5)用去离子水洗涤染色后的凝胶1次,转移凝胶至黑暗处,在装有凝胶的托盘中倒入在冰上遇冷过的显影液,摇床振荡约6 min,待凝胶上出现清晰的条带时,倒入终止液。
(6)将凝胶取出后,去离子水洗涤凝胶3次,在凝胶成像装置中观察DGGE电泳效果并照相。
1.5DGGE条带分析
(1)DGGE中特异条带的回收。
(2)切胶回收后的PCR反应采用不带GC夹的引物NS1-5′-GTA GTC ATA TGC TTG TCT C-3′;Fung-5′-ATT CCC CGT TAC CCG TTG-3′进行扩增。PCR产物片段大小和扩增效果用1%的琼脂糖凝胶进行检测。
(3)连接转化。将胶回收后的PCR产物与pMD-18T质粒连接,转化到Escherichia coli Trans109感受态细胞中,检测转化结果后,由华大基因科技有限公司完成测序工作。
1.6DGGE电泳
运用Quantity One 4.5.2软件对DGGE图谱上的条带进行定量分析,将各个泳道与条带的分析结果进行统计学分析,得到优势度、相似性指数、丰度及香浓系数等参数。
2结果与分析
2.1森林土壤样品基因组DNA提取
采用冻融法破碎真菌细胞壁,SDS裂解法破碎真菌菌体提取土壤中真菌总DNA,并将DNA粗提液用小量琼脂糖凝胶DNA胶回收试剂盒进行纯化,从而获得了高质量的目标基因组DNA片段,如图1所示,目的片段明亮,无弥散,可以进行后续试验。
2.2不同林地土壤总DNA的PCR扩增
以提取到的土壤总DNA为模板,用真菌18 S rDNA通用引物NS1与GCfung进行PCR扩增,电泳检测结果如图2所示。由图可知扩增出的片段大小在350 bp左右,证实为目的条带。目的片段明亮、无弥散、有少许非特异性扩增片段,但不影响后续的DGGE凝胶电泳分析。
2.3不同林型土壤真菌的菌群结构
将不同林型土壤样品的PCR产物取样13 μL,经DGGE电泳后,结果如图3所示。从图可以看出,每个林型土壤样品经过DGGE电泳后都分离出数目不同的条带,且每个条带的迁移率和强度各不相同。各个林型土壤样品中既有共同的条带,也有特异性条带,这说明不同林型土壤样品中既存在着相同的真菌,也存在着各自特有的真菌种类。条带Z1在山杨林土壤中颜色最深,说明此种真菌是山杨林土壤中的优势菌种;条带Z2和Z4在每个林型土壤中都存在,说明每个林型土壤中都存在这2种真菌菌种;条带Z3只存在于杨树林、白桦林和黑桦林土壤中,说明此种菌种只分布于3种林型土壤中;条带Z5和Z6在落叶松林土壤中颜色最深,说明这2种真菌是落叶松林土壤中的优势菌种。
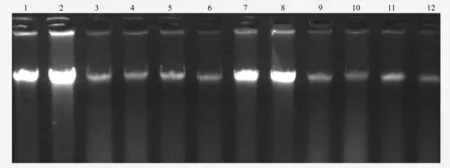
1、2.山杨林;3、4白桦林;5、6黑桦林;7、8、落叶松林;9、10落叶松白桦混交林;11、12樟子松落叶松混交林。
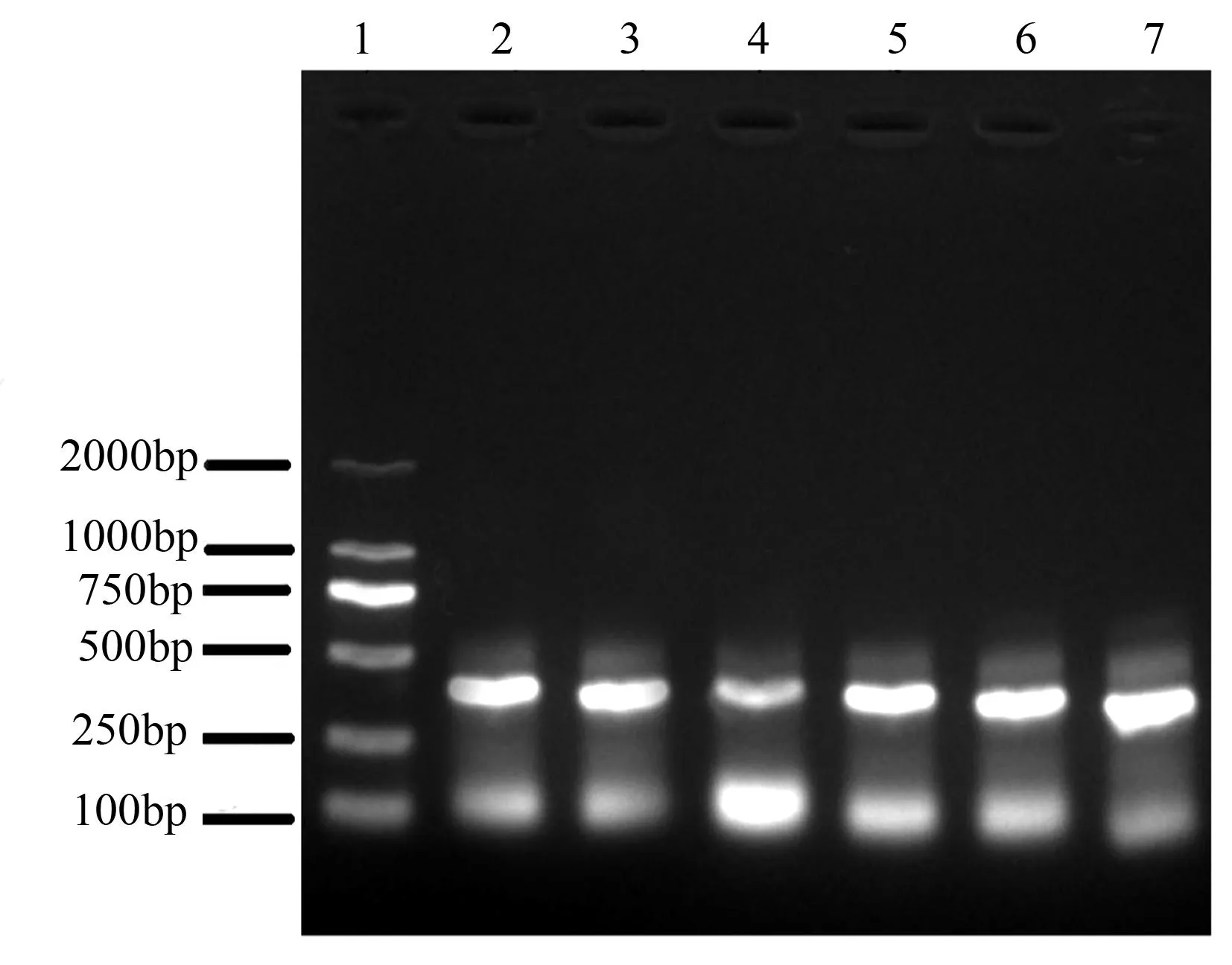
1.Marker;2.山杨林;3.白桦林;4.黑桦林;5.落叶松林;6.落叶松白桦混交林;7.樟子松落叶松混交林。
图2不同林型土壤总18 S rDNA PCR扩增结果

1.山杨林;2.白桦林;3.黑桦林;4.落叶松林;5.落叶松白桦混交林;6.樟子松落叶松混交林。
图3不同林型土壤样品的PCR产物的DGGE图
运用Quantity One 4.5.2软件对DGGE图谱进行分析,得到相似性指数,如图4和表1所示。由图可知,白桦林土壤中真菌菌群的构成与其他林型土壤中真菌菌群的构成具有明显的相关性,与落叶松白桦混交林土壤中真菌组成相似性最高,其次为黑桦、山杨和落叶松林土壤,与樟子松落叶松林土壤中真菌组成的差异性最大。
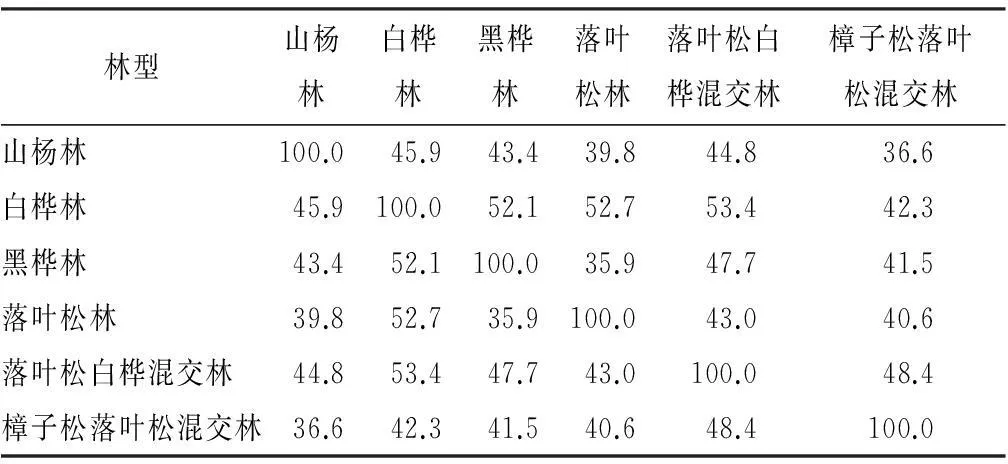
表1 6种林型土壤真菌的相似性聚类分析结果
黑桦林、山杨林、樟子松落叶松混交林、落叶松白桦混交林、落叶松林、白桦林土壤中真菌菌群的香农系数分别为:3.08、3.17、3.20、3.45、3.24、3.13。落叶松白桦混交林中真菌菌群的香农系数最大,表明该林型土壤中真菌多样性最高;黑桦林土壤中真菌菌群的香农系数最小,表明该林型土壤中真菌多样性最低。
2.4DGGE条带的序列
为了对不同林型土壤真菌多样性和分布有更进一步的了解,将DGGE图谱中较为优势的条带切胶回收。对回收的条带进行PCR扩增测序后,通过GenBank数据库进行比对,将其初步鉴定到属,并根据本研究中的菌种特点,在NCBI中索取到菌种Genebank登录号,确定菌种种名并供他人检索如表2。

图4 不同林型土壤真菌的系统发育分析

条带编号GeneBank登录号GenBank数据库中的最相近序列最相近序列的GenBank登录号相似度/%Z1KF776496VolvopluteusearleivoucherMA22816HM56227099Z2KF776497GeomycesdestructansGU35043499Z3KF776498ArxulaterrestrisAB00066398Z4KF776499Penicilliumsolitumstrain20-01JN64222299Z5KF776500HemifridericiaparvaGU90190699Z6KF776501BaculentulustianmushanensisAY03716993
3结论与讨论
在DGGE电泳图谱中,处于不同位置的每条条带都是不同属种微生物的18 S rDNA的核酸片段,其相对亮度代表森林土壤真菌群落中某一特定真菌菌种及其在群落中的相对丰度,电泳条带越亮,表示该菌种的数量越多[10],电泳条带数量越多,说明微生物种群越多[11-12]。根据DGGE图谱中条带数目不同,可以得出各林型土壤真菌多样性高低顺序依次为:落叶松白桦混交林、落叶松林、樟子松落叶松混交林、山杨林、白桦林和黑桦林,进一步利用Quantity One 4.5.2软件对各泳道的香农系数进行分析,得到了相似的结果。根据DGGE图谱中条带亮度不同,可以看出各个林型土壤中优势真菌种类是不相同的,同时对DGGE图谱中优势条带进行切胶回收、克隆、测序进一步验证了这一结论。上述结论表明:森林土壤真菌区系与森林类型密切相关,这与董爱荣等[13]关于凉水自然保护区森林土壤真菌的多样性研究及姚贤民等[14]关于长白山森林土壤真菌区系研究结果相似。
根据系统树和相似性指数数据可以得出:林型相似,土壤中真菌种类越相近,相反,林型差异越大,土壤中真菌种类差异就越大。如白桦林土壤样品中真菌种类与落叶松白桦混交林中真菌种类最相近;阔叶林如白桦林、黑桦林和杨树林土壤真菌种类比较相近,而针叶林如落叶松林和樟子松落叶松混交林土壤真菌种类比较相近。由此可见,生态环境的多样性直接影响了真菌的分布格局[15],同时森林土壤微生物种群的结构比例特征,在对土壤性质的反映上也起到了重要的作用[16]。
参考文献
[1]ZELLES L, BAI Q Y, BECK T, et al.Signature fatty acids in phospholipids and lip-opolysaccharides as indicators of microbial biomass and community structure in agricultural soils[J].Soil Biology and Biochemistry,1992,24(4):317-323.
[2]SKINNER F A, JONES P C, MOLLISON J E.A comparison of a direct and a plate-co-unting technique for the quantitative estimation of soil microorganisms[J].Journal of General Microbiology,1952,6(3/4):261-271.
[3]陈曦.东北地区药用植物根际土壤真菌多样性的研究[D].大连:辽宁师范大学,2010:9-10.
[4]张海燕,王彩虹,龚明福,等.一种简单有效且适于土壤微生物多样性分析的DNA提取方法[J].生物技术通报,2009(8):151-155.
[5]ZHOU J Z, BRUNS M A, TIEDJE J M.DNA recovery from soils of diverse composition[J].Applied and Environmental Microbiology,1996,62(2):316-322.
[6]孙海涛.中国黄土高原苹果产区土壤真菌群落多样性及生防放线菌菌株筛选[D].泰安:山东农业大学,2012:8-10.
[7]吕新,陈丽华,李玥仁.4种不同土壤微生物DNA提取方法对DGGE分析微生物群落的影响[J].福建农业学报,2012,27(4):367-372.
[8]陈谊.利用PCR-DGGE研究膜生物反应器中微生物的群落结构[D].天津:天津大学,2009:27-28.
[9]BRIDGE P, SPOONER B.Soil fungi: diversity and detection[J].Plant and Soil,2001,232(1/2):147-154.
[10]Ellis M B, Ellis J P.Micro fungi on land plants[M].NewYork: Macmillan Publishing Co,1985:370-377.
[11]DUNBAR J, TICKNOR L O, KUSKE C R.Assesment of microbial diversity in four soutlweestem United States Soils by 16S rDNA gene terminal restriction fragment analysis[J].Appl Environ Microbiol,2000,66(7):2943-2950.
[12]MUYZER G, SMALLA K.Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology[J].Antonie van Leeuwenhoek,1998,73:127-141.
[13]董爱荣,吕国忠,吴庆禹,等.小兴安岭凉水自然保护区森林土壤真菌的多样性[J].东北林业大学学报,2004,32(1):8-10.
[14]姚贤民,吕国忠,杨红,等.长白山森林土壤真菌区系研究[J].菌物研究,2007,5(1):43-46.
[15]杨红.长白山自然保护区北坡森林土壤真菌种群及其多样性研究[D].沈阳:沈阳农业大学,2009:5-6.
[16]时雷雷.海南尖峰岭热带山地雨林原始林土壤理化性质空间异质性研究[D].昆明:中国林业科学研究院,2012:57-80.
第一作者简介,赵文静,女,1988年10月生,东北林业大学生命科学学院,硕士研究生。E-mail:786951580@qq.com。 通信作者:赵敏,东北林业大学生命科学学院,教授。E-mail:82191513@163.com。
收稿日期:2014年6月4日。
分类号S714.3
Diversity Analysis of Soil Fungal Community Structure in Erguna National Nature Reserve by PCR-DGGE//
Zhao Wenjing(Northeast Forestry University, Harbin 150040, P.R.China); Zhao Baojun(Inner Mongolia Forest Industry Group); Cui Daizong, Zhao Min(Northeast Forestry University)//
Journal of Northeast Forestry University,2016,44(7):76-79.
We studied the variation of the fungal community structure in the soil of Betula davurica, Populus davidiana, Pinus sylvestris-Larix gmelinii, Larix gmelinii-Betula platyphylla, Larch and Betula platyphylla in Erguna National Nature Reserve by PCR-DGGE.The soil mycobiota were closely related to forest types, and each forest type had its unique species.The diversity ratio of soil fungi in different forest types ranged in descending order of Larix gmelinii-Betula platyphylla, Larch, pinus sylvestris-Larix gmelinii, Populus davidiana, Betula platyphylla and Betula davurica Stands, and each forest type had its unique advantage species.Soil fungi was resemble in similar, but various in different forest soil type.
KeywordsErguna National Nature Reserve; Soil fungi; 18SrDNA; PCR-DGGE; Fungal Diversity
责任编辑:程红。
