普通小麦转录因子基因TaWRKY35的克隆及功能分析
2016-07-18刘自成苗丽丽王景一杨德龙毛新国景蕊莲
刘自成,苗丽丽,王景一,杨德龙,毛新国,景蕊莲
(1甘肃农业大学生命科学技术学院,兰州 730070;2中国农业科学院作物科学研究所/农作物基因资源与基因改良国家重大科学工程/农业部农作物种质资源创新与利用重点开放实验室,北京 100081)
普通小麦转录因子基因TaWRKY35的克隆及功能分析
刘自成1,2,苗丽丽2,王景一2,杨德龙1,毛新国1,2,景蕊莲2
(1甘肃农业大学生命科学技术学院,兰州 730070;2中国农业科学院作物科学研究所/农作物基因资源与基因改良国家重大科学工程/农业部农作物种质资源创新与利用重点开放实验室,北京 100081)
摘要:【目的】非生物逆境是制约小麦生产的主要因素。WRKY转录因子是植物特有的转录因子家族,参与对多种非生物逆境胁迫的应答。普通小麦基因组中至少含有200个WRKY,目前仅对少数成员的功能进行了鉴定。通过克隆小麦 WRKY转录因子 TaWRKY35并揭示其功能,为改良小麦的抗逆性提供基因资源。【方法】利用小麦全长cDNA数据信息,从强抗旱小麦品种旱选10号中克隆逆境胁迫应答转录因子基因TaWRKY35;采用实时定量PCR技术,分析目标基因在不同发育时期、不同组织器官中的表达水平,以及在PEG-6000、NaCl、ABA和低温等逆境胁迫下的表达模式;构建目标基因与GFP融合的表达载体,转化小麦原生质体,以确定目标蛋白在细胞中的作用位置;构建TaWRKY35过表达载体,通过农杆菌介导的方法转化拟南芥,揭示目标基因的功能。【结果】TaWRKY35的开放阅读框全长1 134 bp,编码377个氨基酸的蛋白。TaWRKY35的N端有一个典型的WRKY结构域,C端有一个C2HC型的锌指结构域,属于WRKY家族第Ⅲ亚家族。测序发现TaWRKY35编码区序列非常保守,在32份多态性较高的六倍体小麦材料中未检测到DNA多态性。TaWRKY35在小麦的不同发育时期、不同组织中均有表达,其中在幼苗根基部表达量最高,约为苗期叶片中表达量的26倍;其次为幼芽根基,约为苗期叶片中表达量的21倍;接下来依次为孕穗期茎节、孕穗期穗、芽期根、孕穗期根、苗期根、胚芽、孕穗期叶片,而在幼苗叶片中表达水平最低;同时在ABA、NaCl、PEG和低温胁迫条件下,小麦幼苗TaWRKY35的表达量均显著上升,表明TaWRKY35参与对4种逆境胁迫的应答,但在不同胁迫条件下表达模式差异显著,其中对盐胁迫最敏感。亚细胞定位发现,TaWRKY35特异定位在细胞核上,是典型的核定位蛋白。在高盐胁迫条件下,TaWRKY35过表达转基因拟南芥子叶白化率显著低于对照,TaWRKY35过表达转基因拟南芥的细胞膜稳定性和幼苗存活率均显著高于野生型对照,表明TaWRKY35能增强植物的耐盐性。【结论】TaWRKY35编码蛋白含有WRKY和C2HC结构域,为WRKY家族第Ⅲ亚家族成员。TaWRKY35在小麦发育的不同时期、不同组织中均有表达,且受ABA、PEG、NaCl及低温等逆境胁迫诱导。过表达TaWRKY35能显著提高转基因拟南芥的耐盐性。
关键词:小麦;WRKY转录因子基因;非生物胁迫;耐盐性
联系方式:刘自成,E-mail:liuyao8916@163.com。通信作者杨德龙,Tel:0931-7631875;E-mail:yangdl@gsau.edu.cn。通信作者毛新国,Tel:010-82105829;E-mail:maoxinguo@caas.cn
0 引言
【研究意义】小麦是世界上重要的粮食作物之一,干旱、高盐、极端温度等非生物逆境是制约小麦生产的主要因素。克隆抗逆基因,揭示其功能是利用现代生物技术改良小麦抗逆性的前提和基础。【前人研究进展】遭受逆境胁迫时,植物在分子、细胞和生理水平产生一系列变化以适应不利环境[1-2]。植物体内参与逆境胁迫应答的基因很多,根据基因产物的功能分为两大类:功能蛋白和调节蛋白,其中,功能蛋白主要包括水通道蛋白、LEA蛋白、渗透调节物质合成的酶类以及各种抗氧化酶等;调节蛋白则包括蛋白激酶、蛋白磷酸酶和转录因子等。WRKY转录因子是植物特有的一类转录因子,因其 N端含有高度保守的WRKYGQK基序而得名。WRKY转录因子最早发现于甘薯中[3],随后在野燕麦、荷兰芹、拟南芥、马铃薯、白英、烟草和水稻中相继被发现[4]。WRKY家族成员均含1—2个与DNA结合的WRKY结构域,该结构域由 60个左右高度保守的氨基酸组成,其中WRKYGQK是核心序列,该序列氨基酸突变将导致DNA结合活性的减弱,甚至丧失[5];WRKY成员的C端包含一个锌指结构域,即 C2H2或者 C2HC。根据WRKY结构域的数量和锌指结构域的类型将WRKY家族分成3个亚家族(Group)[4]:亚家族Ⅰ(GroupⅠ)有2个WRKY结构域和1个C2H2(CX4-5CX22-23HX1H)型锌指结构域;亚家族Ⅱ(GroupⅡ)成员只含有 1 个WRKY结构域和1个C2H2结构域;亚家族Ⅲ(GroupⅢ)成员含1个WRKY结构域和1个C2HC型(即CX7CX23HX1C)锌指结构域[6]。WRKY转录因子通过WRKY结构域特异识别目标基因启动子区域的(T)(T)TGAC(C/T)序列(W-box)进而调控目标基因的表达[4,7]。研究发现,WRKY转录因子在植物生长发育过程中起重要作用,并参与抗病应答[8]。近期越来越多的证据表明,WRKY转录因子通过ABA依赖或者非ABA依赖的逆境信号转导途径参与对非生物逆境胁迫的应答[9-11]。过表达WRKY转录因子可以增强植物的抗逆性,例如过表达 AtWRKY25、AtWRKY33能显著增强转基因拟南芥对高温和高盐的耐受性[12-14];过表达BdWRKY36能显著增强烟草对干旱的耐受性[15];过表达GmWRKY13、GmWRKY21和GmWRKY54能增强植物对多种非生物逆境胁迫的耐受性[16];过表达OsWRKY11能够提高水稻对高温、干旱等胁迫的耐受性[17],过表达OsWRKY47能提高水稻的抗旱性[18]。研究推测,普通小麦基因组中至少含有200个WRKY基因[19],但目前仅有少数成员的功能被鉴定,其中 TaWRKY10(GroupⅡ)、TaWRKY44(GroupⅠ)参与对多种非生物逆境胁迫的应答,过量表达能增强植物的抗旱性和耐盐性[20-21];2012年,NIU等[22]在小麦cDNA文库中发现了43个WRKY转录因子,其中TaWRKY2(GroupⅠ)受干旱、盐和ABA诱导表达,过量表达TaWRKY2能提高转基因拟南芥的抗旱、耐盐性。TaWRKY19(GroupⅠ)除受上述3种逆境胁迫诱导外,还参与对冷胁迫的应答,过量表达该基因能提高拟南芥的抗旱、耐盐及耐冻性[22]。2015年,QIN等[23]研究发现TaWRKY93参与对盐胁迫和 ABA处理的应答,过量表达 TaWRKY93 (GroupⅡ)能提高拟南芥对多种非生物逆境的耐受性。【本研究切入点】已有研究证明,小麦 WRKY转录因子参与对非生物胁迫的应答,然而迄今为止,仅对GroupⅠ、GroupⅡ的个别基因进行了功能分析,而关于 Group Ⅲ成员的功能研究尚未见报道。TaWRKY35属于WRKY Group Ⅲ,它是否参与非生物胁迫的应答,以及其在逆境胁迫应答反应的功能目前均不明确。【拟解决的关键问题】本研究克隆了一个新的小麦 WRKY基因,属于 Group Ⅲ,命名为TaWRKY35。对其在不同发育时期、不同组织的特异性表达水平,在ABA、PEG、NaCl、低温逆境胁迫处理的表达模式,以及在细胞中的作用部位进行分析,并通过转化拟南芥鉴定其功能,旨在为利用 WRKY转录因子提高小麦抗逆性提供理论依据。
1 材料与方法
1.1 植物材料及其培养
试验于 2014—2015年在中国农业科学院作物科学研究所进行。利用农业部农作物种质资源创新与利用重点开放实验室保存的强抗旱小麦品种旱选 10号进行基因克隆、基因表达模式分析和目标蛋白的亚细胞定位。挑选大小均匀一致的旱选10号种子放在玻璃培养皿中,在光照培养箱(23℃、光周期12 h·d-1)中水培,待幼苗成长至一叶一心(10 d苗龄)时,分别用50 mmol·L-1ABA、16.1% PEG-6000(-0.5 MP)、250 mmol·L-1NaCl和低温(4℃)进行胁迫处理,在处理0、0.5、1、1.5、2、3、6、12、24、48和72 h取幼苗叶片,-80℃保存,用于分析目标基因逆境胁迫条件下的表达模式。在培养皿上水培旱选10号,芽期取根部、根基部、胚芽;田间种植旱选10号,苗期取幼苗的根、根基和叶片,孕穗期取茎节,孕穗后期取根、叶和幼穗,-80℃保存,用于分析目标基因的组织特异表达。挑选籽粒饱满的小麦种子在培养皿中,用去离子水室温避光培养,培养至一叶一心时,取黄化幼苗叶片的中段制备原生质体,进行目标蛋白的亚细胞定位。
基因染色体定位材料包括普通小麦二倍体供体种材料:乌拉尔图小麦(UR200、UR204、UR205)、拟斯卑尔托山羊草(Y2017、Y2033、Y2035和Y2178)、粗山羊草(Ae38、Ae46和Y57);四倍体材料(DS1、DM49和DM50)和异源六倍体普通小麦材料旱选10号。基因多态性分析材料为农业部农作物种质资源创新与利用重点开放实验室前期利用 SSR标记筛选的32份高多态性材料(表1)。室温条件下水培,取幼苗叶片提取DNA。
1.2 目的基因克隆
利用WRKY保守结构域搜索小麦全长cDNA数据库,得到目标基因的cDNA序列。根据cDNA序列信息设计引物WRKY35-C-F和WRKY35-C-R(表2),以旱选10号小麦cDNA为模板进行PCR扩增目标基因。以目标基因的cDNA序列为源序列,搜索小麦A基因组数据库和D基因组数据库,通过A基因组数据库得到的基因组序列与cDNA序列一致性最高,以该基因组序列为模板,利用Primer Premier 5.0软件设计扩增全长序列的引物TaWRKY35-F和TaWRKY35-R(表2)。
用植物总RNA提取试剂盒(TIANGEN,北京)提取小麦不同胁迫处理的叶片和不同组织的总RNA。以总RNA为模板,用M-MLV Reverse Transcriptase反转录试剂盒(Invitrogen,美国)反转录合成第一链cDNA。以cDNA为模板,利用高保真聚合酶TransStart Fast Pfu(Transgen,北京)进行RT-PCR扩增。反应体系包含模板DNA(50 ng·μL-1)2 μL、5×TransStart Fast Pfu buffer 4 μL、2.5 μmol·L-1dNTP 1.6 μL、DNA Polymerase 0.4 μL、正、反向引物(10 μmol·L-1)各0.8 μL,用ddH2O补足20 μL。反应条件为95℃ 5 min;95℃ 45 s,60℃ 45 s,72℃ 60 s,38个循环;72℃ 10 min。用1.5%琼脂糖凝胶分离PCR产物,将目的片段用 PCR产物纯化试剂盒(生工,上海)回收后,与pEASY-Blunt载体(Transgen,北京)连接,用BigDye Terminator Kit(ABI,美国)测序。
1.3 基因结构和序列分析
用DNAStar软件包中Seqman和MegAlign进行基因序列分析、拼接和比对,用DNAman进行蛋白序列比对分析,用MEGA(Version 6.06)构建系统进化树。
表1 供试小麦材料Table 1 Wheat accessions used in this study

表2 试验中使用的引物Table 2 Primers used in the experiments
1.4 载体构建和亚细胞定位
利用生物信息学网站(http://cello.life.nctu.edu.tw)预测 TaWRKY35在细胞中的定位。为进一步验证预测结果,设计亚细胞定位引物(分别在引物5′和3′端引入了HindⅢ和BamHⅠ酶切位点),将TaWRKY35全长CDS(coding sequence)序列(不含终止密码子)克隆到 pJIT163-GFP载体融合表达,构建 pJIT163-35S::TaWRKY35::GFP载体。用PEG法转化小麦原生质体,室温避光培育15 h,在Zeiss LSM700激光共聚焦显微镜下观察TaWRKY35-GFP在小麦原生质体中的定位。
1.5 目标基因的实时定量RT-PCR检测
根据TaWRKY35全长CDS序列设计qRT-PCR引物WRKY35-RT-F和WRKY35-RT-R,以小麦Actin (JQ269668.1)的引物 Actin F1和 Actin R1为qRT-PCR内参引物(表2)。根据SYBR Premix Ex Taq (Perfect Real Time)试剂盒(TaKaRa,日本)说明书操作,反应体系为1/10 cDNA first-strand 1.0 μL、2× SYBR Premix Ex Taq 10.0 μL、50×ROX Reference Dye 0.4 μL、正、反向引物(5 μmol·L-1)各0.4 μL,用ddH2O补至20 μL。用ABI 7900实时定量PCR仪进行PCR扩增,反应条件为95℃ 30 s;95℃ 5 s,退火30 s,40个循环。数据采用2-△△CT法计算目标基因的相对表达量[24]。
1.6 拟南芥转化及其耐盐性鉴定
利用KpnⅠ和BamHⅠ酶切位点将TaWRKY35全长CDS序列克隆到pCHF3载体上。将构建的载体转化农杆菌GV3101菌株,采用浸花法转化拟南芥。经卡那霉素抗性筛选和PCR检测后获得T3纯合转基因株系,选取目标基因表达量相对较高的3个株系用于耐盐性分析。
将野生型(WT)、转基因株系种子用 10%次氯酸钠溶液消毒处理后,点播于MS培养基上,将培养皿置于人工气候室中垂直培养。将培养7 d的幼苗分成两部分,一部分在MS培养基上进行抗逆表型鉴定,具体操作如下:将长势一致的幼苗10株,分别移至含有 60 μmol·L-1ABA、250 mmol·L-1NaCl和 200 mmol·L-1甘露醇的MS固体培养基上,处理7 d,观察表型,设3次重复。另一部分移到底部有孔的长方形筛盘中(营养土∶蛭石=1∶1),每个株系一行,每行10株幼苗。正常生长3周,然后用200 mmol·L-1NaCl溶液处理(将筛盘放在盐溶液中,直至土壤盐溶液饱和),跟踪观察转基因植株在盐胁迫下的表型,计算植株存活率,重复3次。
细胞膜稳定性测定:利用电导仪分别测定煮沸前与煮沸后幼苗的相对电导率,评价细胞膜稳定性(cell membrane stability,CMS)。从垂直培养的幼苗中取长势一致的幼苗10株,移至含有250 mmol·L-1NaCl 的MS固体培养基上,处理7 d。测定幼苗电导率,10株为一次重复,设3次重复。CMS(%)=(1-煮前电导率/煮后电导率)×100%。CMS值越大,表明细胞膜稳定性越高,植物受损伤程度越低。
2 结果
2.1 目标基因克隆
以普通小麦旱选10号cDNA为模板,利用引物对WRKY35-C-F和WRKY35-C-R扩增目标基因。测序表明,该基因开放阅读框全长1 134 bp,编码377个氨基酸。NCBI数据库比对发现该蛋白的N端有一个典型的WRKY结构域,C端有一个C2HC型锌指结构域。
2.2 TaWRKY35氨基酸序列同源性分析
将TaWRKY35编码的氨基酸序列提交到NCBI网站上进行蛋白质序列比对,发现其与大麦HvWRKY35 (ABL_11228.1)、短柄草BdWRKY2(XP_014757278.1)、水稻 OsWRKY35(NP_001056863.1)、拟南芥AtWRKY35(XP_008660715.1)具有较高的相似性,序列一致性分别为79%、60%、50%和45%,因此将其命名为 TaWRKY35。尽管在序列一致性方面有差异,但TaWRKY35与上述WRKY成员的序列结构相同,均含有一个WRKY结构域和C端锌指结构(图1-A)。小麦TaWRKY35含有一个WRKY结构域和一个C2HC类型的锌指结构域,属于WRKY转录因子GroupⅢ(图1-B)。
2.3 TaWRKY35结构及序列多态性分析
以小麦二倍体供体种(乌拉尔图小麦、拟斯卑尔托山羊草、粗山羊草)、四倍体和六倍体小麦基因组 DNA为模板,利用引物 TaWRKY35-F和TaWRKY35-R扩增目标基因,发现只能在含有A基因组的材料中扩增出 TaWRKY35,说明其可能来自小麦A基因组。测序表明该基因编码区全长2.55 kb,含有3个外显子,2个内含子。用上述引物在32份高多态性的六倍体小麦中扩增 TaWRKY35,测序结果未发现DNA多态性,说明TaWRKY35编码区序列非常保守,预示在小麦生长发育中可能具有极其重要的作用。
2.4 TaWRKY35组织特异性表达
TaWRKY35组织特异性表达模式的分析表明,该基因在小麦发育的不同时期、多种组织器官中均有表达。其中在幼苗根基中表达量最高,约为叶片中表达量的26倍;其次为幼芽根基,约为苗期叶片中表达量的21倍;接下来依次为孕穗期茎节、孕穗期穗、芽期根、孕穗期根、苗期根、胚芽、孕穗期叶片、苗期叶片(图2)。
2.5 逆境胁迫诱导下TaWRKY35的表达模式
在ABA、NaCl、PEG和低温胁迫条件下,小麦幼苗TaWRKY35的表达量均显著上升,表明TaWRKY35参与对4种逆境胁迫的应答。在外源ABA诱导下,TaWRKY35表达模式呈波浪形,在1.5 h达到一个初级峰值,约为对照的8倍,而后逐渐降低,12 h时表达量降至最低,随后又逐步回升,48 h达到最大值(约为对照的13倍),表明TaWRKY35可能在ABA信号通路中起作用(图3-A)。TaWRKY35对低温胁迫比较敏感,低温处理1 h即达到最大值,随后急剧下降(图3-B)。NaCl胁迫下,TaWRKY35的表达在24 h时达到高峰,约为对照的31倍,随后迅速下降(图3-C)。在PEG处理6 h时,TaWRKY35表达量达到第一个高峰,约为对照的4倍,随后下降,在72 h表达量最高,约为对照的5倍(图3-D),说明TaWRKY35受渗透胁迫诱导表达。
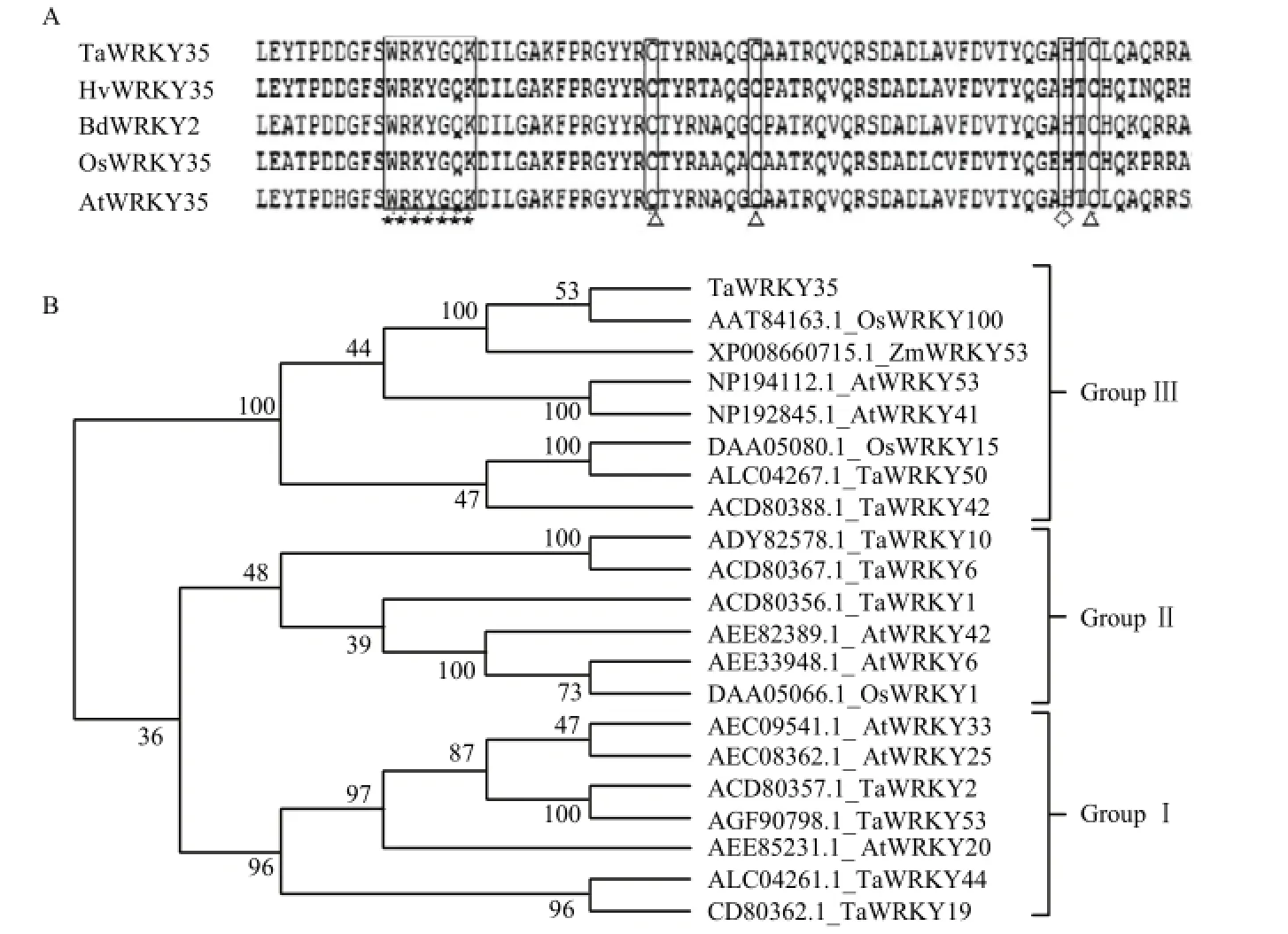
Ta:小麦;Hv:大麦;Bd:短柄草;Os:水稻;At:拟南芥;WRKY:WRKY转录因子。* 代表WRKY结构域中核心序列WRKYGQK所在位置;∆ 代表锌指结构中半胱氨酸所在位置;◇ 代表锌指结构中组氨酸所在位置Ta: Triticum aestivum; Hv: Hordeum vulgare; Bd: Brachypodium distachyon; Os: Oryza sativa; At: Arabidopsis thaliana; WRKY: WRKY-type transcription factor. * indicates the WRKYGQK of WRKY domain; ∆ indicates the cysteine of zinc finger; ◇ indicates the histidine of zinc finger图1 几种植物WRKY序列比对(A)与系统进化分析(B)Fig. 1 Alignment (A) and phylogenetic tree (B) of WRKYs from different plant species
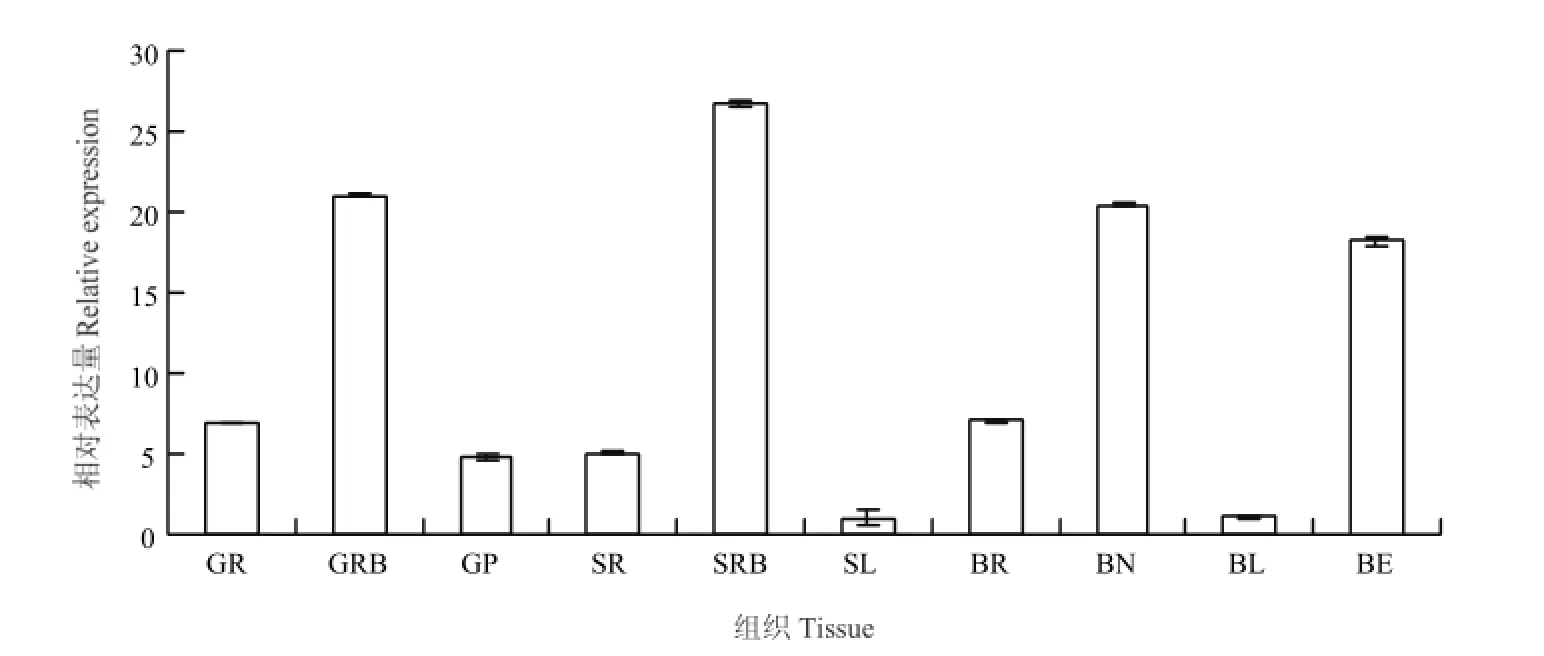
GR:芽期根;GRB:幼芽根基;GP:胚芽;SR:苗期根;SRB:幼苗根基;SL:苗期叶片;BR:孕穗期根;BN:孕穗期茎节;BL:孕穗期叶片;BE:孕穗期穗。以苗期叶片中的TaWRKY35表达量为标准。数据为3次独立试验的平均值 ± SEGR: Germination stage root; GRB: Germination stage root base; GP: Germ; SR: Seeding stage root; SRB: Seeding stage root base; SL: Seeding stage leaf blade;BR: Booting stage root; BN: Booting stage node; BL: Booting stage leaf; BE: Booting stage ear. The expression level of TaWRKY35 in seedling leaf (SL) is assigned a value of 1. Values are the mean ± SE of three biological replicates图2 TaWRKY35在小麦不同发育时期的组织特异性表达Fig. 2 Dynamic expression of TaWRKY35 in multi-tissue at different stages of wheat
2.6 TaWRKY35亚细胞定位
生物信息学预测TaWRKY35定位在细胞核上。用激光共聚焦显微镜观察小麦原生质体,发现TaWRKY35::GFP只在细胞核上有荧光信号,而细胞质和细胞膜中无GFP信号(图4),表明TaWRKY35特异定位在细胞核上。
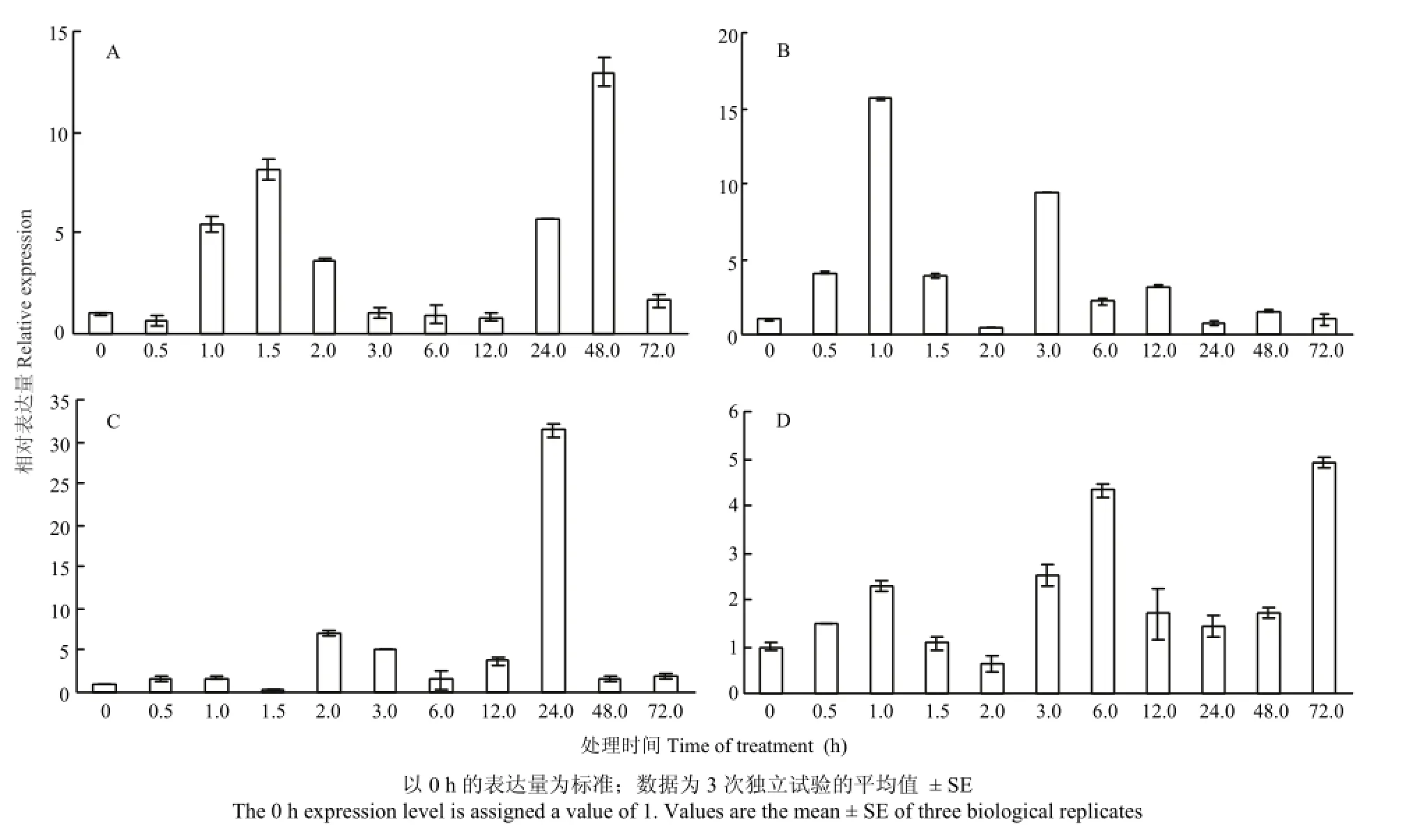
图3 胁迫条件下小麦幼苗TaWRKY35的表达模式Fig. 3 Expression patterns of TaWRKY35 in wheat seedlings under different treatments

图4 TaWRKY35特异定位在小麦细胞核中Fig. 4 TaWRKY35 specifically locates in the nucleus in wheat
2.7 过表达TaWRKY35能提高拟南芥的耐盐性
根据转基因株系中TaWRKY35表达水平,选取表达量相对较高的3个株系L-2、L-4和L-5分析基因功能(图5)。将在MS固体培养基上生长7 d的转基因和野生型拟南芥幼苗移至含有250 mmol·L-1NaCl的MS固体培养基上,培养7 d,部分幼苗子叶开始变白(图6-A)。

以转基因拟南芥株系L-1的表达量作为内参;数据为3次独立试验的平均值 ± SEThe lowest expression of TaWRKY35 in transgenic Arabidopsis line L-1 was regarded as standard. Data represent means ± SE of three replicates图5 TaWRKY35在5个转基因拟南芥株系中的表达水平Fig. 5 Expression levels of TaWRKY35 in five transgenic Arabidopsis lines
将种在筛盘中的拟南芥幼苗用200 mmol·L-1NaCl溶液处理7 d,野生型植株叶片明显黄化,而转基因株系的叶片仅略微黄化;盐处理21 d,近55%的野生型植株死亡,而转基因株系的存活率为70%—75%。为进一步验证 TaWRKY35过表达能增强拟南芥的耐盐性,在土壤中进行了耐盐性鉴定试验,结果和培养皿上的存活率统计结果基本吻合,如图6-B所示。考虑到土壤中的鉴定更接近自然条件下盐胁迫的实际情况,故此处使用了土壤中存活率统计结果。
细胞膜稳定性检测结果表明,TaWRKY35过表达转基因株系细胞膜稳定性显著高于野生型对照(图6-C)。
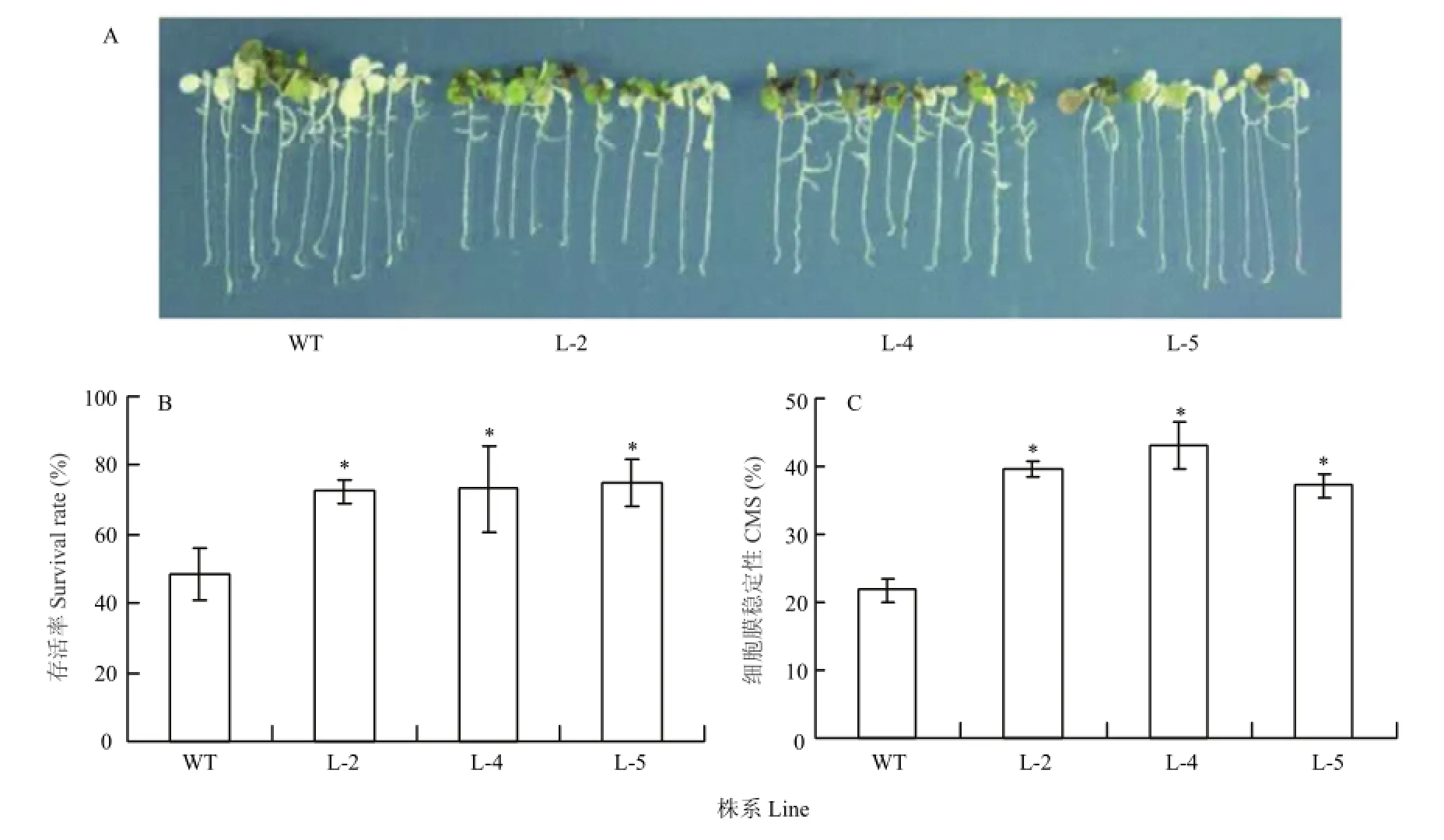
A:NaCl处理7 d的拟南芥表型;B:NaCl处理的拟南芥存活率比较;C:NaCl处理的细胞膜稳定性(CMS)比较,数据为3次独立试验的平均值 ±SE;* 表示转基因株系与野生型在0.05概率水平有显著差异(t-检验)。WT:拟南芥野生型;L-2、L-4和L-5:3个TaWRKY35过表达拟南芥株系A: Arabidopsis seedlings treated with NaCl for 7 days; B: Plant survival rates in soil saturated with NaCl solution; C: Comparison of seedling cell membrane stability (CMS) of Arabidopsis seedlings treated with NaCl for 7 days. Values are the mean ± SE of three biological replicates. * indicates significant difference between the transgenic lines and WT at 0.05 probability level (t-test). Values are the mean ± SE of three biological replicates. WT: Wild type; L-2, L-4 and L-5:Three individual TaWRKY35 transgenic lines图6 TaWRKY35过量表达能提高拟南芥的耐盐性Fig. 6 Overexpression of TaWRKY35 confers enhanced tolerance to salt stress in Arabidopsis
3 讨论
植物基因的时空表达是其参与不同生命过程的直接反映。在不同逆境胁迫条件下,基因的功能不同,其表达模式也千差万别,调节基因通常在逆境胁迫的早期表达,而功能基因的表达则相对滞后[25]。本研究发现TaWRKY35受ABA、PEG、NaCl和低温等多种逆境胁迫诱导表达,同时不同逆境胁迫条件下基因的表达模式差异显著,说明该基因参与对多种逆境胁迫的应答,暗示TaWRKY35在不同逆境应答途径中参与的调节机制可能不同。
蛋白质在细胞中的定位一定程度上反映了其行使功能的部位。生物信息学预测结果表明 TaWRKY35可能定位在细胞核中,亚细胞定位结果进一步证明TaWRKY35-GFP融合蛋白仅出现在细胞核中,该结果与已发表的一些 WRKY转录因子的亚细胞定位结果一致[26],同时也与TaWRKY35作为转录因子参与调控基因的表达相吻合。
在正常生长条件下,TaWRKY35过表达拟南芥和野生型对照在表型上没有差异,但在盐胁迫条件下,转基因拟南芥子叶白化率显著低于对照(图 6-A),同时幼苗存活率显著高于野生型(图 6-C)。细胞膜稳定性是衡量植物抗逆性强弱的重要指标,在高盐条件下植物细胞膜透性通常会增加,但耐盐植物质膜透性变化较小,因此,细胞膜稳定性较高,而盐敏感植物则正好相反。本研究发现转基因植株的细胞膜稳定性显著高于野生型对照(图 6-B),进一步证明TaWRKY35提高了拟南芥的耐盐性。在增强植物的耐盐性方面,TaWRKY35与之前报道的 BcWRKY46、TaWRKY2、TaWRKY19、TaWRKY44和 HvWRKY38的功能相似[20,22,27-29]。
实时定量PCR结果表明,在ABA、PEG胁迫条件下TaWRKY35表达量显著升高。在表型分析时,在MS培养基上对转基因拟南芥和野生型分别进行了ABA(60 μmol·L-1)和甘露醇(200 mmol·L-1)胁迫处理,发现野生型和转基因株系的表型差异不明显,这可能与TaWRKY35在信号调节通路中的作用有关,这一现象与GmWRKY21、GmWRKY54转基因过表达的表型类似[16]。比较TaWRKY35在不同逆境胁迫下的表达水平,发现其在盐胁迫下表达量最高,该结果与其过表达提高植物的耐盐性吻合。
近期人们对 WRKY转录因子的结构、功能以及在其调控胁迫应答反应、信号分子传递、植物衰老以及器官发育等一系列生理活动过程进行了大量的研究,然而对WRKY转录因子所介导的植物应答反应及基因调控网络研究较少,未来应将更多的精力聚焦于解析WRKY在抗逆调控网络中的作用。
4 结论
从小麦品种旱选10号中克隆到WRKY转录因子基因TaWRKY35。TaWRKY35的N端含有典型的WRKY结构域,C端含有 C2HC型锌指结构,属于WRKY家族的亚家族Ⅲ,定位于细胞核中。TaWRKY35在小麦幼苗根基部位表达量最高,其次为幼芽根基部,同时TaWRKY35受ABA、低温、高盐和PEG-6000诱导上调表达;过表达TaWRKY35能显著提高转基因拟南芥的耐盐性。
References
[1] SUZUKI N, RIVERO R M, SHULAEV V, BLUMWALD E,MITTLER R. Abiotic and biotic stress combinations. New Phytologist,2014, 203: 32-43.
[2] MICKELBART M V, HASEGAWA P M, BAILEY-SERRES J. Genetic mechanisms of abiotic stress tolerance that translate to crop yield stability. Nature Reviews Genetics, 2015, 16: 237-251.
[3] ISHIGURO S, NAKAMURA K. Characterization of a cDNA encoding a novel DNA-binding protein, SPF1, that recognizes SP8 sequences in the 5' upstream regions of genes coding for sporamin and beta-amylase from sweet potato. Molecular and General Genetics,1994, 244: 563-571.
[4] EULGEM T, RUSHTON P J, ROBATZEK S, SOMSSICH I E. The WRKY superfamily of plant transcription factors. Trends in Plant Science, 2000, 5: 199-206.
[5] MAEO K, HAYASHI S, KOJIMA-SUZUKI H, MORIKAMI A,NAKAMURA K. Role of conserved residues of the WRKY domain in the DNA-binding of tobacco WRKY family proteins. Bioscience,Biotechnology, and Biochemistry, 2001, 65: 2428-2436.
[6] WU K L, GUO Z J, WANG H H, LI J. The WRKY family of transcription factors in rice and Arabidopsis and their origins. DNA Research, 2005, 12: 9-26.
[7] CIOLKOWSKI I, WANKE D, BIRKENBIHL R P, SOMSSICH I E. Studies on DNA-binding selectivity of WRKY transcription factors lend structural clues into WRKY-domain function. Plant Molecular Biology, 2008, 68: 81-92.
[8] RUSHTON P J, SOMSSICH I E, RINGLER P, SHEN Q J. WRKYtranscription factors. Trends in Plant Science, 2010, 15: 247-258.
[9] RUSHTON D L, TRIPATHI P, RABARA R C, LIN J, RINGLER P,BOKEN A K, LANGUM T J, SMIDT L, BOOMSMA D D, EMME N J, CHEN X, FINER J J, SHEN Q J, RUSHTON P J. WRKY transcription factors: Key components in abscisic acid signalling. Plant Biotechnology Journal, 2012, 10: 2-11.
[10] LI C, LÜ J, ZHAO X, AI X, ZHU X, WANG M, ZHAO S, XIA G. TaCHP: A wheat zinc finger protein gene down-regulated by abscisic acid and salinity stress plays a positive role in stress tolerance. Plant Physiology, 2010, 154: 211-221.
[11] LI H, XU Y, XIAO Y, ZHU Z, XIE X, ZHAO H, WANG Y. Expression and functional analysis of two genes encoding transcription factors, VpWRKY1 and VpWRKY2, isolated from Chinese wild Vitis pseudoreticulata. Planta, 2010, 232: 1325-1337.
[12] JIANG Y, DEYHOLOS M K. Functional characterization of Arabidopsis NaCl-inducible WRKY25 and WRKY33 transcription factors in abiotic stresses. Plant Molecular Biology, 2009, 69: 91-105.
[13] LI S, FU Q, CHEN L, HUANG W, YU D. Arabidopsis thaliana WRKY25, WRKY26, and WRKY33 coordinate induction of plant thermotolerance. Planta, 2011, 233: 1237-1252.
[14] LI S, FU Q, HUANG W, YU D. Functional analysis of an Arabidopsis transcription factor WRKY25 in heat stress. Plant Cell Reports, 2009,28: 683-693.
[15] SUN J, HU W, ZHOU R, WANG L, WANG X, WANG Q, FENG Z,LI Y, QIU D, HE G, YANG G. The Brachypodium distachyon BdWRKY36 gene confers tolerance to drought stress in transgenic tobacco plants. Plant Cell Reports, 2015, 34: 23-35.
[16] ZHOU Q Y, TIAN A G, ZOU H F, XIE Z M, LEI G, HUANG J,WANG C M, WANG H W, ZHANG J S, CHEN S Y. Soybean WRKY-type transcription factor genes, GmWRKY13, GmWRKY21,and GmWRKY54, confer differential tolerance to abiotic stresses in transgenic Arabidopsis plants. Plant Biotechnology Journal, 2008, 6:486-503.
[17] WU X, SHIROTO Y, KISHITANI S, ITO Y, TORIYAMA K. Enhanced heat and drought tolerance in transgenic rice seedlings overexpressing OsWRKY11 under the control of HSP101 promoter. Plant Cell Reports, 2009, 28: 21-30.
[18] RAINERI J, WANG S, PELEG Z, BLUMWALD E, CHAN R L. The rice transcription factor OsWRKY47 is a positive regulator of the response to water deficit stress. Plant Molecular Biology, 2015, 88:401-413.
[19] OKAY S, DERELLI E, UNVER T. Transcriptome-wide identification of bread wheat WRKY transcription factors in response to drought stress. Molecular Genetics and Genomics, 2014, 289: 765-781.
[20] WANG X, ZENG J, LI Y, RONG X, SUN J, SUN T, LI M, WANG L,FENG Y, CHAI R, CHEN M, CHANG J, LI K, YANG G, HE G. Expression of TaWRKY44, a wheat WRKY gene, in transgenic tobacco confers multiple abiotic stress tolerances. Frontiers in Plant Science,2015, 6: 615.
[21] WANG C, DENG P, CHEN L, WANG X, MA H, HU W, YAO N,FENG Y, CHAI R, YANG G, HE G. A wheat WRKY transcription factor TaWRKY10 confers tolerance to multiple abiotic stresses in transgenic tobacco. PLoS One, 2013, 8: e65120.
[22] NIU C F, WEI W, ZHOU Q Y, TIAN A G, HAO Y J, ZHANG W K,MA B, LIN Q, ZHANG Z B, ZHANG J S, CHEN S Y. Wheat WRKY genes TaWRKY2 and TaWRKY19 regulate abiotic stress tolerance in transgenic Arabidopsis plants. Plant Cell and Environment, 2012, 35:1156-1170.
[23] QIN Y, TIAN Y, LIU X. A wheat salinity-induced WRKY transcription factor TaWRKY93 confers multiple abiotic stress tolerance in Arabidopsis thaliana. Biochemical and Biophysical Research Communications, 2015, 464: 428-433.
[24] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T))Method. Methods, 2001, 25: 402-408.
[25] PANDEY S P, SOMSSICH I E. The role of WRKY transcription factors in plant immunity. Plant Physiology, 2009, 150: 1648-1655.
[26] ZOU C, JIANG W, YU D. Male gametophyte-specific WRKY34 transcription factor mediates cold sensitivity of mature pollen in Arabidopsis. Journal of Experimental Botany, 2010, 61: 3901-3914.
[27] WANG F, HOU X, TANG J, WANG Z, WANG S, JIANG F, LI Y. A novel cold-inducible gene from Pak-choi (Brassica campestris ssp. chinensis), BcWRKY46, enhances the cold, salt and dehydration stress tolerance in transgenic tobacco. Molecular Biology Reports, 2012, 39:4553-4564.
[28] XIONG X, JAMES V A, ZHANG H, ALTPETER F. Constitutive expression of the barley HvWRKY38 transcription factor enhances drought tolerance in turf and forage grass (Paspalum notatum Flugge). Molecular Breeding, 2009, 25: 419-432.
[29] XU Y, HU W, LIU J, ZHANG J, JIA C, MIAO H, XU B, JIN Z. A banana aquaporin gene, MaPIP1;1, is involved in tolerance to drought and salt stresses. BMC Plant Biology, 2014, 14: 59.
(责任编辑 李莉)
Cloning and Characterization of Transcription Factor TaWRKY35 in Wheat (Triticum aestivum)
LIU Zi-cheng1, 2, MIAO Li-li2, WANG Jing-yi2, YANG De-long1, MAO Xin-guo1,2, JING Rui-lian2
(1College of Life Science and Technology, Gansu Agricultural University, Lanzhou 730070;2Institute of Crop Science, Chinese Academy of Agricultural Sciences/National Key Facility for Crop Gene Resources and Genetic Improvement/Key Laboratory of Crop Germplasm and Utilization, Ministry of Agriculture, Beijing 100081)
Abstract:【Objective】Abiotic stresses are major limitations to wheat production worldwide. Transcription factors play crucialroles in abiotic stress signaling in plants. It is predicted that there are at least 200 WRKY genes in common wheat genomes, yet only a few of them have been functionally characterized. The aim of this study is to decipher the roles of WRKY transcription factors in abiotic stress signaling and facilitate the utilization of WRKY genes in the improvement of abiotic stress tolerance in wheat. 【Method】A wheat WRKY gene designated TaWRKY35 was cloned via the wheat full-length cDNA libraries. Quantitative real-time PCR was performed to identify the dynamic expression of target gene in different tissues at various developmental stages, and to characterize the transcriptional patterns responding to ABA, PEG, NaCl and low temperature treatments in wheat. To probe the subcellular location of TaWRKY35, the construct encoding TaWRYK35::GFP fusion protein was transferred into wheat protoplast by PEG mediated method. To characterize the function of TaWRYK35, target gene driven by the 35S promoter was delivered into Arabidopsis by Agrobacterium mediated method. 【Result】The cDNA of TaWRYK35 contains an 1 134 bp open reading frame,encoding a 377- amino acid protein. TaWRYK35 possesses a typical WRKY domain in the N-terminal and a C2HC type zinc finger domain in the C-terminal, belonging to group III of WRKY family. In 32 hexaploid wheat materials of highly polymorphic,TaWRKY35 coding region sequence is very conservative. The dynamic expression of TaWRKY35 was identified in different tissues at various developmental stages, and the highest expression occurred in the root base of seedling, while the lowest expression was observed in seedling leaf. Furthermore, its transcript was inducible by ABA, PEG, NaCl and low temperature treatments, yet the expression patterns to difference stress varied significantly. Subcellular localization indicated that TaWRKY35 specifically located in the nucleus. Overexpression of TaWRKY35 resulted in enhanced tolerance to high salinity, supported by improved cell membrane stability and survival rate relative to wild type Arabidopsis. 【Conclusion】TaWRKY35 transcription factor contains a WRKY and a C2HC type zinc finger domain, belonging to subgroup III of the WRKY family. The expression of TaWRKY35 occurs in different tissues at various developmental stages, and TaWRKY35 is an abiotic stress responsive gene. Overexpression of TaWRKY35 confers remarkably enhanced tolerance to high salinity.
Key words:wheat; WRKY transcription factor gene; abiotic stress; salt tolerance
收稿日期:2016-03-04;接受日期:2016-04-15
基金项目:国家高技术研究发展计划(“863”计划)(2011AA100501)、北京市自然科学基金(BJNSF6132030)
