RNA可视化原位杂交技术对感染细胞中猪瘟病毒RNA定位与分布
2016-07-18张玉杰张乾义孙永芳邹兴启朱元源赵启祖宁宜宝
张玉杰,赵 燕,徐 璐,张乾义,陈 锴,孙永芳,邹兴启,朱元源,赵启祖,宁宜宝,王 琴
(中国兽医药品监察所/国家猪瘟参考实验室,北京 100081)
RNA可视化原位杂交技术对感染细胞中猪瘟病毒RNA定位与分布
张玉杰,赵燕,徐璐,张乾义,陈锴,孙永芳,邹兴启,朱元源,赵启祖,宁宜宝,王琴
(中国兽医药品监察所/国家猪瘟参考实验室,北京 100081)
摘要:【目的】为研究CSFV RNA在体外感染细胞中的分布及定位,建立了一种准确、敏感的RNA可视化原位杂交技术。【方法】本研究通过比对GenBank中公布的CSFV、BVDV和BDV全序列,避开BVDV和BDV的同源区,设计了CSFV RNA及内参基因β-actin的特异探针。以CSFV中等致病力毒株(HeBHH1/95)为参考毒株,在PK15细胞中培养病毒,加入RNA可视化原位杂交的特异探针和相应试剂,采用荧光共聚焦显微镜进行成像观察。通过综合分析观测结果、荧光强度、重复性等因素,采用正交试验优化了对原位杂交过程中具有重要影响的蛋白酶K浓度和甲醛固定时间,建立了CSFV RNA可视化原位杂交技术,并与FAT方法比较该技术灵敏度;用我国目前流行的CSFV 1.1、2.1、2.2、2.3基因亚型及BVDV、PPV、PRV和PCV-2病毒进行特异性试验。最终,以CSFV强致病力毒株(SM)接种PK15细胞,病毒感染后0.5、1、3、6、8、10、14、18、24、36、48、72、96h(hours post inoculation,hpi)取样,每个时间点2个重复,采用CSFV RNA可视化原位杂交技术进行检测。为佐证病毒蛋白在细胞中的定位及分布,同时采用FAT方法对SM株E2蛋白在PK15细胞中的表达情况进行动态研究。【结果】采用该技术在荧光共聚焦显微镜下可观察到CSFV RNA在细胞中的定位;当蛋白酶K浓度为1∶1 000、甲醛固定时间为30min时为最优反应条件;灵敏度试验表明该技术对病毒的检测极限为10-8/200µL,比FAT高3.5个数量级;特异性试验结果显示该探针能与CSFV 1.1、2.1、2.2、2.3亚型结合,与BVDV、PPV、PCV-2、PRV无交叉反应。采用该技术对CSFV RNA感染后在靶细胞中的定位与分布研究结果显示∶0.5hpi在胞核和胞浆均能检测到RNA,0.5—6hpi RNA主要分布于胞核内并在核内富集;10hpi胞浆内RNA逐渐增多,胞核内RNA逐渐减少,24hpi RNA主要集中在胞浆内细胞核周围;36hpi核外RNA大量聚集增多,72hpi达到峰值;96hpi RNA总量有所下降。而FAT结果显示:8hpi在少数细胞的胞浆内检测到病毒E2蛋白,10—24hpi蛋白表达数量一直较少;36hpi蛋白表达量不断增多,72hpi达到最大值;96hpi荧光信号由强变弱。病毒蛋白在细胞浆内的聚集数量与细胞浆中RNA含量成正相关。【结论】首次建立了CSFV RNA可视化原位杂交检测技术,并对CSFV强致病力毒株在细胞中的RNA定位分布进行了研究,发现病毒RNA吸附和进入靶细胞的时间早于0.5hpi,观察到CSFV RNA有在细胞核内的生活史。
关键词:猪瘟病毒;定位;分布;RNA可视化原位杂交;荧光抗体试验(FAT)
联系方式:张玉杰,E-mail:zyj-0811@sohu.com。赵 燕,E-mail:zhaoyan82882@126.com。张玉杰、赵燕为同等贡献作者。通信作者王琴,Tel:010-61255400;E-mail:wq551@vip.sina.com
0 引言
【研究意义】猪瘟(classical swine fever, CSF)是由猪瘟病毒(classical swine fever virus, CSFV)引起的猪的高度接触性、致死性传染病[1], 造成了养猪业的极大损失[2-3],被世界动物卫生组织(OIE)列为必须报告的法定传染病之一。致病机理的研究是从根本上控制疫病的途径之一,而病毒的增殖过程又是 CSFV致病机理研究的重要内容之一。病毒成分的合成是病毒增殖过程的关键,包括病毒 RNA的复制和病毒蛋白的合成,因此在病毒RNA水平上研究CSFV的感染及复制机制具有重要的科学价值。本文旨在建立一种新的可视化原位杂交方法,用于研究CSFV RNA在细胞内的定位和分布规律,对阐述CSFV的致病机理具有重要的理论指导依据。【前人研究进展】国内外研究多集中于CSFV感染过程中免疫、病毒载量等领域,已经证实CSFV能在猪体内绝大多数的组织器官中进行感染和复制,病毒载量与不同器官和组织嗜性以及病程密切相关[4]。但是 CSFV在感染细胞后,RNA在细胞内的定位和分布情况仍不十分清楚。RNA可视化检测技术是基于原位杂交技术发展起来的一种新型RNA原位杂交技术。在癌症研究中,Babij等使用原位杂交技术验证了 STK33激酶活性与癌症细胞功能的相关性[5],通过此方法还验证了各受体mRNA的表达动态变化对乳癌的影响[6]。在病毒研究上,HONKAVUORI等也使用RNA原位杂交技术验证了新型小 RNA病毒在土耳其禽类中的存在[7]。最近,NANBO对埃博拉病毒RNA在体外靶细胞中的复制规律进行了研究,发现病毒吸附和进入靶细胞时间早于6 h,12—48 dpi病毒RNA在细胞内完成复制并释放入旁细胞[8]。【本研究切入点】RNA可视化原位杂交检测技术在动物病毒方面的研究还少见报道[9-11],尚未用于对CSFV致病机理的研究。本研究建立了一种RNA可视化原位杂交技术,将CSFV体外感染PK15细胞后,采用该方法对CSFV RNA进行细胞内定位,有助于了解CSFV RNA在靶细胞上的复制与分布动态。【拟解决的关键问题】从RNA 分子水平上对CSFV在感染细胞中的定位和分布动态进行研究,为阐明CSFV致病机制建立一种特异、灵敏和准确的RNA可视化原位杂交技术。
1 材料与方法
试验于2014年6月至2015年5月在中国兽医药品监察所检测技术研究室国家猪瘟参考实验室完成。
1.1 引物与探针
对GenBank公布的CSFV SM株(AF333000)全序列、牛病毒性腹泻病毒(bovine viral diarrhea virus,BVDV)(JQ799141)全序列和绵羊边界病毒(border disease virus, BDV)( KC963426)全序列进行综合比对,避开与BVDV、BDV的同源区,设计了两条特异性CSFV探针(P1位于 5′-UTR 112—210碱基序列,P3位于NS5B 基因11426—11573碱基序列)以及标记探针(P2:标记Cy3红色荧光);同时对猪看家基因β-actin(ACTB)(AK237086)的mRNA序列设计特异性探针作为内参(标记FITC绿色荧光)。CSFV RNA可视化原位杂交探针设计合成由猪瘟参考实验室与北京诺沃安泰生物科技有限公司合作完成。
CSFV 基本探针序列为:
P1: GGA CTA GCA AAC GGA GGG ACTA GCC GTA GTG GCG AGC TCC CTG GGT GGT CTA AGT CCT GAG TAC AGG ACA GTC GTC AAT AGT TCG ACG TGA GCA GGA GC
P2:TAT GAT TTA TTG CAA GCC CAG AGG TAC GGT ATA GAA GAC GGG ATA AAT ATC ACC AAA TCC T
P3: AGG TGG TCA GAC AAC ACT TCT AGT TAC ATG CCG GGG AGA AAT ACA ACC ACA ATC CTA GCT AAA ATG GCC ACA AGG TTA GAT TCC AGT GGT GAG AGG GGT ACC ATA GCA TAT GAG AAA GCA GTA GCA TTC AGC TTC CTG CTG ATG TAC T;
ACTB基本探针序列如下:
TTC CTT CCT GGG TAT GGA ATC CTG TGG CAT CCA CGA AAC TAC CTT CAA CTC AAT CAT GAA
1.2 试验材料
1.2.1 毒株背景 CSFV石门株(SM)、猪瘟兔化弱毒疫苗株(C-Strain)、SXDT/2001株、HeBHH1/95株、HeNBY1/96株均保存于中国兽医药品监察所国家猪瘟参考实验室,经猪瘟荧光抗体染色技术(fluorescent antibody technique,FAT)和电镜观察等鉴定为阳性[12-13];BVDV、PRV(猪伪狂犬病病毒)、PPV09/79株(猪细小病毒)由国家兽医菌种保藏中心提供,保存于中国兽医药品监察所国家猪瘟参考实验室;PCV-2(猪圆环病毒II型)由浙江大学周继勇教授惠赠。样品背景信息如表1。
1.2.2 细胞 PK15、ST、MBDK均保存于中国兽医药品监察所国家猪瘟参考实验室。
1.2.3 试剂和仪器 RNA 可视化原位杂交试剂(蛋白酶、洗液、DAPI等)购自诺沃安泰生物科技有限公司;猪瘟单克隆抗体:WH303,由英国 AHVLA Trevor Drew 教授提供;Lab-Tek II Chamber Slide购自美国Thermo Fisher Scientific公司。荧光共聚焦显微镜:Olympus,型号UltraVIEW VoX。

表1 CSFV RNA可视化原位杂交试验所用样品背景信息Table 1 The samples background information of CSFV RNA visualization ISH test
1.3 实验用参考毒株(HeBHH1/95 株)TCID50测定
在 24孔板中培养 PK15细胞,当细胞密度为70%—80%时,将 HeBHH1/95 株进行梯度稀释(从10-1到10-6),分别接种PK15细胞,每个稀释度4孔,每孔200µL。37℃ 5%CO2培养箱继续培养72h,采用FAT方法测定病毒的TCID50。
1.4 CSFV RNA可视化原位杂交技术的建立
在8孔胶室盖玻片中培养PK15细胞,当细胞密度为 70%—80%时,每孔接种 200µL滴度为 104.5TCID50/200 µL的HeBHH1/95株,37℃ 5%CO2培养箱继续培养72 h。小心吸掉培养基,PBS轻轻洗涤2次,加入4%甲醛溶液室温固定约30 min;PBS洗涤3次,加入Solution I,室温孵育5 min;PBS洗涤2次,加入蛋白酶处理一定时间,加入探针在 40℃经多步孵育并洗涤;最后经荧光探针标记并孵育30 min,加入工作浓度的DAPI 染料着染细胞核,PBS洗细胞2次,最后加入200 µL的PBS后,采用荧光共聚焦显微镜,控制工作温度为5—20℃,波长488/550 nm,在避光的环境中使用20×物镜进行成像。
1.4.1 反应条件优化 采用正交试验优化蛋白酶K浓度和甲醛固定时间,蛋白酶K浓度梯度设置为:1∶500、1∶1 000、1∶2 000、1∶4 000;甲醛固定时间分别设置为:30 min和60 min(表2)。综合考虑检测结果、荧光强度、重复性等因素,确定最优反应条件。

表2 蛋白酶K浓度和甲醛固定时间的优化Table 2 The optimization of proteinase K concentration and the formalin fix time
1.4.2 灵敏度试验 将上述HeBHH1/95毒株(104.5TCID50/200µL)进行10倍梯度稀释(从10-1到10-10),分别接种2组PK15细胞,每个稀释度3孔,每孔200 µL。37℃吸附1 h后补加维持液200 µL。置于37℃ 5%CO2培养箱中72 h后,分别采用CSFV RNA可视化原位杂交技术和CSFV FAT方法进行灵敏度比较试验。
1.4.3 特异性试验 分别取表 1中所列的不同基因亚型CSFV及其它相关病毒BVDV、PPV、PRV和PCV-2质控样本接种各自的靶细胞,每种病毒接种2 组PK15细胞,每组3孔。37℃ 5%CO2培养72 h后,取1组细胞用CSFV RNA可视化原位杂交技术进行检测,在荧光共聚焦显微镜下观察结果;另一组细胞采用每种病毒的相应抗体进行染色。
1.5 CSFV RNA定位及分布规律研究
取200 µL(103.67TCID50/ 200µL)CSFV强致病力SM毒株接种密度为70%—80%的 PK15细胞于8孔胶室盖玻片,病毒感染后0.5、1、3、6、8、10、14、18、24、36、48、72和96 h取样,每个时间点2个重复,采用CSFV RNA可视化原位杂交技术进行检测。
1.6 CSFV E2蛋白的定位及分布规律
取200 µL(103.67TCID50/ 200 µL)CSFV强致病力SM毒株接种密度为70%—80%的PK15细胞于细胞培养皿,同时设不接毒对照,病毒感染后每1 h收集细胞,固定,采用CSFV E2蛋白单克隆抗体(WH303)进行FAT染色。
2 结果
2.1 条件优化
通过对蛋白酶K工作浓度和甲醛固定时间的正交试验,综合考虑荧光强度、重复性等因素,结果显示当蛋白酶K工作浓度为1∶1 000,甲醛固定时间为30 min时,阴性对照背景信号最弱,细胞板中细胞损失最少,对细胞形态影响最小,有最高的信背比(阳性信号与背景干扰信号间的比值)。因此,确定蛋白酶K的工作浓度为1∶1 000,甲醛的固定时间为30 min。
2.2 灵敏度结果比较
通过对CSFV RNA可视化原位杂交方法和FAT方法对HeBHH1/95株的灵敏度比较试验,结果显示:CSFV RNA可视化原位杂交方法检测HeBHH1/95株的极限度为10-8(图1)。FAT方法检测的极限度为10-4.5(图2)。前者比后者高3.5个数量级。

图1 CSFV RNA可视化原位杂交方法灵敏度试验Fig.1 Sensibility test results of CSFV RNA visualization ISH method(200×)
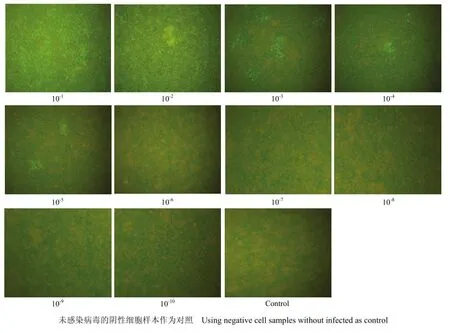
图2 CSFV FAT法灵敏度试验Fig.2 Sensibility test results of CSFV FAT method(100×)
2.3 特异性试验结果
通过对不同基因亚型的CSFV和相关病毒的特异性试验,结果表明CSFV RNA 可视化原位杂交方法能在感染细胞中特异性的检测到SM、C-Strain、SXDT2011、HeBHH1/95、HeNBY1/96的阳性信号(红色荧光,图3);而在BVDV、PPV、PRV、PCV-2的感染细胞则无阳性信号,且BVDV、PPV、PRV、PCV-2均在各自的靶细胞中正常生长(图 4)。说明CSFV RNA可视化原位杂交方法具有良好的特异性。

红色荧光代表CSFV RNA阳性信号,绿色荧光为β-actin信号; 未感染病毒的阴性细胞样本作为对照; “+”代表原位杂交阳性结果,“-”代表原位杂交阴性结果Red fluorescent represents positive siginal of CSFV RNA, green fluorescent represents β-actin siginal;Using negative cell samples without infected as control;“+”represcents ISH test result is positive,“-”represcents ISH test result is negative图3 CSFV RNA 可视化原位杂交方法特异性试验Fig.3 The specificity test results of CSFV RNA visualization ISH(400×)
2.4 CSFV RNA定位及分布动态研究
将按试验方案收集的 SM 株感染细胞用 CSFV RNA可视化原位杂交方法进行检测。结果显示:0.5 hpi就可在胞核与胞浆内均能检测到病毒RNA,0.5—6 hpi病毒RNA主要存在于胞核内并在胞核内聚集增多, 8 hpi胞浆内RNA含量有增多的趋势,直到 18 hpi胞浆内的RNA含量逐渐增多,胞核内RNA逐渐减少;24 hpi病毒RNA主要定位在细胞核周围;36—72 hpi病毒RNA在核外大量积累增多,72 hpi胞浆内RNA含量达到最大值,96 hpi胞浆内RNA总量有所下降(图5)。
2.5 CSFV E2蛋白染色
将按试验方案收集的 SM株感染细胞用 CSFV FAT法进行检测。结果显示,8 hpi可在少数细胞的胞浆内检测到荧光,而在此前检测不到荧光,10—24 hpi被荧光染色的阳性细胞数量一直较少; 36 hpi随着感染时间的延长,感染细胞的数量不断增多,72 hpi阳性细胞数量达到最大值;96 hpi阳性细胞数量有所下降且荧光强度变弱。(图6)
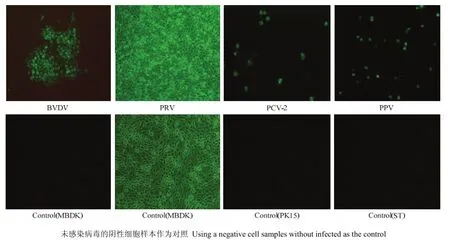
图4 BVDV、PRV、PCV-2和PPV在细胞中正常生长Fig. 4 Regular growth picture of BVDV, PRV, PCV-2 and PPV in cells (100×)
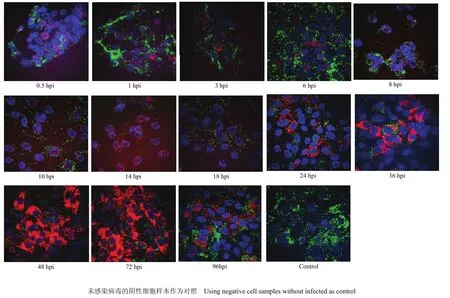
图5 SM株RNA在PK15细胞上的分布动态Fig.5 The distribution dynamics results of SM strain RNA in PK15 cells (200×)
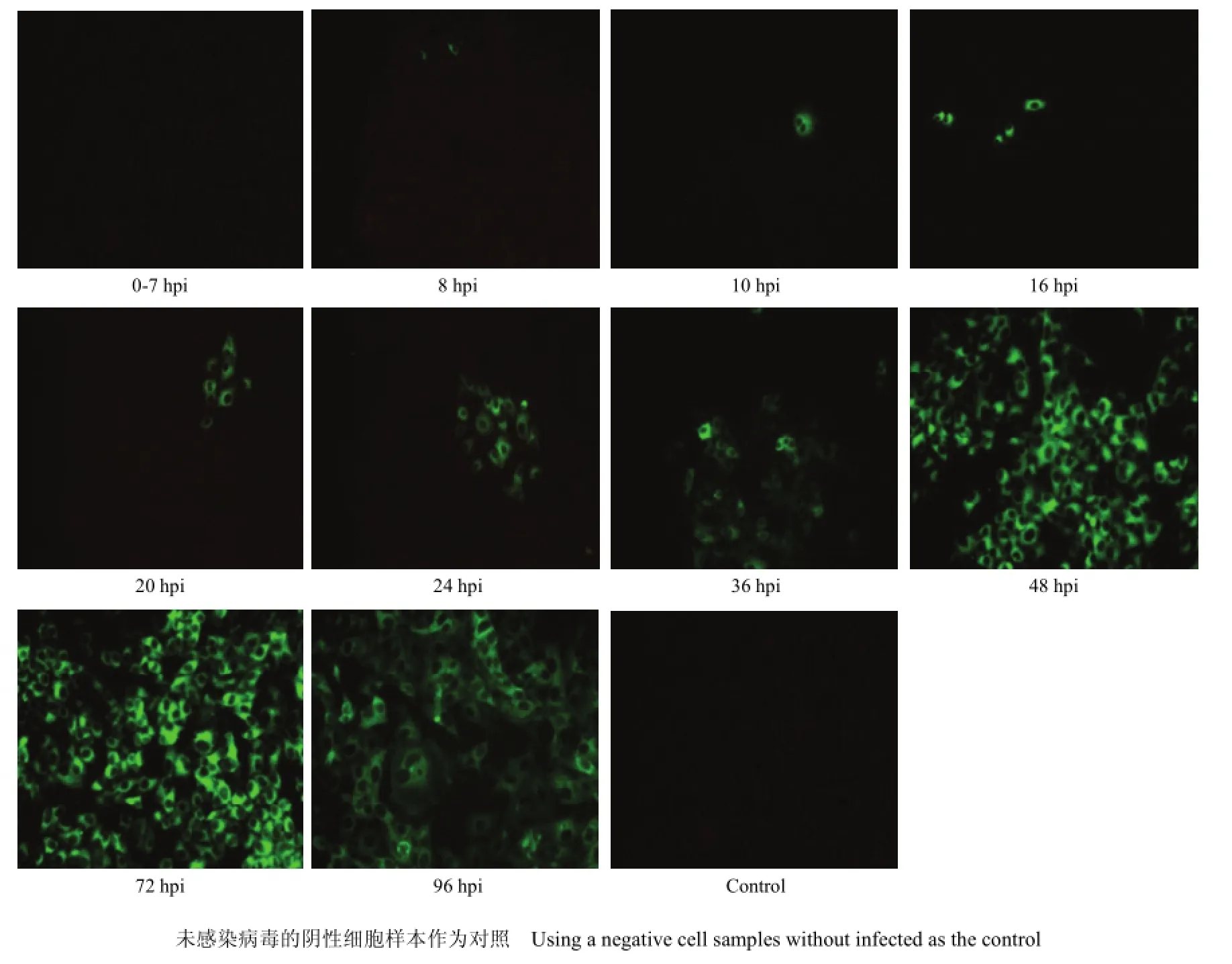
图6 PK15细胞感染SM株后不同时间FAT检测Fig.6 FAT test results of SM strain in infected PK15 cells (100×)
3 讨论
3.1 RNA可视化原位杂交检测技术
RNA可视化原位杂交检测技术的原理是通过设计目的基因“Z”型结构的特异探针,利用“Z”型结构探针的下半部分与经蛋白酶消化后暴露的靶 RNA杂交,之后预杂交液与“Z”型结构探针的上半部分结合,杂交液再与预杂交液结合,通过3次特异性的序列杂交出现信号扩增现象。一个装配完整的信号扩增树有400个探针标签的结合位点,当探针上的所有目标寡核苷酸片段都结合到靶 RNA转录本上时,一个转录样本便可产生8 000倍的放大效应。接着加入碱性磷酸酶,该酶会破坏底物形成红点沉淀表明靶RNA存在,再通过荧光共聚焦显微镜把目标 RNA显影出来。该技术可在不进行RNA分离、反转录和PCR情况下,仅需要样品制备、杂交、检测3个步骤即可完成对某个细胞中特定RNA拷贝数情况进行定位和检测。而传统的电子显微镜只能检测成熟病毒粒子而无法定位病毒RNA,且灵敏性低;荧光定量PCR技术虽能对CSFV RNA总量进行检测,但无法对RNA进行细胞内定位。RNA可视化原位杂交检测技术恰恰弥补了上述缺点,通过收集病毒RNA红色荧光和β-actin看家基因绿色荧光,同时将细胞核染成蓝色,在荧光共聚焦显微镜下进行病毒RNA和细胞核相对位置的观察。荧光共聚焦显微镜由于是激光和荧光 的共聚焦成像以及 Pinhole的应用阻止了焦点以外的干扰衍射光和散射光,使其具有 16bit的高分辨率。多通道采集图像的功能很方便地使共聚焦具有同时获取不同光谱段的荧光信号的功能。并通过Volocity Demo软件处理得到层叠效应图像,克服了传统荧光原位杂交法(fluorescence in situ hybridization, FISH)信号集团强度低、灵敏度差的劣势,其灵敏度显著高于FAT法及荧光定量PCR法。2013年SEIMON等用RNA原位杂交检测技术在濒临灭绝的东北虎脑组织中检测到犬瘟热病毒RNA的存在[15];2012年HURTADO等也采用 RNA原位杂交技术检测到了小鼠舌上皮细胞中 Y受体的分布[16]。以上学者都采用不同的RNA原位杂交技术对组织细胞中的特定病毒进行了定位和检测,说明RNA原位杂交方法具有很好的可行性和特异性,能准确的对体外培养细胞中的RNA进行定位和检测。
3.2 CSFV RNA在感染细胞中的定位及分布
本研究首次建立了CSFV RNA可视化原位杂交检测技术,其灵敏度比FAT方法高3.5个数量级,能特异性的识别中国目前流行的CSFV 1.1、2.1、2.2、2.3基因亚型,且与BVDV、PPV、PRV和PCV-2等常见的猪源病毒 RNA无交叉反应,具有灵敏度高、特异性强、直接在细胞中进行定位观察等特点。通过对强致病力CSFV感染细胞后病毒RNA进行定位研究,表明CSFV接种其易感细胞后迅速侵染细胞,0.5 hpi便可在胞浆与胞核内同时检测到病毒 RNA的存在,随着感染时间的延长,胞浆内的RNA逐渐增多,24 hpi病毒 RNA主要集中在胞核周围,36—72 hpi RNA在胞浆内大量复制增多,72 hpi达到最大值。此次研究首次在体外细胞中对 CSFV RNA进行了定位和分布动态的研究。
众所周知CSFV感染细胞后,病毒RNA主要是在胞浆中复制。而本研究观察到在感染早期,胞核内也检测到病毒 RNA的存在,所以猜测这可能与细胞核内的某种蛋白有关。有文献报道核不均一核糖核蛋白(heterogeneous nuclear ribonucleoprotein,hnRNPs)是存在于核浆中的一类核蛋白,参与病毒 RNA的合成与加工。例如丙型肝炎病毒(HCV)感染宿主细胞时,hnRNp AI参与病毒的复制过程,敲除宿主细胞的hnRNpAI会降低HCV RNA的复制[17];在CSFV感染诱导PK-15细胞蛋白质组研究中也发现细胞中有4种hnRNPs的表达水平发生变化[18],表明CSFV的复制同样需要宿主细胞hnRNPs参与病毒RNA的合成与加工。另外病毒感染宿主细胞后,病毒RNA进入细胞核的现象可能也与CSFV某些蛋白的核定位有关。有研究表明正链和负链 RNA 病毒的蛋白定位到细胞核或者亚细胞结构,特别是核仁结构,有利于病毒的复制和转录[19];也有报道CSFV C蛋白与宿主细胞核仁素蛋白(nucleolin,NCL)在核仁中存在共定位现象。当NCL被抑制后CSFV的复制能力下降[20],CSFV通过作用于细胞核,为病毒的复制或调控提供便利[21]。近几年研究发现黄病毒 C 蛋白的核定位与病毒的复制有关[22-23],C蛋白在合成后几分钟即与病毒基因组 RNA相结合[24],C蛋白的 N端与基因组RNA相结合,C端参与核衣壳的形成。因此CSFV RNA很可能在 C蛋白进行核定位时被 C蛋白携带进入胞核。因此推测,在CSFV感染早期(0.5—10 h),病毒RNA很可能也是被C蛋白一同携带进入细胞核,但是 CSFV RNA是如何进入细胞核并且在胞核中存在还有待深入研究。
为了佐证 CSFV RNA在体外细胞中的定位与分布,同时采用FAT方法对SM株病毒蛋白在PK15细胞中进行增殖动态比较研究,结果显示36—72 hpi病毒 RNA在核外大量聚集,与此同时胞浆内病毒蛋白的检出量也大量增加;72 hpi胞核外的病毒RNA含量达到最大值,而此时病毒蛋白阳性细胞数量达峰值且荧光亮度最强;96 hpi胞核外RNA的含量有所降低,而病毒蛋白阳性细胞数量有所下降且荧光强度变弱。E2蛋白在细胞浆内的聚集数量与细胞浆中RNA含量成正相关。
4 结论
首次建立了可以对病毒 RNA进行细胞内定位的CSFV RNA可视化原位杂交检测技术,证实该技术具有灵敏度高、特异性强等特点。采用该技术对CSFV强致病力SM毒株RNA在靶细胞上进行定位和分布研究,发现病毒RNA吸附和进入靶细胞的时间早于 0.5 hpi,胞核内的RNA含量明显高于胞浆内。随着感染时间的延长,10—18 hpi胞核内RNA量逐渐减少,胞浆内 RNA逐渐增多,24 hpi病毒RNA主要定位在胞浆,观测到CSFV RNA在细胞核内的生活史。
References
[1] 王琴,涂长春. 猪瘟. 北京:中国农业出版社,2015 : 2-4. WANG Q, TU C C. Classical Swine Fever. Beijing:China Agricul True Press, 2015: 2-4. (in Chinese)
[2] EDWARDS S, FUKUSHO A, LEFEVRE P C, LIPOWSKI A,PEJSAK Z, ROEHE P, WESTERGAARD J. Classical swine fever:the global situation . Veterinary Microbiology, 2000, 73(2):103-119.
[3] MEYERS G, THIEL H J, RUMENAPF T. Classical swine fever virus:recovery of infectious viruses from cDNA constructs and generation of recombinant cytopathogenic defective interfering particles. Journal of Virology, 1996, 70(3): 1588-1595.
[4] LIU J, FAN X Z, WANG Q, XU L, ZHAO Q Z, HUANG W, ZHOU YC, TANG B, CHEN L, ZOU X Q, SHA S, ZHU Y Y. Dynamic distribution and tissue tropism of classical swine fever virus in experimentally infected pigs. Journal of Virology, 2011(8): 201.
[5] BABIJ C, ZHANG Y, KURZEJA R J, MUNZLI A, SHEHABEDIN A,FENANDO M, QUON K, KASSNER P D, RUEFLI-BRASSE A A,WATSON V J, FAJARDO F, JACKSON A, ZONDLO J, SUN Y,ELLISON A R, PLEWA C A, MIGUEL T S, ROBINSON J,MCCARTER J, SCHWANDNER R, JUDD T, CARNAHAN J,DUSSAULT I. STK33 kinase activity is nonessential in KRAS-dependent cancer cells. Cancer Research, 2011, 71(17):5818-5826.
[6] CHAE B J, BAE J S, YIM H W, LEE A, SONG B J, JEON H M,CHUN M H, JUNG S S. Measurement of ER and PR status in breast cancer using the QuantiGene2.0 assay. Pathology, 2011, 43(3):248-253.
[7] HONKAVUORI K S, SHIVAPRASAD H L, BRIESE T, STREET C,HIRSCHBERG D L, HUTCHISON S K, LIPKIN W L. Novel picornavirus in Turkey poults with hepatitis, California, USA. Emerging Infectious Diseases, 2011, 17(3): 480.
[8] NANBO A, WATANABE S, HALFMANN P, KAWAOKA Y. The spatio-temporal distribution dynamics of Ebola virus proteins and RNA in infected cells. Scientific Reports, 2013(3):1206.
[9] FEELEY E M, SIMS J S, JOHN S P, CHIN C R, PERTEL T, CHEN L M, GAIHA G D, RYAN B J, DONIS R S, ELLEDGE S J, BRASS A L. IFITM3 inhibits influenza A virus infection by preventing cytosolic entry. PLoS Pathogens, 2011, 7(10):e1002337.
[10] TING D T, LIPSON D, PAUL S, BRANNIGAN B W,AKHAVANFARD S, COFFMAN E J, CONTINO G, DESHPANDE V,JOHN A, LETOVSKY S, RIVERA M N, BARDEESY N,MAHESWARAN S, HABER D A. Aberrant overexpression of satellite repeats in pancreatic and other epithelial cancers. Science,2011, 331(6017): 593-596.
[11] SEIMON T A, MCALOOSE D, RAPHAEL B, HONKAVUORI K S,CHANG T, HIRSCHBERG D L, LIPKIN W L. A novel herpesvirus in 3 species of pheasants: Mountain peacock pheasant (Polyplectron inopinatum), malayan peacock pheasant (Polyplectron malacense),and congo peafowl (Afropavo congensis). Veterinary Pathology, 2011,49(3): 482-491.
[12] 王在时,丘惠深,郎洪武. 猪瘟病毒野外分离物的研究,I猪瘟野毒的分离及初步鉴定. 中国兽药监察所研究报告汇编. 北京: 1999,14: 35-38. WANG Z S,QIU H S, LANG H W. The research of wild isolate of classical swine fever virus, I isolate and identify of classical swine wild strain. Research Report Complie of China Institute of Veterinary Drug Control, Beijing: 1999, 14:35-38. (in Chinese)
[13] 丘惠深,郎洪武,王在时. 猪瘟病毒野外分离物的研究,II猪瘟野毒的纯化及电镜观察. 中国兽药监察所研究报告汇编. 北京: 1999,14: 39-42. QIU H S, LANG H W, WANG Z S. The research of wild isolate of classical swine fever virus, II purification and electro microcopy observation. Research Report CVomplie of China Institute of Veterinary Drug Control, Beijing:1999,14:39-42. (in Chinese)
[14] 刘俊, 王琴, 范学政, 徐璐,赵启祖,黄伟,汤波,沙莎,周远成,陈蕾,邹兴启. 猪瘟病毒野毒株 TaqMan-MGB 荧光定量 PCR 鉴别方法的建立与应用. 中国农业科学, 2009, 42(12):4366-4371. LIU J, WANG Q, FAN X Z, XU L, ZHAO Q Z, HUANG W, TANG B,SHA S, ZHOU Y C, CHEN L, ZOU Q Q. Differentation of wild-type viruses and HCLV vaccine of classical swine fever virus by one-step fluorescent Quantitative PCR using Taq-MGB probe technology. Scientia Agricultura Sinica, 2009, 42(12):4366-4371. (in Chinese)
[15] SEIMON T A, MIQUELLE D G, CHANG T Y, NEWTON A L,KOROTKOVA I, IVANCHUK G, LYUBCHENKO E, TUPIKOV A,SLABE E, MCALOOSE D. Canine distemper virus: an emerging disease in wild endangered amur tigers (Panthera tigris altaica). Mbio,2013, 4(4):16-16.
[16] HURTADO M D, ACOSTA A, RIVEROS P P, BAUM B J,UKHANOV K, BROWN A R, DOTSON C D, HERZOQ H,ZOLOTUKHIN S. Distribution of Y-receptors in murine lingual epithelia. PLoS One, 2012, 7(9):e46358
[17] KIM C S,SEOL S K,SONG O K, PARK J H, JANG S K. An RNA—binding protein,hnRNP A1,and a scaffold protein,sptin 6,facilitate hepatitis C virus replication.Journal of Virology, 2007,81(8):3852-3865.
[18] 孙金福, 史子学, 郭焕成, 涂长春. 猪瘟病毒感染诱导 PK-15 细胞蛋白质组的改变. 中国畜牧兽医学会, 2008年学术年会暨第六届全国畜牧兽医青年科技工作者学术研讨会论文集, 2008. SUN J F,SHI Z X,GUO H C,TU C C. Swine fever virus infection induced PK 15 cells proteome changes. Chinese Association of Animal Science and Veterinary Medicine(CAAV),In 2008 annual meeting and sixth national CAAV youth academic semina conference proceeding, 2008. (in Chinese)
[19] RUBBI C P, MILNER J, MILNER J.Disruption of the nucleolus mediates stabilization of p53 in response to DNA damage and other stresses. EMBO Journal, 2003, 22(22): 6068-6077.
[20] 张鑫, 时洪艳, 徐佳. 猪瘟病毒衣壳蛋白与宿主细胞核仁素蛋白相互作用. 中国预防兽医学报, 2015, 37(6): 430-433.ZHANG X, SHI H Y, XU J. Characteization of interaction between the capsid protein of classical swine fever virus and cellular nucleolin. Chinese Journal of Preventive Veterinary Medicine,2015,37(6):430-433. (in Chinese)
[21] 刘宁, 时洪艳, 陈建飞, 冯力, 张鑫, 胡桂学. 猪瘟病毒衣壳蛋白的细胞核定位研究. 中国预防兽医学报, 2014, 36(1): 71-72. LIU N, SHI H Y, CHEN J F, FENG L, ZHANG X, HU G X. Nucleus localization of classical swine fever virus capsid proteins in cells. Chinese Journal of Preventive Veterinary Medicine, 2014, 36(1):71-72. (in Chinese)
[22] MORI Y, OKABAYASHI T, YAMASHITA T, ZHAO Z, WAKITA T,YASUI K, HASEBE F, TADANO M, KONISHI E, MATSUURA Y. Nuclear localization of Japanese encephalitis virus core protein enhances viral replication. Journal of Virology, 2005, 79(6):3448-3458.
[23] TSUDA Y, MORI Y, ABE T, YAMASHITA T, OKAMOTO T,ICHIMURA T, MORIISHI, MATSUURA Y. Nucleolar protein B23 interacts with Japanese encephalitis virus core protein and participates in viral replication. Microbiology and Immunology, 2006, 50(3):225-234.
[24] 涂亦娴. 猪瘟病毒 E2 基因的克隆及其 DNA 疫苗免疫效果的研究[D]. 乌鲁木齐: 新疆大学, 2005. TU Y X. Research of CSFV E2 genic clone and immune effect of DNA vaccine[D]. Urumqi: Xinjiang University, 2005. (in Chinese)
(责任编辑 林鉴非)
Study of Location and Distribution of Classical Swine Fever Virus RNA in PK15 Cells by Visualization in Situ Hybridization Technology
ZHANG Yu-jie, ZHAO Yan, XU Lu, ZHANG Qian-yi, CHEN Kai, SUN Yong-fang, ZOU Xing-qi,ZHU Yuan-yuan, ZHAO Qi-zu, NING Yi-bao, WANG Qin
(China Institute of Veterinary Drug Control/National Classical Swine Fever Reference Lab, Beijing 100081)
Abstract:【Objective】In order to make researches on the RNA location and distribution of Classical Swine Fever Virus(CSFV)in infected PK15 cells, a rapid, accurate and sensitive in situ hybridization (ISH) technology was established. 【Method】After comparison with the complete sequences of CSFV, BVDV and BDV to avoid the homology regions, a set of specific probes of CSFV RNA andβ-actin were designed and synthesized. A reference strain CSFV (HeBHH1/95) was used to optimize the ISH technology through comparison several parameters such as fluorescence intensity, repeatability, protease K concentration and formalin fixation time. After these parameters were determined, an ISH technology was established. Fluorescent antibody test (FAT)was used to compare the sensitivity with the ISH technology. All the sub genotypes of CSFV (sub genotype 1.1, 2.1,2.2 and 2.3)which are present in China and other normal pig infectious virus (BVDV, PPV, PRV,PCV-2) were used to detect specificity of this ISH technology. A high virulent strain of CSFV (SM) was inoculated in PK15 cells. Infected cells were sampled at 0.5 hours post inoculation (hpi)、1, 3, 6, 8, 10, 14, 18, 24, 36, 48, 72, and 96hpi. Then FAT was performed in parallel to detect the expression and location of CSFV E2 protein. 【Result】CSFV RNA were detected in infected PK15 cells by using the ISH technology. The optimal concentration of protease K was 1:1 000 and the optimal time of formalin fixation was 30 minutes. The minimum of detection is 10-8/200µL which is 3.5 orders of magnitude higher than FAT. The specific tests showed that the ISH technology could react with sub genotypes 1.1, 2.1,2.2 and 2.3 of CSFV in China and has no cross reaction with BVDV, PPV, PRV,PCV-2 viruses. CSFV RNA ISH test results showed that CSFV RNA were firstly detected in nucleus at 0.5 hpi and gathered in nucleus from 0.5hpi to 6hpi; at 10hpi,there were more CSFV RNA gathered in cytolymph than before and less CSFV RNA were detected than before in nucleus;at 24hpi,CSFV RNA mainly gathered in cytolymph around nucleus; at 36hpi, more and more CSFV RNA gathered in cytolymph and got maximum at 72hpi; at 96hpi, the amount of CSFV RNA declined. The results of FAT showed that little E2 protein was detected in cytolymph at 8hpi. From 10hpi to 24 hpi, only few cells were detected positive. After 36hpi, the expression of E2 protein increased gradually and got to the maximum at 72hpi. But after 96hpi, the amount of E2 protein declined. The trend of E2 protein expression was in accord with CSFV RNA. 【Conclusion】 A visualization in situ hybridization technology was firstly established to detect CSFV RNA. The location and distribution of CSFV RNA was studied by using this technology. The results proved that CSFV RNA enters cell early than 0.5hpi and CSFV RNA has ever existed in nucleus.
Key words:Classical Swine Fever Virus (CSFV); location; distribution; RNA in situ hybridization (ISH); Fluorescent antibody test (FAT)
收稿日期:2015-12-31;接受日期:2016-03-28
基金项目:国家自然科学基金(31372434)
