家兔不同发育阶段和组织中内参基因的稳定性分析
2016-07-13杨晓农曾婉秋
陈 瑞,杨晓农,曾婉秋,文 娟
(西南民族大学生命科学与技术学院,成都 610041)
家兔不同发育阶段和组织中内参基因的稳定性分析
陈瑞,杨晓农*,曾婉秋,文娟
(西南民族大学生命科学与技术学院,成都 610041)
摘要:本研究旨在筛选出新西兰白兔不同时期不同器官中表达最稳定的内参基因。以胚胎期(E28.5)、幼龄期(10d)和成年期(A)3个阶段的新西兰白兔的肾、心和肝3种组织作为研究对象,按照RT-qPCR试验最低要求试验信息量(MIQE)的指导,运用RT-qPCR和在线内参基因稳定性评价工具RefFinder,比较6个常用的内参基因(GAPDH、HPRT1、18S rRNA、B2M、Sdha、β-actin)mRNA的表达稳定性。结果表明,新西兰白兔的肝和肾组织中的6个内参基因在3个发育阶段最稳定的3个内参基因依次是β-actin、HPRT1、GAPDH,而在心组织中最稳定的3个内参基因依次是β-actin、HPRT1、Sdha。在新西兰白兔胚胎期的3种组织中,最稳定的3个内参基因依次是GAPDH、β-actin、HPRT1,而在幼龄期和成年期最稳定的3个内参基因依次是HPRT1、GAPDH、β-actin。对新西兰白兔的3个发育阶段和3种组织的内参基因稳定性结果分析表明,最稳定的3个内参基因依次是β-actin、HPRT1、GAPDH。本研究证实,新西兰白兔不同的发育时期和组织中,最稳定的内参基因不同。推荐在进行新西兰白兔的相关试验时,可以选择上述表达最稳定的至少3个内参基因的几何平均数作为标准化相关RT-qPCR数据的指标,以确保试验组个体间的差异最小,和小变量的准确数据分析。
关键词:内参基因;表达稳定性;MIQE;RefFinder
实时定量PCR基于荧光信号,可以用于定量基因转录水平的研究,是用来了解基因表达量和功能的技术。它是研究基因差异表达最常用的技术,具有特异、灵敏、快速和高通量等优点,在生命科学研究中被广泛应用。在某一点cDNA扩增产生的可接受荧光信号会转化成cyclethreshold(Ct),take-offpoint(TOP)或者quantificationcyclenumber(Cq)[1-2]。在相对定量检测中,对来自不同样本中特定基因的mRNA表达量水平进行比较分析时,理想的条件是用于制备RNA的各样品起始细胞数相同,RNA的提取效率及反转录效率相同。但实际上这些条件不可能同时得到满足,许多因素可能导致测量不一致,例如测量的组织细胞不同的数量和类型、RNA提取效率、mRNA处理技术、mRNA完整性、反转录的方法和效率、分析检测方法等[3-4]。为了确定结果的准确性、特异性和标准化,必须控制这些因素[5]。
内参基因是一种维持细胞最低限度功能不可缺少的基因。理想的内参基因在不同类型细胞和组织中的表达应无显著差异,且不受试验和临床条件的影响。由于内参基因的表达具有物种及组织特异性,所以其表达的稳定性是相对的。为了获得有意义的试验数据,研究者会根据特殊的组织及试验条件来选择合适的内参基因[6-7]。2009年,RT-qPCR试验最低要求试验信息量(MinimumInformationforPublicationofQuantitativeReal-timePCRExperiments,MIQE)的发表给所有RT-qPCR数据报道确定了一个准则[8-9]。这个指南的应用确保了发表的RT-qPCR数据更为详细(包括关键参数,例如RNA完整度、RT-qPCR质量控制和反应效率),同时确定了候选内参基因的重要性。
本研究严格按照MIQE的指导,检测在胚胎期、幼龄期和成年期3个阶段新西兰白兔的3个器官(肾、心、肝)中6个常用的内参基因(GAPDH、HPRT1、18S rRNA、Beta-2-microglobulin(B2M)、Sdha、β-actin)的表达稳定性[10]。使用最新的内参基因稳定性在线评价软件RefFinder(http://www.leonxie.com/referencegene.php) 确定最适合的内参基因,即受年龄发育状态影响和不同组织影响最小的基因[11]。RefFinder是基于4种常用算法(geNorm、BestKeeper、NormFinder和comparativedelta-CT)的几何平均数的指定权重法来排列基因稳定性的[12-15]。
1材料与方法
1.1试验材料
试验所有组织来自9只新西兰白兔。分为胚胎期(E28.5d)、幼龄期(出生后10d)和成年期(A)3组,每组3只。每只采集肾、心、肝部组织,置于-80 ℃低温冰箱保存备用。
1.2总RNA提取和cDNA合成
肾、心、肝50mg剪碎后加入1mLRNAisoReagent(TaKaRa,日本),进行总RNA提取。获得的RNA用50μLDEPC水稀释,-80 ℃保存。使用分光光度计(BioSpec-nano,日本)测定RNA的浓度和质量。并用琼脂糖凝胶电泳测定28S/18S核糖体RNA的比值,对RNA完整度进行评估。所有RNA样品的OD260nm/OD280nm比值都为1.7~2.0,而且RNA的完整度较高。cDNA的合成按RTReagents试剂盒(TaKaRa,日本)说明书进行。获得的cDNA10倍稀释,置于-80 ℃保存。
1.3引物的特异性鉴定及阳性标准品的制备
引物[16](表 1)由生工生物工程有限公司合成(上海,中国)。使用cDNA作为模板进行PCR,扩增6个内参基因的目的片段,反应条件:95 ℃ 5min; 95 ℃变性40s,60 ℃退火40s,72 ℃延伸30s;72 ℃总延伸10min;16 ℃结束反应。循环数为30个。总PCR反应体系为25μL:Mix(TaKaRa,日本)12.5μL,上下游引物各1μL(10μmol·mL-1),cDNA2μL,ddH2O8.5μL。PCR产物经2%琼脂糖凝胶电泳鉴定,100V、80mA电泳30min。使用DNA胶回收试剂盒(Axygen,美国)回收目的片段,溶于20μLddH2O。与pMD19-TVector质粒载体(TaKaRa,日本)连接。连接体系为SolutionⅠ 5μL,DNA4μL,PMD19-TVector1μL,时间为1h。转化大肠杆菌DH5α(TaKaRa,日本)。提取质粒(Axygen,美国),送生工生物工程有限公司进行测序验证(上海,中国)。质粒浓度测定基于全质粒,用分光光度计测量浓度(BioSpec-nano,日本),作为阳性标准品。10倍梯度稀释阳性质粒,采用10-2、10-3、10-4、10-5、10-6、10-7、10-87个浓度梯度构建标准曲线。

表1 扩增6个候选内参基因所用的引物
1.4RT-qPCR扩增
使用CFX96(BioRad,美国)荧光定量PCR仪进行两步法扩增。每个阶段的家兔组织都有3个生物学重复,每个样品中各内参基因都进行3次RT-qPCR扩增,分别得出各样本的各内参基因Ct值。RT-qPCR前先使用梯度PCR确定最佳退火温度。10倍梯度稀释阳性标准品,确定各基因的扩增率和最低检测下限。RT-qPCR反应使用20μL反应体系:SYBRGreenⅡ(TaKaRa,日本) 10μL,上下游引物各0.4μL,cDNA2μL,ddH2O7.2μL。RT-qPCR反应条件:95 ℃ 3min;95 ℃ 15s;60 ℃ 30s;循环数为40个。熔解曲线为55~95 ℃,每15s增加0.5 ℃,用以确定扩增特异性。
1.5表达数据分析
通过CFXManager软件获得各样本的原始数据,输入Excel进一步分析。用RefFinder分析所有基因表达的稳定性。
2结果
2.1内参基因引物的特异性
对GAPDH、HPRT1、18S rRNA、B2M、Sdha和β-actin 6个基因PCR的扩增产物进行2%的琼脂糖电泳(图1)。6对引物特异性均较好,无引物二聚体,胶回收产物及克隆测序结果均与目的基因序列完全一致,能够满足试验需要。
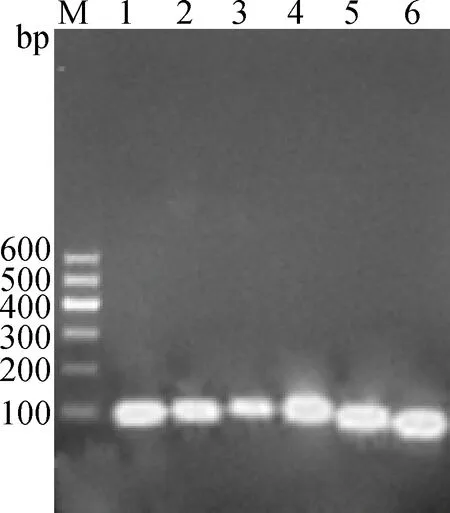
M.Marker;1.GAPDH;2.HPRT1;3.18S rRNA;4.B2M;5. Sdha;6.β-actin图1 目的片段的琼脂糖凝胶电泳结果Fig.1 Agarose gel electrophoresis of target products
2.2阳性标准品的制备及标准曲线的构建
采用分光光度计测定质粒OD260nm/OD280nm,计算质粒浓度和检测下限(表1)。将10倍梯度稀释的标准品作为模板,进行RT-qPCR扩增。熔解曲线分析结果显示,RT-qPCR产物只有一个特异峰,无非特异性扩增峰和无引物二聚体。GAPDH、HPRT1、18S rRNA、B2M、Sdha和β-actin 6个内参基因的熔解温度分别为(84.75±0.25)、(82.25±0.25)、86.50、(82.25±0.25)、84.00、(84.75±0.25)℃。以获得的Ct值为纵坐标,拷贝数对数值LogCO为横坐标做线性回归曲线。由图2和图3可知,各基因在系列稀释梯度内有良好的线性关系。
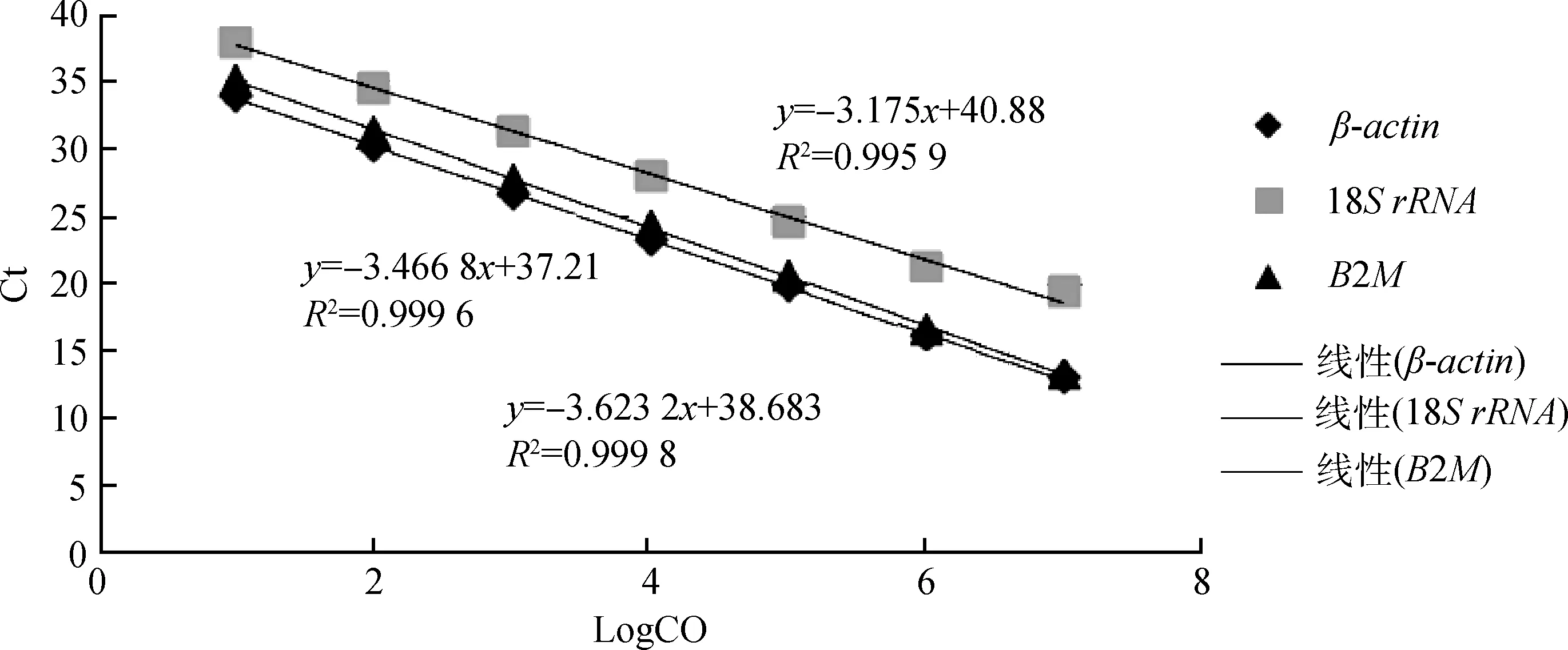
图2 18S rRNA、B2M和β-actin的标准曲线Fig.2 Standard curves of 18S rRNA,B2M and β-actin

图3 GAPDH、HPRT1和Sdha的标准曲线Fig.3 Standard curves of GAPDH,HPRT1 and Sdha
2.3内参基因在各组织不同时期的表达稳定性
通过对心、肝、肺组织中6个内参基因在胚胎期、幼龄期、成年期的表达稳定性分析发现,肾和肝中最稳定的3个内参基因依次是β-actin、HPRT1、GAPDH(图4),而在心组织中最稳定的3个内参基因依次β-actin、HPRT1、Sdha。在心和肝中,18S rRNA是表达最不稳定的内参基因,但在肾中,表达最不稳定的内参基因是B2M。
2.4内参基因在各时期不同组织的表达稳定性
在家兔胚胎期的心、肝和肺,最稳定的3个内参基因依次是GAPDH、β-actin、HPRT1(图5),而在幼龄期和成年期最稳定的3个内参基因依次是HPRT1、GAPDH、β-actin。B2M是胚胎期和幼龄期表达最不稳定的内参基因,18S rRNA是成年期表达最不稳定的内参基因。
2.5内参基因在不同时期不同组织的表达稳定性
分析家兔3个发育阶段和3种组织的内参基因稳定性结果表明,表达最稳定的3个内参基因依次是β-actin、HPRT1、GAPDH,而18S rRNA显示最差的稳定性(图6)。
3讨论
已经证实,由于内参基因标准化过程中的夸大和缩小效应,微小的差异很难通过RT-qPCR来定量[12]。内参基因的稳定表达对RT-qPCR的标准化是很重要的,但目前并没有发现一个内参基因在所有细胞、组织、时期或在不同的试验处理下都能够稳定地表达[17]。在某个组织或时期表达最稳定的内参基因在其他组织或时期的表达可能相对不稳定。其原因可能是动物不同组织在不同时期具有不同的功能和代谢特征,内参基因作为维持机体最低限度功能的基因其表达存在差异。为了获得有意义的试验数据,研究者应根据各自特殊的组织及试验条件来选择合适的内参基因。

a.家兔心的合适内参基因分析;b.家兔肾的合适内参基因分析;c.家兔肝的合适内参基因分析a.Expression stability of reference genes in different development periods of Oryctolagus cuniculus heart;b.Expression stability of reference genes in different development periods of Oryctolagus cuniculus kidney;c.Expression stability of reference genes in different development periods of Oryctolagus cuniculus liver图4 内参基因在各组织不同时期的表达稳定性Fig.4 Expression stability of reference genes in different development periods of Oryctolagus cuniculus tissues
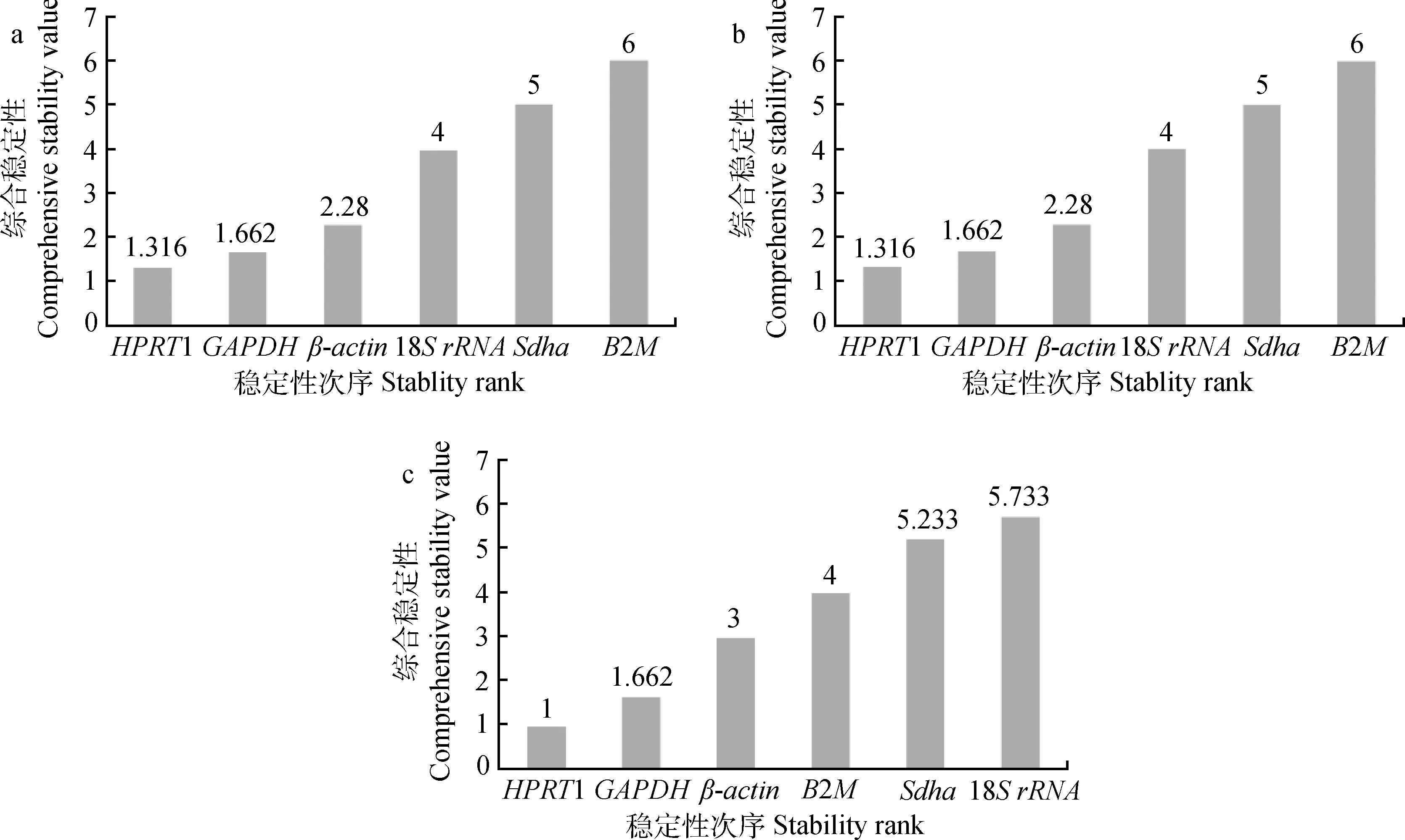
a.家兔胚胎期合适内参基因分析;b.家兔幼龄期合适内参基因分析;c.家兔成年期合适内参基因分析a.Expression stability of reference genes in Oryctolagus cuniculus tissues of embryonic period;b.Expression stability of reference genes in Oryctolagus cuniculus tissues of juvenile period;c.Expression stability of reference genes in Oryctolagus cuniculus tissues of juvenile adult period图5 内参基因各时期不同组织的表达稳定性Fig.5 Expression stability of reference genes in Oryctolagus cuniculus tissues in different development periods
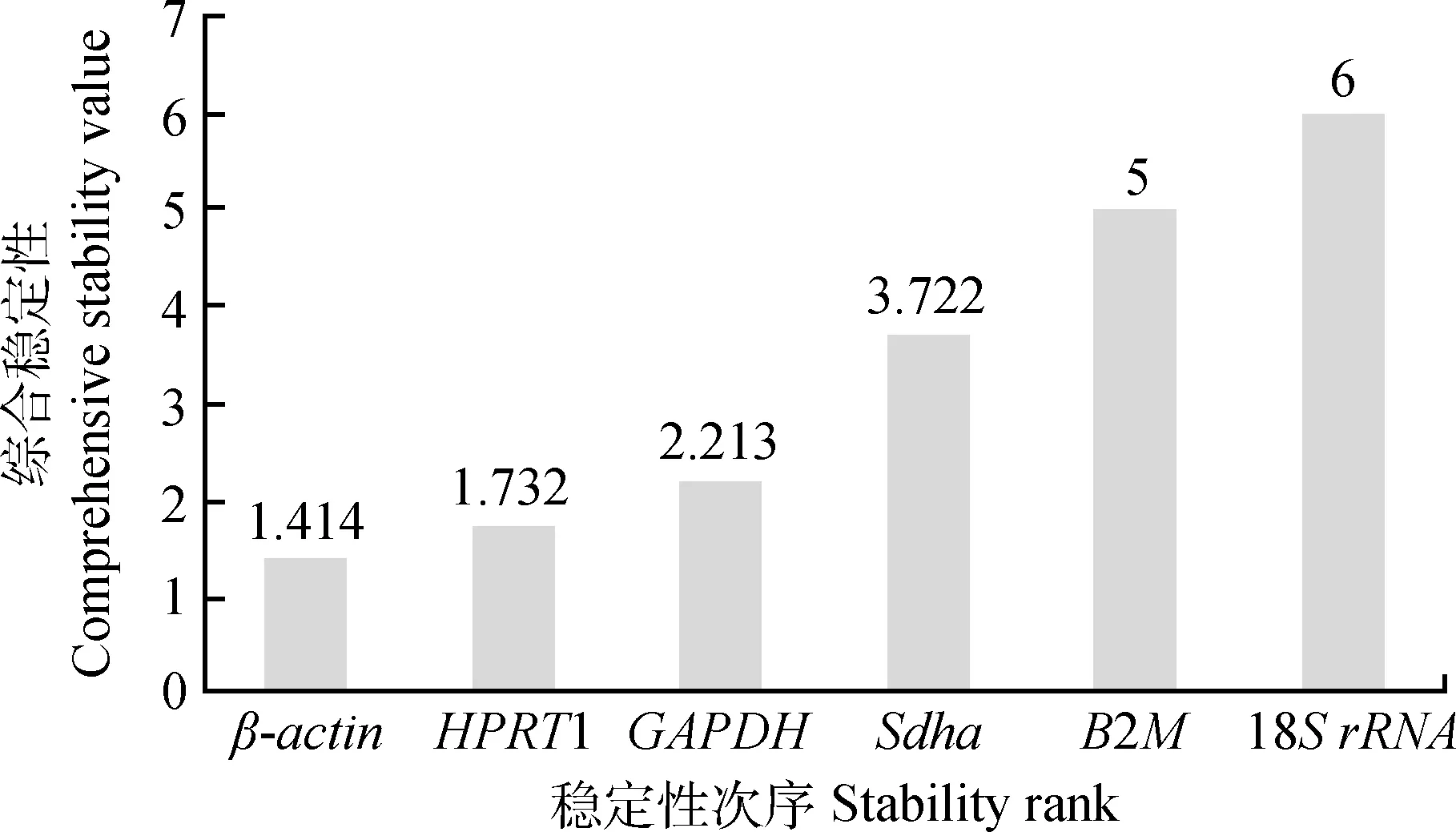
图6 内参基因在不同时期不同组织的表达稳定性Fig.6 RefFinder analysis comparing the selected reference genes for all timings and all tissues
本研究检测了新西兰白兔胚胎期、幼龄期和成年期3个发育阶段的肾、心、肝3种组织中6个内参基因的稳定性。分析结果表明,新西兰白兔的肾和肝组织中的6个内参基因在胚胎期、幼龄期、成年期最稳定的3个内参基因依次是β-actin、HPRT1、GAPDH,而在心组织中最稳定的3个内参基因依次是β-actin、HPRT1、Sdha。在新西兰白兔胚胎期的心、肝和肺组织中,最稳定的3个内参基因依次是GAPDH、β-actin、HPRT1,而在幼龄期和成年期最稳定的3个内参基因依次是HPRT1、GAPDH、β-actin。对新西兰白兔的3个发育阶段和3种组织的内参基因稳定性结果分析表明,最稳定的3个内参基因依次是β-actin、HPRT1、GAPDH。本研究证实不同的时期和器官中,最稳定的内参基因不同。理想的内参基因应该在不同组织、个体、时间和试验条件下稳定表达,但目前没有单个基因满足所有这些条件。有报道显示,使用最稳定的至少3个内参基因的几何平均数是标准化RT-qPCR数据更准确的方法[8-9]。因此,本研究推荐研究者在进行新西兰白兔的相关试验时,可以根据各自试验的特点,选择以上表达最稳定的3个内参基因的几何平均数作为标准化相关RT-qPCR数据的指标,以确保试验组个体间的差异最小,和微小变量的准确数据分析。
β-actin是一种真核细胞的细胞骨架蛋白,也是横纹肌肌纤维中的一种主要蛋白质成分和肌肉细丝的主要成分[18-20]。在细胞分泌、迁移及细胞质的流动和分离过程中起着重要作用,且在不同的物种间高度保守。A.B.Nygard等[21]证明了β-actin在猪的不同组织表达水平的稳定性。E.Jursza等[22]从猫科动物子宫内膜的7个内参基因中发现表达最稳定的是β-actin。但β-actin作为理想内参基因的稳定性也一直被质疑。M.J.Najafpanah等[23]在筛选山羊不同组织中稳定表达的内参基因时发现β-actin的表达变异最大。S.Mamo等[24]研究6个不同发育阶段的单独胚胎中12个常用内参基因的稳定性,发现不同阶段和培养条件下Ppia、H2afz和HPRT1基因是最稳定的,而经典的内参基因β-actin显示最低的稳定性。β-actin的表达稳定性存在较大差异,可能是由于研究所用的样本及样本间的处理不同,所选择的共同研究的内参种类不同和物种间的差异等。甘油醛-3-磷酸脱氢酶(GAPDH)是糖酵解、糖异生及光合作用过程中的关键酶,在生物进化的早期就已经出现,是最为常用的内参基因之一。W.Nachar等[25]报道,兔左心室舒张功能紊乱模型中10个内参基因中GAPDH和HPRT1基因有高度的稳定性。在最近的研究中,次黄嘌呤磷酸核糖基转移酶(HPRT1)的稳定性得到更多的认可,常被选作内参基因进行研究[24-25]。X.Feng等[26]在研究不同日龄梅山猪和约克夏猪骨骼肌11个内参基因的表达稳定性时发现HPRT1稳定性较好。
4结论
MIQE指南的应用为RT-qPCR分析提供了一个标准,对提高定量转录数据的再现性和精密分析具有重要意义。本研究检测了新西兰白兔胚胎期、幼龄期和成年期3个发育阶段的肾、心、肝3种组织中6个内参基因的表达稳定性。结果证实,不同的发育时期和组织中,最稳定的内参基因不同。因此,本研究推荐研究者根据各自试验的特点,选择表达最稳定的3个内参基因的几何平均数作为标准化相关RT-qPCR数据的指标,以确保试验组个体间的差异最小和小变量的准确数据分析。
参考文献(References):
[1]D’HAENEB,VANDESOMPELEJ,HELLENMANSJ.Accurateandobjectivecopynumberprofilingusingreal-timequantitativePCR[J].Methods,2010,50(4):262-270.
[2]DERVEAUXS,VANDESOMPELEJ,HELLENMANSJ.Howtodosuccessfulgeneexpressionanalysisusingreal-timePCR[J].Methods,2010,50(4):227-230.
[3]BUSTINSA,NOLANT.Pitfallsofquantitativereal-timereverse-transcriptionpolymerasechainreaction[J].J Biomol Technol,2004,15(3):155-166.
[4]IMBEAUDS,GRAUDENSE,BOULANGERV,etal.TowardsstandardizationofRNAqualityassessmentusinguser-independentclassifiersofmicrocapillaryelectrophoresistraces[J].Nucleic Acids Res,2005,33(6):e56.
[5]KOWALEWSKAM,DANSKA-BIDZINSKAA,BAKULA-ZALEWSKAE,etal.Identificationofsuitablereferencegenesforgeneexpressionmeasurementinuterinesarcomaandcarcinosarcomatumors[J].Clin Biochem,2012,45(4-5):368-371.
[6]HUGGETTJ,DHEDAK,BUSTINS,etal.Real-timeRT-PCRnormalisation;strategiesandconsiderations[J].Genes Immun,2005,6(4):279-284.
[7]HO-PUN-CHEUNGA,CELLIERD,LOPEZ-CRAPEZE.ConsiderationsfornormalisationofRT-qPCRinoncology[J].Ann Biol Clin(Paris),2008,66(2):121-129.
[8]BUSTINSA,BENESV,GARSONJA,etal.TheMIQEguidelines:minimuminformationforpublicationofquantitativereal-timePCRexperiments[J].Clin Chem,2009,55(4):611-622.
[9]JOHNSONG,NOURAA,NOLANT,etal.Minimuminformationnecessaryforquantitativereal-timePCRexperiments[J].Methods Mol Biol,2014,1160:5-17.
[10]MAJEROWICZD,ALVES-BEZERRAM,LOGULLOR,etal.Lookingforreferencegenesforreal-timequantitativePCRexperimentsinRhodniusprolixus(Hemiptera:Reduviidae)[J].Insect Mol Biol,2011,20(6):713-722.
[11]THOMASKC,ZHENGXF,GARCESSUAREZF,etal.EvidencebasedselectionofcommonlyusedRT-qPCRreferencegenesfortheanalysisofmouseskeletalmuscle[J].PLoS One,2014,9(2):e88653.
[12]VANDESOMPELEJ,DEPRETERK,PATTYNF,etal.Accuratenormalizationofreal-timequantitativeRT-PCRdatabygeometricaveragingofmultipleinternalcontrolgenes[J].Genome Biol,2002,3(7):RESEARCH0034.
[13]PFAFFLMW,TICHOPADA,PROGMETC,etal.Determinationofstablehousekeepinggenes,differentiallyregulatedtargetgenesandsampleintegrity:BestKeeper-Excel-basedtoolusingpair-wisecorrelations[J]. Biotechnol Lett,2004,26(6):509-515.
[14]ANDERSENCL,JENSENJL,ØRNTOFTTF.Normalizationofreal-timequantitativereversetranscription-PCRdata:amodel-basedvarianceestimationapproachtoidentifygenessuitedfornormalization,appliedtobladderandcoloncancerdatasets[J].Cancer Res,2004,64(15):5245-5250.
[15]SILVERN,BESTS,JIANGJ,etal.Selectionofhousekeepinggenesforgeneexpressionstudiesinhumanreticulocytesusingreal-timePCR[J].BMC Mol Biol,2006,7:33.
[16]PENGXX,ZHAORL,SONGW,etal.Selectionofsuitablereferencegenesfornormalizationofquantitativereal-timePCRincartilagetissueinjuryandrepairinrabbits[J].Int J Mol Sci,2012,13(11):14344-14355.
[17]JAINM,NIJHAWANA,TYAGIAK,etal.Validationofhousekeepinggenesasinternalcontrolforstudyinggeneexpressioninricebyquantitativereal-timePCR[J].Biochem Biophys Res Commun,2006,345(2):646-651.
[18]LIL,JIANGJ,WANGL,etal.Expressionpatternsofperoxisomeproliferator-activatedreceptorgamma1versusgamma2,andtheirassociationwithintramuscularfatingoattissues[J].Gene,2013,528(2):195-200.[19]ZHUH,PARKS,SCHEFFLERJM,etal.Porcinesatellitecellsarerestrictedtoaphenotyperesemblingtheirmuscleorigin[J].J Anim Sci,2013,91(10):4684-4691.
[20]HOLFORDNC,SANDHUHS,THAKKARH,etal.Stabilityofbeta-actinmRNAinplasma[J].Ann N Y Acad Sci,2008,1137:108-111.
[21]NYGARDAB,JØRGENSENCB,CIRERAS,etal.SelectionofreferencegenesforgeneexpressionstudiesinpigtissuesusingSYBRgreenqPCR[J].BMC Mol Biol,2007,8:67.
[22]JURSZAE,SKARZYNSKIDJ,SIEMIENIUCHMJ.Validationofreferencegenesinthefelineendometrium[J].Reprod Biol,2014,14(4):302-306.
[23]NAJAFPANAHMJ,SADEGHIM,BAKHTIARIZADEHMR.Referencegenesselectionforquantitativereal-timePCRusingRankAggregmethodindifferenttissuesofCaprahircus[J].PLoS One,2013,8(12):e83041.
[24]MAMOS,GALAB,BODOS,etal.Quantitativeevaluationandselectionofreferencegenesinmouseoocytesandembryosculturedin vivoandin vitro[J].BMC Dev Biol,2007,7:14.
[25]NACHARW,BUSSEUILD,SHIY,etal.Optimisationofreferencegenesforgene-expressionanalysisinarabbitmodelofleftventriculardiastolicdysfunction[J].PLoS One,2014,9(2):e89331.
[26]FENGX,XIONGY,QIANH,etal.SelectionofreferencegenesforgeneexpressionstudiesinporcineskeletalmuscleusingSYBRgreenqPCR[J].J Biotechnol,2010,150(3):288-293.
(编辑郭云雁)
ExpressionStabilityAnalysisofReferenceGenesinDifferentDevelopmentPeriodsandTissuesinOryctolagus Cuniculus
CHENRui,YANGXiao-nong*,ZENGWan-qiu,WENJuan
(College of Life Science and Technology,Southwest University for Nationalities,Chengdu 610041,China)
Abstract:ThepurposeofthisstudywastoscreenthemoststablereferencegenesindifferentdevelopmentperiodsandtissuesinNewZealandWhiterabbit.Quantitativereal-timePolymeraseChainReaction(RT-qPCR),alatestonlinereferencegenesstabilityassessmenttoolRefFinderandMinimumInformationforPublicationofQuantitativeReal-timePCRExperiments(MIQE)guidelinewereusedtocomparetheexpressionlevelsof6commonlyusedreferencegenes(GAPDH,HPRT1,18S rRNA,B2M,Sdha,β-actin)mRNAin3tissues(heart,liver,kidney)ofrabbitatembryo(E28.5),juvenile(10d)andadultstages(A).TheresultshowedthatthemoststablereferencegenesinliverandkidneyofNewZealandWhiterabbitin3developmentperiodswereβ-actin, HPRT1andGAPDH,successively.Butforheart,theywereβ-actin,HPRT1, Sdha.ThemoststablereferencegenesinembryoperiodofNewZealandWhiterabbitin3tissueswereGAPDH,β-actinandHPRT1,successively.Forjuvenileandadultstages,theywereHPRT1,GAPDHandβ-actin.Toall3periodsandall3tissuesofNewZealandWhiterabbit,themoststablereferencegenesweresuccessivelyβ-actin,HPRT1andGAPDH.ThisstudyconfirmedthatthemoststablereferencegenesweredifferentindifferentperiodsandtissuesofNewZealandWhiterabbit.Toensureminimalvariationbetweenindividualsinanexperimentalgroupandtheaccuratestatisticalanalysisofsmallfoldchanges,werecommendnormalizingRT-qPCRdatatothegeometricmeanofatleast3validatedreferencegenesasabovewhenrelatedNewZealandWhiterabbittrialswereperformed.
Keywords:referencegenes;expressionstability;MIQE;RefFinder
doi:10.11843/j.issn.0366-6964.2016.03.008
收稿日期:2015-03-04
基金项目:西南民族大学研究生学位点建设项目 (2014XWD-S0906)
作者简介:陈 瑞(1989-),男,安徽涡阳人,硕士生,主要从事临床兽医研究,E-mail: 18010619617@163.com *通信作者:杨晓农,教授,E-mail:yangxn058@163.com
中图分类号:S829.1;S813.3
文献标志码:A
文章编号:0366-6964(2016)03-0477-07
