巴马小型猪OCA2基因序列、组织表达分析及载体构建研究
2016-07-13洪潜龙曹春伟王宪龙赵建国
洪潜龙,张 颖,曹春伟,王宪龙,赵建国*
(1.中国科学院动物研究所 干细胞与生殖生物学国家重点实验室,北京 100101; 2.安徽大学生命科学学院,合肥 230601)
巴马小型猪OCA2基因序列、组织表达分析及载体构建研究
洪潜龙1,2,张颖1,曹春伟1,王宪龙1,赵建国1*
(1.中国科学院动物研究所 干细胞与生殖生物学国家重点实验室,北京 100101; 2.安徽大学生命科学学院,合肥 230601)
摘要:旨在为猪的毛色调控机制和模拟与OCA2基因相关的人类疾病模型的研究建立基础。本研究将GenBank公布的猪OCA2基因序列(NC_010457.4)所缺失的5个外显子(20~24号外显子)进行了克隆和划分,在基因组水平和cDNA水平上采用PCR扩增和Sanger测序方法对划分结果进行了验证;并对OCA2基因进行生物信息学分析,同时在广西巴马小型猪中对OCA2基因外显子的多态位点进行了筛选。采用实时荧光定量PCR技术(Q-PCR)分析了OCA2基因在各组织中的表达规律,并以皮肤全长cDNA为模板构建重组表达质粒,并利用XhoⅠ和SacⅡ限制性内切酶进行双酶切验证。在已公布的猪OCA2基因中成功划分和克隆了所缺失的外显子,并在OCA2基因所有外显子中共发现了10个SNPs位点,其中7个为同义突变,3个为错义突变(R124H、G509D、R573H);猪OCA2基因在多种组织中广泛表达,其中,肺、大脑、小脑相对高表达,脾、胃、盲肠和皮肤中度表达,而在心和肝中表达量较低;OCA2基因重组表达质粒经酶切和测序鉴定证明构建成功。本研究初步探讨了猪OCA2基因的序列信息及组织表达规律,并构建了OCA2基因重组表达质粒,为该基因下一步的功能学研究及疾病模型的研究奠定了基础。
关键词:猪;OCA2基因;荧光定量PCR ;重组表达质粒;单核苷酸多态性
白化病(Albinism)是一组与黑色素合成相关的基因发生突变,导致眼、皮肤、毛发或全身黑色素缺乏的一种单基因遗传性疾病[1]。医学上白化病分为3种类型,其中,眼、皮肤、毛发均有色素缺乏的称为眼皮肤白化病(Oculocutaneous albinism,OCA),仅有眼色素减少或缺乏的称眼白化病(Ocular albinism,OA),另外一种是除具有一定程度的眼皮肤白化病表现外,同时还具有免疫功能低下的Chediak-Higashi综合征[2]和具有出血症状的Hermansky-Pudlak综合征[3]。眼皮肤白化病分为4种亚型(OCAⅠ、OCAⅡ、OCAⅢ、OCAⅣ),其致病基因分别为TYR、OCA2、TYRP1、SLC45A2[4]。据统计,世界范围内白化病的发病率约为1/17 000[5],其中由OCA2基因突变引起的眼皮肤白化病Ⅱ型发生频率最高。
人类OCA2基因(NM_000275.2)定位于染色体15q11.2-15q12,基因组DNA全长345 kb,mRNA长3.4 kb,包含24个外显子。OCA2基因编码产物是一个由838个氨基酸残基组成的跨膜蛋白,含有12个跨膜区域,主要在黑色素细胞表达[6]。近年来关于眼皮肤白化病的研究相继被报道,I.Yuasa等[7]报导了眼皮肤白化病人OCA2基因的14号外显子内发现G-A转换,C.Rooryck等[8]在3个没有血缘关系的眼皮肤白化病人的OCA2基因中发现了184 kb的缺失。目前对于眼皮肤白化病动物模型研究相关的报道较少,且主要研究啮齿类小型试验动物模型。猪在遗传、代谢、生理生化特性等方面比小鼠更接近于人类,因此在作为遗传、发育、疾病模型以及提供异种器官移植等方面具有不可替代的优势[9]。另外猪皮肤的组织学与生化性质与人类非常类似[10],眼睛具有与人类类似的脉管系统和光感受器,已广泛用于人类眼睛的外科手术[11],本研究以猪为模型对OCA2基因进行相关的研究。
猪OCA2基因(NC_010457.4)位于15号染色体,目前NCBI数据库中GenBank上公布的基因序列只包括19个外显子,而将19个外显子拼接分析发现,其总长要比mRNA(NM_214094)短,故推断NCBI所公布的OCA2基因序列不够完整。虽然已报道的五指山猪全基因组测序研究提示猪基因组中存在缺失的序列,但是目前尚未有研究在基因组DNA和mRNA水平上将OCA2基因的外显子序列进行精细定位和验证。本研究旨在完善OCA2基因的序列信息并对其序列进行生物信息学分析,并通过Q-PCR研究猪OCA2基因的组织表达特点和规律,并构建重组表达质粒,为下一步基因功能和眼皮肤白化病疾病模型研究建立基础。
1材料与方法
1.1试验材料和试剂
1.1.1试验动物试验动物为广西巴马小型猪,来自于中国科学院动物研究所北方大动物研究基地。饲养环境温度控制在18~25 ℃,相对湿度为 40%~70%,每日饲喂2次,每日饲料量为体重的3%。按照北京市试验动物管理条例规范试验操作。
1.1.2主要试剂TIANamp Genomic DNA 试剂盒、2×Taq PCR Master Mix、DNA MarkerⅡ和Ⅲ、FastQuant cDNA第一链合成试剂盒、快速质粒小提试剂盒、卡那霉素等试剂购自天根生化科技有限公司,Trizol试剂购自Invitrogen公司,DL10000 DNA Marker、6×Loading Buffer、SYBR®Premix Ex TaqTM(Tli RNaseH Plus)、E.coliDH5α Competent Cells购自宝生物工程(大连)有限公司,pEGFP-N1质粒购自Clontech公司,XhoⅠ和SacⅡ限制性内切酶、Phusion超保真PCR Master Mix、T4 DNA连接酶购自NEB公司,快捷型琼脂糖凝胶DNA回收试剂盒Ⅱ型购自北京百泰克生物技术有限公司。
1.2试验方法
1.2.1样本采集取100头巴马小型猪的耳组织用于提取基因组DNA,ddH2O溶解,-20 ℃保存备用。采集3头8月龄成年猪的心、肝、脾、肺、肾、大脑、小脑、胃、盲肠、皮肤等组织样本提取RNA,样品采集后立即放入液氮速冻,-80 ℃保存,用于Q-PCR和克隆。
1.2.2DNA提取 按照TIANamp Genomic DNA 试剂盒的说明书进行DNA的提取,溶于ddH2O,用Nanodrop 2000分光光度计测定浓度和纯度后,-20 ℃保存备用。
1.2.3PCR引物设计与扩增利用Primer Premier 5.0设计引物,扩增巴马猪OCA2基因的24个外显子,引物由Invitrogen公司合成,序列见表1。PCR反应体系为50 μL:2×Taq PCR Master Mix 25 μL,上下游引物各1 μL,DNA模板1 μL,ddH2O补足50 μL。PCR反应程序:94 ℃预变性3 min;94 ℃变性30 s,最适温度退火30 s,72 ℃延伸30 s,30个循环;72 ℃延伸10 min。PCR产物用1.5%琼脂糖凝胶电泳检测,剩余样本送往Invitrogen公司测序,用Chromas软件分析测序峰图和碱基序列。
1.2.4引用的数据库猪基因组信息系统( Pig Genomic Informatics System,PigGIS)(http://pig.genomics.org.cn/)对猪的基因进行了准确的注释,人类基因突变数据库(The Human Gene Mutation Database)(http://www.hgmd.cf.ac.uk/ac/index.php)收录了文章报导的人类遗传性疾病的突变位点,NCBI中的dbSNP 数据库(http://www.ncbi.nlm.nih.gov/snp/?term=)对猪中存在的多态位点进行了汇总。
1.2.5生物信息学分析用Clustal 2.1分析软件将猪和黑猩猩、恒河猴、人、小鼠、大鼠、斑马鱼的OCA2基因的编码区氨基酸序列进行同源性分析;通过NCBI CDD(http://www.ncbi.nlm.nih.gov/cdd)在线预测保守结构域;用SMART(http://smart.embl-heidelberg.de/)在线软件对猪OCA2蛋白结构域进行预测。
1.2.6RNA的提取及cDNA反转录总RNA的提取方法参照Invitrogen公司的Trizol试剂说明书进行,提取的总RNA用RNase-free水充分溶解。根据天根生化科技有限公司的FastQuant cDNA第一链合成试剂盒里的说明书进行cDNA合成,保存于-20 ℃。
1.2.7RT-PCR和荧光定量PCR根据GenBank公布的OCA2基因的mRNA序列(NM_214094)设计普通RT-PCR引物和荧光定量PCR引物,内参选择GAPDH基因,具体信息见表2。RT-PCR反应体系为20 μL: 2×Taq PCR Master Mix 10 μL,上下游引物各1 μL,DNA模板1 μL,ddH2O补足20 μL。PCR反应程序:94 ℃预变性3 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,40个循环;72 ℃延伸10 min。荧光定量PCR试验利用Agilent Technologies公司的Stratagene Mx3005P仪器完成,反应步骤:95 ℃ 60 s;95 ℃ 10 s,60 ℃ 40 s,40个循环;熔解曲线的制作:95 ℃ 60 s,60 ℃ 30 s,95 ℃ 30 s。根据倍比稀释法制作内参基因和目标基因的标准曲线,目标基因样本和GAPDH内参样本重复3次进行扩增,且均有空白对照组,采用2-△△CT方法进行统计。
1.2.8OCA2基因重组表达质粒的构建与鉴定以皮肤总RNA反转录的cDNA为模板,根据OCA2的mRNA序列(NM_214094)设计引物,引物序列见表2。PCR反应体系20 μL:2×Phusion HF PCR Master Mix 10 μL,上下游引物各1 μL,cDNA模板1 μL,DMSO 0.6 μL,ddH2O补足20 μL。PCR反应程序:98 ℃预变性30 s;98 ℃变性10 s,64 ℃退火30 s,72 ℃延伸1 min 30 s,40个循环;72 ℃延伸10 min。 PCR产物用快捷型琼脂糖凝胶DNA回收试剂盒Ⅱ型切胶回收,用XhoⅠ和SacⅡ限制性内切酶对纯化的PCR产物和pEGFP-N1质粒进行双酶切后,将目的基因和载体用T4 DNA连接酶连接,将连接载体转化感受态细胞,涂布于预先加入卡那霉素的LB培养基中,37 ℃培养过夜,挑单克隆摇菌经酶切鉴定后送Invitrogen公司测序。
2结果
2.1猪OCA2基因结构分析及缺失外显子的位置划分

表1 OCA2基因PCR扩增引物信息
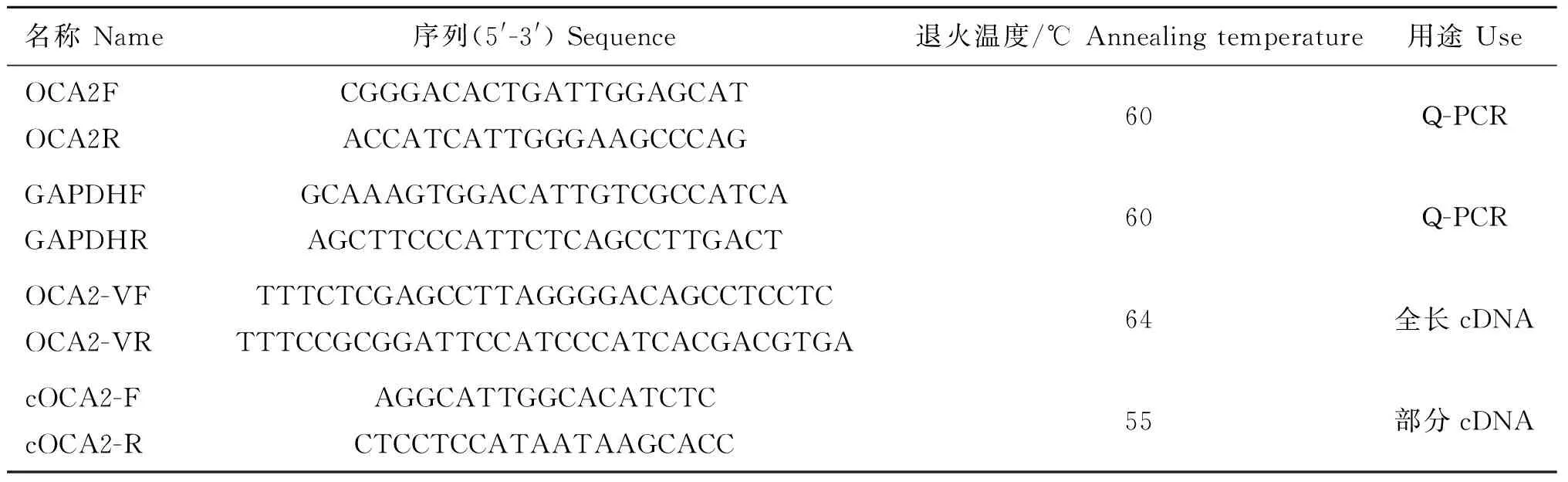
表2 猪OCA2基因的cDNA PCR扩增的引物信息
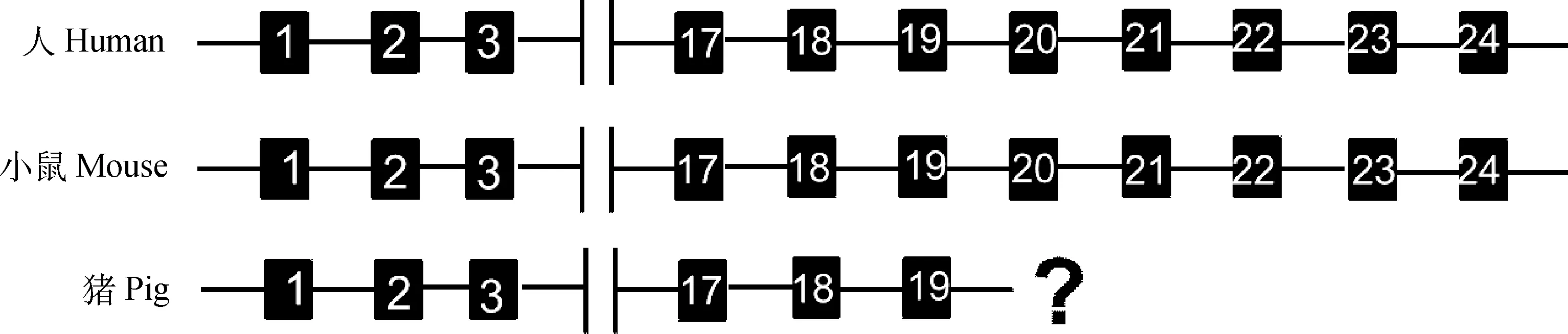
图1 人、小鼠、猪OCA2基因结构Fig.1 The structure of OCA2 gene in human,mouse and pig
小鼠和人的OCA2基因均由24个外显子组成,而目前NCBI数据库中的GenBank公布的猪OCA2基因仅包括19个外显子,且将其拼接分析发现总长要比全长mRNA短(NM_214094)(图1),故推断NCBI所公布的OCA2基因序列不够完整。根据五指山猪的基因组序列[12]和GenBank公布的OCA2基因cDNA序列(NM_214094),将猪OCA2基因序列(NC_010457.4)所缺失的5个外显子(20~24号外显子)的位置进行了划分(图2)。
2.2猪OCA2基因未注释的外显子在基因组水平上的验证
根据GenBank公布的猪OCA2基因19个外显子和上述划分的5个外显子设计引物并进行PCR扩增,结果如图3和图4所示。24个外显子的PCR扩增产物特异性均较好,并经Sanger测序进行验证,发现20~24号外显子的大小以及在基因组的位置与预测的一致。
2.3猪OCA2基因未注释的外显子在cDNA水平上的验证
由于GenBank公布的猪OCA2基因的cDNA序列(NM_214094)是通过试验获得并公布的[13],故根据基因组20~24号外显子区域所对应的cDNA设计引物(表2)并进行PCR扩增。1.5%琼脂糖凝胶电泳(图5)和Sanger测序结果同样验证了上述在基因组中对20~24号外显子序列划分的准确性。
2.4猪OCA2基因生物信息学分析
将NCBI所公布的猪(NC_010457.4)和黑猩猩(NC_006482.3)、恒河猴(NC_007864.1)、人(NC_000015.10)、小鼠(NC_000073.6)、大鼠(NC_005100.4)、斑马鱼(NC_007117)的OCA2基因的氨基酸序列进行同源性比对,相对于人而言,除了灵长类外,猪与其同源性最高(表3); CDD检测结果显示,OCA2蛋白中具有ArsB/NhaD permease 超家族和Citrate transporter两个结构域(图6),利用SMART程序进行的蛋白结构域分析表明,猪OCA2具有12个跨膜结构域(图7)。
2.5猪OCA2基因外显子多态位点的分析
获得了猪OCA2基因的全部外显子后,在群体中随机选择了100头巴马猪,提取耳组织DNA,分别根据表1中所设计的24个外显子的引物扩增PCR并进行测序,结果发现巴马猪OCA2基因中存在 10个单碱基突变位点,其中7个为同义突变,3个为错义突变(图8):外显子4中的c.371G>A转换,外显子15的c.1526G>A转换和外显子16的c.1718G>A转换,3个错义突变位点在NCBI中猪的SNPs数据库均有已知的SNPs ID号(表4),而在人的HGMD数据库中所收录的82个OCA2基因单碱基突变的位点中并未发现与本次研究中同源对应的SNPs位点。

下划线为外显子,省略号为内含子underline represent exons,apostrophe represent introns图2 猪OCA2基因20~24号外显子的定位Fig.2 The location of exons 20-24 in OCA2 gene of pig
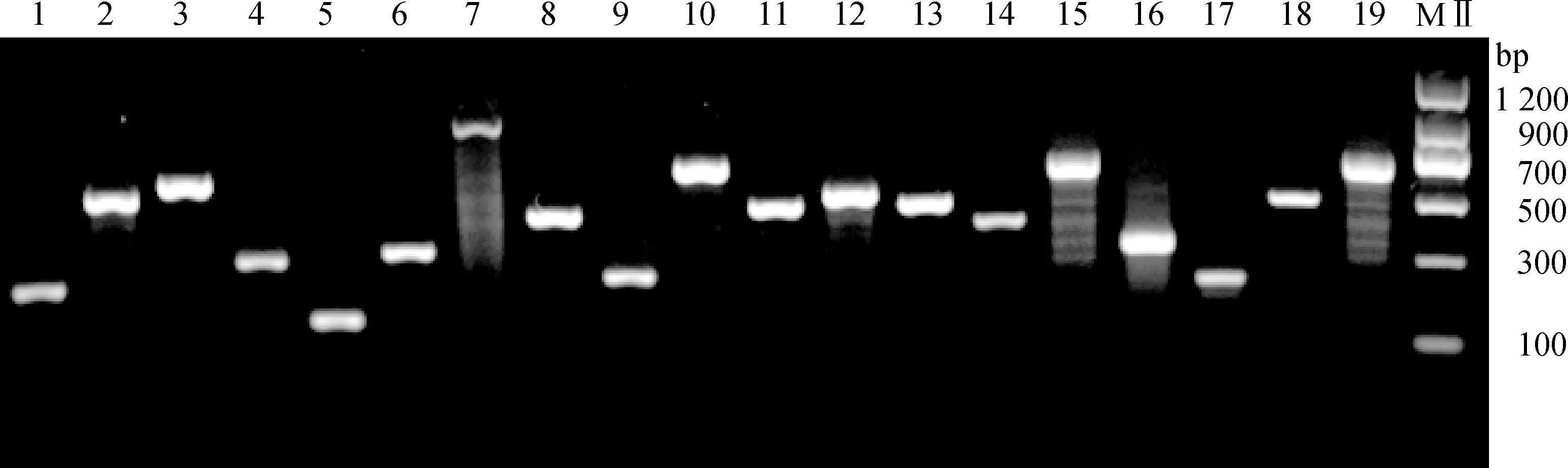
1~19.分别表示1~19号外显子;M.DNA相对分子质量标准1~19.Represent exon 1-19;M.Marker Ⅱ图3 OCA2基因第1~19外显子的PCR扩增结果Fig.3 The PCR results of exons 1-19 in OCA2 gene
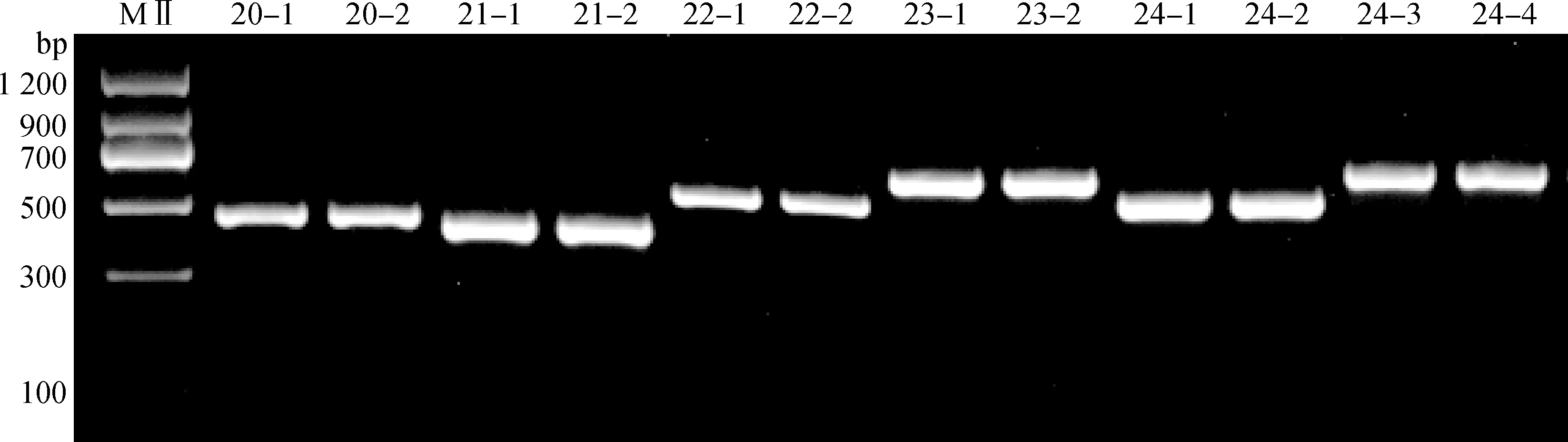
20-1、20-2.外显子20;21-1、21-2.外显子21;22-1、22-2.外显子22;23-1、23-2.外显子23;24-1、24-2、24-3、24-4.外显子24,每个样本两个重复;M.DNA相对分子质量标准20-1,20-2.Exon 20;21-1,21-2.Exon 21;22-1,22-2.Exon 22;23-1,23-2.Exon 23;24-1,24-2,24-3,24-4.Exon 24,twice per sample;M.Marker Ⅱ图4 OCA2基因第20~24外显子的基因组PCR验证结果Fig.4 The validation results of exons 20-24 in OCA2 gene by PCR

1~4.RT-PCR产物,代表4个样本;M.DNA相对分子质量标准1-4.RT-PCR products for 4 samples;M.Marker Ⅱ图5 OCA2基因第20~24外显子cDNA的PCR产物Fig.5 The PCR amplification products of exon 20-24 for cDNA of OCA2 gene
2.6猪OCA2基因mRNA水平组织表达分析
利用相对实时荧光定量PCR(Q-PCR)方法分别检测了OCA2基因在巴马猪的心、肝、脾、肺、大脑、小脑、胃、盲肠、皮肤中mRNA的表达情况(图9)。结果表明,在巴马猪中,OCA2基因在不同组织中均有不同程度的表达。其中,肺、大脑、小脑相对高表达,脾、胃、盲肠和皮肤中度表达,而在心和肝中表达量较低。
2.7OCA2基因重组表达质粒的构建与鉴定
以猪皮肤组织的cDNA为模板,扩增得到与预期全长CDS序列片段大小相符的特异性片段。回收的PCR产物与pEGFP-N1连接的重组质粒经XhoⅠ和SacⅡ限制性内切酶双酶切后,琼脂糖凝胶电泳可见与预期相符的两条带:分别为2 638 bp的目的片段和4 733 bp的载体片段(图10),且经Sanger测序验证结果正确。
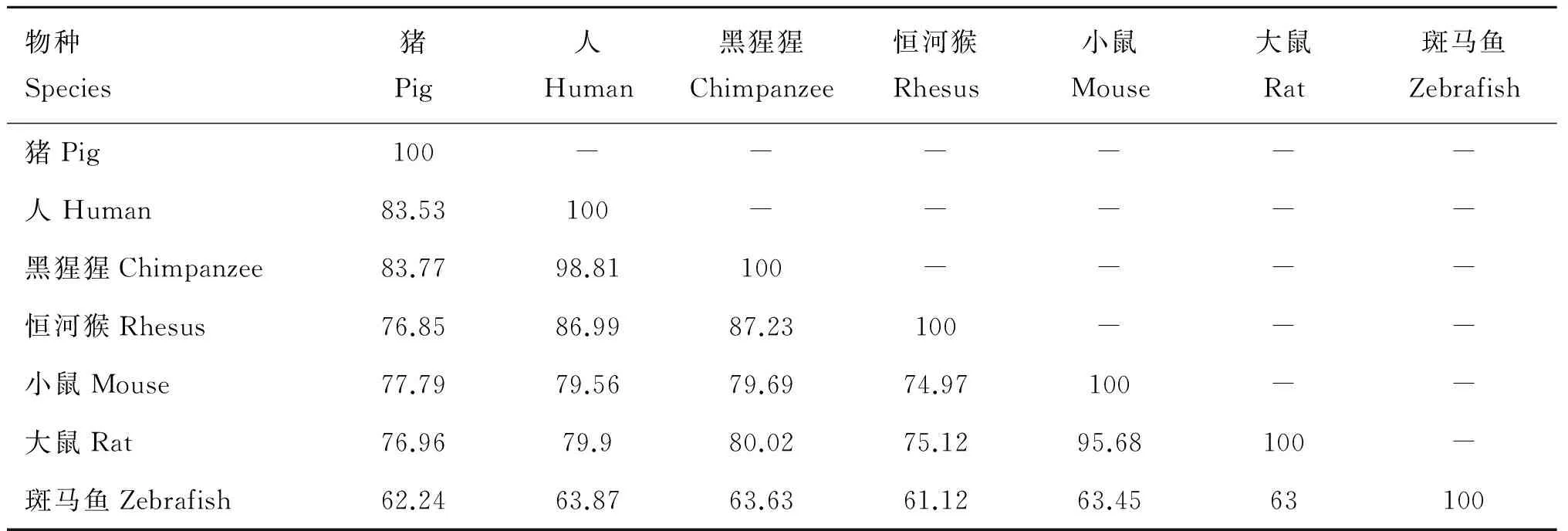
表3 巴马小型猪与6个物种间OCA2基因氨基酸序列同源性比较

图6 猪OCA2保守结构域Fig.6 The conserved domains of pig OCA2
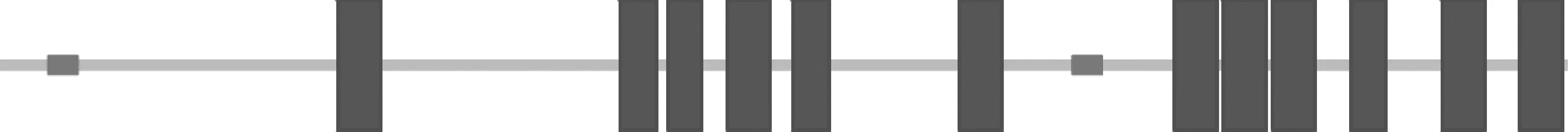
图7 猪OCA2蛋白质跨膜结构域Fig.7 The transmembrane domains of pig OCA2
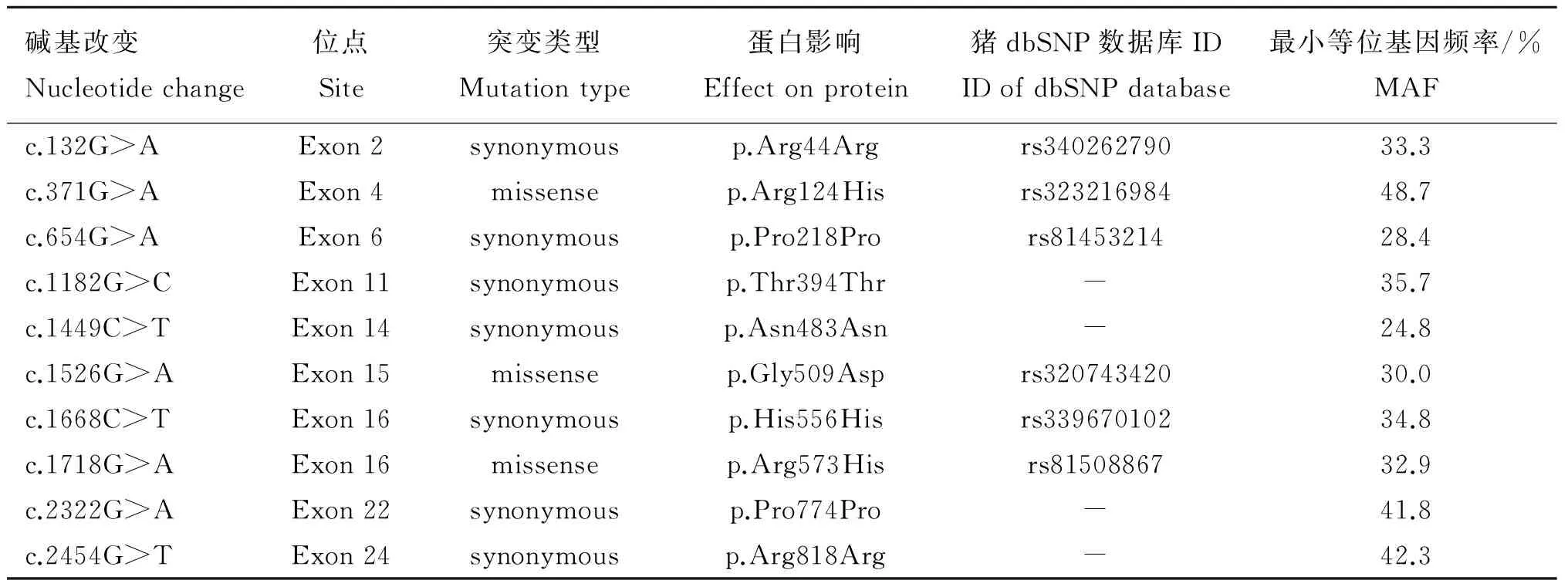
表4 巴马小型猪OCA2基因的多态位点信息
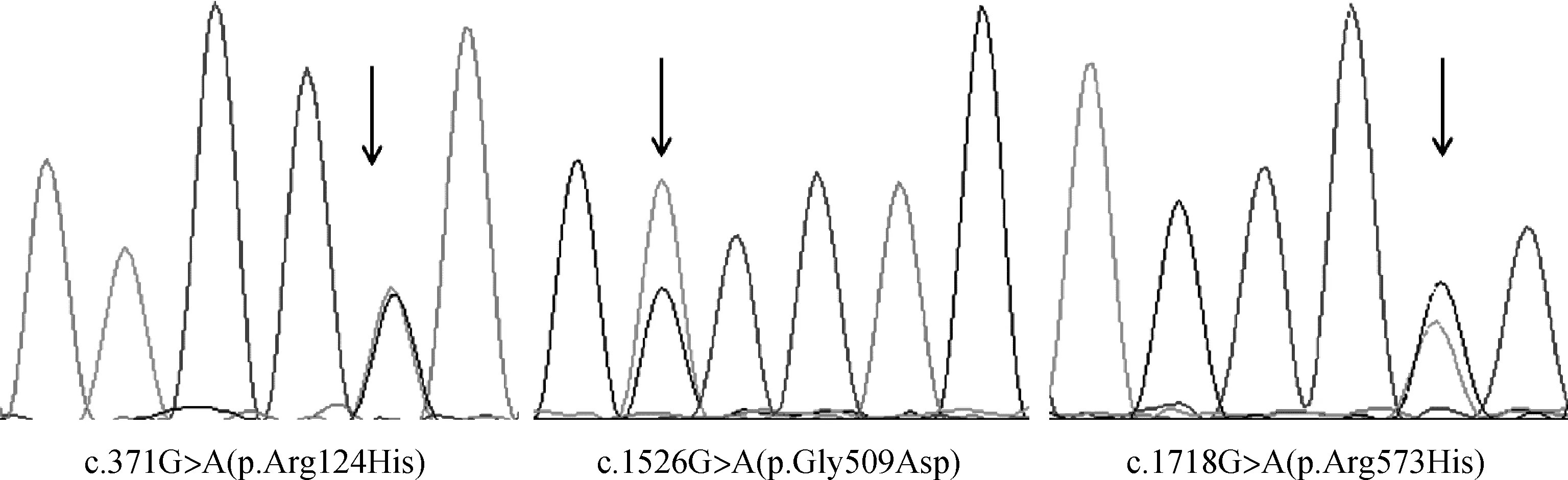
图8 OCA2基因的错义突变位点测序图谱Fig.8 The sequencing chromatograms of missense sites of OCA2 gene
3讨论
本研究以巴马小型猪为模型完善了OCA2基因外显子序列信息,并检测和分析了OCA2基因在各组织中的表达情况,成功构建了OCA2基因载体,为后续功能学研究及疾病模型构建建立了基础。目前对眼皮肤白化病的研究较少,且主要研究啮齿类小型试验动物模型[14],由于小型哺乳动物体型过小,可操作性差,更为重要的是与人的解剖结构相差甚远,所以限制了人类疾病模型的创制。巴马小型猪由于体型小、适应性强等特点,增加了试验的可操作性以及节省了饲养成本,与体型大的猪相比较具有不可替代的优势。本研究通过对OCA2基因的氨基酸序列同源性分析发现,猪与人的同源性较高,这一结果无疑增添了猪作为该类疾病模型的研究具有优势的科学依据。
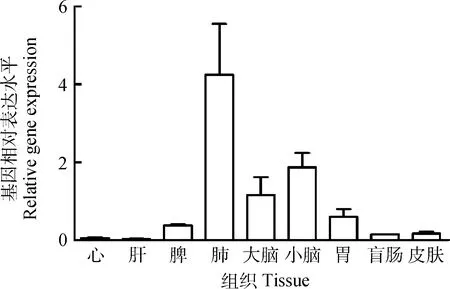
图9 猪OCA2基因在mRNA水平上的多组织表达谱分析Fig.9 The tissue expression profile of OCA2 gene at the mRNA level in pig
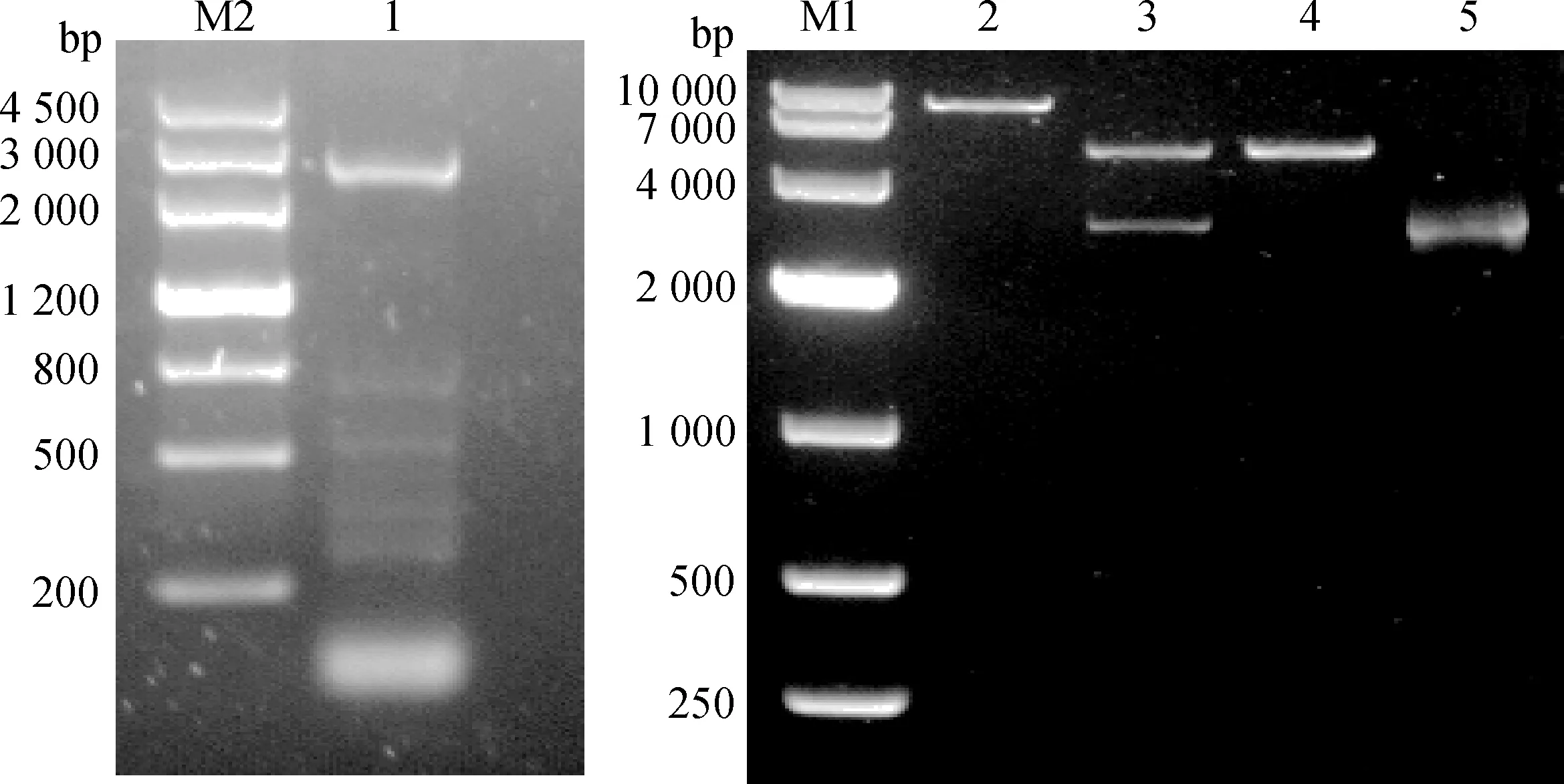
1.CDS序列全长;2.单酶切产物;3.双酶切产物;4.空载体;5.CDS序列全长;M1、M2.DNA相对分子质量标准1.Full-length CDS;2.SAFLP;3.Double digestion;4.Empty vector;5.Full-length CDS;M1.DL10000 DNA marker;M2.Marker Ⅲ图10 猪OCA2基因CDS全长的PCR扩增和pEGFP-N1-OCA2表达载体酶切鉴定Fig.10 PCR product of OCA2 gene and digestion of pEGFP-N1-OCA2
基因序列信息完整性是一切后续的基因功能研究的基本前提,而目前NCBI数据库中GenBank公布的OCA2基因序列不够完整。虽然五指山猪全基因组测序研究提示存在缺失的序列,但目前尚未有研究通过试验进行精细地分析和确认,因此完善猪OCA2基因信息具有极大的必要性。而且,这也是以基因组修饰创制猪眼皮肤白化病Ⅱ型模型研究的基础。为此,本研究将OCA2基因未注释的外显子部分进行了划分。通过在这些划分的序列周围设计引物,同时在基因组和cDNA水平上对所缺失区域进行PCR扩增和测序分析,最终证实巴马猪基因组中确实存在以上序列。本研究获得了猪OCA2基因的全部外显子后,在巴马猪群体中筛选SNPs位点,结果发现,巴马猪OCA2基因外显子中存在 10个单碱基突变位点,其中3个为错义突变,且在猪的SNPs数据库中均已报道。截止目前,HGMD数据库中共收录了82个OCA2基因单碱基突变位点,例如,W.S.Oetting等[15]发现了人S86R、C112F、A368V、T592I、A724P 和 A787V与眼皮肤白化病Ⅱ型相关,W.S.Oetting等[16]报导了2个错义突变位点(Ile646Val 和 Val519Ala)会导致人眼皮肤白化病Ⅱ型,然而以上所有位点均与本次研究中发现的SNPs位点不同,说明在巴马猪自然群体中并未发现与人类OCA2基因突变引发的眼皮肤白化病Ⅱ型相关的模型,同时反映出未来通过对巴马猪基因组修饰来获得眼皮肤白化病Ⅱ型模型的必要性。
由于OCA2基因组织表达的相关报道较少,且在不同组织中发挥的生物学功能未知。利用相对实时荧光定量PCR(Q-PCR)方法分别检测了OCA2基因在巴马猪各组织的表达情况。结果表明,在巴马猪中,OCA2基因在不同组织中均有不同程度的表达。其中,肺、大脑、小脑相对高表达,脾、胃、盲肠和皮肤中度表达,而在心和肝中表达量较低。通过GeneCards数据库得知,OCA2基因在人类组织器官中也具有广泛表达的特性,而且也在皮肤中相对高表达。OCA2基因的多组织表达表明其不仅在皮肤和眼睛中发挥主要作用,在其他组织中也可能扮演了重要的角色。此外,本研究成功构建了OCA2基因重组表达质粒,为该基因下一步的功能学研究建立了基础。
找到合适的动物模型来研究人类疾病,已经成为生物医学研究和临床药物开发和治疗研究的关键。猪作为大动物模型已经广泛应用于人类的多种复杂的疾病模型中,比如心血管疾病模型等,而眼部和皮肤相关的疾病模型在大动物中研究较少。基因组编辑技术(比如TALEN[17]、CRISPR-Cas9技术[18])由于相对经济和高效,已经在动物疾病模型构建中兴起,然而由于基因组的信息不完善从不同程度上阻碍了疾病模型的创建。本研究以巴马猪为研究对象,初步探讨了OCA2基因序列信息、组织表达规律和载体的构建,为OCA2基因的功能研究提供了基础。
4结论
本研究完善了OCA2基因的序列信息,并通过Q-PCR研究了猪OCA2基因的组织表达特点和规律,并成功构建了重组表达质粒,为下一步基因功能和眼皮肤白化病疾病模型的研究建立了基础。
参考文献(References):
[1]OETTING W S,KING R A.Molecular basis of albinism:mutations and polymorphisms of pigmentation genes associated with albinism[J].HumMutat,1999,13(2):99-115.
[2]INTRONE W J,WESTBROEK W,GOLAS G A,et al.GeneReviews[M].Chediak-Higashi Syndrome.Seattle,University of Washington,1993.
[3]GAHL W A,HUIZING M.Gene Reviews[M].Hermansky-Pudlak Syndrome.Seattle,University of Washington,1993.
[4]KAMARAJ B,PUROHIT R.Mutational analysis of oculocutaneous albinism:a compact review[J].BiomedResInt,2014,2014:905472.
[5]GRØNSKOV K,EK J,BRONDUM-NIELSEN K.Oculocutaneous albinism[J].OrphanetJRareDis,2007,2:43.
[6]LEE S T,NICHOLLS R D,JONG M T,et al.Organization and sequence of the human P gene and identification of a new family of transport proteins[J].Genomics,1995,26(2):354-363.
[7]YUASA I,UMETSU K,HARIHARA S,et al.OCA2 481Thr,a hypofunctional allele in pigmentation,is characteristic of northeastern Asian populations[J].JHumGenet,2007,52(8):690-693.
[8]ROORYCK C,MORICE-PICARD F,LASSEAUX E,et al.High resolution mapping of OCA2 intragenic rearrangements and identification of a founder effect associated with a deletion in Polish albino patients[J].HumGenet,2011,129(2):199-208.
[9]PRATHER R S,HAWLEY R J,CARTER D B,et al.Transgenic swine for biomedicine and agriculture[J].Theriogenology,2003,59(1):115-123.
[10]JACOBI U,KAISER M,TOLL R,et al.Porcine ear skin:aninvitromodel for human skin[J].SkinResTechnol,2007,13(1):19-24.
[11]GHOSH F,WONG F,JOHANSSON K,et al.Transplantation of full-thickness retina in the rhodopsin transgenic pig[J].Retina,2004,24(1):98-109.
[12]FANG X,MOU Y,HUANG Z,et al.The sequence and analysis of a Chinese pig genome[J].Gigascience,2012,1(1):16.
[14]HAGIWARA N,KLEWER S E,SAMSON R A,et al.Sox6 is a candidate gene for p100H myopathy,heart block,and sudden neonatal death[J].ProcNatlAcadSciUSA,2000,97(8):4180-4185.
[15]OETTING W S,GARDNER J M,FRYER J P,et al.Mutations of the human P gene associated with Type II oculocutaneous albinism(OCA2).Mutations in brief no.205.Online[J].HumMutat,1998,12(6):434.
[16]OETTING W S,GARRETT S S,BROTT M,et al.P gene mutations associated with oculocutaneous albinism type II(OCA2)[J].HumMutat,2005,25(3):323.
[17]GAJ T,GERSBACH C A,BARBAS C F 3rd.ZFN,TALEN,and CRISPR/Cas-based methods for genome engineering[J].TrendsBiotechnol,2013,31(7):397-405.
[18]GRATZ S J,WILDONGER J,HARRISON M M,et al.CRISPR/Cas9-mediated genome engineering and the promise of designer flies on demand[J].Fly(Austin),2013,7(4):249-255.
(编辑郭云雁)
Analysis of Sequence,Tissue Expression and Vector Construction ofOCA2 Gene in Bama Miniature Pigs
HONG Qian-long1,2,ZHANG Ying1,CAO Chun-wei1,WANG Xian-long1,ZHAO Jian-guo1*
(1.StateKeyLaboratoryofStemCellandReproductiveBiology,InstituteofZoology,ChineseAcademyofSciences,Beijing100101,China;2.SchoolofLifeSciences,AnhuiUniversity,Hefei230601,China)
Abstract:In order to establish scientific basis for farm animal models associated with coat color mechanism and human diseases inOCA2 gene,we carried out this experiment.The exons 20-24 of porcineOCA2 gene(NC_010457.4) in absence in GenBank were cloned and mapped.The mapping result was confirmed at the genomic and cDNA levels using PCR amplification and Sanger sequencing simultaneously.Bioinformatics analysis was performed forOCA2 gene.Polymorphic loci of all exons forOCA2 gene were screened in Guangxi Bama Miniature pigs.Meanwhile,the mRNA expression in different tissues was detected by real-time fluorescence quantitative PCR(Q-PCR).A full-length skin cDNA was used as a template to construct recombinant expression plasmid,and thenXhoⅠandSacⅡrestriction enzyme double digestion was carried out.We mapped and cloned the porcineOCA2 gene in GenBank successfully.A total of 10 SNP loci were found in all exons,including 7 synonymous mutations and 3 missense mutations(R124H,G509D,R573H).PorcineOCA2 gene was widely expressed in various tissues.The expression in lung,brain and cerebellum was at relatively higher level compared to that in spleen,stomach,caecum and skin,which exhibited moderate expression level,and the expression level in heart and liver was lower.pEGFP-N1-OCA2 recombinant plasmid was constructed successfully after confirmation of restriction enzyme double digestion and sequencing.This study explored porcineOCA2 gene sequence information and mRNA expression preliminarily,and recombinant expression plasmid ofOCA2 gene was constructed,which provide the foundation for the study of disease model and gene biological function in future.
Key words:pig;OCA2 gene;Q-PCR;recombinant expression plasmid;SNP
doi:10.11843/j.issn.0366-6964.2016.03.004
收稿日期:2015-03-30
基金项目:973国家科技重大专项(2011CBA01005);国家高技术研究发展计划(2012AA020602)
作者简介:洪潜龙(1988-),男,安徽潜山人,硕士,主要从事大动物遗传修饰研究,E-mail:hongqianlong.1988@163.com *通信作者:赵建国,研究员,E-mail:zhaojg@ioz.ac.cn
中图分类号:S828;S813.3
文献标志码:A
文章编号:0366-6964(2016)03-0439-10
