文蛤HDAC1基因克隆、时空表达及生长相关SNP位点筛查
2016-06-29高晓艳董迎辉姚韩韩林志华浙江万里学院浙江省水产种质资源高效利用技术研究重点实验室宁波315100
高晓艳 董迎辉 姚韩韩 林志华(浙江万里学院,浙江省水产种质资源高效利用技术研究重点实验室,宁波 315100)
文蛤HDAC1基因克隆、时空表达及生长相关SNP位点筛查
高晓艳 董迎辉 姚韩韩 林志华
(浙江万里学院,浙江省水产种质资源高效利用技术研究重点实验室,宁波 315100)
摘要:为探索HDAC1基因在文蛤生长发育中的作用,研究通过已构建的文蛤转录组文库,利用SMART RACE技术扩增得到文蛤HDAC1(Mm-HDAC1)基因的cDNA全长序列,分析了其生物信息学、组织及发育阶段表达特征,并用直接测序法在外显子中筛查生长相关的SNP位点。结果显示,Mm-HDAC1基因的cDNA全长3065 bp,开放阅读框1599 bp,编码532个氨基酸; 氨基酸序列比对发现,文蛤与其他物种同源性为74.3%—78.7%。荧光定量PCR(qRT-PCR)结果表明,Mm-HDAC1基因在文蛤6个组织中均有表达,其中外套膜中的表达量相对最高,并与其他组织间有极显著差异(P<0.01); 不同发育时期的表达差异结果表明,Mm-HDAC1基因在壳顶幼虫期表达量最高,显著高于其他发育时期(P<0.05)。SNP位点筛查结果表明,在Mm-HDAC1基因的外显子区域发现了19个SNP位点,其中有3个SNP位点(627A>T、924T>C和1266T>C)与文蛤生长相关。
关键词:文蛤; 组蛋白去乙酰化酶1; cDNA; 荧光定量; SNP
组蛋白去乙酰化酶1(Histone deacetylase 1,HDAC1)是HDACs家族中的重要一员,在卵母细胞成熟、胚胎发育、神经系统发生、骨骼分化、心脏发育、表皮分化、干细胞分化等过程中都发挥重要作用[1]。HDAC1是发现的第一个哺乳动物组蛋白脱乙酰化酶,推测其可能是基因转录的关键调节因子[2],可介导位点特异DNA结合的转录抑制子[3]。在高等脊椎动物中,Sun等[4]纯化并鉴定了鸡HDAC1蛋白,刘峰等[5]、柳小春等[6]证明猪HDAC1基因与生长及胴体性状相关,刘芳等[7]研究表明HDAC1与藏羚羊(Pantholops hodgsonii)的高原适应性可能有关,Pillai等[8]证明HDAC1基因与斑马鱼(Danio rerio)颅面软骨及鱼鳍的形成有关,李慧等[9]证明HDAC1基因在牙鲆(Paralichthys olivaceus)变态前以及变态阶段均有表达。而在海洋贝类中,仅在泥蚶(Tegillarca granosa)中见有基因克隆及时空表达研究的报道[10]。
文蛤(Meretrix meretrix)作为我国重要的经济养殖贝类之一,近些年来在分子遗传和功能基因研究方面已经取得了重要研究进展。免疫相关基因的报道有血清补体C1q[11]、脂多糖诱导的肿瘤坏死因子(LITAF)[12]、铜锌超氧化物歧化酶(SOB)[13]、金属硫蛋白(MT)[14]、脂多糖和β-1,3葡聚糖结合蛋白(LGBP)[15]、富含半胱氨酸小肠蛋白(CRIP)[16]等;生长相关基因方面有铁蛋白(Ferritin)[17]、组织蛋白酶B(Cathepsin B)[18]、磺基转移酶(SULT)[19]和果糖-1,6-二磷酸醛缩酶(FBA)[20]等,但HDAC1基因却未见报道。本研究首次克隆获得文蛤HDAC1(Mm-HDAC1)的cDNA全长,研究了其在不同组织、不同发育阶段中的表达特征,并在基因外显子中筛选与生长相关的SNP位点,以期为文蛤生长相关基因资源的挖掘利用奠定基础。
1 材料与方法
1.1 实验材料
文蛤亲贝取自宁波市鄞州区丹艳水产养殖基地,性腺发育成熟后进行催产,通过隔离产卵和人工授精技术获得同步发育的未受精卵、受精卵、4细胞胚胎、囊胚、原肠胚、担轮幼虫、D形幼虫、壳顶幼虫、眼点幼虫和稚贝10个发育时期样品,液氮速冻,存于-80℃冰箱,用于不同发育时期总RNA的提取。取3粒成贝,解剖取6个组织(外套膜、闭壳肌、内脏团、斧足、水管和鳃)样品,用于不同组织总RNA提取。用于SNP位点筛选的文蛤自然群体共500粒,随机取样100粒,分别测量总重、壳宽、壳长和壳高等生长性状,解剖取闭壳肌,液氮速冻,备用。
1.2 实验方法
Mm-HDAC1基因cDNA全长克隆 用Trizol法提取文蛤闭壳肌总RNA,微量紫外分光光度仪检测RNA浓度,1%琼脂糖凝胶检测RNA完整性。根据SMART RACE试剂盒((Clontech)说明,合成cDNA第一链,用于全长cDNA 3′和 5′端序列快速扩增(RACE)的模板。根据文蛤转录组文库(登录号:SRA021052)的注释信息,检索到Mm-HDAC1基因的EST片段,据此分别设计3′-RACE和5′-RACE特异性引物HDAC1-F和HDAC1-R(表 1),采用touch down PCR,分别进行3′-和5′-RACE扩增。PCR产物进行割胶纯化,纯化产物与pMD18-T载体(TaKaRa)连接,并将重组质粒转化到大肠杆菌DH-5α感受态细胞(TIANGEN)中,涂平板,进行蓝白斑筛选,阳性克隆经菌液PCR鉴定后送上海桑尼生物技术有限公司测序。
为保证Mm-HDAC1基因cDNA全长的准确性,以拼接3′-和5′-RACE测序结果获得的片段设计引物HDAC1-F1和HDAC1-R1,2×Taq MasterMix(北京康为世纪生物科技有限公司)进行PCR扩增,产物经割胶纯化,连接转化后测序,具体步骤同上。
Mm-HDAC1基因序列特征分析 将5′-RACE片段和3′-RACE片段序列进行拼接,用DNAMAN软件预测氨基酸序列,并推测氨基酸基本理化性质; ORF Finder程序搜索开放阅读框; 信号肽区域用SignalP4.1(http://www.cbs.dtu.dk/services/ SignalP/)预测; NetPhos2.0工具(http//www.cbs.dtu.dk/services/NetPhos/)预测氨基酸磷酸化位点; Prot Scale软件(http://web.expasy.org/protscale/)分析蛋白质疏水性; TMHMM软件(http://www.cbs.dtu.dk/services/TMHMM)预测蛋白质跨膜区; SMART 4.0(http://smart.emblheidel-berg.de/)预测蛋白结构域;Swiss Model软件(http://swissmodel.expasy.org/ workspace/)预测蛋白质高级结构; MEGA 6.0软件NJ法构建系统进化树; Clustal X进行氨基酸多序列比对。

表 1 实验所用引物及其序列Tab.1 Primers used in the study
Mm-HDAC1基因不同组织、不同发育时期的表达差异分析 Trizol法提取上述文蛤6个组织和10个发育时期样品的总RNA,反转录合成cDNA。由已获得的cDNA全长序列设计特异性引物Real-H-F和Real-H-R(表 1),18S rRNA 基因作为内参,利用ABI 7500Fast进行qRT-PCR,每个样本做3个平行。数据处理采用相对值2-ΔΔCt进行计算,SPSS 20.0进行统计分析。
Mm-HDAC1基因外显子SNP位点筛选 提取随机取样的100个文蛤样品的总RNA,反转录合成cDNA。根据获得的Mm-HDAC1基因的cDNA全长,设计3对特异性PCR引物HDAC1-F2和HDAC1-R2、HDAC1-F3 和HDAC1-R3、 HDAC1-F4和HDAC1-R4,以反转录合成的100个文蛤样本的cDNA为模板,进行PCR扩增,PCR产物直接送公司测序。结果用MEGA6.0进行序列比对,查找SNP位点,按照突变位点相对于起始密码子的位置分别命名,并用SPSS 20.0统计分析SNP位点与生长的相关性。
2 结果
2.1 Mm-HDAC1基因cDNA全长克隆
Mm-HDAC1基因cDNA全长3065 bp(GenBank登录号:KP250877),开放阅读框1599 bp,编码532个氨基酸,5′UTR长35 bp,3′UTR为1431 bp,包含一个终止密码子TAG,加尾信号AATAAA和26 bp的polyA尾巴。软件预测显示,Mm-HDAC1基因编码蛋白质的分子量为60.16 kD,理论等电点为5.55,表现为亲水性,没有明显的信号肽和跨膜区域。
SMART软件预测显示,Mm-HDAC1蛋白含有一段HDAC1高度保守的氨基酸序列(79 Ile-311Cys)。蛋白质高级结构预测显示,Mm-HDAC1蛋白由17个α-螺旋和17个β-折叠片组成,β-折叠片基本平行排列,这种排列方式可能对维持蛋白的稳定性具有重要作用(图 1)。NetPhos2.0软件预测显示,Mm-HDAC1蛋白有21个丝氨酸磷酸化位点,11个苏氨酸磷酸化位点和9个酪氨酸磷酸化位点。
氨基酸多序列比对显示,Mm-HDAC1基因与其他物种的同源性较高,为74.3%—78.7%,N端去乙酰化酶活性结构域(54Try-325Ala)与其他物种的同源性则高达92%以上(表 2)。从构建的系统进化树来看,文蛤与同为无脊椎动物的紫色球海胆(Strongylocentrotus purpuratus)亲缘关系较近,脊椎动物中的哺乳纲、鸟纲、两栖纲和鱼纲聚成一大支,与文蛤亲缘关系较远(图 2)。
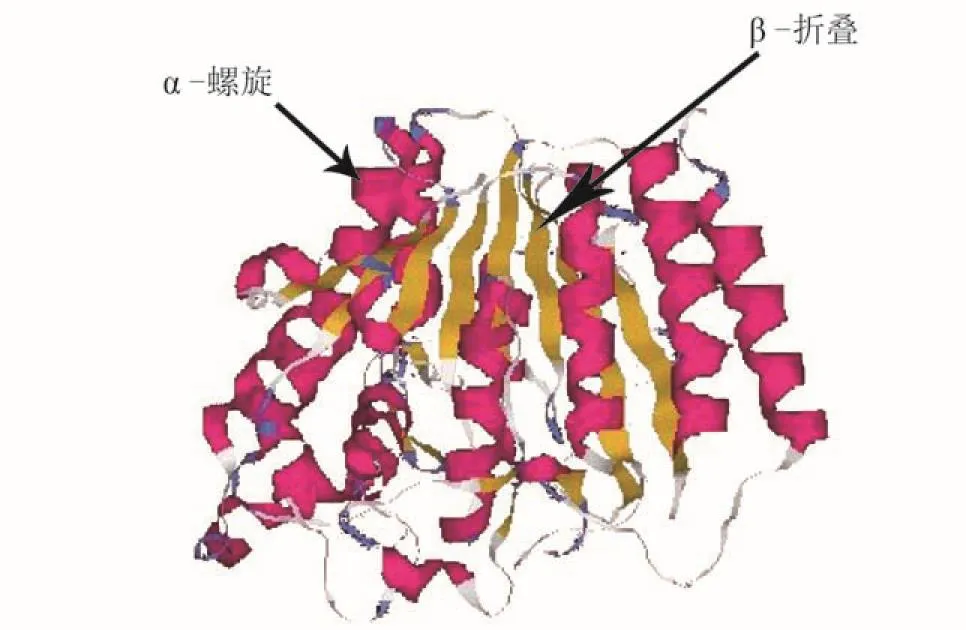
图 1 文蛤Mm-HDAC1蛋白的二级结构预测
2.2 Mm-HDAC1基因在文蛤不同组织和不同发育时期的表达差异分析
利用qRT-PCR技术,检测了Mm-HDAC1基因在6个组织和10个发育时期的时空表达特征。结果表明,Mm-HDAC1基因在文蛤6个组织中均有表达,其中外套膜中的表达量相对最高,并与其他组织之间有极显著性差异(P<0.01),足中的表达量仅次于外套膜,与其他组织之间也有极显著差异(P<0.01)(图3)。
Mm-HDAC1基因在文蛤幼虫的10个发育时期均有表达,从未受精卵开始表量呈逐渐上升的趋势,在壳顶幼虫中达到最高,并显著高于其他发育时期(P<0.05),到眼点幼虫和稚贝时期表达量降低,并与其他时期之间存在显著差异(P<0.05),但两组之间并无显著差异(P>0.05)(图 4)。
2.3 Mm-HDAC1基因外显子SNP位点筛查与分析

表 2 文蛤Mm-HDAC1氨基酸序列与其他物种HDAC1的同一性比对Tab.2 Comparison of amino acid sequences of Mm-HDAC1 of M.meretrix with others
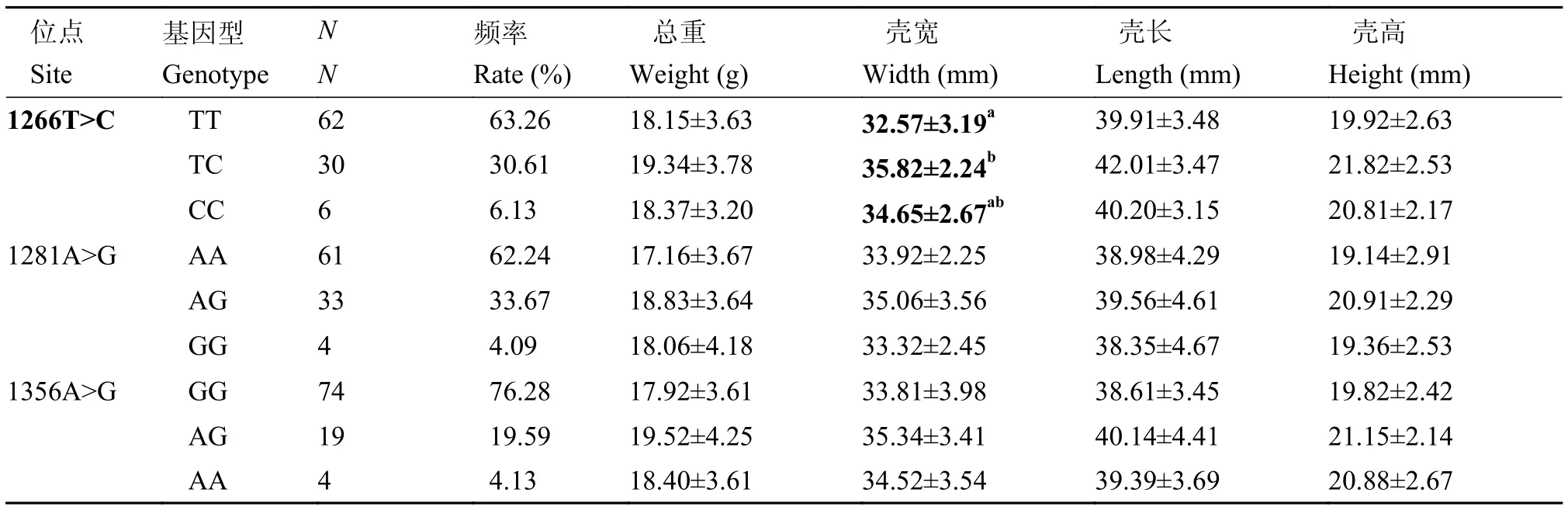
经序列比对,共筛查到19个SNP位点,将这些位点分别与生长性状(总重、壳长、壳高、壳宽)进行相关性分析。结果发现,3个位点627A>T、924T>C和1266T>C与生长性状显著相关,其中位点627A>T 的AT型个体总重和壳宽均显著高于AA型个体(P<0.05),位点924T>C的CC型个体壳宽显著高于TT型个体(P<0.05),位点1266T>C的TC型个体壳宽显著高于TT型个体(P<0.05),位点231T>G、255A>T/G、588A>T/C、591A>G、1114A>C、1158A>G、1194C>T、1221T>C、1266T>C、1281A>G和1356A>G中,杂合子的各生长性状均高于纯合子的平均值,但两组数据间无显著差异(P>0.05)(表 3)。

图 2 用MEGA6.0软件邻接法构建的文蛤与其他物种HDAC1的系统进化树
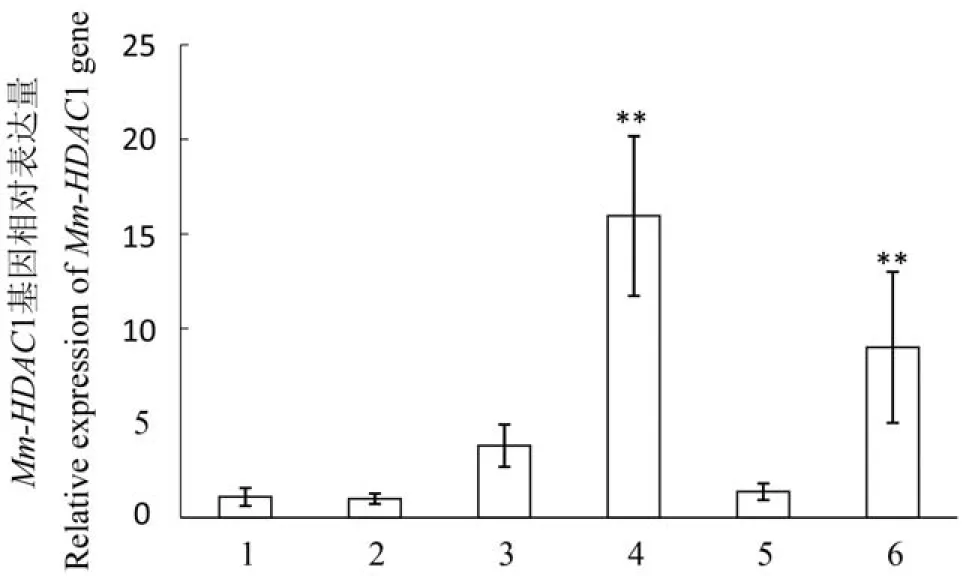
图 3 Mm-HDAC1基因在文蛤不同组织中的表达差异分析
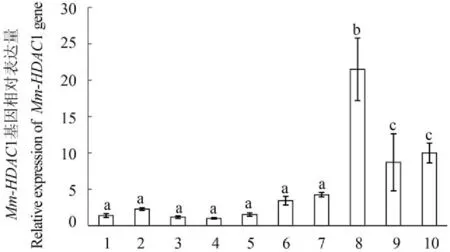
图 4 Mm-HDAC1基因在文蛤不同发育时期的表达差异分析
3 讨论
HDAC1基因可通过调节染色质结构和抑制特异性转录因子活性来调控细胞生长和分化,故在动物各个组织中均有表达。本研究通过qRT-PCR技术检测了Mm-HDAC1基因在文蛤各组织中的表达情况,结果表明,Mm-HDAC1基因在各组织中均有表达,其中外套膜中表达量最高,文蛤外套膜是由内外两层上皮细胞及其间的结缔组织和肌肉纤维组成[21],有研究表明HDAC1基因对哺乳动物上皮细胞分化起到很重要的作用[2 2],因此推断Mm-HDAC1基因在文蛤外套膜中上皮细胞的分化中发挥作用。另外Mm-HDAC1在足中表达量也很高,仅次于外套膜,并与其他组织间有显著差异。有研究显示,组蛋白乙酰化酶(HATs)和组蛋白去乙酰化酶(HDACs)以重塑染色质的方式直接或间接影响成肌分化抗原(MyoD)或肌细胞增强因子2(MEF2)及其下游基因的表达,进而调节肌肉的生肌过程[23,24],而文蛤足中肌肉含量很高,导致HDAC1在该组织中大量表达。
HDAC1基因在多种动物胚胎发育中的表达情况都有研究,研究表明几乎在所有物种的早期胚胎中都有表达。敲除HDAC1基因或HDAC1碳末端发生突变,都会导致小鼠早期胚胎发育异常,表现为胚胎生长阻滞、头部和尿囊发育异常,胚胎在妊娠10.5d死亡[25,26],说明HDAC1基因在生物的发育过程中发挥着重要的作用。本研究结果表明,Mm-HDAC1基因在各发育时期中均有表达,在未受精的卵子中就有表达,说明HDAC1基因是母源性的,Dufourcq等[27]在模式动物线虫的早期胚胎发育过程中发现HDAC1基因是母源的,胚胎基因组开始转录时就能检测到。而在研究动物后期胚胎发育的过程中,发现HDAC1基因在果蝇、斑马鱼、两栖动物的头部区域,包括中枢神经系统和大脑发育过程中都有所表达[28—30]。Mm-HDAC1基因在文蛤壳顶幼虫时期表达量达到最高,可能是壳顶幼虫时期是文蛤快速生长的时期,细胞分裂和组织器官分化速度加快,生命活动旺盛[31],需要更多HDAC1基因来调节胚胎发育过程中的基因转录。到眼点幼虫和稚贝时期,Mm-HDAC1的表达量又逐渐下降,可能是由于此时文蛤各组织、器官已基本形成,Mm-HDAC1基因的表达量逐渐达到稳定。
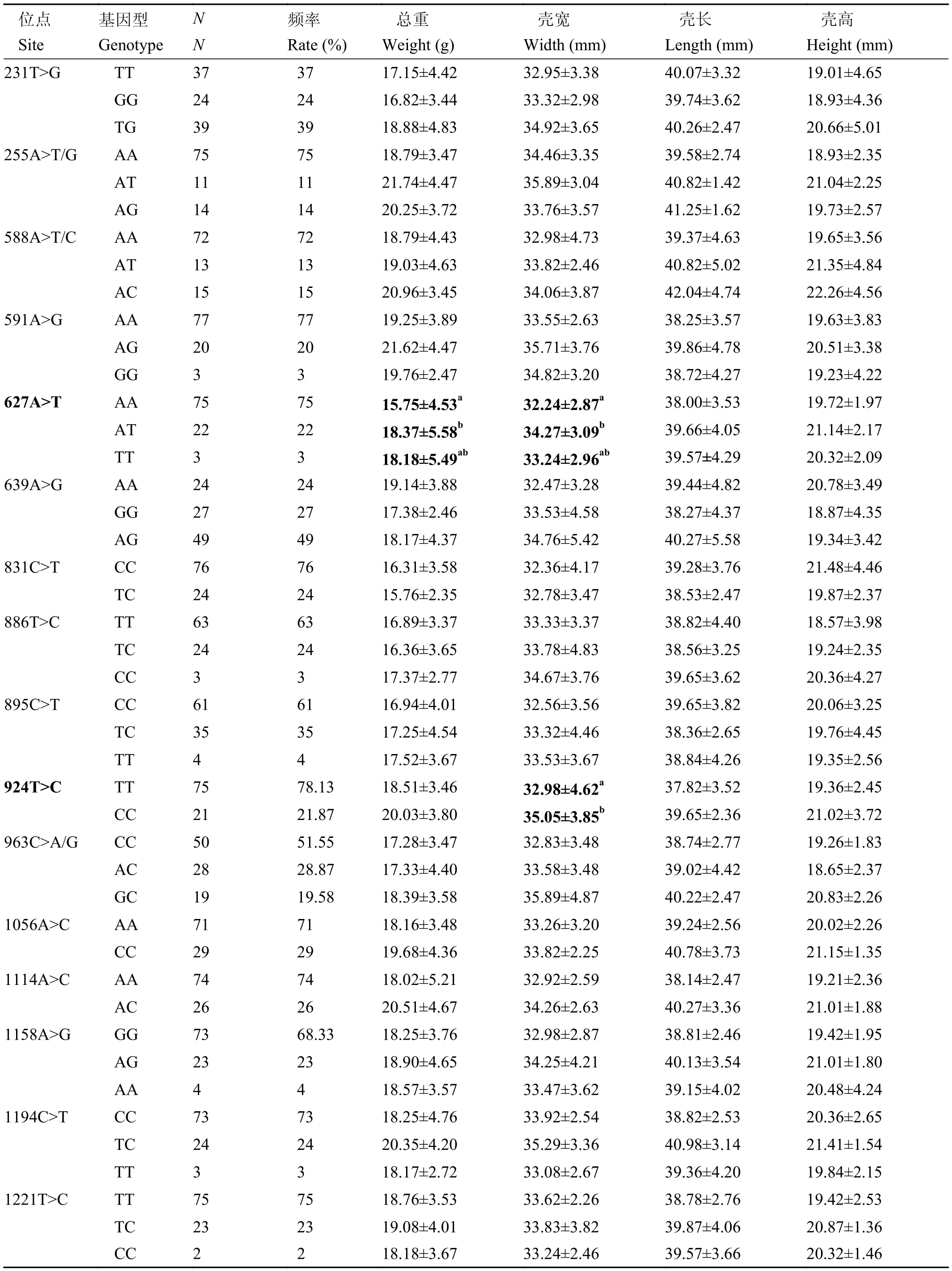
表 3 不同基因型文蛤的生长性状Tab.3 Growth traits of M.meretrix with different genotypes
续表
本研究利用直接测序的方法在Mm-HDAC1基因的外显子区域共筛查到19个SNP位点,3个位点(627A>T、924T>C 和1266T>C)与生长性状有相关性,其中位点627A>T 的AT型个体总重、壳宽和壳高均显著高于AA型个体(P<0.05),位点924T>C的CC型个体壳宽显著高于TT型个体(P<0.05),位点1266T>C的TC型个体壳宽显著高于TT型个体(P<0.05),提示HDAC1基因可能是贝类生长相关的潜在基因。有研究表明,在杜洛克猪种的HDAC1基因中,GG基因型的生长速度和生长性能指数明显优于AA型,等位基因G对生长具有正效应[5]。进一步分析发现,位点627A>T和924T>C位于Mm-HDAC1的乙酰化酶活性结构区域,该位点的突变可能与HDAC1发挥其活性有关,但其具体机制还需进一步深入研究。
参 考 文 献:
[1]Wang Z W.Function of HDAC1gene during the development of bovine oocytes and cloned embryos[D].Thesis for Doctor of Science,Jilin University,Changchun.2011[王中伟.HDAC1基因在牛卵母细胞和早期克隆胚胎发育中的作用.博士学位论文,吉林大学,长春.2011]
[2]Taunton J,Hassig C A,Schreiber S L.A mammalian histone deacetylase related to the yeast transcriptional regulator Rpd3p[J].Science,1996,272(8):408—411
[3]Hassiga C A,Fleischer T C,Billin AN,et al.Histone deacety-lase activity is required for full transcriptional repression by mSin3A[J].Cell,1997,89(3):341
[4]Sun J M,Chen H Y,Moniwa M,et al.Purification and characterization of chicken erythrocyte histone deacetylase 1[J].Biochemistry,1999,38(18):5939—5947
[5]Liu F.Study of on effects of HDAC1 gene and PIT-1 gene on partial economic traits in pigs[D].Thesis for Master of Science,Hunan Agricultural University,Changsha.2005[刘峰.HDAC1基因、PIT-1基因与猪部分经济性状相关性研究.硕士学位论文,湖南农业大学,长沙.2005]
[6]Liu X C,Xiao T Y,He W G,et al.Genetic effect of three loci related with growth and carcass traits in swine[J].Scientia Agricultura Sinica,2009,42(2):742—747[柳小春,肖调义,何伟光,等.猪生长及胴体性状3个相关基因座遗传效应.中国农业科学,2009,42(2):742—747]
[7]Liu F,Ge R L.Cloning and sequencing of HDAC1 coding cDNA sequence from Tibetan antelope(Pantholops hodgsonii)[J].Acta Theriologica Sinica,2011,31(4):396—403[刘芳,格日力.藏羚羊组蛋白去乙酰化酶1基因编码区的克隆与序列分析.兽类学报,2011,31(4):396—403]
[8]Pillai R,Coverdale L E,Dubey G,et al.Histone deacetylase 1(HDAC-1)required for the normal formation of craniofacial cartilage and pectoral fins of the zebrafish[J].Developmental Dynamics,2004,231(3):647—654
[9]Li H,Xu Y P,Bao Y P.Different expression pattern of HDAC1 before and during the process of metamorphosis in Paralichthys olivaceus[J].Journal of Shanghai Ocean University,2014,23(5):641—649[李慧,徐义平,鲍宝龙.牙鲆胚后发育阶段HDAC1基因的空间表达.上海海洋大学学报,2014,23(5):641—649]
[10]Zhou X L.Development and application of SSR and cloning of growth-related gene HDAC1 in Tegillarca granosa[D].Thesis for Master of Science,Shanghai Ocean University,Shanghai.2013[周小龙.泥蚶微卫星分子标记的开发与应用及生长相关基因HDAC1的克隆.硕士学位论文,上海海洋大学,上海.2013]
[11]Sui L J,Liu W D,Li Y F,et al.Molecular cloning,mRNA expression and bioassay of recombinant protein of C1q gene in hard clam(Meretrix meretrix)[J].Fisheries Science,2012,31(6):311—319
[12]Li H J,Yang Q,Gao X G,et al.Identification and expression of a putative LPS-induced TNF-a factor from Asiatic hard clam Meretrix meretrix[J].Molecular BiologyReports,2012,39(2):865—871
[13]Zhu D.Molecular cloning and mRNA expression and recombinant protein of an Cu/Zn-superoxide dismutase(Cu/Zn-SOD)gene from hard clam(Meretrix meretrix)[D].Thesis for Master of Science,Liaoning Normal University,Dalian.2011[朱丹.文蛤胞内铜锌超氧化物歧化酶的基因克隆与表达及重组蛋白活性的测定.硕士学位论文,辽宁师范大学,大连.2011]
[14]Gao X G,He C B,Li Y F,et al.Analysis on cloning and sequence Characteristics of metallothionein gene in hard clam Meretrix meretrix[J].Chinese Journal Fisheries,2009,22(4):8—11[高祥刚,赫崇波,李云峰,等.文蛤金属硫蛋白基因的克隆与分析.水产学杂志,2009,22(4):8—11]
[15]Liu S X,Qi Z H,Zhang J J,et al.Lipopolysaccharide and β-1,3-glucan binding protein in the hard clam(Meretrix meretrix):molecular characterization and expression analysis[J].Genetics and Molecular Research,2014,13(3):4956—4966
[16]Chen H,Yang X,Tang T,et al.The involvement of cysteine-rich intestinal protein in early development and innate immunity of Asiatic hard clam,Meretrix meretrix[J].Fish and Shellfish Immunology,2014,40(2):435—440
[17]Wang X M.Molecular cloning and functional analysis of genes involved in larval development in clam Meretrix meretrix[D].Chinese Academy of Sciences,Institute of Oceanology,Qingdao.2008[王晓梅.文蛤(Meretrix meretrix)幼虫生长发育相关基因的克隆和功能分析.博士学位论文,中国科学院海洋研究所,青岛.2008]
[18]Yao X L,Zhang J Q,Sun J S,et al.Recombinant expression,characterization and expressional analysis of clam Meretrix meretrix cathepsin B,an enzyme involved in nutrient digestion[J].Molecular Biology Reports,2011,38(3):1861—1868
[19]Wang C,You Y N,Wang H X,et al.Genetic diversity of the sulfotransferase-like gene and one nonsynonymous SNP associated with growth traits of clam,Meretrix meretrix[J].Molecular Biology Reports,2012,39(2):1323—1331
[20]Wang C,Wang H X,Li Y,et al.Identification of a fructose-1,6-bisphosphate aldolase gene and association of the single nucleotide polymorphisms with growth traits in the clam Meretrix meretrix[J].Molecular Biology Reports,2012,39(4):5017—5024
[21]Cheng L,Wang K L.Studies on the morphology and ultrastructure of Meretrix meretrix Linnaeus[J].Zoological Research,1997,18(4):351—357[程量,王康乐.文蛤形态学及其亚显微结构的研究.动物学研究,1997,18(4):351—357]
[22]Tou L,Liu Q,Shivdasani R A.Regulation of mammalian epithelial differentiation and intestine development by class I histone deacetylases[J].Molecular and Cellular Biology,2004,24(8):3132—3139
[23]Puri P L,Iezzi S,Stiegler P,et al.Class I histone deacetylases sequentially interact with MyoD and pRb during skeletal myogenesis[J].Molecular Cell,2001,8(4):885—897
[24]Mal A,Harter M L.MyoD is functionally linked to the silencing of a muscle-specific regulatory gene prior to skeletal myogenesis[J].Proceedings of the National Academy of Sciences of the USA,2003,100(4):1735—1739
[25]Lagger G,O'Carroll D,Rembold M,et al.Essential function of histone deacetylase 1 in proliferation control and CDK inhibitor repression[J].The EMBO Journal,2002,21(11):2672—2681
[26]Montgomery R L,Davis C A,Potthoff M J,et al.Histone deacetylases 1 and 2 redundantly regulate cardiac morphogenesis,growth,and contractility[J].Genes and Development,2007,21(14):1790—1802.
[27]Dufourcq P,Victor M,Gay F,et al.Function requirement for histone deacetylase 1 in Caenorhabditis elegans gonadogenesis[J].Molecular Cellular Biology,2002,22(9):3024—3034
[28]Mannerrvik M,Levine M.The Rpd3 histone deacetylaseis required for segmentation of the Drosophila embryo[J].Proceedings of the National Academy of Sciences of the USA,1999,96(12):6797—6801
[29]Nambiar R M,Henion P D.Sequential antagonism of early and late Wnt-signaling by zebrafish colgate promotes dorsal and anterior fates[J].Developmental Biology,2004,267(1):165—180
[30]Damjanovski S,Sachs L M,Shi Y B.Multiple stage-dependent roles for histone deacetylases during amphibian embryogenesis:implications for the Involvement of extracellular matrix remodeling[J].International Journal of Developmental Biology,2000,44(7):769—776
[31]Wang R C,Wang Z P.Science of Marine Shellfish Culture[M].Qingdao:China Ocean University Press.2008,28—29[王如才,王昭萍.海水贝类养殖学.青岛:中国海洋大学出版社.2008,28—29]
CLONING,SPATIOTEMPORAL EXPRESSION AND SNPS IDENTIFICATION OF HDAC1 GENE IN HARD CLAM MERETRIX MERETRIX
GAO Xiao-Yan,DONG Ying-Hui,YAO Han-Han and LIN Zhi-Hua
(Key Laboratory of Aquatic Germplasm Resource of Zhejiang,Zhejiang Wanli University,Ningbo 315100,China)
Abstract:Mm-HDAC1 cDNA was cloned by SMART RACE techniques using 454 cDNA library of Meretrix meretrix.The bioinformatics and expression profiles in different tissues and developmental stages were analyzed,and SNPs were screened.The results indicated that the full length cDNA of Mm-HDAC1 gene was 3065 bp,containing a complete 1599 bp ORF that encodes 532 amino acids.Comparison of animal acid sequence,M.meretrix shared 74.3%—78.5% identity with others,suggesting that Mm-HDAC1 was highly conservative.The result of qRT-PCR showed that Mm-HDAC1 expressed in all six tissues,and its expression was significantly higher in mantle than other tissues(P<0.01).Moreover,the expression of Mm-HDAC1 gradually increased with the process of the development with the highest level in umbo larvae stage(P<0.01).19 SNPs in exons of Mm-HDAC1 were identified and 3 SNPs were potentially associated with M.meretrix growth(627A>T,924T>C and 1266T>C).
Key words:Meretrix meretrix; Histone deacetylase1(HDAC1); cDNA; qRT-PCR; SNP
中图分类号:Q785; S968.3
文献标识码:A
文章编号:1000-3207(2016)03-0601-08
doi:10.7541/2016.81
收稿日期:2015-07-14;
修订日期:2015-12-20
基金项目:国家自然科学基金项目(31372527); 国家现代贝类产业技术体系项目(CARS-48); 浙江省重大科技专项(2012C12907-4); 国家水产种质资源平台项目(2015DKA30470)资助[Suppprted by National Natural Science Foundation of China(31372527); Modern Agro-industry Technology Research Systerm(CARS-48); Zhejiang Major Program of Science and Technology(2012C12907-4)and National Infrastructure of Fishery Germplasm Resource Programme(2015DKA30470)]
作者简介:高晓艳(1989—),女,河南安阳人; 硕士研究生; 主要从事海洋贝类遗传育种研究。E-mail:yanyuninan6264@163.com
通信作者:董迎辉(1980—),副研究员; 硕导; 主要从事贝类遗传育种研究。E-mail:dongyinghui118@126.com; 林志华(1965—),研究员;博导; E-mail:zhihua9988@126.com
