来源于大肠杆菌的NMN转移酶的克隆表达与酶学性质研究
2016-06-24段琳琳李红梅
段琳琳, 李红梅
上海理工大学医疗器械与食品学院,上海 200093
来源于大肠杆菌的NMN转移酶的克隆表达与酶学性质研究
段琳琳,李红梅*
上海理工大学医疗器械与食品学院,上海 200093
摘要:在生物体内,NMN(烟酰胺单核苷酸)转移酶能够催化NMN生成NAD。本研究通过构建重组表达质粒pET30α(+)-Nmnat,成功实现来源于大肠杆菌的NMN转移酶基因(Nmnat)的原核表达。从大肠杆菌基因组中克隆得到的NMN转移酶基因长度为1 245 bp,所编码的重组酶分子量 45 kDa。对重组酶的酶学性质进行分析,结果显示该酶最适反应温度和pH分别为37 ℃和7.5。4 ℃下,该酶的热失活半衰期可长达990.2 min。Mn2+、Fe2+对该酶的酶活的激活作用显著,而EDTA对酶活能造成明显的抑制作用。酶动力学分析结果显示,该酶对底物NMN催化的Km和Vmax分别为16.89 mmol/L和2.46 μmol/(L·min)。该NMN转移酶基因在大肠杆菌宿主中的成功表达,为NAD生物合成应用研究奠定了基础。
关键词:大肠杆菌; NMN转移酶; 克隆表达; 酶学性质
烟酰胺腺嘌呤二核苷酸(NAD,简称辅酶I),作为一种转递质子辅酶[1],参与生物体内的多种 氧化反应,如细胞物质代谢、能量合成、细胞DNA修复等[2-3],在细胞生命活动起着至关重要的作用[4-5]。在生物代谢途径中, NAD的合成主要以烟酰胺单核苷(NMN) 和ATP为前体,在NMN转移酶催化作用下,生成NAD和焦磷酸盐。
研究发现NMN转移酶是唯一一种能够催化细胞核内NAD合成的生物酶,其类型有3种,分别是Nmnat1、Nmnat2和Nmnat3,且其分子量大小,性质以及特征随着在组织细胞中的分布位置不同出现差异[6-7]。NMN转移酶对细胞的存活也十分重要,可以降低对由基因引起的轴突变性[8-11],加快真核细胞的有丝分裂, 从而降低生物细胞的衰老和死亡[12],也可作为生物药剂的靶标用于提高抗肿瘤药物的生物活性[13-14]。
目前NMN转移酶研究主要集中于酶活测定、酶分子结构、催化反应机制和体内代谢途径等方面,而对酶基因进行重组表达的报道却相对较少[15]。本文运用PCR技术从大肠杆菌中获取NMN转移酶基因,构建表达NMN转移酶的重组菌,以期获得具有良好催化性能的NMN转移酶,并对重组酶的酶学性质进行研究,为NAD生物合成应用奠定基础。
1材料与方法
1.1材料
1.1.1菌株、质粒和试剂
大肠杆菌BL21(DE3)、质粒pET30α(+)由实验室保存。
1.1.2工具酶和试剂
TaqDNA聚合酶、限制性内切酶BamHI和NdeI、T4DNA连接酶、蛋白质标准分子量、DNA标准分子量购于TaKaRa公司。异丙基硫代-β-D-半乳糖苷(IPTG)购自上海生工生物工程公司。其他试剂均为国产分析纯。
1.1.3培养基
种子培养基:采用LB培养基。磷酸盐缓冲液(g/L):KH2PO423.1,K2HPO4125.4。121 ℃下灭菌20 min。发酵培养基:采用TB培养基。121 ℃下灭菌20 min。冷却至60 ℃后再添加磷酸盐缓冲液。固体培养基中加2%的琼脂。培养基中按需添加终浓度为30 μg/mL的硫酸卡那霉素。
1.2方法
1.2.1NMN转移酶基因的克隆
以大肠杆菌的基因组DNA为模板(GI:61676796),设计引物PCR扩增NMN转移酶(Nmnat)编码基因。上游引物P1:5’-CATCATATG TAGTCCTTATGCCTCCTCT-3;下游引物P2:5’-GTAGGATCCTAGAGCTGTC CGGAGAAGGTCT-3’,引物两端分别引入BamHI和NdeI酶切位点,具体操作参见文献[16]。PCR扩增条件:95 ℃,3 min,50 ℃,2 min, 72 ℃,1 min,35个循环,72 ℃延伸2 min。PCR产物跑电泳切胶回收目的片段。
1.2.2大肠杆菌表达载体的构建
将目的基因片段用BamHI和NdeI双酶切后切胶回收,再与双酶切后切胶回收的pET30α(+)载体片段在16 ℃下连接过夜,连接产物转化E.coliBL21(DE3)感受态细胞,涂布抗性平板,37 ℃下培养过夜。挑取阳性转化子提质粒并送上海生工测序,含正确克隆序列的质粒命名为pET30α(+)-Nmnat。
1.2.3目的基因在大肠杆菌中的诱导表达
将含有重组表达质粒pET30α(+)-Nmnat的E.coliBL21(DE3)单菌落接种于10 mL的LB液体管(含30 μg/mL的硫酸卡那霉素)中,37 ℃,200 r/min振荡培养过夜,然后以1%接种量接入100 mL TB液体培养基(含30 μg/mL硫酸卡那霉素)中,37 ℃,200 r/min振荡培养至OD600为0.5~0.7时,加入IPTG诱导剂至终浓度30 μg/mL,在25 ℃下诱导表达8 h,离心收集菌体用50 mmol/L的磷酸盐缓冲溶液(pH 7.5)重悬,冰上超声破碎(4 s,间隔6 s,60次,功率300 W),然后4 ℃,12 000 r/min下离心20 min去除细胞碎片,取上清进行SDS-PAGE分析并测酶活。
1.2.4SDS-PAGE分析
制备10%的分离胶和5%的浓缩胶,在80 V和120 V的电压下进行不连续垂直平板电泳,凝胶用考马斯亮蓝染色。
1.2.5NMN转移酶活力测定
NAD酶活力的检测原理:

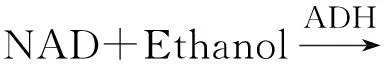
反应体系:1 mol/L pH 7.5 Tris-HCl缓冲液,300 mmol/L NMN,50 mmol/L ATP,50 mmol/L Mn2+,乙醇2%,适量酶液,7U的ADH(乙醇脱氢酶),混匀,置37 ℃水浴中振荡反应2 h,加0.155 mol/L的EDTA终止酶反应,5 min后冰浴冷却,8 000 r/min下离心1 min,上清液在340 nm处测定吸光值,以OD340代表NMN转移酶酶活力大小。酶活单位(U)定义:37 ℃,pH 7.5条件下,1 min催化1 μmol NMN所需的酶量。比酶活用U/mg或U/g表示。
1.2.6重组酶性质分析
酶的最适温度研究:分别于20 ℃、30 ℃、37 ℃、45 ℃、50 ℃、55 ℃、60 ℃、70 ℃、80 ℃下测定酶活,确定酶的最适反应温度。
酶的热失活曲线与失活动力学和热力学参数计算:
将酶分别置30 ℃、37 ℃、50 ℃下保温,每隔30 min冰浴冷却离心取样测定残余酶活,绘制酶热失活曲线。根据公式(1)~(3)计算出热失活速率常数Ki、半衰期t1/2和吉布斯自由能ΔG。
(1)
失活半衰期公式t1/2=ln2/Ki
(2)
吉布斯自由能的计算公式[15]为
ΔG=-2.303RTlg(Kih/KBT)
(3)
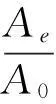
酶的最适pH研究:在不同pH(pH 4.0~10.0,1 mol/L的缓冲液)下测定酶活,确定酶的最适反应pH。
酶的pH稳定性:酶在37 ℃下,1 mol/L的缓冲液(pH 4.0~10.0)中保温2 h后冰浴冷却5 min测定残余酶活。
金属离子和EDTA对酶活力的影响:在最适反应温度和最适反应pH下,分别添加不同金属离子和EDTA(终浓度50 mmol/L),分别测定其对酶促反应的影响。以不加金属离子或EDTA作为对照。
反应动力学常数Km及Vmax的测定:调整反应体系中底物NMN的浓度为1 mmol/L、2 mmol/L、3 mmol/L、4 mmol/L和5 mmol/L,测定NMN转移酶的酶活,根据底物浓度和相应的反应初速率,用Michaelis-Menten方程计算米氏常数Km和最大反应速率Vmax值。
2结果与分析
2.1目的基因克隆和重组表达质粒构建
从大肠杆菌基因组中PCR扩增出NMN转移酶编码基因,阳性转化子提取质粒双酶切验证(图1),正确者送测序,结果显示克隆片段长1 245 bp,与大肠杆菌的Nmnat基因序列同源性为99%,表明重组表达质粒构建成功。
将重组质粒pET30α(+)-Nmnat转入E.coliBL21(DE3)感受态细胞中,进行目的基因诱导表达,将表达后的细胞破碎液上清进行SDS-PAGE分析。如图2所示,以空载质粒为对照,在45 kDa处出现特征条带,且与目标蛋白的理论计算值吻合,说明目的基因在大肠杆菌宿主中成功实现了异源表达。
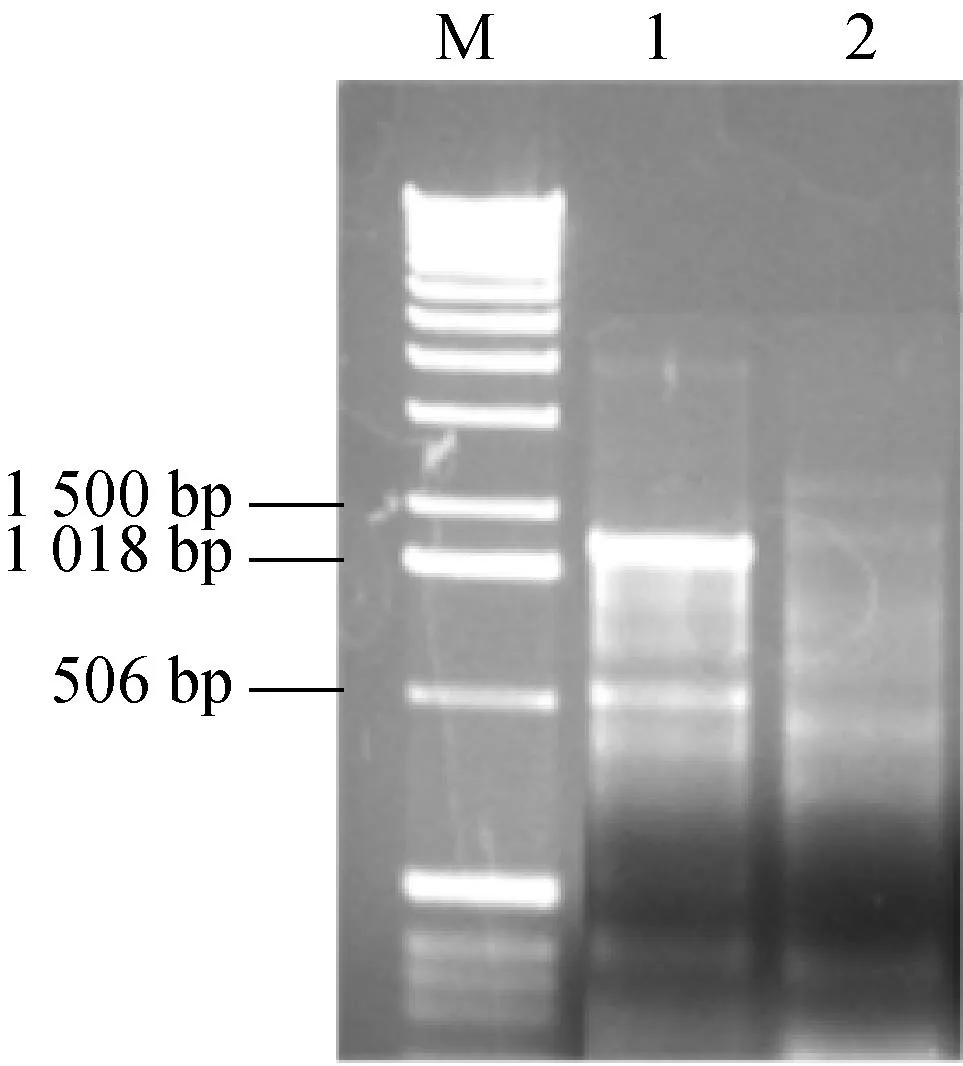
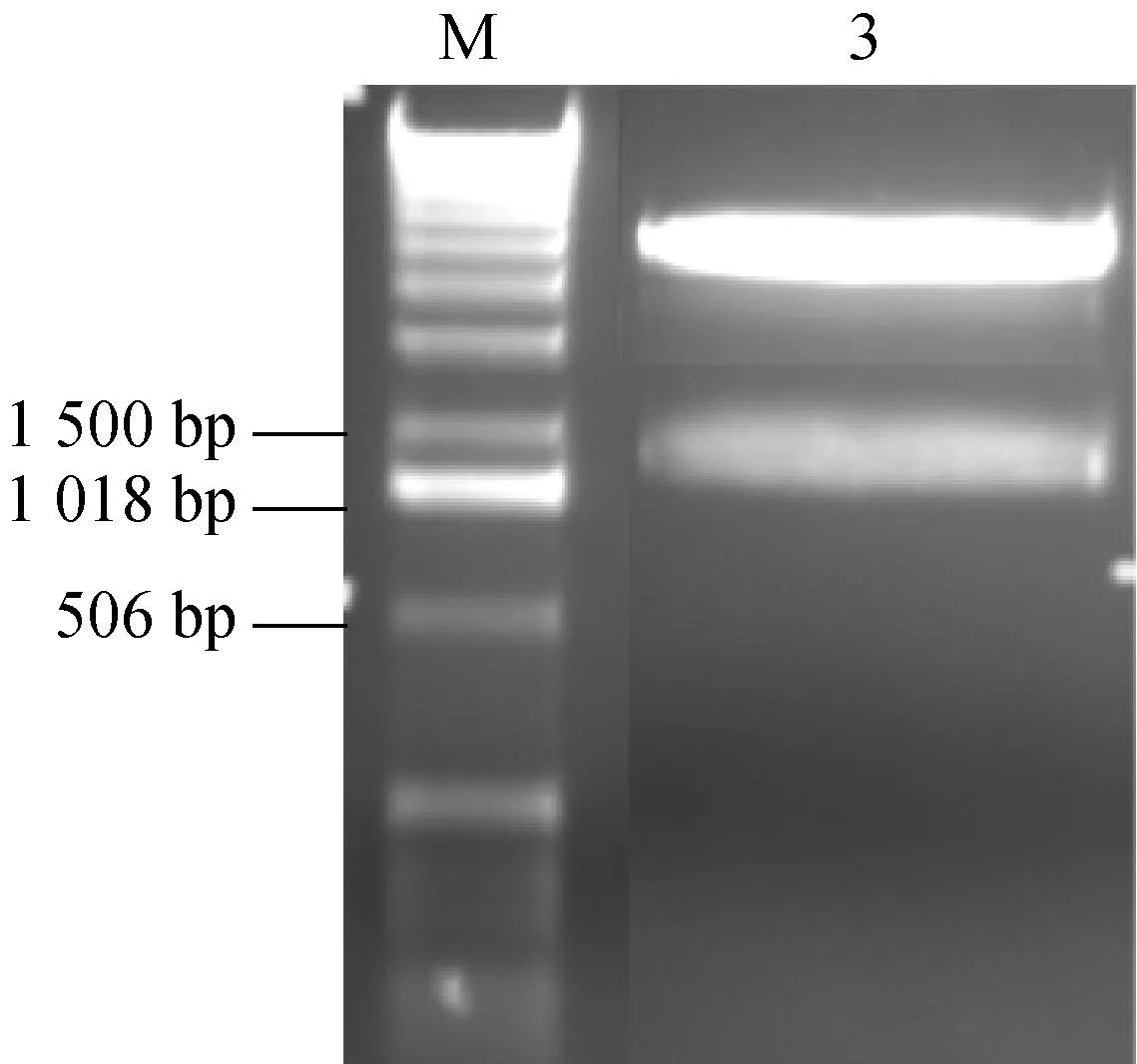
M: DNA相对分子质量标准;1:PCR产物;2:阴性对照;3:重组表达质粒pET30α(+)-Nmnat经BamHI和NdeI双酶切后的产物
图1琼脂糖电泳检测DNA
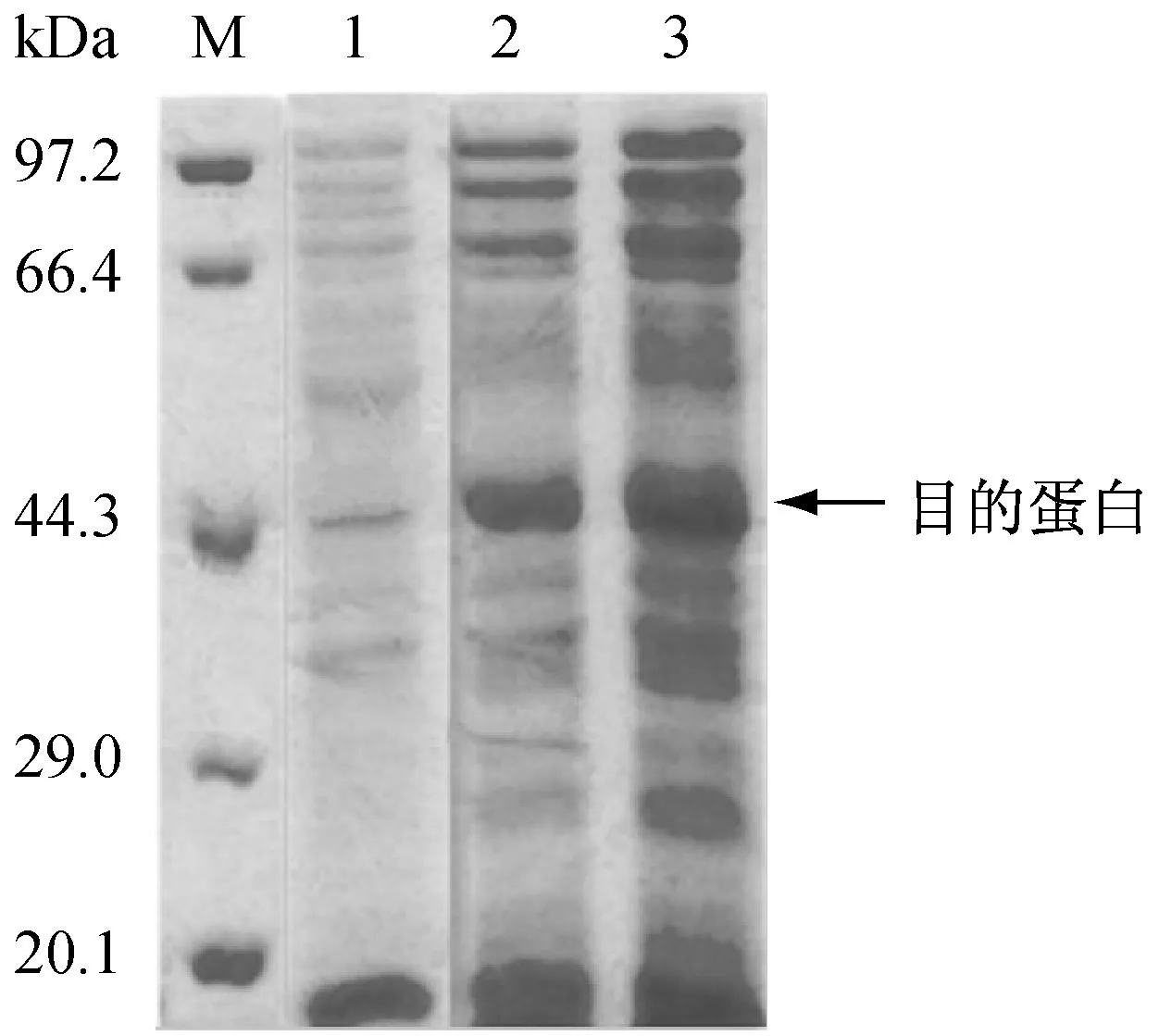
泳道M:蛋白质相对分子质量标准;泳道1:E.coliBL21(DE3)经超声破碎后的上清;泳道2:稀释10倍诱导表达8 h后,含pET30α(+)的E.coliBL21(DE3)经超声破碎后的上清;泳道3:稀释5倍诱导表达8 h后,含pET30α(+)-Nmnat的E.coliBL21(DE3)经超声破碎后的上清
图2目的基因在E.coliBL21(DE3)中的表达
2.2重组酶的酶学性质研究
2.2.1重组酶的最适反应温度
如图3所示,在20 ℃~37 ℃内,随温度升高,酶活性逐渐升高;当反应温度超过37 ℃,酶活性迅速下降。因此确定该酶最适反应温度为37 ℃,与已报道的其它来源的NMN转移酶的最适温度相同[ 17-18]。

图3 重组酶的最适反应温度
2.2.2重组NMN转移酶的热失活动力学和热力学分析
结果如图4和图5所示。从图中可看出,重组NMN转移酶的失活符合一级失活反应规律。热失活速率常数和半衰期自由(ΔG)的结果如表1所示。该酶在4 ℃时半衰期长达990.2 min,而60 ℃时,酶的稳定性较差,半衰期仅为2.5 min。文献数据显示,吉布斯自由能的数值大小与酶的热稳定性相关。ΔG越高,代表酶的稳定性越好[15]。因此,从吉普斯自由能角度可发现,在20 ℃、30 ℃、37 ℃时,重组NMN转移酶的ΔG较高,这也进一步说明,在低温时该酶的稳定性较好,当温度高于37 ℃时,ΔG开始降低,表明该酶的稳定性下降。
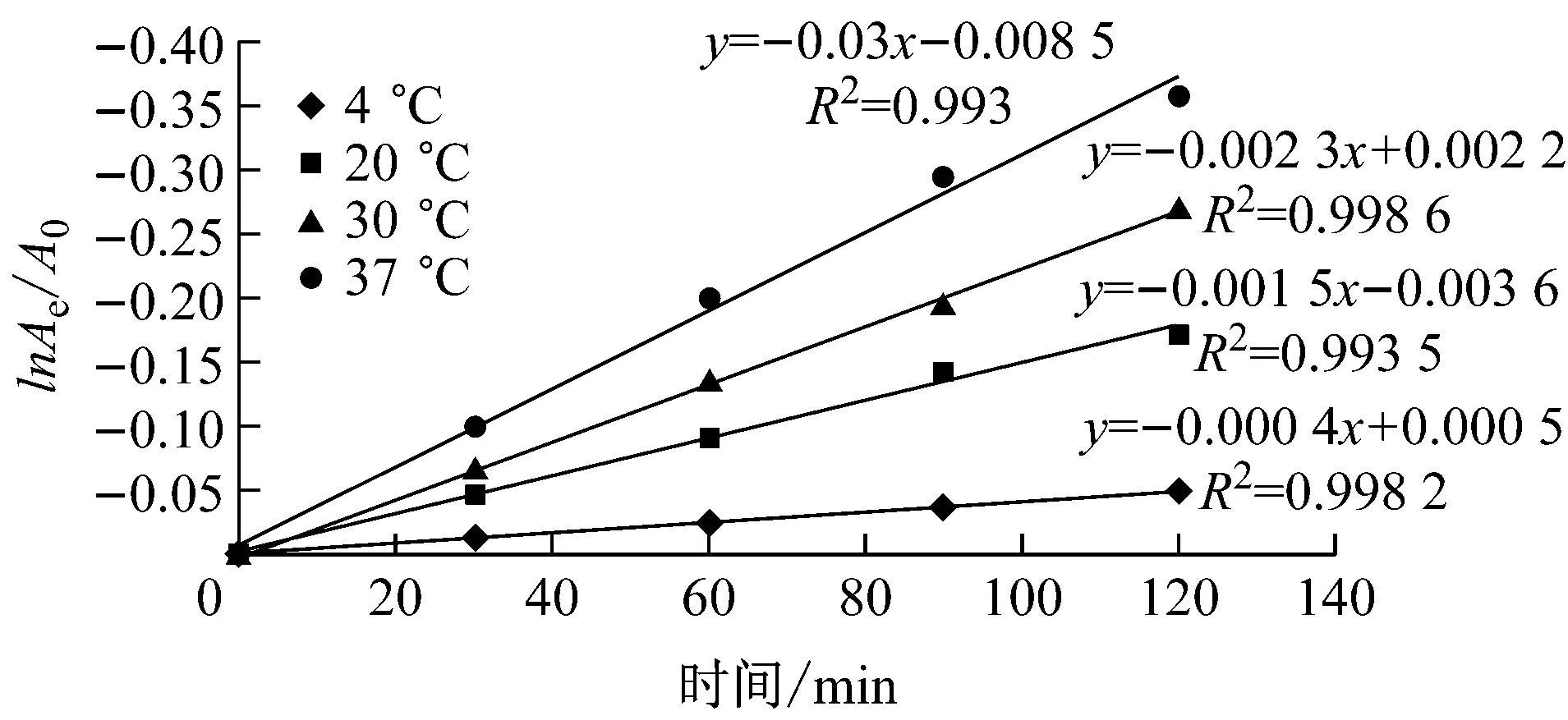
纵坐标:相对残留酶活力的对数;曲线斜率:热失活的一级速率常数Ki
图4重组NMN转移酶的热稳定性
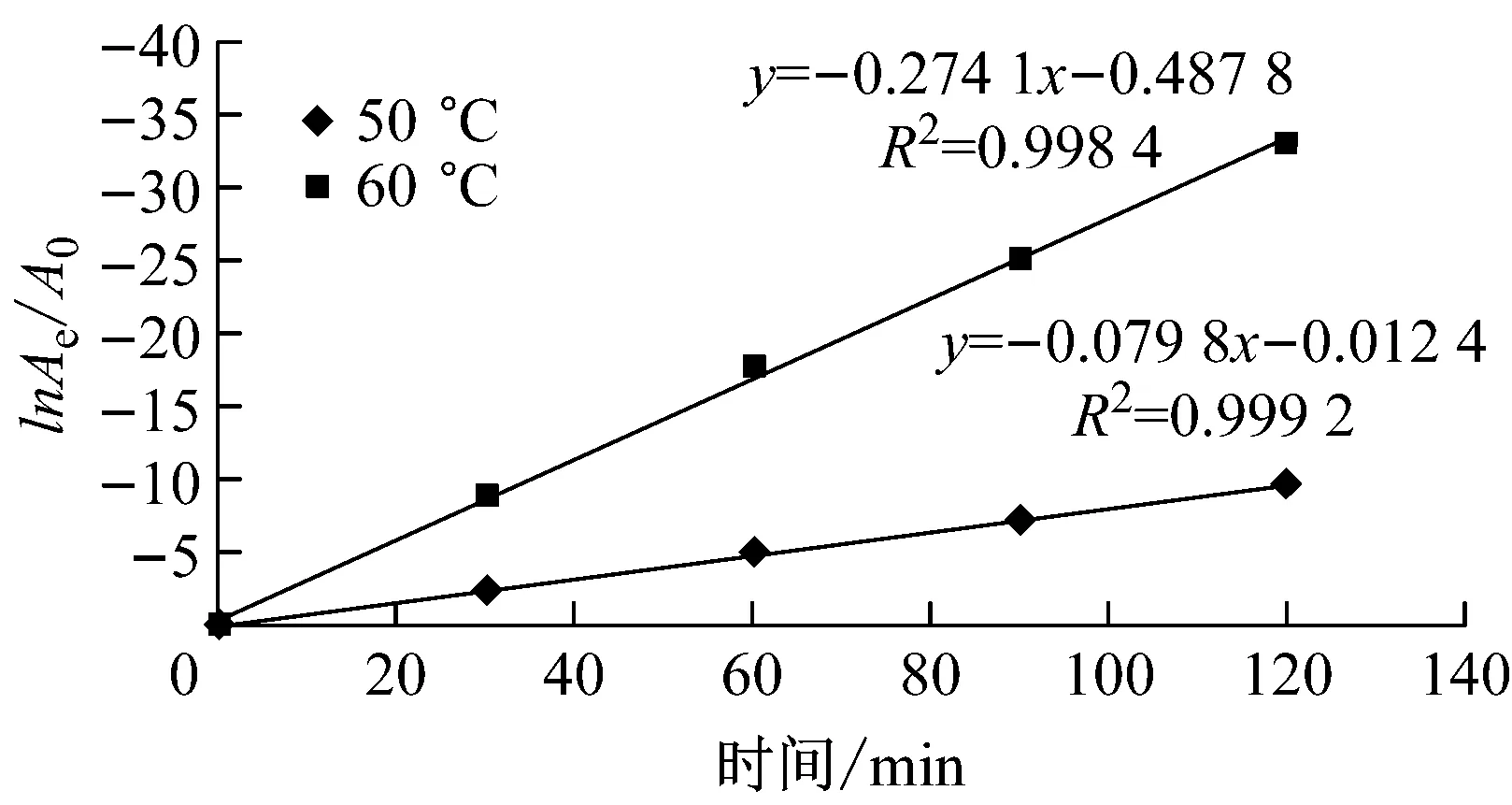
ln Ae/A0:相对酶活力的对数;曲线斜率:热失活的一级速率常数Ki
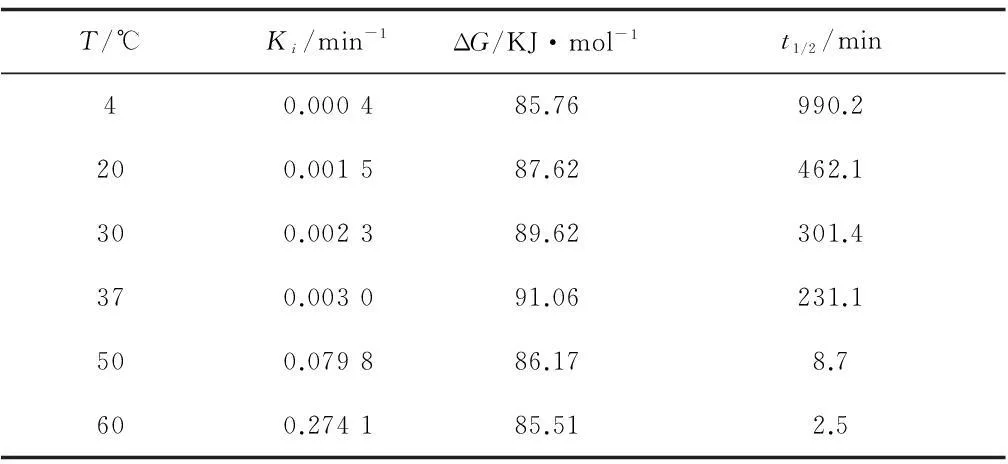
T/℃Ki/min-1ΔG/KJ·mol-1t1/2/min40.000485.76990.2200.001587.62462.1300.002389.62301.4370.003091.06231.1500.079886.178.7600.274185.512.5
2.2.3重组酶的最适反应pH
环境pH可影响酶催化活性位点上相关基团的解离及底物的解离状态,进而影响酶的催化性能。本研究考察了该重组NMN转移酶在不同的pH值下催化NMN转化为NAD的变化情况。如图6所示,重组酶最适反应pH为7.5,当pH大于8时酶活迅速降低。该重组酶的最适反应pH与炭疽杆菌细胞NMN转移酶最适pH相同[19]。
2.2.4重组NMN转移酶的pH稳定性
反应体系的pH可以影响酶的空间构象和稳定性[19]。重组NMN转移酶的pH稳定性如图7所示,pH 7.0~8.0时酶的稳定性较好,酶活性保持在80%以上;当pH低于5.5或高于9时,酶的活性明显降低。这可能是由于在偏酸或偏碱的环境中,重组酶的空间构象发生改变,导致酶活降低。

图6 重组NMN转移酶的最适反应pH
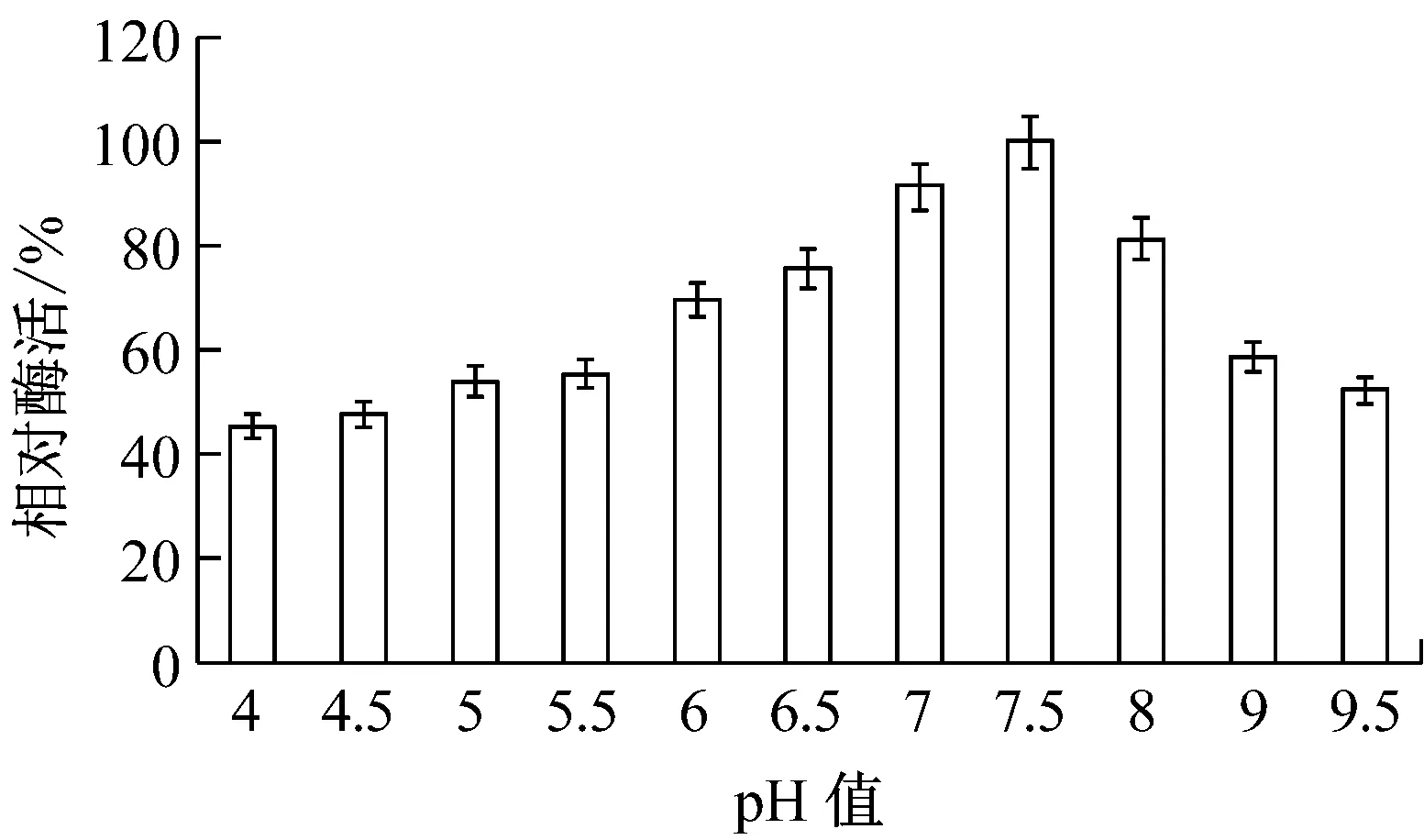
图7 pH对重组NMN转移酶稳定性的影响
2.2.5金属离子及EDTA对酶活的影响
某些酶需要金属离子作为辅助因子[20]。本研究考察了不同的金属离子及EDTA对酶活的影响。如图8所示,Fe2+和Mn2+能显著提高重组NMN转移酶的活性;而Ca2+、Zn2+、Mg2+对酶活促进作用不明显;Fe3+起抑制酶活的作用;当在反应体系中添加金属螯合剂EDTA,酶活显著下降。
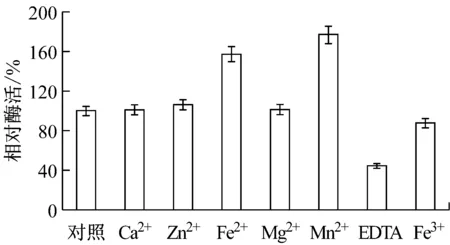
图8 金属离子及EDTA对酶活的影响
2.2.6重组NMN转移酶的反应动力学常数测定
本研究采用双倒数法测定重组NMN转移酶的Km及Vmax,结果如图9所示。该重组酶的Km为16.89 mmol/L。该重组酶的Km值与来源于哺乳动物细胞核内的NMN转移酶的Km接近[2],但低于来源于人体组织的NMN转移酶的Km[7]及来源于炭疽杆菌细胞的NMN转移酶的Km[18](表2)。
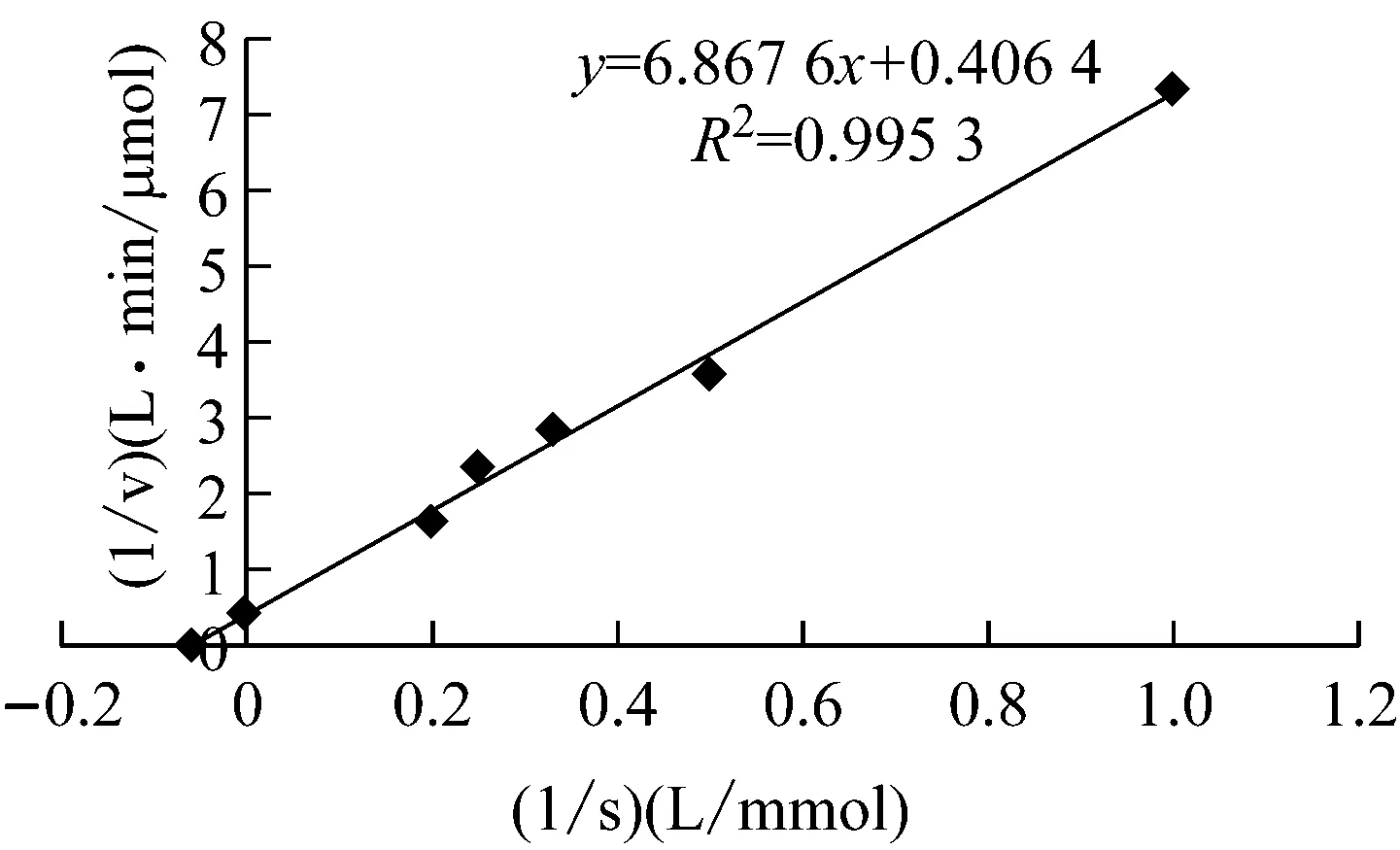
图9 重组NMN转移酶的反应动力学常数测定

来源最适反应pH最适反应温度/℃Kcat/s-1Km分子量Mr/kDa大肠杆菌7.53712.1316.8945哺乳动物细胞核人体啤酒酵母炭疽杆菌细胞7.56.0-9.05.0-9.07.53737373710.73--16.22±2/20.67±1.914.660.023/0.091-0.5435[2]31.9/34.4[7]48[17]49[18]
3结论
(1) 本研究利用基因工程技术将重组表达质粒pET30α(+)-Nmnat转入E.coliBL21(DE3)中进行目的基因的异源表达。SDS-PAGE分析显示,经IPTG诱导8 h后,目的基因成功表达,重组酶分子量45 kDa。
(2) NMN转移酶有三种同源体,不同来源的性质差异显著。本文深入研究来源于大肠杆菌的重组NMN转移酶的酶学性质,重组酶最适反应温度和pH分别为37 ℃、7.5。Mn2+和Fe2+对该酶具有明显的激活作用,而EDTA则可以显著抑制酶活性。另外,通过动力学分析热失活常数、pH稳定性及酶动力学参数测定,以及与不同来源的NMN转移酶进行比较,结果表明,该重组酶对底物NMN的亲和力水平与来源于哺乳动物细胞核的NMN转移酶相近,但远低于来源于人体的NMN转移酶,后续可考虑采用蛋白质工程技术进一步增强该酶的底物亲和力。
本研究成功构建了能高效表达大肠杆菌NMN转移酶的基因工程菌,并对该重组酶的酶学性质进行了系统研究,为后续NAD生物合成提供了有价值的技术参考。
参考文献
[1]Penberthy WT. Nicotinamide adenine dinucleotide biology and disease.CurrPharmaceut Design, 2009,15: 1-2.
[2]吕小群,张偌瑜,徐学文等. 烟酰胺单核苷酸腺苷转移酶的表达、纯化及活性测定[J].第二军医大学学报, 2010,11(31):1251-1254.
[3]Koubova J. Guarente L. How does calorie restriction work.GeneDev, 2003,17(3):313-321.
[4]Hassanain HH, Chon SY, Gupta SL. Differential regulation of human indoleanine 2,3-dioxygenase gene expression by interferons-gamma and-alpha. Analysis of the regulatory reginon of the gene and identification of an interferon-gamma-inducible DNA-binding factor.J Biol Chem,1993,268(7):5077-5084.
[5]Yahata N,Y uasa S,Araki T. Nicotinamide mononucleotide adenylyltransferase expression in mitochondrial matrix delays wallerian degeneration. J Neurosci, 2009, 29: 6276-6284.
[6]Zhang T, Berrocal JG, Frizzell KM,etal. Enzymes in the NAD+salvage pathway regulate sirt1 activity at target gene promoter. J Biol Chem, 2009,284: 20408-20417.
[7]Berger F, Lau C, Ziegler M. Regulation of poly(ADP - ribose) polymerase1 activity by the phosphorylation state of the nuclear nad biosynthetic enzyme NMN adenylyltransferase 1. PNAS,2007,104: 3765-3770.
[8]Nadia Raffaelli, Leonardo Sorci, Adolfo Amici,etal. Identification of a novel human nicotinamide mononucleotide adenylyltransferase.Biochemical and Biophysical Research Communications, 2002, 297: 835-840.
[9]Barile M, Passarella S, Danese G,etal. Rat liver mitochondria can synthesize nicotinamide adenine dinucleotide from nicotinamide mononucleotide and ATP via a putative matrix nicotinamide mononucleotide adenylyltransferase. Biochem Mol Biol Int,1996, 38(2):297-306.
[10]Arau jo WL, Nunes-Nesi A, Trenkamp S,etal. Inhibition of 2-oxoglutarate dehydrogenase in potato tuber suggests the enzyme is limiting for respiration and confirms its importance in nitrogen assimilation. Plant Physiol, 2008, 148(4):1782-1796.
[11]Burgos ES, Schramm VL. Weak coupling of ATP hydrolysis to the chemical equilibrium of human nicotinamide phosphoribosyl-transferase. Biochemistry, 2008,47(42):11086-11096.
[12]Berger F, Lau C, Dahlmann M,etal. Subcellular compart-mentation and differential catalytic properties of the three human nicotinamide mononucleotide adenylyltransferase Isoform. J Biol Chem, 2005,280(43):36334-36341.
[13]Magni G, Amici A, Emanuelli M,etal. Enzymology of NAD+homeostasis in man.CellMol Life Sci,2004,61(1):19-34.
[14]Berger F, Lau C, Dahlmann M,etal. Subcellular compart-mentation and differential catalytic properties of the three hum-annicotinamide mononucleotide adenylyltransferase isoforms[J].J Biol Chem, 2005,280(43):36334-36341.
[15]Thierry L, CharlesG, Georges F. Psychrophilicenzymes:Re-vising the thermodynamic parameters of activation may explain local flexibility.BiochimBiophysActa,2000,1543(1):1-10.
[16]Sambrook J, Frisch EF, Maniatis T. Molecular Cloning. A Laboratory Manual[M]. New York: Cold Spring Harbor Laboratory Press, 1989.
[17]Monica Emanuelli, Francesco Carnevali, Maria Lorenzi,etal.Identification and characterization of YLR328W, the Saccharomyces cerevisiae structural gene encoding NMN adenylyltransferase.Expression and characterization of the recombinant enzyme.FEBS Letters,1999,455:13-17.
[18]Valerie C Sershon, Bernard D, Santarsiero,etal. Mesecar Kinetic and X-Ray Structural Evidence for Negative Cooperativity in Substrate Binding to Nicotinate Mononucleotide Adenylyl-transferase (NMAT) from Bacillus anthracis J. Mol. Biol,2009, 385: 867-888.
[19]Catello Di Martino, Maria Luigia Pallotta. Mitochondria-localized NAD biosynthesis by nicotinamide mononucleotide adenylyl-transferase in Jerusalem artichoke (Helianthus tuberosus L.) heterotrophic tissues.Planta, 2011, 234:657-670.
[20]陈清西.酶学及其研究技术[M]. 厦门: 厦门大学出版社, 2010.
Cloning, expression and characterization of NMNadenylyltransferase fromEscherichiacoli
DUAN Lin-lin, LI Hong-mei
University of Shanghai for Science and Technology, School of Medical Instrument and Food Engineering, Shanghai 200093, China
AbstractNMN (Nicotinamide mononucleotide) adenylyltransferase can catalyze NMN to generate NAD in vivo. The gene encoding NMN adenylyltransferase was amplified from the genomic DNA of E. coli and was overexpressed in E. coli BL21(DE3) by using the recombinant expression plasmid pET30a(+). The results showed that a gene fragment of 1 245 bp and its encoding protein of 45 kDa were obtained. The optimal reaction temperature and pH of recombinant enzyme was 37 ℃ and 7.5, respectively. The thermo-inactivation half-life of the enzyme at 4 ℃ was up to 990.2 min. Mn2+and Fe2+stimulated the enzyme, while EDTA remarkably suppressed the enzyme activity. When NMN was used as substrate, Km and Vmaxof the enzyme were 16.89 mmol/L and 2.46 μmol/(L·min), respectively. The successful overexpression of NMN adenylyltransferase gene in E. coli could lay the foundation for application of NAD through biosynthesis method.
Key wordsEscherichia coli; NMN adenylyltransferase; cloning and expression; enzymatic properties
doi:10.3969/j.issn.1001-6678.2016.02.006
作者简介:段琳琳(1990~),女,硕士研究生。研究方向: 微生物资源综合利用。E-mail: duanlinlin0303@163.com。 *通讯作者:李红梅,博士,硕士生导师。Tel:021-55271117,E-mail:sunnysand@126.com。
