水稻泛素结合酶基因家族的生物信息学与表达分析
2016-06-16刘鑫张恒阚虎飞周立帅黄昊宋林林翟焕趁张君鲁国东
刘鑫 张恒 阚虎飞 周立帅 黄昊 宋林林 翟焕趁 张君 鲁国东
(福建农林大学 功能基因组学研究中心, 福州 350002; *通讯联系人, E-mail: gdlufafu@163.com)
水稻泛素结合酶基因家族的生物信息学与表达分析
刘鑫张恒阚虎飞周立帅黄昊宋林林翟焕趁张君鲁国东*
(福建农林大学 功能基因组学研究中心, 福州 350002;*通讯联系人, E-mail: gdlufafu@163.com)
刘鑫, 张恒, 阚虎飞, 等. 水稻泛素结合酶基因家族的生物信息学与表达分析. 中国水稻科学, 2016, 30(3): 223-231.
摘要:泛素/蛋白酶体系统在植物的生长发育、形态建成和抗病反应等过程中起着重要的作用。近年的研究表明,某些病原菌能够模拟寄主植物泛素/蛋白酶体系统组分,从而达到利用该系统为病原菌服务的目的。泛素结合酶是泛素化反应过程中的第二个酶,对植物泛素/蛋白酶体系统的正常运行不可或缺。已有的研究表明,水稻基因组数据库中存在48个预测的泛素结合酶基因。为了初步揭示这些泛素结合酶基因在植物抗病防御反应中的功能,研究其与植物抗病性的关系。本研究通过生物信息学、RNA-seq和qRT-PCR的方法,分析了水稻泛素结合酶基因家族的特征及其表达模式。系统进化树分析表明,48个水稻泛素结合酶基因可分为3个大组,总共7个亚组。蛋白结构域分析表明,水稻泛素结合酶基因主要由一个泛素结合酶催化结构域组成。电子表达谱分析表明,大多数水稻泛素结合酶基因能被稻瘟病菌诱导表达。启动子区顺式作用元件分析表明,4个抗病相关顺式作用元件和1个过敏性反应相关顺式作用元件在48个水稻泛素结合酶基因的启动子区有很高的分布。稻瘟病菌接种亲和性和非亲和性水稻单基因系的RNA-seq结果表明,处理36 h读取到的水稻泛素结合酶基因为44个,其中高表达基因数量超过读取到的泛素结合酶基因总数的50%。qRT-PCR分析结果表明稻瘟病菌的侵染在亲和和非亲和组合中都能诱导部分水稻泛素结合酶基因的表达。在非亲和组合中水稻泛素结合酶基因的表达倾向于受到抑制。
关键词:水稻; 泛素结合酶; 泛素/蛋白酶体系统; 生物信息学; 稻瘟病菌; RNA-seq; qRT-PCR
植物泛素/蛋白酶体系统是细胞内蛋白质降解的主要途径,在植物的生长发育、形态建成和抗病反应等过程中起着重要的作用[1]。近年的研究又表明,某些病原菌能够模拟寄主植物泛素/蛋白酶系统组分,从而达到利用该系统为病原菌服务的目的[2]。泛素/蛋白酶体途径主要由泛素激活酶E1、泛素结合酶E2、泛素蛋白连接酶E3、蛋白酶体和去泛素化酶(DUBs)组成。反应的过程从泛素分子在ATP提供能量的情况下被E1激活开始,激活的泛素分子以硫酯键连接到E1上,E1进一步与E2结合,通过交酯作用将泛素分子转移到E2上,E2再与E3结合,而E3主要负责招募底物,就这样底物被泛素标记上了,之后被蛋白酶体降解成小段多肽。同时,在去泛素化酶的作用下,泛素分子重新回收利用[3,4]。泛素化在植物的生物和非生物胁迫响应过程中扮演了重要角色[2-5]。
稻瘟病菌(Magnaportheoryzae)引起的稻瘟病是水稻的主要病害之一,其发生导致水稻产量大幅减少,严重时甚至颗粒无收[7]。Zeng等[8]的研究结果表明,Spl11是一个拥有泛素蛋白连接酶E3活性的植物细胞死亡和防御反应的负调节子,非亲和及亲和的水稻-稻瘟病菌互作都能诱导Spl11的表达。Park等[9]的研究表明,稻瘟病菌效应蛋白AvrPiz-t通过作用于RING型E3泛素连接酶APIP6来抑制病原相关分子模式(PAMPs)触发的PTI反应。蒋春苗等[10]从疣粒野生稻中克隆到一个泛素结合酶基因OmE2,并证明这个基因受白叶枯病菌诱导表达。这些研究说明,泛素/蛋白酶体系统在水稻抗病防御中有着重要的作用,但目前已有的研究多集中于E3泛素连接酶的功能。胡婷丽等[11]综述了不同类型E3泛素连接酶介导的泛素化在植物抗病中的作用。杨玖霞等[12]近来对E3泛素连接酶在植物抗病分子机理方面的调节作用进行了专题论述。相比之下,对于泛素结合酶E2在植物抗病防御反应中功能的研究则很少。我们前期的研究发现过表达水稻的一个泛素结合酶基因能够增强水稻对稻瘟病菌的抗性,相应的RNAi株系对稻瘟病菌的抗性减弱[13]。而已有研究表明,水稻基因组数据库中存在48个预测的泛素结合酶基因,其中只有39个含有有活性的半胱氨酸位点[14,15]。拟南芥AtUBC1和AtUBC2基因参与开花抑制基因FLC的激活并抑制开花[16]。过表达拟南芥AtUBC32基因使植株对盐胁迫的响应敏感度下降[17]。在拟南芥中过表达绿豆VrUBC1基因、花生AhUBC2基因或大豆GmUBC2基因能增强植株的抗旱性[18-20]。在拟南芥中异位表达野生稻OgUBC基因增强了植株对灰葡萄孢及紫外线UV-B辐射的抗性[21]。为了初步探讨水稻泛素结合酶基因在水稻抗病反应中扮演的角色,本研究通过生物信息学、RNA-seq及qRT-PCR的方法对该家族基因的特征及表达模式进行了初步研究。
1材料与方法
1.1水稻泛素结合酶基因序列的获取
根据Hansol等[14]的补充材料信息,从TIGR数据库(http://rice.plantbiology.msu.edu/)中下载到48个水稻泛素结合酶基因的碱基序列和氨基酸序列[14,22]。
1.2水稻泛素结合酶基因系统进化树的构建
48个水稻OsUBC基因的蛋白氨基酸序列通过MEGA 5.05软件的ClustalW算法比对,其中(gap-open)和(gap-extension penalties)值分别设置为10和0.1。比对完的序列用邻接法,bootstrap 10 000次抽样来构建系统树。
1.3水稻泛素结合酶基因的结构域分析
结构域分析由Pfam 27.0 (http://pfam.xfam.org/) 在线完成[23]。输入的是蛋白氨基酸序列,参数设置如下:Cut-off选Use E-value,E-value值为默认的1.0。
1.4水稻泛素结合酶基因的电子表达谱
电子表达谱数据来自NCBI中的GEO Profiles(http://www.ncbi.nlm.nih.gov/geoprofiles/)数据库中的microarray结果,人工统计分析。
1.5水稻泛素结合酶基因启动子区顺式作用元件聚类分析
起始密码子上游1 500 bp的序列来自TIGR数据库,应用的是水稻基因组浏览网页(http://rice.plantbiology.msu.edu/cgi-bin/gbrowse/rice/),找到相应基因位点后,缩放到合适大小,然后点击Go按钮,在下载出来的序列中寻找对应基因的起始密码子序列并手动截取起始密码子上游1500 bp的序列。启动子区的顺式作用元件分析在PLACE网站(http://www.dna.affrc.go.jp/PLACE/signalscan.html)完成[24,25]。聚类分析由Cluster 3.0和Java TreeView软件共同完成[26]。
1.6稻瘟病菌接种水稻后的转录组分析
稻瘟病菌菌株Guy11孢子悬浮液,浓度为1×105个/mL,喷雾接种到3叶1心期的Pid2和Pid3单基因系上。于接种后36 h取样,提取植物总RNA,将符合要求的样品送至北京六合华大基因科技股份有限公司进行测序。RPKM(reads per kb per million reads)代表的是每百万reads中来自于某基因每千碱基长度的reads数[27]。
1.7稻瘟病菌侵染情况下水稻泛素结合酶基因的表达分析
将浓度约为1×105个/mL的Guy11孢子悬浮液接种到3叶1心期的Pid2和Pid3单基因系水稻幼苗上,于接种前(即0 h), 及接种后1 h,3 h,6 h,12 h,24 h取样。提取植物总RNA,反转录,稀释到合适浓度进行qRT-PCR定量分析。数据统计运用SPSS软件进行,显著性分析采用的是最小显著差数法(LSD),显著性水平0.05。
2结果与分析
2.1水稻泛素结合酶基因的系统进化树分析
系统进化树分析结果表明水稻泛素结合酶主要分为3个大组,其中Ⅰ大组包含4个亚组(Ⅰ-a,Ⅰ-b,Ⅰ-c,Ⅰ-d),Ⅱ大组包括3个亚组(Ⅱ-a,Ⅱ-b,Ⅱ-c),Ⅲ大组可分成2个亚组——Ⅲ-a和Ⅲ-b(图1)。第Ⅱ-a亚组中只有2个基因,可能在进化过程中由同一个基因复制而来。
2.2蛋白结构域分析
蛋白结构域分析结果表明,除了OsUBC38之外,其余47个水稻泛素结合酶基因都含有一个大的泛素结合酶催化结构域(UBCc domain)。除此之外,还零星分布有Low complexity结构域。
2.3电子表达谱分析
电子表达谱数据统计选取了生物胁迫因素1个,即稻瘟病菌的侵染;非生物胁迫因素1个,即盐胁迫;激素处理因素3个,即脱落酸、赤霉素、细胞分裂素;激发子1个,即寡聚糖几丁质。结果如表1所示,从中可以看出,除了OsUBC9、OsUBC14、OsUBC15和几个数据不可用的基因外,其他都能被稻瘟病菌的侵染诱导表达,这说明水稻泛素结合酶很可能在抵抗稻瘟病菌的侵染过程中发挥重要的作用。
2.4启动子区顺式作用元件的分析
启动子区的顺式作用元件分析选取抗病相关顺式作用元件9个(S000042,S000056,S000232,S000390,S000391,S000430,S000443,S000447, S000453);激发子相关顺式作用元件3个(S000142,S000200,S000492);苯丙氨酸解氨酶相关顺式作用元件3个(S000137,S000400,S000444);过敏性反应相关顺式作用元件1个(S000198)。值得一提的是,4个抗病相关顺式作用元件(S000390,S000430,S000447,S000453)和1个过敏性反应相关顺式作用元件S000198在水稻泛素结合酶基因的启动子区有很高的分布(图2)。这说明水稻泛素结合酶很可能参与了稻瘟病菌侵染过程中的抗病防御反应。
2.5稻瘟病菌接种水稻后的转录组数据分析
2.5.1RNA-seq测序质量报告
测序结果中, clean reads数占总数据的98.02%。以reads数量作为横坐标,识别到的基因百分数为纵坐标,进行测序饱和度的测验。结果表明,随着reads数的增多,识别到的基因百分数增长速度趋于平缓,说明识别到的基因数趋于饱和,测序数据可用于后续的分析(图3)。
2.5.2RNA-seq数据中水稻泛素结合酶基因的表达情况分析
RNA-seq数据中读取到水稻泛素结合酶基因44个。根据一般的标准,RPKM值在0.1到3.75之间的为低表达基因,3.75到15之间的为中等表达基因,大于15的为高表达基因[27]。RNA-seq读取到的水稻泛素结合酶基因中超过50%的基因属于高表达基因。值得一提的是OsUBC16在Pid2(亲和组合)和Pid3(非亲和组合)中的RPKM值分别为558.43和675.23,差别明显。OsUBC17的RPKM值则在Pid2中为150.60,高于Pid3中的124.43。相比之下,OsUBC18和OsUBC25在Pid2和Pid3中未表现出太大差别(图4)。
2.6稻瘟病菌诱导情况下几个水稻泛素结合酶基因表达情况分析
为了进一步验证RNA-seq数据的可靠性,选取了OsUBC16、OsUBC17、OsUBC18和OsUBC25来做qRT-PCR分析。结果表明Guy11接种Pid2单基因系水稻能诱导OsUBC16、OsUBC18的表达。接种24 h后,OsUBC16基因的表达显著高于0 h。非亲和组合中,OsUBC16、OsUBC17在接种后1 h就被显著诱导表达,3 h之后OsUBC16的表达就开始受到抑制,而12 hOsUBC17的表达达到一个高峰值。OsUBC25基因的表达量在接种后1 h开始就受到极显著的抑制(图5)。

图1水稻泛素结合酶基因家族系统进化树
Fig. 1. Phylogenetic tree of rice UBC gene family.
表1水稻泛素结合酶基因家族电子表达谱
Table 1. Expression pattern of rice UBC gene family in silico.
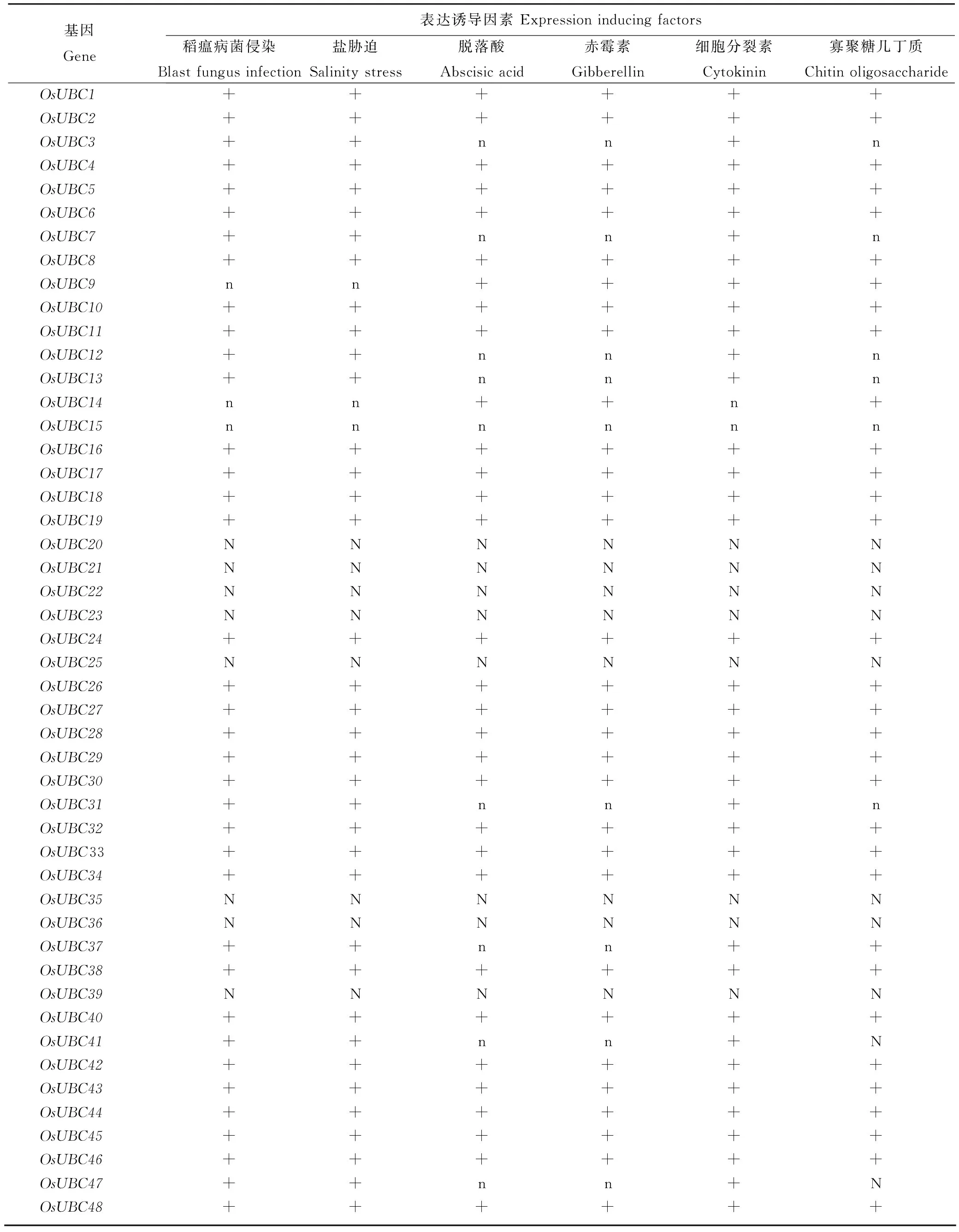
基因 Gene 表达诱导因素Expressioninducingfactors稻瘟病菌侵染Blastfungusinfection盐胁迫Salinitystress脱落酸Abscisicacid赤霉素Gibberellin细胞分裂素Cytokinin寡聚糖几丁质ChitinoligosaccharideOsUBC1++++++OsUBC2++++++OsUBC3++nn+nOsUBC4++++++OsUBC5++++++OsUBC6++++++OsUBC7++nn+nOsUBC8++++++OsUBC9nn++++OsUBC10++++++OsUBC11++++++OsUBC12++nn+nOsUBC13++nn+nOsUBC14nn++n+OsUBC15nnnnnnOsUBC16++++++OsUBC17++++++OsUBC18++++++OsUBC19++++++OsUBC20NNNNNNOsUBC21NNNNNNOsUBC22NNNNNNOsUBC23NNNNNNOsUBC24++++++OsUBC25NNNNNNOsUBC26++++++OsUBC27++++++OsUBC28++++++OsUBC29++++++OsUBC30++++++OsUBC31++nn+nOsUBC32++++++OsUBC33++++++OsUBC34++++++OsUBC35NNNNNNOsUBC36NNNNNNOsUBC37++nn++OsUBC38++++++OsUBC39NNNNNNOsUBC40++++++OsUBC41++nn+NOsUBC42++++++OsUBC43++++++OsUBC44++++++OsUBC45++++++OsUBC46++++++OsUBC47++nn+NOsUBC48++++++
“+”代表可诱导表达,“n”代表无相关数据,“N”表示无数据可用。
‘+’ , Inducible; ‘n’ , No related data; ‘N’ , Unavailable.

图2水稻泛素结合酶基因启动子区顺式作用元件聚类分析
Fig. 2. Cluster analysis of cis-element in the promoter region of rice UBC genes.

图3水稻RNA-seq测序饱和度分析
Fig. 3. Saturation analysis of rice RNA-seq data.
3讨论
Zeng等[3]早在2006年就强调泛素/蛋白酶体系统可能在植物和微生物互作的过程中发挥了重要的作用,并且提供了很多间接的证据。而后在2010年Dielen等[2]发表在《分子植物病理学》上的一篇综述更是强调“植物防御反应的每一步都涉及到泛素/蛋白酶体系统”。同时,作者还提到“泛素/蛋白酶体系统不光是寄主植物用来防御的武器,也是一些病原菌攻击的靶标,从而抑制这个系统的正常运行或者利用这个系统为病原菌服务”。因此,我们不难提出设想:病原菌在侵染植物的过程中扰乱了寄主植物的泛素/蛋白酶体系统,从而也打破了寄主植物细胞体内的稳态,从而有利于病菌的侵染;还有一种模式就是:病原菌分泌效应蛋白进入寄主植物细胞中,模拟寄主植物泛素/蛋白酶体系统的组分,从而利用寄主的泛素蛋白酶体系统以利于病菌的成功侵染。
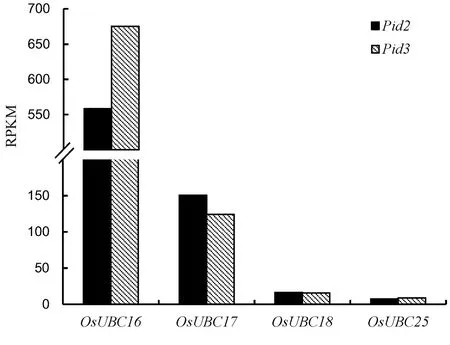
图4四个水稻泛素结合酶基因在Pid2和Pid3单基因系水稻中表达情况比较
Fig. 4. Expression comparison of four rice ubiquitin-conjugating enzyme gene in Pid2 and Pid3 monogenic lines.
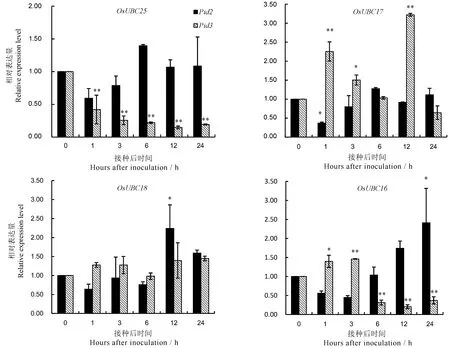
*,**分别表示表达量显著和极显著上调。
*,** indicate significantly or extremely significantly upregulated expression levels,respectively.
图5接种稻瘟病菌后水稻泛素结合酶基因的表达量变化
Fig. 5. Expression level of rice ubiquitin-conjugating enzyme genes after Magnaporthe oryzae inoculation.
从48个水稻泛素结合酶基因的系统进化树来看,水稻泛素结合酶家族基因在进化过程中存在复制事件,即同一家族的两个基因存在片段重复或者串联重复。例如:OsUBC25和OsUBC26在碱基序列上存在片段重复,但是功能方面是否有冗余还有待进一步论证。另外,水稻泛素结合酶家族基因在进化过程中存在功能的分化现象。在水稻泛素结合酶基因家族中存在9个泛素结合酶变体(UEV, ubiquitin E2 variants)[28,29]。这些UEV是否还有泛素结合酶活性暂且没有报道,但是半胱氨酸-巯基位点作为泛素结合酶与泛素分子的结合位点在E2发挥功能的过程中至关重要。由此可见,至少这9个UEV可能趋于行使泛素结合酶以外的功能。而水稻泛素结合酶家族基因在进化中的复制事件则说明在进化过程中不断有新的泛素结合酶呈现。
从电子表达谱来看,水稻泛素结合酶大多数能被稻瘟病菌的侵染诱导表达。从启动子区顺式作用元件来看,4个抗病相关顺式作用元件和1个过敏性反应相关顺式作用元件在水稻泛素结合酶基因的启动子区有较高的分布。本实验室前期的研究结果也表明,稻瘟病菌和JA处理能够诱导OsUBC26的表达[13]。这也让我们更有理由相信水稻的泛素结合酶参与了稻瘟病菌侵染信号途径的响应,并且很可能与JA信号途径有关。E等[15]的研究也表明水稻泛素结合酶的表达至少能被IAA,6-BA,GA,ABA中的两种诱导。虽然Hansol等[14]对40个水稻E2s和17个水稻ARM-U-box E3s的互作模式做了探讨,但是,在水稻中E2和E3的互作网络还鲜有报道,大多数水稻泛素蛋白连接酶的底物还处于未知阶段。目前,可以确定的是水稻泛素结合酶参与了水稻与稻瘟病菌的互作过程。
qRT-PCR的结果说明不同水稻泛素结合酶在水稻响应稻瘟病菌侵染的过程中扮演了不同的角色。这可以从水稻泛素结合酶基因的表达模式在稻瘟病菌与水稻的亲和互作和非亲和互作中表现出很大的差别看出来。这些差别说明在稻瘟病菌侵染的过程中水稻泛素结合酶之间可能存在系统的调控。已有的研究表明泛素结合酶E2主要决定泛素链的长度和拓扑结构,而不同拓扑结构的泛素链标记的蛋白分子将行使不同的功能,例如以K48相连的泛素链标记的蛋白分子主要被送往26S蛋白酶体进行降解,而以K63相连的泛素链标记的蛋白分子则主要在信号转导方面起作用[30]。因此,我们可以推测那些在稻瘟病菌侵染初期就被诱导高表达的水稻泛素结合酶基因很可能在水稻抗病防御反应的信号转导方面起了作用,而被诱导表达较晚的水稻泛素结合酶基因则可能主要在水稻启动防御反应方面起了作用,例如通过泛素化途径降解掉一些防御反应相关的抑制蛋白,从而启动水稻的抗病防御反应。
参考文献:
[1]Hershko A. The ubiquitin system for protein degradation and some of its roles in the control of the cell division cycle.CellDeathDiffer, 2005, 12: 1191-1197.
[2]Dielen A, Badaoui S, Candresse T, et al. The ubiquitin/26S proteasome system in plant-pathogen interactions: A never-ending hide-and-seek game.MolPlantPathol, 2010, 11(2): 293-308.
[3]Zeng L R, Miguel E V, Zhu T, et al. Ubiquitination-mediated protein degradation and modification: An emerging theme in plant-microbe interactions.CellRes, 2006, 16: 423-426.
[4]Stone S L. The role of ubiquitin and the 26S proteasome in plant abiotic stress signaling.FrontPlantSci, 2014, 5.
[5]Marino D, Peeters N, Rivas S. Ubiquitination during plant immune signaling.PlantPhysiol, 2012, 160(1): 15-27.
[6]Smalle J, Vierstra R D. The ubiquitin 26S proteasome proteolytic pathway.AnnuRevPlantBiol, 2004, 55: 555-590.
[7]Madden L V, Wheelis M. The threat of plant pathogens as weapons against U.S. crops.AnnuRevPhytopathol, 2003, 41(4): 155-176.
[8]Zeng L R, Qu S H, Bordeos A, et al.Spottedleaf11, a negative regulator of plant cell death and defense, encodes a U-Box/Armadillo repeat protein endowed with E3 ubiquitin ligase activity.PlantCell, 2004, 16: 2795-2808.
[9]Park C, Chen S B, Shirsekar G, et al. TheMagnaportheoryzaeeffector AvrPiz-t targets the RING E3 ubiquitin ligase APIP6 to suppress pathogen-associated molecular pattern-triggered immunity in rice.PlantCell, 2012, 24: 4748-4762.
[10]蒋春苗, 黄兴奇, 付坚, 等.疣粒野生稻泛素结合酶基因的全长cDNA序列克隆与分析. 作物学报,2012,38(5): 808-813.
Jiang C M, Huang X Q, Fu J, et al. Cloning and analysis on full-Length cDNA sequence of ubiquitin-conjugating enzyme gene fromOryzameyerianaBaill.ActaAgronSin, 2012, 38(5): 808-813. (in Chinese with English abstract)
[11]胡婷丽, 李魏, 刘雄伦, 等. 泛素化在植物抗病中的作用. 微生物学通报, 2014, 41(6): 1175-1179.
Hu T L, Li W, Liu X L, et al. The role of ubiquitination in plant disease resistance.MicrobiolChina, 2014, 41(6): 1175-1179. (in Chinese with English abstract)
[12]杨玖霞, 张浩, 王志龙, 等. E3泛素连接酶调控植物抗病分子机理研究进展. 植物保护,2015,41(4): 1-8.
Yang J X, Zhang H, Wang Z L, et al. Recent progresses in the regulation mechanism of E3 ligases in plant disease resistance.PlantProtect, 2015, 41(4): 1-8. (in Chinese with English abstract)
[13]林艺娟.OsRBCS和OsUBC2在水稻抗病防御中的作用机制. 福州:福建农林大学,2014.
Lin Y J. The mechanism ofOsRBCsandOsUBC2 in rice disease resistance. Fuzhou: Fujian Agriculture and Forestry University, 2014.(in Chinese with English abstract)
[14]Hansol B, Woo T K. Classification and interaction modes of 40 rice E2 ubiquitin-conjugating enzymes with 17 rice ARM-U-box E3 ubiquitin ligases.BiochemBiophysResCommun, 2014, 444: 575-580.
[15]E Z G, Zhang Y P, Li T T, et al. Characterization of the ubiquitin-conjugating enzyme gene family in rice and evaluation of expression profiles under abiotic stresses and hormone treatments.PLoSONE, 2015, 10(4).
[16]Xu L, Ménard R, Berr A, et al. The E2 ubiquitin-conjugating enzymes, AtUBC1 and AtUBC2, play redundant roles and are involved in activation ofFLCexpression and repression of flowering inArabidopsisthaliana.PlantJ, 2009, 57(2): 279-288.
[17]Cui F, Liu L J, Zhao Q Z, et al.Arabidopsisubiquitin conjugase UBC32 is an ERAD component that functions in brassinosteroid-mediated salt stress tolerance.PlantCell, 2012, 24(1): 233-244.
[18]Chung E, Cho C W, So H A, et al. Overexpression ofVrUBC1, a mung bean E2 ubiquitin-conjugating enzyme, enhances osmotic stress tolerance inArabidopsis.PLoSONE, 2013, 8(6).
[19]Wan X R, Mo A Q, Liu S, et al. Constitutive expression of a peanut ubiquitin-conjugating enzyme gene inArabidopsisconfers improved water-stress tolerance through regulation of stress-responsive gene expression.JBiosciBioengin, 2011, 111(4): 478-484.
[20]Zhou G A, Chang R Z, Qiu L J. Overexpression of soybean ubiquitin-conjugating enzyme geneGmUBC2 confers enhanced drought and salt tolerance through modulating abiotic stress-responsive gene expression inArabidopsis.PlantMolBiol, 2010, 72(4-5): 357-367.
[21]Jeon E H, Pak J H, Kim M J, et al. Ectopic expression of ubiquitin-conjugating enzyme gene from wild rice,OgUBC1, confers resistance against UV-B radiation andBotrytisinfection inArabidopsisthaliana.BiochemBiophysResCommun, 2012, 427(2): 309-314.
[22]Kawahara Y, Bastide M D L, Hamilton J P, et al. Improvement of theOryzasativaNipponbare reference genome using next generation sequence and optical map data.Rice, 2013, 6: 4.
[23]Finn R D, Bateman A, Clements J, et al. The Pfam protein families database.NuclAcidsRes, 2014, Database Issue 42: D222-D230.
[24]Higo K, Ugawa Y, Iwamoto M,et al. Plant cis-acting regulatory DNA elements (PLACE) database.NuclAcidsRes, 1999, 27: 297-300.
[25]Prestridge D S. SIGNAL SCAN: A computer program that scans DNA sequences for eukaryotic transcriptional elements.ComputerApplBiosci, 1991, 7: 203-206.
[26]de Hoon M J L, Imoto S, Nolan J, et al. Open source clustering software.Bioinformatics, 2004, 20(9): 1453-1454.
[27]Mortazavi A, Williams B A, Mccue, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq.NatMethods, 2008, 5(7): 621-628.
[28]van Demark A P, Hofmann R M, Tsui C, et al. Molecular insights into polyubiquitin chain assembly: Crystal structure of the Mms2/Ubc13 heterodimer.Cell, 2001, 105(6): 711-720.
[29]Andersen P L, Zhou H, Pastushok L, et al. Distinct regulation of Ubc13 functions by the two ubiquitin-conjugating enzyme variants Mms2 and Uev1A.JCellBiol, 2005, 170(5): 745-755.
[30]Ye Y, Rape M. Building ubiquitin chains: E2 enzymes at work.NatRevMolCellBiol, 2009, 10(11): 755-764.
Bioinformatic and Expression Analysis of Rice Ubiquitin-conjugating Enzyme Gene Family
LIU Xin, ZHANG Heng, KAN Hu-fei, ZHOU Li-shuai, HUANG Hao, SONG Lin-lin, ZHAI Huan-chen,ZHANG Jun, LU Guo-dong*
(TheFunctionalGenomicsCenter,FujianAgricultureandForestryUniversity,Fuzhou350002,China;*Corresponding author, E-mail:gdlufafu@163.com)
LIU Xin, ZHANG Heng, KAN Hufei, et al. Bioinformatic and expression analysis of rice ubiquitin-conjugating enzyme gene family. Chin J Rice Sci, 2016, 30(3): 223-231.
Abstract:The ubiquitin/proteasome system plays an important role in plant growth and development, morphogenesis and disease resistance. Recent studies have shown that some pathogens can mimic the host plant ubiquitin/proteasome system components to achieve their own purposes. Ubiquitin-conjugating enzyme is the second enzyme in the ubiquitination process and is indispensable for the plant ubiquitin/proteasome system. Previous studies showed that there are 48 predicted ubiquitin-conjugating enzyme genes in rice genome. In order to preliminarily elucidate the functions of rice ubiquitin-conjugating enzyme genes in plant disease resistance, bioinformatic, RNA-seq and qRT-PCR methods were used to analyze characteristics and expression patterns of rice ubiquitin-conjugating enzyme gene family. Phylogenetic tree analyses indicate that the 48 rice ubiquitin-conjugating enzyme genes can be divided into 3 groups, 7 sub-groups in total. Protein domain analysis showed that ubiquitin-conjugating enzyme genes mainly consist of a big ubiquitin-conjugating enzyme catalytic domain. Expression analysis in silico suggested that most of the rice ubiquitin-conjugating enzymes can be induced by blast fungus infection. Plant cis-acting elements analysis indicated that four pathogen resistance cis-acting elements and one hypersensitivity reaction cis-acting element have high distribution in the promoter region of the 48 rice ubiquitin-conjugating enzyme genes. RNA-seq data from compatible and incompatible monogenic rice after rice blast fungus infection showed that 44 rice ubiquitin-conjugating enzyme genes were expressed at 36 hours after treatment, among which more than 50% were highly expressed genes. qRT-PCR analysis showed that expression of some ubiquitin-conjugating enzyme genes can be induced by the inoculation of rice blast fungus both in compatible and incompatible monogenic rice. However, in incompatible rice the expression of rice ubiquitin-conjugating enzyme genes tends to be inhibited after rice blast fungus inoculation.
Key words:rice; ubiquitin-conjugating enzyme; ubiquitin/proteasome system; bioinformatics; Magnaporthe oryzae; RNA-seq; qRT-PCR
DOI:10.16819/j.1001-7216.2016.5177
收稿日期:2015-11-30; 修改稿收到日期: 2016-03-16。
基金项目:国家科技支撑计划资助项目(2013BAD19B03)。
中图分类号:Q755; S511.01
文献标识码:A
文章编号:1001-7216(2016)03-0223-09
中国水稻科学(Chin J Rice Sci),2016,30(3):223-231
http://www.ricesci.cn
