全细胞的核酸适配体筛选的研究进展
2016-05-12刘品多
刘品多, 屈 锋
(北京理工大学生命学院, 北京 100081)
全细胞的核酸适配体筛选的研究进展
刘品多,屈锋*
(北京理工大学生命学院, 北京 100081)
摘要:核酸适配体(aptamer)是从人工合成的随机单链DNA(ssDNA)或RNA文库中筛选得到的,能够高亲和力、高特异性地与靶标结合的ssDNA或RNA。核酸适配体的靶标范围广,可包括小分子、蛋白质、细胞、微生物等多种靶标。其中以细胞为靶标的适配体在生物感应、分子成像、医学诊断、药物传输和疾病治疗等领域有很大的应用潜能。但全细胞的核酸适配体筛选过程复杂,筛选难度大,筛选的适配体性能不佳是导致目前可用的适配体非常有限的主要原因。由于细胞表面蛋白质在提取纯化过程中分子结构和形态会发生改变,故以膜表面蛋白质为靶标筛选的适配体很难应用于识别整体细胞。以全细胞为靶标的核酸适配体筛选则不需要准确了解细胞表面的分子结构,筛选过程中可保持细胞的天然状态,以全细胞为靶标筛选出的核酸适配体有望直接用于全细胞识别。本文总结了2008~2015年全细胞的核酸适配体筛选的研究进展,介绍了靶细胞的分类、核酸库的设计、筛选条件和方法以及核酸适配体的亲和力表征方法等。并列出全细胞靶标的核酸适配体序列。
关键词:核酸适配体;细胞;筛选;指数富集的配基系统进化技术;综述
核酸适配体是从人工合成的随机单链DNA(ssDNA)或RNA文库中筛选得到的以高亲和力、高特异性与靶标结合的ssDNA或RNA。核酸适配体通常由10~100个碱基组成,这些碱基可通过分子内碱基配对形成稳定的二级结构如发夹(hairpin)、假结(pseudoknot)、G-四链体(G-quartet)等,或在靶标的诱导下形成稳定的二级结构。核酸适配体作为配体,其结合的靶标范围广泛,包括离子、小分子、多肽、蛋白质乃至完整的细胞和微生物。目前,由于缺乏可高效识别细胞表面分子的特异性工具,故对细胞表面的分子水平研究还受到很大的限制。而基于全细胞筛选的核酸适配体有望成为细胞的识别分子,用于对其进行快速分析检测。
以全细胞为靶标的核酸适配体筛选(cell-SELEX,或whole cell-SELEX)并非针对细胞中分离纯化出来的蛋白质组分,而是以完整细胞为靶标,利用单链核酸与完整细胞表面的分子间的相互作用进行分子识别。基于全细胞的核酸适配体筛选,不需要准确了解细胞表面的分子及其结构信息,且筛选后的核酸适配体可直接用于全细胞识别。例如,通过Cell-SELEX有望筛选出可识别未分化和分化的细胞、正常细胞和癌细胞、感染和未感染的细胞等的适配体。因此全细胞的核酸适配体筛选及应用对于疾病的诊断和治疗有重要的研究意义和应用前景。
全细胞核酸适配体的筛选及细胞识别研究促进了核酸适配体在早期临床诊断和疾病治疗等生物医学研究领域的开发与应用。谭蔚泓教授课题组在国际上率先开始全细胞核酸适配体的研究,发表了一系列研究论文[1-8]和综述[9-13]。近期出版的whole-cell-SELEX[14]一书系统总结了相关工作。他们通过cell-SELEX筛选出多种癌细胞的适配体,指出核酸适配体可用于特异性鉴别肿瘤类型,改进肿瘤治疗方法。通过辨识肿瘤细胞表面膜蛋白还可以发现肿瘤标志物。此外,McKeague等[15]对1990~2013年公开发表的2 334种适配体序列及相关文章做了一系列的统计,包括核酸库的类型、适配体长度、靶标种类、筛选条件及筛选方法等,其中以细胞为靶标的适配体占到6%。Shamah等[16]总结的复合靶标的筛选方法可以排除无特异性或低特异性的核酸序列。他们指出,一次实验中既可以筛选出特异性结合不同靶位的多种适配体,也可以筛选出对多种靶位有结合作用的同一个适配体。根据筛选目的的不同可应用不同的复合筛选方法。赵仲麟等[17]综述了cell-SELEX技术在活病原微生物、癌细胞、生物标志物筛选等方面的进展,还介绍了适配体用于细胞摄取药物及给药方式方面的研究。
与生物体内的天然抗体相比,核酸适配体完全通过体外筛选获得,可通过化学方法合成及修饰,实验成本低,无需动物免疫及分离纯化。核酸适配体有较好的组织渗透性,易进入细胞,且无免疫原性。还可根据应用条件筛选特定靶标的适配体,通过筛选过程控制其与靶标的亲和力和特异性。已有研究表明,核酸适配体作为识别分子,在细胞生物学、细胞成像、癌症诊断和疾病治疗等方面有广阔的应用前景,但因核酸适配体筛选过程复杂、繁琐、人力和财力消耗大,实际筛选过程难度很大,筛选的成功率并不令人满意。面对重大疾病的细胞识别和治疗药物发现的巨大需求,目前可用的核酸适配体还非常有限,远远不能满足实际需求。而盲目地进行核酸适配体筛选常常无功而返。已有多篇综述重点介绍了2008~2015年全细胞的核酸适配体的研究进展和应用[1-13,16,17],但针对细胞的适配体筛选方法的总结和分析还少见报道。针对全细胞靶标的适配体筛选方法进行归纳总结无疑将为研究者提供系统的信息和参考,有助于提供适配体的筛选效率。本文重点针对细胞的核酸适配体的筛选方法进行总结,包括靶细胞的分类、核酸库的设计、筛选条件和方法及核酸适配体的亲和力表征方法等。另外总结了2008年以来全细胞筛选适配体序列(见表1)。
1靶细胞分类
癌症是当今世界第一大威胁人类生命健康的疾病,癌症的诊断、治疗及预后监测等一直是生物医学研究的热点和难题。目前,以细胞为靶标的适配体筛选主要针对不同类型的癌细胞,也有少量针对炎症细胞、体内正常细胞及感染细胞的筛选。
1.1癌细胞
针对人类各系统,可将癌细胞进行如下分类。
1.1.1呼吸系统癌细胞
呼吸系统的癌症主要包括鼻咽癌、喉癌、气管癌和肺癌,而肺癌的发病率远高于其他几种癌症,是发病率和死亡率最高的癌症种类。目前对呼吸系统癌细胞适配体的筛选也主要针对各类肺癌细胞。Kunii等[37]筛选出小细胞肺癌细胞SBC3的适配体,这种适配体与其他几种肺癌细胞如DMS53、A549、NCI-H69及RERF-LC-MA的结合能力极低,说明该适配体特异性极强,有望用于肺癌类型诊断。Chen等[38]以小细胞肺癌细胞NCI-H128为靶细胞筛选适配体。当在人的全血中加入NCI-H128细胞,并与筛选出的适配体进行孵育后,通过用流式细胞仪可检测适配体与全血中的NCI-H128细胞有结合力,表明该适配体可在人的血液环境中与NCI-H128细胞有较强的结合,说明该适配体可用于血液环境中NCI-H128细胞的检测。
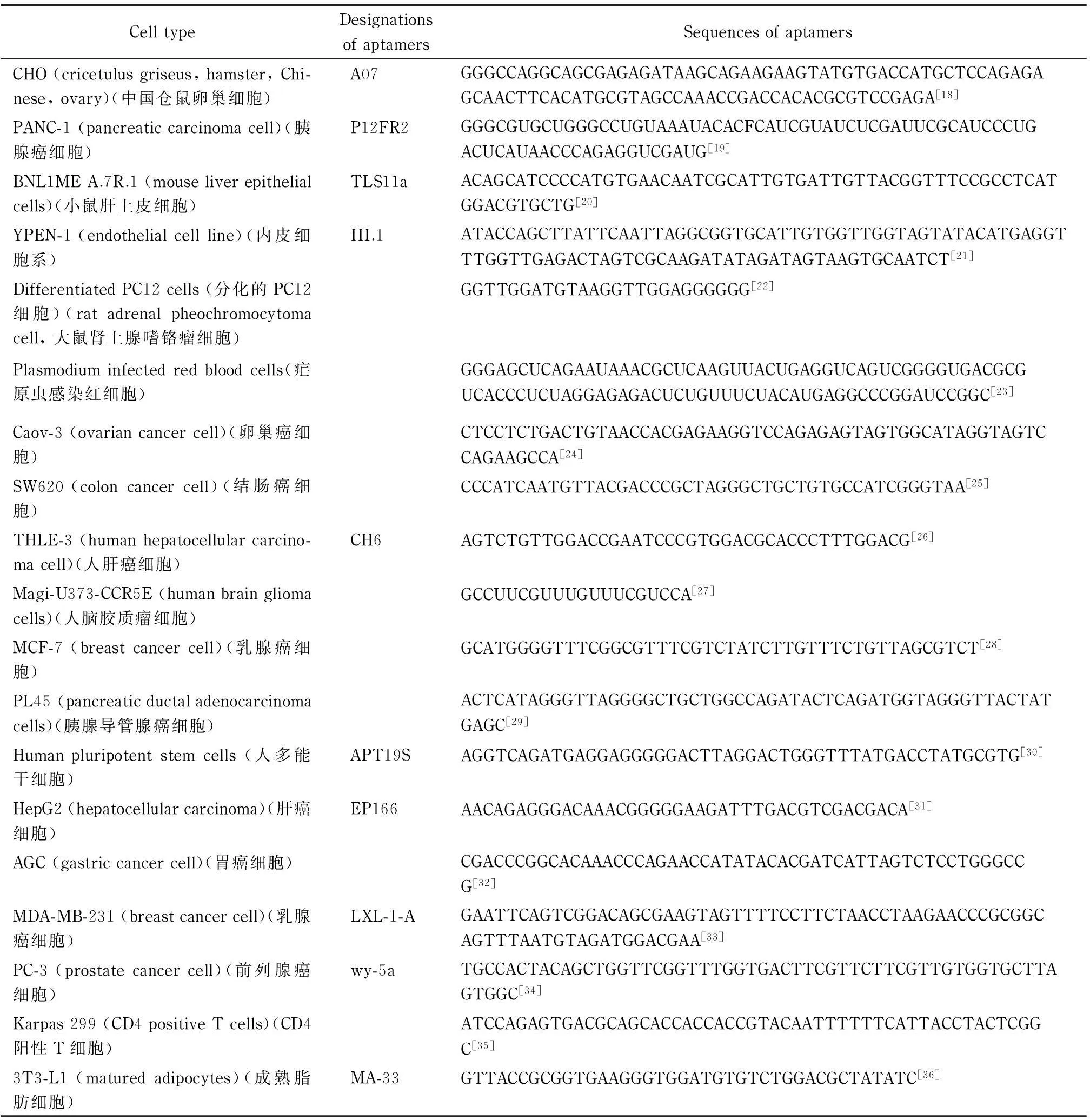
表 1 细胞靶标的适配体序列
1.1.2消化系统癌细胞
消化系统的癌症包括食道癌、胃癌、肝癌、结肠癌和直肠癌等。胃癌、肝癌及结肠直肠癌等的适配体筛选均有报道。李真真等[39]通过链霉亲和素磁珠分离和PCR扩增条件的优化,筛选出胃癌细胞SGC-7901的适配体。Ninomiya等[40]筛选了肝癌细胞HepG2细胞适配体,该适配体与正常肝细胞不结合,在靶向治疗肝癌时可避免伤及正常肝细胞,从而减少副作用。Li等[41]筛选出以转移性直肠癌LoVo为靶细胞的适配体,并且在肿瘤切片上进行原位杂交,结果表明该适配体对肿瘤细胞结合能力很强,说明该适配体具有靶向治疗直肠癌的潜力。Li等[42]筛选出乳腺癌细胞MDA-MB-231适配体,为验证适配体的结合部位是膜表面蛋白,用胰蛋白酶进行消化使细胞表面膜蛋白减少,将消化过的细胞与修饰荧光标签的适配体孵育,用流式细胞仪检测荧光密度,结果显示细胞经消化后与适配体的结合力明显下降,证明适配体是与细胞表面膜蛋白结合。Wu等[29]筛选出了胰腺导管腺癌(PDAC)PL45细胞的高亲和力和特异性的适配体。对荷瘤小鼠分别进行静脉注射适配体和核酸库后,利用IVIS Lumina II活体成像系统发现,仅需5 min适配体就可大量结合在肿瘤部位,说明该适配体可以在体内迅速结合到肿瘤,具有靶向治疗肿瘤的潜力。Kim等[31]以过表达上皮黏附分子(EpCAM)的肝癌细胞HepG2为筛选细胞,以普通HepG2细胞为阴性细胞,筛选出EpCAM的适配体。因EpCAM为多种肿瘤细胞的生物标志物,其适配体具有初步诊断癌症的潜质。Cao等[32]筛选了与胃癌细胞AGS的适配体。分别用AGS细胞、SW620细胞(直肠腺癌细胞)、HepG2细胞(肝癌细胞)对大鼠进行荷瘤实验,荷瘤成功后静脉注射AGS适配体缓冲液,通过活体成像系统可见适配体与AGS细胞瘤体结合程度很高,而与其他两种细胞瘤体几乎没有结合,证明该适配体对胃癌细胞AGS具有高亲和力和高特异性,有望于胃癌细胞的识别。
1.1.3生殖系统癌细胞
男性和女性罹患的生殖系统癌症分别主要为前列腺癌和宫颈癌及卵巢癌。针对卵巢癌及宫颈癌已筛选出多种相关癌细胞的适配体。Simaeys等[43]筛选的卵巢癌细胞TOV-21的适配体,不与卵巢癌细胞CAOV-3和宫颈癌细胞Hela结合,表明它对TOV-21有高特异性,在肿瘤早期诊断中有鉴别特定卵巢癌的潜力。Graham等[44]以未感染人乳头瘤病毒的宫颈癌细胞Hela为靶细胞筛选的适配体,不能与感染此类病毒的宫颈癌细胞结合,因此,使用该适配体可以判断癌症的种类及癌症亚型,有助于对患癌种类的预测。Benedetto等[24]筛选的针对恶性卵巢癌细胞Caov-3的高亲和力适配体则对良性卵巢癌细胞SK-OV-3没有结合,故可用于卵巢癌良恶性的判断。Lu等[28]筛选的乳腺癌干细胞Mammosphere适配体,与正常乳腺细胞MCF-A以及乳腺癌细胞MCF-7均无结合,具有诊断早期乳腺癌的应用潜能。Hung等[45]建立芯片cell-SELEX(on-chip cell-SELEX)的方法,在磁珠表面修饰上皮细胞抗体并与靶细胞孵育,使磁珠能够结合在靶细胞表面,结合磁珠的靶细胞与核酸库孵育后,用磁珠分离出磁珠靶细胞适配体结合物并进行PCR扩增,最终筛选出与卵巢癌细胞TOV21G具有高亲和力的适配体。Li等[25]筛选出对乳腺癌SW620细胞有特异性识别的适配体,证明在4 ℃和37 ℃孵育时,适配体与该细胞的结合无明显差异。也有关于前列腺癌细胞的适配体筛选报道。Wang等[34]筛选的前列腺癌细胞PC3的适配体,对肝癌细胞SMMC-7721、宫颈癌细胞Hela及前列腺癌细胞22RV-1则没有明显的结合,证明该适配体与PC3细胞有高特异性识别,可望用于前列腺癌的早期鉴别诊断。
1.1.4血液系统癌细胞
血液系统的癌症主要包括各类白血病、多发性骨髓瘤以及恶性淋巴瘤。Sefah等[46]筛选出Hl60细胞的适配体,可用于急性骨髓性白血病的早期诊断。陈鑫等[47]用急性早幼粒白血病细胞NB4作靶细胞,以慢性髓系白血病细胞株K562细胞为阴性细胞,筛选出NB4细胞适配体,该适配体能够鉴别急性早幼粒白血病和慢性髓性白血病。
1.2炎症细胞
炎症是常见的基本病理过程,它与肿瘤及多种代谢类疾病如糖尿病、动脉粥样硬化等的发生发展关系密切。有效阻断炎症的发展通路是医学界研究的热点。陈亮等[48]用小鼠胚胎成纤维细胞NIH3T3作为靶细胞,以IL-17RA表达缺陷性的NIH3T3为阴性细胞,筛选了NIH3T3的适配体。他们的研究表明IL-17/IL-17RA介导的信号通路与炎症因子IL-1、IL-6和IL-8的表达密切相关,因此有可能利用该适配体治疗骨关节炎或类风湿关节炎。汪江波等[49]将THP-1巨噬细胞诱导为泡沫细胞,即以巨噬细胞源性泡沫细胞作为靶细胞,以巨噬细胞和平滑肌细胞作为阴性细胞,筛选出巨噬细胞源性泡沫细胞适配体。由于巨噬细胞源性泡沫细胞是动脉粥样硬化中最早出现的标识性病理细胞,故利用其适配体有可能利用巨噬细胞源性泡沫细胞作为动脉粥样硬化治疗靶点,该适配体有可能用于早期心脑血管的诊断和靶向治疗。
1.3正常体细胞
筛选正常体细胞的适配体的意义在于可利用适配体追踪体内正常的生理过程及模拟病理过程,便于深入探讨发病机理,寻找有效的防病治病方法。Liang等[26]筛选出成骨细胞的适配体,并将其制备成功能性脂质纳米微粒,探讨了骨形成的相关机制。Zhou等[27]用表达CCR5的人脑神经胶质瘤细胞U373-Magi-CCR5E细胞作为靶细胞,以不表达CCR5的U373-Magi细胞作为阴性细胞筛选出适配体,由于HIV病毒的结合位点为T细胞表面CCR5受体,如果该适配体能与CCR5结合,就能有效阻断HIV病毒与T细胞的结合,该适配体作为药物则具有治疗艾滋病的潜力。Hou等[30]筛选出人类多能干细胞适配体,该适配体与几种干细胞包括小鼠胚胎肝细胞、猕猴胚胎肝细胞、人胚胎肝细胞及人诱导型多能干细胞结合能力都很强,但与几种非干细胞包括人B细胞淋巴瘤细胞、人胚胎成纤维细胞、人胚肾细胞和人滋养细胞的结合能力很弱,说明该适配体能特异性地结合干细胞而不能结合已分化细胞,对研究干细胞的分化过程有很大帮助。Zhao等[35]以CD4阳性T细胞Karpas 299为靶细胞,以不表达CD4的B淋巴细胞CA46为阴性细胞筛选出的适配体可与CD4因子结合,因而阻碍GP120病毒与CD4的结合,从而防止对GP120病毒的感染。Kim等[36]筛选成熟了的白色脂肪细胞的适配体,因白色脂肪细胞的主要功能是储存体内多余的脂肪,若将该适配体与药物分子连接,再结合到脂肪细胞,则有望有效治疗肥胖病。
1.4感染细胞
近年来,全球传染病流行态势十分严峻。传染病仍对人类健康构成很大威胁。特别是不少传染病以暴发流行的形式出现,在短期内可使数以千计的人罹患或死亡。因此研究有效防止传染病疾病的形成十分必要。当细胞受到病毒或病原微生物感染后,细胞膜表面的某些蛋白质结构会发生变化。筛选感染细胞的适配体对于诊断一些有潜伏期的传染性疾病具有重大意义。Birch等[50]建立惯性微流SELEX(i-SELEX)的方法,即基于惯性聚焦螺旋微流控通道,运用惯性将细胞和未与细胞结合的核酸分离,筛选出被恶性疟原虫感染的红细胞的适配体,这种适配体对恶性疟原虫的有效识别对于疟疾的诊断和治疗有重大意义。筛选感染细胞的适配体时,通常选择受感染的细胞作为阳性细胞,而将同类型的非感染的细胞作为阴性细胞用于负筛选。有时也可选用未感染细胞为靶细胞而将感染的细胞作为阴性细胞,如Graham等[44]选用未被人乳头瘤病毒感染的宫颈癌细胞为靶细胞,而将易感细胞作为阴性细胞,筛选出未感染人乳头瘤病毒的宫颈癌细胞的适配体。该适配体不能与感染了此类病毒的宫颈癌细胞结合,可用于宫颈癌早期诊断中鉴别宫颈癌细胞是否被乳头瘤病毒感染。
2核酸库的设计
针对细胞筛选时所用的核酸库与其他靶标所用的核酸库相似,也是由大量随机合成的单链DNA或RNA分子序列组成。理论上,一定碱基数的核酸库中存在的核酸分子序列应有4n种。这些单链DNA或RNA链的两端含有引物序列,中间为随机序列。随机序列长度一般为15~80 mer。两端的引物长度总和为30~50 mer。与蛋白质和小分子靶标筛选适配体不同,全细胞筛选时,细胞与核酸序列的结合位点具有不确定性,因此筛选所用的核酸库通常未对单链DNA或RNA的骨架进行过修饰,以减少筛选试验成本。
3筛选条件和方法
细胞是由多种生物大分子和小分子组成、能够进行新陈代谢的生命体。细胞膜表面包含蛋白质、多糖、脂类等多种物质,也会因周围环境变化而做出一定的形态变化。全细胞作为适配体筛选的靶标,它的复杂成分和生物活性导致全细胞筛选过程复杂、难度大。细胞在生物体内处在一个复杂的新陈代谢环境中,温度为37 ℃左右。但目前报道的筛选过程多在体外进行,因此筛选出的适配体有可能不会在体内环境下有效识别和结合靶细胞,从而导致筛选出的适配体性能变差。细胞表面的很多蛋白质种类相同,筛选出的适配体如果结合了非某种细胞特定的蛋白质,则不能保证适配体的特异性。为了解决这个问题,在筛选过程中需加入负筛选的步骤。根据McKeague等[15]统计的1990年以来的细胞靶标适配体的筛选仅占6%,下文中将系统介绍筛选过程,并对重要条件进行讨论。
全细胞适配体筛选过程包括几个步骤:核酸库与靶细胞的孵育,离心分离细胞与未结合的核酸序列,洗脱结合在细胞上的核酸序列,对这些结合序列进行负筛选。将负筛选后得到的核酸序列进行PCR扩增,得到次级库用于下轮筛选。次级库通过上述循环进行多轮筛选。将最后筛选的具有高亲和力的核酸序列测序,即得到适配体。其中负筛选的过程是可与靶标结合的序列与阴性细胞孵育,目标是去掉同时可与阴性细胞结合的核酸序列,以提高筛选序列的特异性。是否进行负筛过程,需要根据具体筛选的目的决定。最后筛选出的适配体还需要进行亲和力测定。根据筛选流程,下面重点介绍孵育条件、负筛选和分离方法。
3.1孵育条件
目前,绝大多数细胞靶标与适配体是在4 ℃孵育。Shamah等[16]提到4 ℃下可减少适配体的降解,而37 ℃筛选出的适配体结合能力更低。但Sefah等[46]则认为4 ℃下筛选的适配体在人体体温条件下结合能力会下降,因此应在37 ℃下筛选适配体。而Simaeys等[43]则通过实验证明两个温度下筛选出的适配体的结合能力并无明显差异。理论上,蛋白质结构随温度有所变化,但具体的变化程度因蛋白质而异,并不相同。由于全细胞适配体筛选并不针对具体的某种蛋白质,因此认为适配体结合的蛋白质在温度变化下结构改变很小,故筛选温度的影响可忽略不计。但如果已知适配体结合的蛋白质在不同温度时结构变化较大,则筛选温度会对其有很大的影响,应予以关注。4 ℃时,核酸的结构相对稳定,孵育时间可设置0.5~2 h不等。而37 ℃时核酸易降解,孵育时间不宜过长,通常为0.5 h。
由于操作简单,实验成本低,适配体与靶细胞孵育通常在体外进行。但也有将核酸库注入小鼠体内,研究其结合作用。Mi等[51]在小鼠肝脏内注射肿瘤细胞做成移植肝癌小鼠,饲养12天后,通过静脉注射随机RNA库,RNA长度为80 mer,20 min后处死小鼠取出瘤体,分离与瘤体结合的RNA分子,进行反转录扩增,再将次级库注射到抑制肝癌小鼠体内,经过多轮筛选最终得到适配体。体内筛选的最大的优势在于核酸适体可直接识别原位的潜在靶细胞,筛选在生理环境下进行,筛选出的适配体便于实际应用。因此,通过体内筛选开发的高亲和力和高特异性的核酸适配体,可用于鉴别不同组织及抑制体内靶细胞中特定靶分子,该种筛选方法具有极其重要的应用前景。
3.2负筛选
在靶细胞外选择其他细胞作为负筛选的阴性对照细胞,目的是除去非特异性结合的核酸序列,有效提高靶细胞的适配体的特异性。负筛选的阴性细胞要根据靶细胞的环境条件及筛选目的进行选择。已知靶细胞表面具有某些特定蛋白质时,需要选择不含这些蛋白质的细胞类型。如Kim等[31]以过表达上皮黏附分子(EpCAM)的肝癌细胞HepG2作为靶细胞,而以普通HepG2细胞作为阴性细胞。筛选特定癌细胞类型时,需要选择人体同一系统的其他癌细胞作为阴性细胞。如陈鑫等[47]用急性早游离白血病细胞NB4细胞作为筛选细胞,以慢性髓系白血病细胞株K562细胞作为阴性细胞。
3.3分离方法
细胞的适配体筛选主要采用离心分离,此外还有链霉亲和素磁珠分离法和惯性微流SELEX(i-SELEX)的方法。
离心法因操作简单而应用最普遍。先采用低速离心(1 000 r/min),使靶细胞和结合核酸物沉降,弃去未结合核酸的上清液。沉降物中加入缓冲液后置于94 ℃水浴中10 min,随后再通过高速离心(12 000 r/min),收取上清液,即为与靶细胞结合的核酸。该核酸扩增后作为次级库用于后续筛选。链霉亲和素磁珠法多用于蛋白质筛选,但也可用于全细胞适配体筛选,如Hung等[50]在建立芯片cell-SELEX的方法中,采用了联袂亲和素磁珠法分离靶细胞核酸结合物。具体为在磁珠表面修饰上皮细胞抗体,该抗体能与靶细胞特异性结合,从而在与靶细胞孵育后,磁珠能结合在靶细胞表面。当结合磁珠的靶细胞与核酸库孵育后,用磁珠分离出磁珠靶细胞适配体结合物。微流SELEX[50]分离方法是建立聚焦螺旋微流控通道,当靶细胞结合适配体后质量发生变化,运用惯性将靶细胞核酸结合物与未结合细胞的核酸分离,该方法的优势在微流控通道中可以运行单个细胞,有利于后续的收集及PCR扩增。
4细胞适配体的亲和力表征方法
筛选的细胞的适配体需要表征其亲和力。重要表征方法有流式细胞仪、共聚焦显微镜或荧光显微镜检测,此外还有活体成像系统。
利用流式细胞仪是全细胞适配体筛选中最常用的表征法。它的优势在于能够计算出靶细胞与适配体结合的平衡解离常数,一般用于检测最后筛选出的适配体与靶细胞的结合能力。利用共聚焦显微镜和荧光显微镜可定性地观察适配体与靶细胞及其他对照细胞的结合能力,可用来检测适配体的特异性,由于操作相对简单,一般用于筛选过程中每一轮筛选结束后检测次级库与靶细胞的结合能力,以此决定筛选何时终止,或是检测最终筛选出的适配体与靶细胞的亲和力和适配体与其他几种细胞的亲和力进行对比。活体成像系统主要用于直接检测体内的核酸适配体,可以通过图像直接观察适配体结合到肿瘤的过程,具体操作过程是将带有荧光标签的适配体溶液静脉注射到荷瘤大鼠或小鼠体内,将大鼠或小鼠放置在仪器内,然后观察屏幕上的图像,能够直接观测到适配体与肿瘤的结合速度和亲和力大小。
5总结与展望
利用全细胞的适配体的细胞识别作用,可在生物传感、分子成像、临床诊断、药物传输和疾病治疗等领域有很大的发展空间。针对目前的适配体筛选的低效率等瓶颈问题,有必要对筛选过程和筛选机理进一步研究。由于细胞表面的膜蛋白或多肽具有特殊的形态,将这些膜蛋白或多肽分离纯化后进行适配体筛选,无疑它们的形态会有所改变,不同于它们在细胞表面时的天然形态,因此提取细胞表面蛋白质后筛选的适配体有可能在实际细胞检测中是无效。而用全细胞靶标筛选适配体,针对完整细胞的天然形态和表面分子结构的筛选条件和环境更接近实体状态,有助于筛选的适配体在实际环境下的应用。全细胞的适配体大部分用于活体生命,而筛选过程则在体外进行。为提高筛选成功率和实际的可应用性,筛选过程应尽量接近体内环境。相比体外筛选,体内环境下进行适配体筛选则能够完全模拟适配体识别和结合靶标的过程,更有利于实际应用,但现有文献报道很少。目前的全细胞适配体筛选报道虽日渐增多,但在实际应用中还面临亲和力和特异性以及稳定性不好等不足。探索和建立高效、实用的全细胞适配体筛选方法十分必要,还需进一步深入进行研究。
参考文献:
[1]Liang C, Guo B H, Wu H, et al. Nat Med, 2015, 21(3): 288
[2]Wang J, Sefah K, Tan W H, et al. Chem Asian J, 2013, 8(10): 2417
[3]Sefah K, Bae K M, Tan W H, et al. Int J Cancer, 2013, 132(11): 2578
[4]Liu J, Liu H X, Tan W H, et al. PLoS One, 2012, 7(5): e37789
[5]Jimenez E, Sefah K, Tan W H, et al. PLoS One, 2012, 7(10): e46222
[6]Parekh P, Tang Z W, Tan W H, et al. Anal Chem, 2010, 82(20): 8642
[7]Sefah K, Meng L, Tan W H, et al. PLoS One, 2010, 5(12): e14269
[8]Simaeys D, Lopez C D, Tan W H, et al. PLoS One, 2010, 5(11): e13770
[9]Fang X H, Tan W H. Accounts Chem Res, 2010, 43(1): 48
[10]Mao Y, Hu J, Peng M Y, et al. Int J Mol Sci, 2012, 13(3): 3341
[11]Meng H M, Fu T, Zhang X B, et al. Natl Sci Rev, 2015, 2(1): 71
[12]Zhu H J, Li J, Tan W H. Chemmedchem, 2015, 10(1): 39
[13]Zhu G Z, Ye M, Tan W H, et al. Chem Commun, 2012, 48(85): 10472
[14]Tan W H, Fang X H. Aptamers Selected by Cell-SELEX for Theranostice. Berlin: Springer, 2015
[15]McKeague M, McConnell E M, Toledo J C, et al. J Mol Evol, 2015, 81(5): 150
[16]Shamah S M, Healy J M, Cload S T. Chem Inform, 2008, 39(18): 13
[17]Zhao Z L, Li Y, Yuan C. Biotechnology Bulletin, 2014(5): 52
赵仲麟, 李燕, 袁超. 生物技术通报, 2014(5): 52
[18]Ohuchi S P, Ohtes T, Nakamua Y, et al. Biochimie, 2006, 88(7): 897
[19]Kim Y H, Sung H J, Kim S, et al. Cancer Lett, 2011, 313(1): 76
[20]Shangguan D H, Meng L, Tan W H. Anal Chem, 2008, 80(3): 721
[21]Blank M, Weinschenk T, Priemer M et al. Biol Chem, 2001, 276(19): 16464
[22]Wang C L, Zhang M, Yang G, et al. J Biotechnol, 2003, 102(1): 15
[23]Zhao N X, Pei S N, Parekh P, et al. Int J Biochem Cell B, 2014, 51(6): 10
[24]Benedetto G, Hamp T J, Wesselman P J, et al. Nucleic Acid Ther, 2015, 25(3): 162
[25]Li X L, An Y, Jin J, et al. Anal Chem, 2015, 87(9): 4941
[26]Liang C, Guo B S, Wu H, et al. Nat Med, 2015, 21(3): 288
[27]Zhou J H, Satheesan S, Burnett J C, et al. Chem Biol, 2015, 22(3): 379
[28]Lu M, Zhou L, Zheng X H, et al. Cancer Biomark, 2015, 15(2): 163
[29]Wu X Q, Zhao Z L, Bai H R, et al. Theranostics, 2015, 5(9): 985
[30]Hou Z G, Meyer S, Propson N E, et al. Cell Res, 2015, 25(3): 390
[31]Kim J W, Kim E Y, Kim S Y, et al. Mol Cells, 2014, 37(10): 742
[32]Cao H Y, Yuan A H, Chen W, et al. BMC Cancer, 2014, 14: 699
[33]Li X L, Zhang W Y, Liu L, et al. Anal Chem, 2014, 87(13): 6596
[34]Wang Y Y, Luo Y, Bing T, et al. PLoS One, 2014, 9(6): e100243
[35]Zhao N X, Pei S N, Parekh P, et al. Int J Biochem Cell B, 2014, 51(6): 10
[36]Kim E Y, Kim J W, Kim W K, et al. PLoS One, 2014, 9(5): e97747
[37]Kunii T, Ogura S I, Mie M, et al. Analyst, 2011, 136: 1310
[38]Chen H W, Medley C D, Sefah K, et al. Chemmedchem, 2008, 3(6): 991
[39]Li Z Z, Han Y W, Liu L L, et al, Biotechnology, 2009, 19(3): 42
李真真, 韩跃武, 刘玲玲, 等. 生物技术, 2009, 19(3): 42
[40]Ninomiya K, Kaneda K, Kawashima S, et al. Bioorg Med Chem Lett, 2013, 23(6): 1797
[41]Li W M, Bing T, Wei Z Z, et al. Biomaterials, 2014, 35(25): 6998
[42]Li X H, Zhang W Y, Liu L, et al. Anal Chem, 2014, 86(13): 6596
[43]Simaeys D V, Sefah K, Sutphen R, et al. PLoS One, 2010, 5(11): e13770
[44]Graham J C, Zarb H. PLoS One, 2012, 7(4): e36103
[45]Hung L Y, Wang C H, Hsu K F, et al. Lab Chip, 2014, 14(20): 4017
[46]Sefah K, Tang Z W, Shangguan D H, et al. Leukemia, 2009, 23(2): 235
[47]Chen X, Li W B, Wang K Y, et al. Chinese Journal of Clinical Laboratory Science, 2012, 30(7): 518
陈鑫, 李卫滨, 王开宇, 等. 临床检验杂志, 2012, 30(7): 518
[48]Chen L, Tang F, Liang J Y, et al. Chinese Journal of Joint Surgery, 2013, 7(6): 827
陈亮, 唐芳, 梁津瑜, 等. 中华关节外科杂志, 2013, 7(6): 827
[49]Wang J B, Li Y H, Yan P K, et al. China Medicine and Pharmacy, 2012, 2(24): 25
汪江波, 李艳红, 严鹏科, 等. 中国医药科学, 2012, 2(24): 25
[50]Birch C M, Hou H M, Han J Y, et al. Sci Rep, 2015, 5: 11347
[51]Mi J, Liu Y M, Rabbani Z N, et al. Nat Chem Biol, 2010, 6(1): 22
Research advances of aptamer selection for whole cell
LIU Pinduo, QU Feng*
(School of Life Science, Beijing Institute of Technology, Beijing 100081, China)
Abstract:Aptamers are single stranded DNA (ssDNA) or RNA that may bind to small molecules, proteins, cells, microorganisms, and other targets. Typically, aptamers are generated by a selection process referred to as systematic evolution of ligands by exponential enrichment (SELEX). Aptamers that bind with high affinity and specificity to proteins that reside on the cell surface have potential utility in the fields of biosenser, molecular imaging, medical diagnosis, drug delivery, and disease treatment. However, till now, available aptamers are very limited mainly due to the complex and difficulty screening of aptamer and their poor properties. Using purified cell surface proteins as target has the disadvantage of changing structure. When target proteins are present in a modified state, the isolated aptamers might not recognize the natural structure of some proteins. Aptamers screening targeting whole cell has the potential to be used as cell probe for directing whole cell analysis. It does not need to understand the molecular structure of the cell surface protein and maintain the natural state of cells in the screening process. This review mainly focuses on the advances of aptamer selection for whole cell from 2008 to 2015. The contents include the classification of target cells, design of library, methods for cell-ssDNA complex separation and affinity characterization. The sequences of aptamers reported are listed.
Key words:aptamers; cells; selection; systematic evolution of ligands by exponential enrichment (SELEX); review
中图分类号:O658
文献标识码:A
文章编号:1000-8713(2016)04-0382-07
基金项目:国家自然科学基金项目(21175011,21375008);国家“973”计划项目(2012CB910603).
*收稿日期:2015-12-14
DOI:10.3724/SP.J.1123.2015.12015
多尺度靶标的核酸适配体筛选研究进展专栏
·专论与综述
*通讯联系人.Tel:(010)68918015,E-mail:qufengqu@bit.edu.cn.
Foundation item: National Nature Science Foundation of China (Nos. 21175011, 21375008); National “973” Project (No. 2012CB910603).
