小分子靶标的核酸适配体筛选的研究进展
2016-05-12赵新颖石冬冬
王 勇, 赵新颖,2, 石冬冬, 杨 歌, 屈 锋*
(1. 北京理工大学生命学院, 北京 100081; 2. 北京市理化分析测试中心, 有机材料检测技术
与质量评价北京市重点实验室, 北京 100089; 3. 中国农业科学院饲料研究所, 北京 100081)
小分子靶标的核酸适配体筛选的研究进展
王勇1,赵新颖1,2,石冬冬3,杨歌1,屈锋1*
(1. 北京理工大学生命学院, 北京 100081; 2. 北京市理化分析测试中心, 有机材料检测技术
与质量评价北京市重点实验室, 北京 100089; 3. 中国农业科学院饲料研究所, 北京 100081)
摘要:核酸适配体(aptamer)是通过指数富集配体系统进化(SELEX)技术筛选得到的核糖核酸(RNA)或单链脱氧核糖核酸(ssDNA)。核酸适配体通过高亲和力特异性地识别小分子、蛋白质、细胞、微生物等多种靶标,在生物、医药、食品和环境检测等领域的应用日渐增多。但目前实际可用的核酸适配体有限,其筛选过程复杂,筛选难度大,制约了其应用。与生物大分子、细胞和微生物等靶标不同,小分子靶标与核酸分子的结合位点少、亲和力弱,且靶标通常需要固定在载体上。此外,小分子靶标结合核酸形成的复合物与核酸自身的大小、质量、电荷性质等方面差异较小,二者的分离难度大。故小分子靶标的核酸适配体筛选过程与大分子和细胞等复合靶标相比有明显差异,筛选难度更大。因此需要根据其自身结构特点和核酸适配体的应用目的选定靶标或核酸库的固定方法,优化靶标核酸复合物的分离方法。本文介绍了不同类型小分子(具有基团差异的单分子、含相同基团分子和手性分子等)靶标的选择及其核酸适配体的筛选方法,并对核酸库的设计、与靶标结合的核酸的分离方法和亲和作用表征方法进行了介绍,列出了自2008年以来报道的40余种小分子靶标的核酸适配体序列和复合物的平衡解离常数(Kd)。
关键词:核酸适配体;小分子靶标;筛选;综述
核酸适配体(aptamer)是通过链内碱基间的氢键作用折叠形成稳定的发卡、茎环、假结、口袋、凸环和G-四链体等二级或三级结构,并与靶标产生空间结构匹配的高亲和力和特异性结合的核糖核酸(RNA)和单链脱氧核糖核酸(ssDNA)[1,2]。核酸适配体一般由几十个核苷酸组成,相对分子质量小、易穿透细胞膜、性质稳定、容易制备和修饰,其结合的靶标范围广,包括离子、小分子、蛋白质、细胞和微生物等。目前,蛋白质、细胞和微生物等靶标的核酸适配体主要应用于疾病诊断和治疗、药物研发、蛋白质组学以及基因表达调控的机理研究[3]等。药物、氨基酸、抗生素等小分子靶标的核酸适配体因其可特异性识别和区分小分子间极微小的结构差异,通常作为化学和生物传感器的识别分子用于食品和环境检测以及生物分析等领域。理论上,任何靶标都可以筛选到核酸适配体,但目前实际可用的核酸适配体数量仍然非常有限。核酸适配体筛选过程复杂,筛选难度大,解决各种靶标的高效筛选问题是加快其广泛应用的关键一步[4]。
核酸适配体可以通过指数富集配体系统进化(SELEX)技术从随机寡核苷酸库(核酸库)中筛选得到。SELEX过程包括核酸库的建立、核酸库与靶标相互作用的判定、与靶标结合的核酸分子的分离和扩增、循环筛选、筛选后的核酸序列测定以及序列与靶标的亲和作用表征等步骤。完整的SELEX过程相当繁杂,需耗费大量的时间、人力和物力,成功率低,筛选效果常不能令人满意。目前的核酸适配体筛选难以实现自动化,还没有标准化的程序适用于不同类型的靶标。
与生物大分子、细胞和微生物等复合靶标不同,小分子靶标(Mr<1 000 )的结构简单,与核酸的结合位点少、亲和力弱。小分子靶标与核酸结合的复合物与核酸分子自身的大小、质量、电荷等性质差异较小,二者的分离难度大,通常需将靶标固定在载体上进行筛选,而小分子靶标可用于键合固定的基团选择性小。故小分子靶标的核酸适配体筛选过程与大分子和细胞等复合靶标相比有明显差异,筛选难度更大。因此,需要根据小分子靶标的自身结构特点和核酸适配体的应用目的,选定靶标或核酸库的固定方法,优化靶标-核酸复合物的分离方法。目前国内外关于小分子靶标的核酸适配体的综述主要针对核酸适配体作为传感分子在食品安全分析检测中的应用进展[5-8],专门针对筛选方法的总结报道不多。本文介绍了小分子靶标的选择及其核酸适配体的筛选方法、核酸库的设计、复合物的固相/液相分离方法和亲和作用表征方法。统计了40余种小分子靶标的核酸适配体序列和平衡解离常数(Kd)。
1小分子靶标的类型
核酸适配体的筛选需考虑不同的研究和应用目的,选择不同特性的靶标。蛋白质靶标的核酸适配体主要识别其氨基酸序列,细胞或微生物靶标的核酸适配体主要识别其表面的特定蛋白质,小分子靶标的核酸适配体主要识别其基团或空间结构。小分子靶标通常包括不同基团组成的分子、含相同基团的同类分子和基团相同空间结构不同的手性分子。
1.1不同基团组成的分子
以某种分子为靶标筛选的核酸适配体可用于构建识别该分子的化学和生物传感器。由于核酸适配体对其他杂质分子或靶标的类似物没有响应或仅有弱响应,故可实现对该分子靶标的高选择性识别。1994年,Jenison等[9]用琼脂糖亲和柱筛选得到茶碱的RNA 核酸适配体,Kd为100 nmol/L。茶碱与咖啡因的区别虽仅是N7原子上的一个甲基,但该核酸适配体与茶碱的亲和力比咖啡因高10 000倍。相同条件下,即使是茶碱抗体的区分因子也只有1 000倍,说明该RNA 核酸适配体对官能团有微小差异的茶碱和咖啡因具有极高的识别和区分能力。
核酸适配体筛选通常需将靶标分子固定在载体上,而固定靶标的载体也可能与核酸库中的核酸分子作用,因此有必要进行负筛选,即将核酸库与载体预先混合孵育,扣除与载体结合的核酸分子,然后用余下的核酸库与靶标作用。负筛选可以排除载体与核酸库的结合,提高核酸适配体筛选效率和特异性。Jo等[10]筛选双酚A(BPA)的DNA 核酸适配体,在第3轮后以乙醇胺修饰的环氧琼脂糖凝胶6B载体为靶标进行负筛选,去除载体结合的DNA,最后得到BPA 核酸适配体,用荧光法测定Kd为8.3 nmol/L。该核酸适配体可以区分与BPA只有1个甲基差异的双酚B,也可以区分有2个甲基差异的4,4′-双酚。
为了提高核酸适配体的特异性还可以进行反筛选。即将核酸库和与靶标具有相似结构的分子混合孵育,扣除可能识别靶标相似结构的分子,用余下的核酸库与靶标进行筛选,以减小核酸适配体对相似结构分子的结合,提高核酸适配体对靶标的特异性识别。Zhou等[11]通过环氧树脂磁珠筛选链霉素的DNA 核酸适配体,每2轮筛选后都以修饰基团乙醇胺为靶标进行1次反筛选,经过8轮筛选和3轮反筛选得到核酸适配体 STR1。液相色谱法测定其Kd为199.1 nmol/L。STR1对链霉素的结合率为76.5%,而对新霉素和卡那霉素等其他含相似结构的氨基苷类抗生素的结合率均低于10%。
1.2含相同基团的同类分子
以含有相同基团的一类分子为靶标筛选的核酸适配体能识别该基团,对含有该基团的一类分子具有亲和力,而对不含该基团的分子亲和力低或无亲和力。为了提高特异性,可采用切换筛选,即分别以含相同官能团和不含官能团的分子为靶标进行筛选。切换筛选是在不同的目标靶间进行切换,是同时针对多目标靶进行筛选的一种方法。Derbyshire等[12]以庆大霉素和阿泊拉霉素、卡那霉素和妥布霉素、巴龙霉素和新霉素、链霉素和二双氢链霉素4组氨基糖苷类抗生素为靶标,通过切换筛选得到可识别氨基糖苷类抗生素的RNA 核酸适配体。表面等离子共振法(SPR)测定该核酸适配体对氨基糖苷类靶标的Kd为10 nmol/L。Mei等[13]以对硝基苯磺酰基修饰的L-赖氨酸为靶标,将其固定在琼脂糖微珠上,在第12~20轮筛选中以硝基苯甲酰修饰末位氨基的L-赖氨酸为靶标进行反筛选,得到2条氨烷基类分子的核酸适配体M6b-M14和M13a。等温滴定量热法测定其Kd分别为(3.98±0.72)和(4.59±1.56) μmol/L,它们对对硝基苯甲酰修饰末位氨基的L-赖氨酸和L-赖氨酸的亲和力较弱。目前报道含相同官能团的分子靶标核酸适配体有氨基糖甙类、四环素类、麦角生物碱类等。
1.3手性分子
以R或S型分子筛选的核酸适配体可以结合该型的手性分子,区别其空间结构不同的对映体。手性分子的核酸适配体可用作手性分子分离的拆分剂。Kim等[14]以物质的量比为1∶1混合的异丁苯丙酸外消旋体布洛芬为靶标,筛选得到5条DNA序列。其中3条DNA序列对外消旋体和R-异丁苯丙酸都有亲和力,是R-异丁苯丙酸的核酸适配体。利用平衡过滤法测定3条DNA序列复合物的Kd分别为3.0、5.2和3.2 μmol/L;另外2条DNA序列对外消旋体和S-异丁苯丙酸都有亲和力,是S-异丁苯丙酸的核酸适配体,其Kd分别为1.5和3.8 μmol/L。以上5条核酸适配体可用于药物动力学分析中体内布洛芬的检测以及布洛芬手性药物的分离、纯化等。Yang等[15]将L-色氨酸固定在琼脂糖微珠上,选用的核酸库先以L-酪氨酸为靶标进行反筛选,再与L-色氨酸相互作用。经过12轮筛选得到特异性识别L-色氨酸的DNA 核酸适配体 Trp3a-1,其含有34个碱基,可用于D/L-色氨酸的手性拆分。荧光法测定其复合物的Kd为1.757 μmol/L。可以预见,手性分子的核酸适配体将在生物和化学制药行业中的天然多糖及衍生物、大环抗生素、手性大环配体、配体交换复合物、手性表面活性剂等的手性分离和检测方面有巨大的应用潜力[16]。
针对这些小分子靶标筛选的核酸适配体,可以特异性识别特定的基团、分子结构及手性。核酸适配体可以是ssDNA或RNA。ssDNA因其性质稳定,筛选成本低,是广泛应用的小分子核酸适配体。目前报道的以单分子为靶标筛选的核酸适配体以ssDNA为主(见表1)。

表 1 小分子靶标的核酸适配体序列及其平衡解离常数

表 1 (续)

表 1 (续)
* ‘t’ indicates 5-(2-(6-(arginamido)hexylamino)-2-oxoethyl)-dU.
2核酸库的设计
无论是生物大分子,还是细胞、微生物等复合靶标,SELEX筛选中均需要设计合理的核酸库。核酸库可选择RNA或ssDNA库。选择RNA库时,需要将RNA分子反转录为DNA才能进行扩增。ssDNA库比RNA库稳定性高,不需进行转录,容易合成和纯化。使用ssDNA库筛选,筛选步骤较少,对操作环境的要求较低。对核酸库进行修饰可以增加核酸库的稳定性,同时也利于形成特定的结构以增加对靶标的亲和力[4,59-61],如戊糖2′位的取代(2′-氨基、2′-甲氧基、2′-氟)、磷酸修饰(磷酸酯、磷酰胺、吗啉)、肽核酸(PNA)、锁核酸(LNA)等。此外,核酸库的设计还可以借助计算机辅助完成。以AutoNock为代表的多种分子模拟软件已经可以完成基于结构的药物设计、先导化合物优化、虚拟筛选、组合库设计、化学机制研究等多种工作,并且大部分都有可视化的效果图。核酸库设计的合理性、靶标和核酸库结合亲和力的预测、核酸库修饰效果的评价以及模拟的筛选路线等,都有望通过计算机辅助设计得以实现[62]。
小分子的核酸适配体筛选较大分子等更为困难,所以对核酸库的选择和设计要求更高。Liu等[63]将总长为103个碱基、随机区域为59个碱基的ssDNA核酸库转录为RNA核酸库,然后与固定血红素的亲和色谱柱孵育。再用含血红素的淋洗液洗脱,获得与血红素有特异性结合的RNA序列。然后再反转录为DNA序列,经PCR扩增,获得新一级的ssDNA核酸库。如此反复筛选,可得到血红素的RNA 核酸适配体。该课题组[64]还建立了用偶氮苯修饰的腺苷酸取代全部腺苷酸的修饰RNA核酸库,经过8轮筛选得到在可见光照射下与血红素结合而在紫外光照下与血红素解离的光敏RNA 核酸适配体,该研究为光敏小分子核酸适配体的筛选提供了重要的参考。然而由于修饰的核酸分子不能直接进行PCR扩增,反复筛选无疑增加了筛选的复杂程度,所以选用修饰的核酸库更需要高效的筛选方法,以减少筛选步骤[44]。计算机辅助设计的推广有望为解决小分子靶标的核酸适配体筛选问题提供新的途径,但目前的报道还不多。
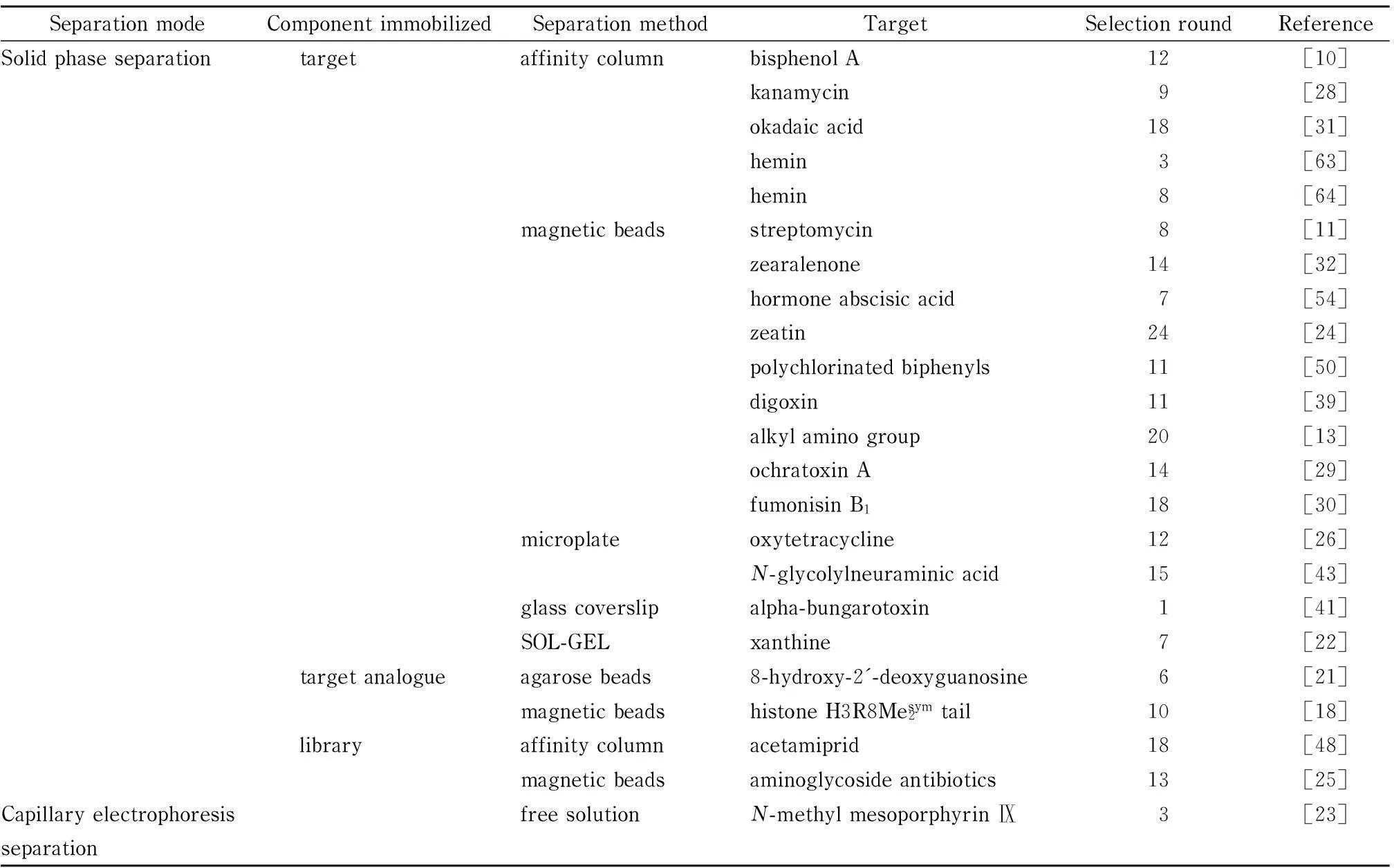
表 2 部分小分子靶标的核酸适配体筛选轮次与分离方法
3与靶标结合的核酸的分离方法
在SELEX筛选过程中,简单、快速地得到与靶标结合的核酸是提高筛选效率的关键。固相分离法将靶标或核酸库通过化学键合固定到载体上,利用不同的溶剂分别去除和清洗未与靶标结合的核酸,再通过溶剂洗脱使与靶标结合的核酸解离下来。该方法操作简单,技术成熟,已被广泛使用。此外,利用毛细管电泳(CE)法可以实现靶标-核酸复合物与未结合核酸在自由溶液中的快速分离,得到的靶标-核酸复合物可直接用于后续的PCR扩增。小分子靶标的核酸适配体筛选循环通常在3~20轮不等,而使用CE法进行筛选一般仅需1~4轮,是高效的筛选方法。部分小分子靶标的核酸适配体筛选和分离方法见表2。
3.1固相分离
3.1.1固定靶标
琼脂糖亲和色谱柱是常见的固相分离载体。早在SELEX技术发展之初,Ellington等[1]就是通过交联琼脂糖珠亲和色谱柱筛选得到与代谢辅因子结构相似的染料辛巴蓝3GA、活性红120、活性黄86、活性棕10、活性绿19和活性蓝4的RNA 核酸适配体。利用这种方法已经筛选得到多种小分子的核酸适配体,如ATP[65,66]、S-腺苷同型半胱氨酸[67]、L-精氨酸[68]和辅酶A[69]等。Song等[28]利用溴化氰活化的琼脂糖亲和色谱柱固定卡那霉素,经过9轮筛选得到DNA 核酸适配体,荧光法测定的Kd为78.8 nmol/L。应用琼脂糖亲和色谱柱一般经过3~18轮筛选可以得到小分子靶标的核酸适配体。用琼脂糖亲和色谱柱固定小分子靶标技术较成熟,但所需样品量大,且靶标和载体的修饰方法选择性不多,固定靶标的反应时间较长。
磁珠是近年来广泛使用的固相载体,它对靶标用量少,分离操作简便快速,且磁珠表面的复合物无需洗脱就可直接进行PCR扩增。此外,磁珠载体的表面修饰形式种类较多,如环氧基活化磁珠[11]、链霉亲和素包被磁珠[27,58]、羧基修饰磁珠[30]、氨基修饰磁珠[32]、甲苯磺酰基修饰磁珠[55,70]等。利用磁珠固相载体进行分离,一般经过8~24轮筛选可以得到小分子靶标的核酸适配体。McKeague等[30]利用羧酸修饰磁珠固定伏马菌素B1,经过18轮筛选得到了6条DNA序列,其中核酸适配体 FB139的Kd为(100±30) nmol/L。Chen等[32]利用氨基修饰磁珠固定玉米烯酮,经过14轮筛选,得到DNA 核酸适配体 8Z(31)的Kd为(41±5) nmol/L。Joeng等[40]利用氨基和羧基反应将双氯芬酸固定在含氨基的磁珠M-270表面,通过Flu-Mag SELEX从容量为2.4×1014的DNA库中筛选到56条可与其结合的DNA 核酸适配体。Niazi等[55]以氧四环素为正筛选靶标、四环素和多西环素作为反筛选靶标,利用甲苯磺酰基活化磁珠筛选了7条四环素特异的DNA 核酸适配体,其Kd为63~483 nmol/L。Kiani等[39]将生物素酰化的地高辛包覆在链霉亲和素磁珠表面,经过11轮筛选得到地高辛的DNA 核酸适配体,荧光法测定的Kd为8.2 nmol/L。Grozio等[58]将生物素修饰的脱落酸固定在链霉亲和素磁珠表面,经过7轮筛选,得到其DNA 核酸适配体,Kd为(0.80±0.07) μmol/L。
微孔板是酶联适配体分析法(ELAA)常用的反应载体,可用于固定小分子靶标。先将靶标连接到介质蛋白质上,再通过蛋白质包被微孔板将靶标固定到微孔板表面。该法样品用量少、操作简单,但存在孵育时接触面积小、所需时间长的不足。Kim等[26]以牛血清白蛋白为介质蛋白质,间接将氧四环素固定到微孔板上,经过12轮筛选得到其核酸适配体,通过ELAA测定Kd为4.7 nmol/L。Gong等[43]将N-糖神经氨酸与羟基丁二酰亚胺反应活化后连接到牛血清白蛋白的N-末端,利用微孔板载体筛选了N-糖神经氨酸的核酸适配体。载玻片也可以作为固相载体。Lauridsen等[41]提出一步筛选α-金环蛇毒素DNA 核酸适配体的方法。靶标通过N-羟基丁二酰亚胺活化聚乙二醇固定到载玻片上,经过1轮选择成功得到DNA 核酸适配体,表面等离子共振法测定Kd为7.58 μmol/L。
溶胶-凝胶(SOL-GEL)是室温下合成的硅酸盐材料,通过水解和缩聚反应形成纳米级的空洞和微米级的通道[71],通常用来固定蛋白质、酶等生物大分子。利用SOL-GEL的空洞和通道可简单地固定小分子靶标而不需化学键合反应,固定后的小分子仍然可与核酸分子接触产生相互作用。Bae等[22]利用SOL-GEL经过7轮筛选到灵敏度低至1 μmol/L的黄嘌呤特异性的核酸适配体,Kd约为10 μmol/L,而将黄嘌呤通过共价键固定到聚合树脂进行SELEX却不能筛选到特异的核酸适配体。该研究显示SOL-GEL法固定靶标黄嘌呤筛选小分子靶标的核酸适配体具有更好的效果。
3.1.2固定靶标类似物或核酸库
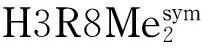
当小分子靶标难以固定时,也可将核酸库固定到载体上。这种方法需要在核酸库的随机序列中设计一段用于固定的碱基序列作为“桥接”序列,同时在载体上设计一段互补序列。利用该“桥接”序列与载体上的互补序列杂交,通过氢键作用将核酸库固定在载体上。当核酸库中的核酸分子与靶标结合时,“桥接”序列构象发生变化,从载体表面释放出来。Stoltenburg等[25]将ssDNA核酸库固定在磁珠表面,利用Capture-SELEX经过13轮筛选得到卡那霉素A的核酸适配体。He等[48]通过在琼脂糖亲和色谱柱表面固定ssDNA核酸库筛选了啶虫脒的核酸适配体。这种固定核酸库的方法可以解决难固定的小分子靶标的核酸适配体的筛选问题,同时保持了靶标的天然结构不受破坏。然而,当核酸结合靶标后,如不能产生构象变化,结合的序列则不能脱离载体,也不能与核酸库中其他序列分离,不能实现筛选。此外,由于核酸分子的碱基互补配对的作用力较弱,使载体表面固定的核酸结合力不如化学键合强,因此也可能导致部分未结合靶标的核酸也在洗脱过程中脱落下来,产生假阳性,降低筛选效率。
3.2毛细管电泳分离
靶标和核酸库在自由溶液中相互作用时有利于保持二者的天然结构,也可以模拟生物体环境,因此在自由溶液中实现靶标结合核酸的分离是最为理想的方法。目前,仅有CE法可在自由溶液中实现靶标与核酸的相互作用以及靶标-核酸复合物与未结合核酸的同步分离。CE法中可将迁移率有明显差异的靶标-核酸复合物与未结合核酸进行分离,同时可以在毛细管出口端定时收集靶标-核酸复合物,该过程为CE-SELEX。一般CE-SELEX只需要1~4轮筛选就可获得蛋白质等生物大分子的核酸适配体。但是由于小分子靶标结合核酸形成的复合物与未结合的核酸相比迁移率差异很小,在CE中难以分离,通常认为CE-SELEX并不适合小分子靶标的核酸适配体筛选。最近,Yang等[23]提出靶标-核酸复合物与未结合核酸虽不能完全分离,但二者的迁移时间仍有差异,通过对未结合核酸电泳峰的前段组分进行收集,仍可进行小分子核酸适配体的筛选。他们在25 mmol/L Tris-192 mmol/L 甘氨酸-5 mmol/L KH2PO4溶液(pH 8.3)中经过3轮筛选得到小分子N-甲基卟啉二丙酸(NMM)的DNA 核酸适配体,荧光法测定的Kd为(0.88±0.12) μmol/L。该核酸适配体与初始核酸库相比,亲和力提高了50倍以上。该结果证明了CE-SELEX用于筛选小分子靶标的核酸适配体的可行性。本课题组也利用相似方法筛选了克伦特罗的核酸适配体,并取得了较好的结果(文章待发表)。
4亲和作用表征
靶标与随机核酸库中的核酸分子具有高亲和性、特异性的结合是SELEX筛选核酸适配体的基础。经过多轮筛选,靶标与次级库的亲和力逐渐增加,强于与初始核酸库的作用。因此,靶标与核酸库和次级库的结合力可用于定性评价每轮筛选的效果,毛细管区带电泳和亲和毛细管电泳法都可用于快速、定性地表征亲和力的相对大小。此外,多轮筛选得到的核酸适配体与靶标结合形成的复合物的Kd通常用于表示亲和力强弱。计算复合物的Kd可用组分分离法,即使溶液中的靶标、靶标-核酸复合物与未结合核酸完全分离,依据其平衡浓度求算Kd,所用方法有透析法、超速离心法、超滤法、凝胶电泳法、毛细管电泳法和液相色谱法[11,72-75]等。也可以不经分离,直接利用靶标、靶标-核酸复合物与未结合核酸混合溶液测定,所用方法有荧光各向异性/偏振、圆二色谱、SPR等。对小分子靶标进行亲和力表征时,所用方法有超滤法[11,21,55]、荧光光谱法[28-31]、荧光各向异性/偏振法[42]、紫外光谱法[13,17,24]、ELAA法[26,43]、SPR法[12,18,19]等。荧光光谱法灵敏度高、操作简单、定量准确,是测定Kd的主要方法。例如Barthelmebs等[29]利用羧基荧光素标记的DNA 核酸适配体与固定于磁珠表面的赭曲霉毒素A相互作用,利用荧光光度计测定结合的核酸适配体,通过拟合饱和曲线得到核酸适配体的Kd为96 nmol/L。SPR法也是亲和力测定的常用方法。Rouah-Martin[56]等将生物素修饰的靶标固定到链霉亲和素包被的SPR芯片上,通过测定不同浓度的核酸适配体流经芯片表面时的响应值,测定了麦角生物碱核酸适配体的Kd为44 nmol/L。此外,紫外光谱法、ELAA法测定Kd的应用也较多。
5展望
小分子靶标的核酸适配体可用作各种传感器的识别分子,解决实际样品中小分子快速、灵敏检测的问题。但目前可应用的小分子靶标的核酸适配体的数量和效果远远不能满足实际需要。深入理解小分子靶标的核酸适配体筛选的本质和SELEX过程的特点,克服筛选过程中的困难,提高小分子靶标的核酸适配体的亲和力和特异性,是小分子靶标的核酸适配体得以广泛应用的关键。纳米材料、高通量测序和计算机辅助设计等多种新技术的快速发展有望为小分子靶标的核酸适配体的高效筛选提供新方法和新思路,也是核酸适配体筛选的发展方向。
参考文献:
[1]Ellington A D, Szostak J W. Nature, 1990, 346(6287): 818
[2]Tuerk C, Gold L. Science, 1990, 249(4968): 505
[3]Proske D, Blank M, Buhmann R, et al. Appl Microbiol Blot, 2005, 69(4): 367
[4]Klussmann S. The Aptamer Handbook: Functional Oligonucleotides and Their Applications. Weinheim: Wiley-VCH, 2006
[5]Zhou C L, Xu H X. Chinese Journal of Frontier Health and Quarantine, 2011, 34(4): 276
周成林, 许化溪. 中国国境卫生检疫杂志, 2011, 34(4): 276
[6]Liang M, Liu R, Su R X, et al. Progress in Chemistry, 2012, 24(7): 1378
梁淼, 刘锐, 苏荣欣, 等. 化学进展, 2012, 24(7): 1378
[7]Xu D M, Wu M, Zhou Y, et al. Chinese Journal of Analytical Chemistry, 2011, 39(6): 925
徐敦明, 吴敏, 邹远, 等. 分析化学, 2011, 39(6): 925
[8]Wu J, Zhu Y, Xue F, et al. Microchim Acta, 2014, 181(5/6): 479
[9]Jenison R D, Gill S C, Pardi A, et al. Science, 1994, 263(5152): 1425
[10]Jo M, Ahn J Y, Lee J, et al. Oligonucleotides, 2011, 21(2): 85
[11]Zhou N, Wang J, Zhang J, et al. Talanta, 2013, 108: 109
[12]Derbyshire N, White S J, Bunka D H J, et al. Anal Chem, 2012, 84(15): 6595
[13]Mei H, Bing T, Yang X, et al. Anal Chem, 2012, 84(17): 7323
[14]Kim Y S, Hyun C J, Kim I A, et al. Bioorg Med Chem, 2010, 18(10): 3467
[15]Yang X, Bing T, Mei H, et al. Analyst, 2011, 136(3): 577
[16]Kalikova K, Riesova M, Tesarova E. Cent Eur J Chem, 2012, 10(3): 450
[17]Liu M, Kagahara T, Abe H, et al. Bioorg Med Chem Lett, 2009, 19(5): 1484
[18]Hyun S, Lee K H, Han A, et al. Nucleic Acid Ther, 2011, 21(3): 157
[19]Han S R, Yu J, Lee S W. Biochem Bioph Res Co, 2014, 448(4): 397
[20]Boese B J, Corbino K, Breaker R R. Nucleos Nucleot Nucl, 2008, 27(8): 949
[21]Miyachi Y, Shimizu N, Ogino C, et al. Bioorg Med Chem Lett, 2009, 19(13): 3619
[22]Bae H, Ren S, Kang J, et al. Nucleic Acid Ther, 2013, 23(6): 443
[23]Yang J, Bowser M T. Anal Chem, 2013, 85(3): 1525
[24]Qi C, Bing T, Mei H, et al. Biosens Bioelectron, 2013, 41: 157
[25]Stoltenburg R, Nikolaus N, Strehlitz B. J Anal Methods Chem, 2012: 415697
[26]Kim C H, Lee L P, Min J R, et al. Biosens Bioelectron, 2014, 51: 426
[27]Wochner A, Menger M, Orgel D, et al. Anal Biochem, 2008, 373(1): 34
[28]Song K M, Cho M, Jo H, et al. Anal Biochem, 2011, 415(2): 175
[29]Barthelmebs L, Jonca J, Hayat A, et al. Food Control, 2011, 22(5): 737
[30]McKeague M, Bradley C R, De Girolamo A, et al. Int J Mol Sci, 2010, 11(12): 4864
[31]Eissa S, Ng A, Siaj M, et al. Anal Chem, 2013, 85(24): 11794
[32]Chen X, Huang Y, Duan N, et al. Anal Bioanal Chem, 2013, 405(20): 6573
[33]Ma X, Wang W, Chen X, et al. Eur Food Res Technol, 2014, 238(6): 919
[34]Ma X, Wang W, Chen X, et al. Food Control, 2015, 47: 545
[35]Malhotra S, Pandey A K, Rajput Y S, et al. J Mol Recognit, 2014, 27(8): 493
[36]Chen X, Huang Y, Duan N, et al. Microchim Acta, 2014, 181(11/12): 1317
[37]McKeague M, Velu R, Hill K, et al. Toxins, 2014, 6(8): 2435
[38]Chen X, Huang Y, Duan N, et al. J Agric Food Chem, 2014, 62(42): 10368
[39]Kiani Z, Shafiei M, Rahimi-Moghaddam P, et al. Anal Chim Acta, 2012, 748: 67
[40]Joeng C B, Niazi J H, Lee S J, et al. Bioorg Med Chem Lett, 2009, 17(15): 5380
[41]Lauridsen L H, Shamaileh H A, Edwards S L, et al. PLoS One, 2012, 7(7): e41702
[42]Martin J A, Parekh P, Kim Y, et al. PLoS One, 2013, 8(3): e53419
[43]Gong S, Ren H L, Tian R Y, et al. Biosens Bioelectron, 2013, 49: 547
[44]Bawazer L A, Newman A M, Gu Q, et al. Acs Nano, 2014, 8(1): 387
[45]Wu Y, Zhan S, Wang L, et al. Analyst, 2014, 139(6): 1550
[46]Rajendran M, Ellington A D. Anal Bioanal Chem, 2008, 390(4): 1067
[47]Kim M, Um H J, Bang S, et al. Environ Sci Technol, 2009, 43(24): 9335
[48]He J, Liu Y, Fan M, et al. J Agric Food Chem, 2011, 59(5): 1582
[49]Reinemann C, Stoltenburg R, Strehlitz B. Anal Chem, 2009, 81(10): 3973
[50]Xu S, Yuan H, Chen S, et al. Anal Biochem, 2012, 423(2): 195
[51]Elshafey R, Siaj M, Zourob M. Biosens Bioelectron, 2015, 68: 295
[52]Cho Y S, Lee E J, Lee G H, et al. Bioorg Med Chem Lett, 2015, 25(23): 5536
[53]Spiga F M, Maietta P, Guiducci C. Acs Comb Sci, 2015, 17(5): 326
[54]Vanschoenbeek K, Vanbrabant J, Hosseinkhani B, et al. J Steroid Biochem, 2015, 147: 10
[55]Niazi J H, Lee S J, Gu M B. Bioorg Med Chem Lett, 2008, 16(15): 7245
[56]Rouah-Martin E, Mehta J, van Dorst B, et al. Int J Mol Sci, 2012, 13(12): 17138
[57]Ohsawa K, Kasamatsu T, Nagashima J, et al. Anal Sci, 2008, 24(1): 167
[58]Grozio A, Gonzalez V M, Millo E, et al. Nucleic Acid Ther, 2013, 23(5): 322
[59]Jayasena S D. Clin Biochem, 1999, 45(9): 1628
[60]Kusser W. J Biotechnol, 2000, 74(1): 27
[61]Eulberg D, Klussmann S. Chembiochem, 2003, 4(10): 979
[62]Cissé C, Mathieu S V, Abeih M B O, et al. Acs Chem Biol, 2014, 9(12): 2779
[63]Liu M, Kagahara T, Abe H, et al. Nucleic Acids Symp, 2008, 52(1): 513
[64]Liu M, Jinmei H, Abe H, et al. Bioorg Med Chem Lett, 2010, 20(9): 2964
[65]Huizenga D E, Szostak J W. Biochemistry, 1995, 34(2): 656
[66]Sassanfar M, Szostak J W. Nature, 1993, 364(5): 550
[67]Gebhardt K, Shokraei A, Babaie E, et al. Biochemistry, 2000, 39(24): 11
[68]Harada K D, Frankel A. EMBO J, 1995, 14(23): 5798
[69]Burke D H, Hoffman D C. Biochemistry, 1998, 37(13): 4653
[70]Bruno J G, Carrillo M R, Cadieux C L, et al. J Mol Recognit, 2009, 22(3): 197
[71]Ahn J, Cho M, Lee S, et al. Mol Cell Toxicol, 2008, 4(2): 100
[72]Boles T C, Hogan M E. Biochemistry, 1987, 26(2): 367
[73]De Cian A, Mergny J L. Nucleic Acids Res, 2007, 35(8): 2483
[74]Berezovski M, Krylov S N. J Am Chem Soc, 2002, 124(46): 13674
[75]Krylov S N, Berezovski M. Analyst, 2003, 128(6): 571
Research advances of aptamers selection for small molecule targets
WANG Yong1, ZHAO Xinying1,2, SHI Dongdong3, YANG Ge1, QU Feng1*
(1. School of Life Science, Beijing Institute of Technology, Beijing 100081, China;2. Beijing Centre for Physical and Chemical Analysis, Beijing Key Laboratory of Organic Materials Testing Technology & Quality Evaluation, Beijing 100089, China;3. Feed Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081, China)
Abstract:Aptamers are ribonucleic acid (RNA) or single-stranded deoxyribonucleic acid (ssDNA) selected by systematic evolution of ligands by exponential enrichment (SELEX). Aptamers can identify small molecules, proteins, cells, microorganisms and other targets with high affinity and specificity, and have been widely applied in biology, medicine, food and environmental monitoring. However, available aptamers of practical use are limited. The complex and difficult screening of aptamers are the key to restrict its wide application. Differing from biomacromolecules, cells and microorganisms, small molecules have less binding sites and weaker affinity with nucleic acids. And they usually need to be immobilized on substrates. In addition, due to the tiny differences of size, weight and charge of the target-ssDNA/RNA complex and ssDNA/RNA, their separation is difficult. Therefore, the aptamer selection of small molecules is more difficult than biomacromolecules or cells. The selection of methods for immobilizing the targets or library and the optimization of separation process proceed mainly based on the structure characteristics and applications of aptamers. In this paper, the screening methods for molecules with different groups, molecules containing the same group and chiral molecules are introduced. Also, the library design, the methods for separating targets-ssDNA complex and characterizing affinity interaction are discussed. The sequences and dissociation constants (Kd) of about 40 aptamers reported since 2008 are listed.
Key words:aptamer; small molecule targets; screening; review
中图分类号:O658
文献标识码:A
文章编号:1000-8713(2016)04-0361-09
基金项目:国家自然科学基金项目(21175011,21375008);北京市科学技术研究院青年骨干计划项目;“十二五”农村领域国家科技计划课题(2011BAD26B040406).
*收稿日期:2015-05-05
DOI:10.3724/SP.J.1123.2015.05001
多尺度靶标的核酸适配体筛选研究进展专栏
·专论与综述
*通讯联系人.E-mail:qufengqu@bit.edu.cn.
Foundation item: National Natural Science Foundation of China (Nos. 21175011, 21375008); Youth Plan of Beijing Academy of Science and Technology; Twelfth Five-Year-Plan in National Science and Technology for the Rural Development (No. 2011BAD26B040406).
