曲张链菌素产生菌壮观链霉菌NRRL2494菌株遗传操作体系的建立
2016-04-12宋姣姣康前进张连茹吴莹莹白林泉
宋姣姣,康前进,张连茹,吴莹莹,3*,白林泉
( 1.厦门大学生命科学学院,天然产物源靶向药物国家地方联合工程实验室,福建厦门361102; 2.上海交通大学生命科学技术学院,微生物代谢国家重点实验室,上海200240; 3.中国科学院上海生命科学研究院植物生理生态研究所,合成生物学重点实验室,上海200032)
曲张链菌素产生菌壮观链霉菌NRRL2494菌株遗传操作体系的建立
宋姣姣1,康前进2,张连茹1,吴莹莹1,3*,白林泉2
( 1.厦门大学生命科学学院,天然产物源靶向药物国家地方联合工程实验室,福建厦门361102; 2.上海交通大学生命科学技术学院,微生物代谢国家重点实验室,上海200240; 3.中国科学院上海生命科学研究院植物生理生态研究所,合成生物学重点实验室,上海200032)
摘要:曲张链菌素属于安莎类抗生素,具有显著的生物活性.在从壮观链霉菌NRRL2494菌株的发酵产物中分离和鉴定了多组分曲张链菌素的基础上,探索和优化了外源DNA通过接合转移进入壮观链霉菌NRRL2494菌株的操作方法和培养条件.以游离型质粒pJTU1278为载体,在体外构建了一个曲张链菌素酰胺合酶基因svaF敲除的质粒,通过接合转移转入到壮观链霉菌NRRL2494野生型菌株中,所获得的svaF基因缺失突变株失去了产生曲张链菌素的能力.该遗传操作体系的成功建立和优化,使得在体内分析和鉴定曲张链菌素生物合成基因的功能成为可能,同时也为建立其他类似放线菌的遗传操作体系提供了参考.
关键词:壮观链霉菌;遗传操作体系;曲张链菌素;生物合成;酰胺合酶
放线菌所产生的次级代谢产物是天然活性物质的重要来源.曲张链菌素( streptovaricin)属较早发现的安莎类抗生素,其组分A~E由Siminoff等[1]和Ravina[2]于1957年从壮观链霉菌( Streptomyces spectabilis) NRRL2494菌株的发酵产物中首先分离,其中曲张链菌素A和C的基本化学结构在1968年得到纯化和鉴定[3].此后,科学家们还从其他壮观链霉菌株中先后分离和鉴定了组分F,G,K,J,U等数个曲张链菌素的同系物[4-5],发现这些化合物都有共同的芳香核-萘醌环结构(图1粗体部分).对曲张链菌素的生物合成进行初步研究发现,其以3-氨基-5-羟基苯甲酸( 3-amino-5-hydroxybenzoic acid,AHBA)为前体,经Ⅰ型聚酮合酶( polyketide synthaseⅠ,PKSⅠ)催化的10步缩合反应延伸形成聚酮脂肪长链,最终形成大环内酰胺结构并释放[6],这一聚酮链骨架的化学结构和合成过程与抗结核临床药物利福霉素相同.
由于曲张链菌素具有抑制RNA聚合酶和反转录酶( DNA聚合酶)的活性[7-9],目前已发现其对结核分枝杆菌( Mycobacterium tuberculosis)和非结核分枝杆菌如堪萨斯分枝杆菌( M.kansasii)、鸟杆菌( M.avium)等都有显著的抗菌活性[10-11],并有抗病毒活性和优于环孢菌素A的免疫抑制功能[4],但由于毒性较大而较少使用.随着合成生物学的学科发展和技术创新,通过组合生物合成的策略对目标抗生素进行结构优化成为可能[12-14].基于曲张链菌素良好的药物开发前景,我们试图发掘生物活性更好的新结构类似物并增加其产量,从而适应于工业化开发.成功建立抗生素产生菌的遗传操作体系是对抗生素进行生物合成研究和结构改造的前提,但关于壮观链霉菌的遗传操作体系目前还未见报道.
以曲张链菌素产生菌壮观链霉菌NRRL2494菌株为对象,本文在了解其抗生素敏感性和生长速度快等特点的基础上,发现缩短常规接合转移时间和使用半营养培养基等方法对接合转移有促进作用,并利用该遗传操作体系成功实现了对曲张链菌素生物合成基因的敲除突变.

图1曲张链菌素的化学结构式Fig.1 Chemical structures of streptovaricins
1材料与方法
1. 1材料
1. 1. 1菌株和质粒
产生曲张链菌素的壮观链霉菌菌株NRRL2494、用于接合转移的游离型质粒载体pJTU1278[15]、作为阿泊拉霉素抗性基因来源的质粒pJTU472[16],由上海交通大学微生物代谢国家重点实验室提供;构建克隆用载体pBluescriptⅡSK ( + )、宿主大肠杆菌( Escherichia coli)菌株TOP10、接合转移的质粒供体大肠杆菌菌株ET12567/pUZ8002[17],由第一作者单位保存.
1. 1. 2培养基
Luria-Bertani ( LB)培养基( g/L) :胰蛋白胨10,酵母抽提物5,氯化钠10; pH 7.0.
TSBY培养基( g/L) :胰酪胨大豆肉汤培养基( TSB) 30,酵母抽提物10,蔗糖103; pH 7.2.
黄豆饼粉浸汁( SFM)培养基( g/L) :黄豆饼粉20,加入自来水灭菌后用4层纱布过滤,再加入甘露醇20; pH 7.5.
高氏一号培养基( g/L) :可溶性淀粉20,氯化钠0.5,硝酸钾1,硫酸亚铁0.001,磷酸氢二钾0.1,七水硫酸镁0.5; pH 7.2.
Waksman培养基( g/L) :硫酸铵0.2,磷酸氢二钾3.0,七水硫酸镁0.5,七水氯化钙0.126; pH 7.2.
壮观链霉菌发酵( ZM)培养基[4]( g/L) :黄豆饼粉10,葡萄糖25,酵母粉2.5,氯化钾4,磷酸氢二钾0.1,硫酸铵5,牛肉浸膏1,碳酸钙3.
ISP2和ISP3培养基配方见参考文献[18].
以上对应的固体培养基按20 g/L加入琼脂.
1. 1. 3酶和主要试剂
酶和DNA分子质量标准购自TaKaRa公司,DNA回收试剂盒购自Omega公司,质粒回收试剂盒购自生工生物工程股份有限公司,其他常规试剂均为国产分析纯产品.各种抗生素购自国内试剂公司,贮存质量浓度为:氨苄青霉素( ampicillin) 100 mg/mL,氯霉素( chloramphenicol) 25 mg/mL,卡那霉素( kanamycin) 25 mg/mL,硫链丝菌素( thiostrepton) 10 mg/mL,阿泊拉霉素( apramycin) 30 mg/mL,萘啶酮酸( nalidixic acid) 25 mg/mL,链霉素( streptomycin) 25 mg/mL.N-tris-(羟甲基)甲基-2-氨基乙磺酸( TES)缓冲液: TES终浓度0.05 mol/L,加入1 nmol/L氢氧化钠溶液调节pH至8.0.PCR引物由上海英潍捷基贸易有限公司合成,DNA测序由上海立菲公司完成.
1. 2方法
1. 2. 1壮观链霉菌NRRL2494菌株的抗生素敏感性检测
采用SFM培养基,配制添加氨苄青霉素、氯霉素、卡那霉素、硫链丝菌素、阿泊拉霉素、链霉素和萘啶酮酸7种抗生素的系列平板,终质量浓度梯度分别为5,25,50 mg/L.取新鲜的菌丝体划线展开于各种抗生素浓度梯度的固体平板上,置于28℃培养箱中培养,从第3天开始至第7天进行生长情况观察.
1. 2. 2酰胺合酶基因svaF敲除重组质粒pWY5构的建
壮观链霉菌NRRL2494菌株的基因组DNA参考文献[19]的方法进行快速提取.根据所获得酰胺合酶基因svaF的DNA序列,在基因的左右两侧各1 100 bp处分别设计2对引物,即左同源臂引物SvaFLF( 5'-AAAGGTACCGGCTCAGGGACGCCACCA-3',引入BamHⅠ酶切位点)和SvaFLR ( 5'-AAAAAGCTTGTCCGGCGACTGGGCGACA-3',引入HindⅢ酶切位点),右同源臂引物SvaFRF( 5'-AAAAAGCTTGTCCTGCGCCGCGCCGTTGT-3',引入HindⅢ酶切位点)和SvaFRR( 5'-AAAGGATCCGCCAGTCGCAGGGGAGCGG-3',引入KpnⅠ酶切位点).以曲张链菌素产生菌NRRL2494野生型菌株的基因组DNA为模板,分别用上述2对引物进行PCR,扩增出两侧同源交换臂片段;再根据两端引入的限制性内切酶位点分别进行双酶切,将这2个片段与经同样酶切处理后的pBluescriptⅡSK( +)载体连接,构建重组质粒pWY1.测序验证后以KpnⅠ和BamHⅠ双酶切pWY1,回收2.2 kb包含双臂序列的片段,与经过相同酶处理的载体pJTU1278连接,构建重组质粒pWY4.用HindⅢ酶切质粒pJTU472,回收1.4 kb左右含有转移原点oriT和阿泊拉霉素抗性基因aac( 3)Ⅳ的HindⅢ片段,连接至同样经HindⅢ处理过的pWY4上,构建最后用于基因置换的质粒pWY5.将pWY5转化到大肠杆菌ET12567/pUZ8002中,获得大肠杆菌ET12567/pUZ8002/pWY5,作为接合转移的供体菌株.
1. 2. 3大肠杆菌供体菌株和壮观链霉菌NRRL2494菌株的接合转移实验
E.coli ET12567/pUZ8002/pWY5在6 mL含有相应终质量浓度的卡那霉素、氯霉素、阿泊拉霉素和氨苄青霉素的LB液体培养基中于37℃生长至600 nm下吸光度( OD600)为0.4~0.6时,离心收集菌体( 3 220 g,10 min),用LB洗涤菌体2~3次后重悬于1 mL LB培养基中,作为接合转移的供体菌株.壮观链霉菌NRRL2494菌株于SFM固体培养基上生长120 h,将孢子收集到TES缓冲液中,充分震荡并过滤后以3 220 g离心5 min去除上清,将孢子重悬于5 mL TES缓冲液中,50℃热激10 min,冷却后加入等体积的预萌发液,37℃培养萌发2.5 h,3 220 g离心10 min收集菌体,悬浮于10 mL LB液体培养基中,作为接合转移的受体菌株.将上述供体菌株和受体菌株分别进行显微计数,按大肠杆菌细胞与壮观链霉菌孢子数量相当的比例各取相应体积的悬液混合均匀,涂布于不含任何抗生素的半营养SFM固体培养基上,吹干,置于37℃培养8 h,用1 mL含有30 μg/mL阿泊拉霉素和25 μg/mL萘啶酮酸的无菌水均匀覆盖整个平板,吹干表面水分后置于28℃培养,3~5 d后可观察到接合子.
1. 2. 4壮观链霉菌NRRL2494双交换突变株的获得
接合转移平板上长出的接合子分别影印到含有硫链丝菌素和阿泊拉霉素的SFM固体培养基上,28℃培养3~5 d后,选取对硫链丝菌素敏感而对阿泊拉霉素不敏感的菌株,提取基因组DNA,以阿泊拉霉素的抗性基因引物Apr-F( 5'-GCTCATCGGTCAGCTTCTCA-3')和Apr-R( 5'-TCGCATTCTTCGCATCCC-3')进行PCR扩增.为进一步验证该突变株,在所敲除基因序列的上下游设计检测引物MTF( 5'-ACCACCTCATGGCGATCCCGTACAAC-3')和MTR( 5'-GCTGTGCGGTGAGTTCGTGCGGTT-3'),通过PCR结果判断接合子的基因型(单交换或双交换突变株).
1. 2. 5壮观链霉菌NRRL2494野生型菌株及其突变株的发酵和产物分析
NRRL2494野生型菌株或其突变株接种于SFM固体培养基上,28℃培养6 d后,刮取孢子接种于ZM液体培养基中,28℃、220 r/min摇床培养68~72 h.按照培养物∶培养基比例为1∶50的接种量转接于新鲜的ZM液体培养基中,28℃、220 r/min继续培养5 d,得到菌株的液体发酵物.发酵液经离心,调节上清pH至7.0,加等体积的乙酸乙酯提取2次,菌丝体经丙酮浸泡24 h,过滤、减压浓缩至无丙酮水溶液,乙酸乙酯萃取2次.合并乙酸乙酯萃取液,用旋转蒸发仪得到浓缩物样品,溶于500 μL甲醇中,进行高效液相色谱( HPLC)检测.检测条件如下:流动相A为纯水,流动相B为甲醇,流速为0.6 mL/min,检测波长为254 nm.梯度洗脱程序: 0~6 min,20%~75%(体积分数,下同) B相; 7~10 min,75% B相; 11~15 min,75%~95% B 相; 16~20 min,95% B相; 21~22 min,95%~20% B相,23~37 min,20% B相.
2结果和分析
2. 1壮观链霉菌NRRL2494菌株的抗生素敏感性实验
为了确定壮观链霉菌遗传操作系统所需的遗传筛选标记,我们检测了NRRL2494菌株对多种抗生素的敏感性.结果表明,NRRL2494菌株对卡那霉素、阿泊拉霉素和硫链丝菌素较敏感,而对氨苄青霉素、氯霉素、链霉素和萘啶酮酸都表现出不同程度的抗性(表1).据此,我们以阿泊拉霉素作为NRRL2494突变株的筛选标记,并将萘啶酮酸作为接合转移实验中大肠杆菌的抑制剂.
2. 2壮观链霉菌NRRL2494菌株与大肠杆菌接合转移实验方法和条件的摸索
大肠杆菌与链霉菌的接合转移方法由于条件温和,操作方便和菌株稳定,是目前遗传操作中最常用的方法[20].鉴于壮观链霉菌NRRL2494菌株产生的孢子比较丰富,我们选择利用该菌株的孢子,通过接合转移的方法来探索其遗传操作体系的建立.为收集大量孢子以进行遗传操作,采用SFM、ISP2、ISP3、ISP4 和Waksman 5种固体培养基对NRRL2494菌株进行培养,发现其在SFM平板上的产孢情况最佳,故选用SFM培养基作为NRRL2494菌株的产孢培养基.由于链霉菌孢子的质量和数量是影响接合转移成功与否的关键因素,我们采用插片法观察NRRL2494菌株在SFM培养基上生长的菌丝和孢子的形态,并使用血球计数板对孢子进行计数,结果显示:培养24 h后NRRL2494菌株开始产生少量孢子,48 h后开始产生色素,而96~120 h孢子形态和数量达到最佳状态,168 h后孢子开始呈现老化状态,故选取120 h作为孢子收集的时间.

表1壮观链霉菌NRRL2494菌株对不同抗生素的敏感性Tab.1 The sensitivities of S.spectabilis NRRL2494 to various antibiotics
鉴于NRRL2494菌株较其他种属链霉菌生长速度更快,我们在接合转移实验中设置了一系列供体菌与受体菌的接合时间梯度,由常规链霉菌使用的12~20 h缩短到6~14 h,发现结合时间在8 h左右效果最好.也正是基于该菌株生长速度快的特点,我们分别采用SFM完全培养基和半营养培养基进行接合转移实验,比较两者差异发现,在抗生素覆盖接合转移平板后,完全培养基表面在48 h内就生长出菌苔,而半营养培养基上则在3~5 d后才出现分散的菌落.这一结果说明:完全培养基上链霉菌的生长速度过快,抑制了大肠杆菌的生长,导致接合转移过程受阻;而半营养培养基限制了链霉菌的生长,保证了大肠杆菌的生长速率,使得大肠杆菌中的外源重组质粒能够进入链霉菌,提高了接合效率.因此,我们选择SFM半营养培养基作为NRRL2494菌株与大肠杆菌接合转移的筛选培养基.

图2 svaF基因失活突变株的构建及pWY5质粒验证Fig.2 Construction of svaF inactive mutant and confirmation of plasmid pWY5
2. 3 svaF基因失活突变株的筛选
根据1.2.2小节中的方法,我们构建了用于NRRL2494菌株中酰胺合酶基因svaF敲除的重组质粒pWY5(图2( a) ).为验证其正确性,将pWY5以KpnⅠ和BamHⅠ双酶切,得到9.2和3.6 kb的两个片段,用HindⅢ单酶切,得到12.8 kb的单个片段,结果均符合预期(图2( b) ).
以大肠杆菌ET12567/pUZ8002/pWY5作为供体菌,壮观链霉菌NRRL2494菌株的孢子作为受体进行接合转移,培养3~5 d后,在每个9 cm平板上都有20个以上的接合转移子出现.挑取单菌落分别影印至含有硫链丝菌素和阿泊拉霉素的平板上培养,并对可能的单交换接合子在无抗性的固体培养基上进行2轮松弛,筛选出4个突变株,分别为SW1-2、SW1-3、SW1-10和SW1-12.以这4个突变株的基因组DNA为模板,用阿泊拉霉素抗性基因引物( Apr-F 和Apr-R)及检测引物( MTF和MTR)分别进行PCR扩增.如果是双交换菌株,可得到0.73 kb的阿泊拉霉素抗性基因片段和1.7 kb的检测引物扩增产物;野生型菌株则只能得到1.0 kb的检测引物扩增产物,而不含阿泊拉霉素抗性基因片段.PCR结果表明所得到的4个突变株均为双交换菌株(图3),对检测引物扩增产物的测序结果进一步验证了突变株的正确性.
2. 4 svaF基因失活突变株的发酵产物分析
将所获得的双交换突变株进行发酵培养,同时以NRRL2494野生型菌株作为对照,用HPLC-质谱( MS)联用的方法检测发酵产物.结果显示,野生型菌株的发酵产物(图4( a) )在保留时间为31.29和37.60 min时,分别检测到曲张链菌素组分C([M+H]+( m/z: 769.8),[M+Na]+( m/z: 791.8) ;图4 ( c) )和组分G([M+H]+( m/z: 786.4),[M+Na]+( m/z: 808.3) ;图4 ( d) ),而突变株发酵产物(图4( b) )未检出相应的组分,即突变株不再产生野生型菌株中主要的曲张链菌素组分C和G,说明位于重组质粒pWY5上的外源DNA通过接合转移从大肠杆菌中导入到了壮观链霉菌NRRL2494菌株中,并发生了预期的同源重组,成功地获得了酰胺合酶基因svaF缺失的双交换突变株.酰胺合酶属于NAT ( arylamine N-acetyltransferase)家族,具有保守的Cys-His-Asp三联体活性位点,与聚酮链的释放环化有关[21].由于这些突变株不再产生曲张链菌素的组分,表明酰胺合酶与曲张链菌素的生物合成相关.
3讨论
链霉菌属于放线菌中的最大类群,是一类具有重要经济价值的革兰氏阳性菌,能够产生多种抗生素和生物活性物质.为实现对其生物合成途径及代谢调控的人工控制,需要稳定的遗传操作系统.通过接合转移的方式,链霉菌遗传操作体系的发展相对成熟,但对壮观链霉菌的遗传操作系统仍未见报道.壮观链霉菌能够产生大观霉素、曲张链菌素、硝本吡喃酮、巴弗洛霉素等多种抗生素[22],其中曲张链菌素是一种极具前景的候选药物.因此,建立壮观链霉菌的遗传操作体系,利用组合生物合成的方法对其改造,有望获得具有良好活性的新型化合物.

图3 svaF基因失活突变株PCR产物的凝胶电泳分析Fig.3 Gel electrophoresis analyses of PCR products from svaF inactive mutants
在常规的放线菌基因敲除实验中,接合转移过程中供体菌和受体菌的接合时间一般都在12 h以上,鲜见低于12 h的报道.我们按照常规链霉菌使用的接合时间12~20 h,多次尝试壮观链霉菌NRRL2494菌株的接合转移都没能成功获得接合子.插片法观察的结果表明NRRL2494菌株的生长速度较普通链霉菌快,因此我们尝试缩短接合时间至8 h,同时选择半营养培养基用于接合转移,最终成功建立了外源DNA通过接合转移进入壮观链霉菌NRRL2494菌株的遗传操作体系,并实现对其所产生曲张链菌素的生物合成基因进行体内敲除的遗传操作,获得了曲张链菌素酰胺合酶基因svaF失活的双交换突变株,使该突变株不再产生曲张链菌素.此外,生物信息学分析表明,该基因与利福霉素生物合成基因簇中的rifF同源性较高;而RifF是利福霉素生物合成中的关键酶,负责安莎聚酮链的释放[23].我们的实验结果暗示曲张链菌素中的酰胺合酶SvaF在体内可能具有与RifF相似的功能.
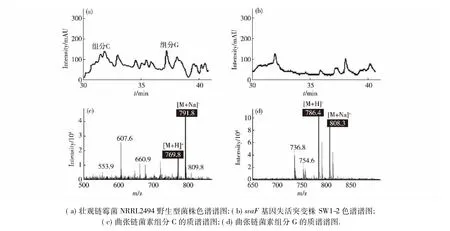
图4 svaF基因失活突变株发酵产物的高效液相色谱-质谱分析Fig.4 Analyses of fermentation products from svaF inactive mutants by HPLC-MS
综上所述,本研究成功建立了壮观链霉菌NRRL2494菌株的遗传操作体系,为进一步研究曲张链菌素的生物合成和获取曲张链菌素的新结构类似物奠定了基础,同时也为其他生长速度较快的链霉菌属菌种的相关研究提供了参考.
参考文献:
[1]SIMINOFF P,SMITH R M,SOKOLSKI W T,et al.Streptovaricin.Ⅰ.Discovery and biologic activity[J].Am Rev Tuberc,1957,75( 4) : 576-583.
[2]RAVINA A.A new anti-tuberculosis drug: streptovaricin[J].Presse Med,1957,65( 35) : 834.
[3]YAMAZAKI H.Studies on antimicrobial substance B 44 P ( streptovaricin) produced by a strain of actinomycetes.Ⅰ.Production,extraction and characteristics of substance B 44 P and the identity of the substance with streptovarcin [J].J Antibiot,1968,1( 3) : 204-208.
[4]胡海峰,朱宝泉,张琴,等.微生物来源的免疫抑制活性化合物SIPI-18-1和SIPI-18-2的研究[J].中国天然药物,2003,1( 2) : 116-119.
[5]KNOLL W M,RINEHART K L JR,WILEY P F,et al.Streptovaricin U,an acyclic ansamycin[J].J Antibiot,1980,33 ( 2) : 249-51.
[6]STALEY A L,RINEHART K L.Biosynthesis of the streptovaricins: 3-amino-5-hydroxybenzoic acid as a precursor to the meta-C7N unit[J].J Antibiot,1991,44( 2) : 218-224.
[7]SANCHEZ-HIDALGO M,NUNEZ L E,MENDEZ C,et al.Involvement of the beta subunit of RNA polymerase in resistance to streptolydigin and streptovaricin in the producer organisms Streptomyces lydicus and Streptomyces spectabilis[J].Antimicrob Agents Chemother,2010,54( 5) : 1684-1692.
[8]HOROSZEWICZ J S,LEONG S S,CARTER W A.Differential susceptibility of spleen focus-forming virus and murine leukemia viruses to ansamycin antibiotics[J].Antimicrob Agents Chemother,1977,12( 1) : 4-10.
[9]HOROSZEWICZ J S,RINEHART K L,LEONG S S,et al.Activity of pure streptovaricins and fractionated streptovaricin complex against Friend virus[J].Antimicrob Agents Chemother,1975,7( 3) : 281-284.
[10]DESHMUKH P V,KAKINUMA K,AMEEL J J,et al.Letter: protostreptovaricinsⅠ-Ⅴ[J].J Am Chem.Soc,1976,98( 3) : 870-872.
[11]RINEHART K L,ANTOSZ F J,DESHMUKH P V,et al.I-dentification and preparation of damavaricins,biologically active precursors of streptovaricins[J].J Antibiot,1976,29 ( 2) : 201-203.
[12]ZHOU H,XIE X,TANG Y.Engineering natural products using combinatorial biosynthesis and biocatalysis[J].Curr Opin Biotechnol,2008,19( 6) : 590-596.
[13]HOPWOOD D A.Genetic contributions to understanding polyketide synthases[J].Chem Rev,1997,97 ( 7 ) : 2465-2498.
[14]白林泉,邓子新.微生物次级代谢产物生物合成基因簇与药物创新[J].中国抗生素杂志,2006,31( 2) : 80-86.
[15]HE Y,WANG Z,BAI L,et al.Two pHz1358-derivative vectors for efficient gene knockout in streptomyces[J].J Microbiol Biotechnol,2010,20( 4) : 678-682.
[16]JANSSEN G R,BIBB M J.Derivatives of pUC18 that have BglⅡsites flanking a modified multiple cloning site and that retain the ability to identify recombinant clones by visual screening of Escherichia coli colonies[J].Gene,1993,124( 1) : 133-134.
[17]KIESER H M,KIESER T,HOPWOOD D A.A combined genetic and physical map of the Streptomyces coelicolor A3 ( 2) chromosome[J].J Bacteriol,1992,174 ( 17) : 5496-5507.
[18]SHIRLING E B,GOTTLIEB D.Methods for characterization of Streptomyces species[J].Int J Syst Bacteriol,1966,16: 313-340.
[19]WU Y,LU C,QIAN X,et al.Diversities within genotypes,bioactivity and biosynthetic genes of endophytic actinomycetes isolated from three pharmaceutical plants[J].Curr Microbiol,2009,59( 4) : 475-482.
[20]BIERMAN M,LOGAN R,O' BRIEN K,et al.Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp[J].Gene,1992,116( 1) : 43-49.
[21]KUBIAK X,DAIROU J,DUPRET J M,et al.Crystal structure of arylamine N-acetyltransferases: insights into the mechanisms of action and substrate selectivity[J].Expert Opin Drug Metab Toxicol,2013,9( 3) : 349-362.
[22]金燕华,裘娟萍,何景昌.壮观链霉菌产生抗生素的多样性[J].中国医药工业杂志,2006,37( 12) : 849-855.
[23]FLOSS H G,YU T W.Rifamycin-mode of action,resistance,and biosynthesis[J].Chem Rev,2005,105( 2) : 621-632.
Gene Manipulation System for Streptovaricin Producer Streptomyces spectabilis NRRL2494
SONG Jiaojiao1,KANG Qianjin2,ZHANG Lianru1,WU Yingying1,3*,BAI Linquan2
( 1.State-Province Joint Engineering Laboratory of Targeted Drugs from Natural Products,School of Life Sciences,Xiamen University,Xiamen 361102,China; 2.State Key Laboratory of Microbial Metabolism,School of Life Sciences and Biotechnology,Shanghai Jiaotong University,Shanghai 200240,China; 3.Key Laboratory of Synthetic Biology,Institute of Plant Physiology and Ecology,Shanghai Institutes for Biological Sciences,Chinese Academy of Sciences,Shanghai 200032,China)
Abstract:In order to enable the streptovaricin biosynthetic study by in vivo gene disruption,it is crucial to develop a genetic modification system for the streptovaricin producer Streptomyces spectabilis NRRL2494.In this study,we aimed to establish a method of conjugation to transfer exogenous DNA into Streptomyces spectabilis NRRL2494.A putative streptovaricin amide synthase gene svaF was disrupted in vitro,and the constructed plasmid was transferred into Streptomyces spectabilis NRRL2494 by conjugation through a double-crossover recombination event.The resulting svaF mutant lost the ability to produce streptovaricin.Based on the above work,we developed a genetic manipulation system for Streptomyces spectabilis NRRL2494,enabling the functional characterization of streptovaricin biosynthetic genes in vivo,and offering a positive example for other fast-growing Actinobacteria which lack an appropriate genetic manipulation system.
Key words:Streptomyces spectabilis; genetic manipulation system; streptovaricin; biosynthesis; amide synthase
*通信作者:wuyingying@ xmu.edu.cn
基金项目:国家自然科学基金( 31100027) ;高等学校博士学科点专项科研基金( 20110121120013)
收稿日期:2015-02-06录用日期: 2015-04-20
doi:10.6043/j.issn.0438-0479.2016.01.011
中图分类号:Q 933
文献标志码:A
文章编号:0438-0479( 2016) 01-0060-07
引文格式:宋姣姣,康前进,张连茹,等.曲张链菌素产生菌壮观链霉菌NRRL2494菌株遗传操作体系的建立[J].厦门大学学报(自然科学版),2016,55( 1) : 60-66.
Citation: SONG J J,KANG Q J,ZHANG L R,et al.Gene manipulation system for streptovaricin producer Streptomyces spectabilis NRRL2494[J].Journal of Xiamen University( Natural Science),2016,55( 1) : 60-66.( in Chinese)
