青稞遗传多样性及其农艺性状与SSR标记的关联分析
2016-03-03孟亚雄孟祎林汪军成司二静张海娟任盼荣马小乐李葆春王化俊
孟亚雄 孟祎林 汪军成 司二静 张海娟 任盼荣马小乐 李葆春 杨 轲 王化俊*
1甘肃省干旱生境作物学重点实验室 / 甘肃省作物遗传改良与种质创新重点实验室, 甘肃兰州 730070;2甘肃农业大学农学院, 甘肃兰州 730070;3甘肃农业大学生命科学技术院, 甘肃兰州 730070
青稞遗传多样性及其农艺性状与SSR标记的关联分析
孟亚雄1,2孟祎林1,2汪军成1,2司二静1,2张海娟1,2任盼荣1,2马小乐1,2李葆春1,3杨轲1,2王化俊1,2*
1甘肃省干旱生境作物学重点实验室 / 甘肃省作物遗传改良与种质创新重点实验室, 甘肃兰州 730070;2甘肃农业大学农学院, 甘肃兰州 730070;3甘肃农业大学生命科学技术院, 甘肃兰州 730070
摘要:利用92个SSR标记对108份青稞亲本材料进行多态性扫描, 分析其遗传多样性, 旨在寻找与农艺性状相关联的分子标记, 为青稞杂交组合的配制及分子标记辅助育种提供依据。挑选48个多态性标记进行群体遗传结构分析,在此基础上采用Tassel 2.1 GLM (general linear model)和MLM (mixed linear model)方法进行标记与农艺性状的关联分析。共检测出156个等位变异, 每个位点2~6个等位变异。供试群体的Shannon指数为0.6727~1.1368, 材料间遗传相似系数为0.2250~1.0000, 平均0.7585。通过群体遗传结构分析将供试材料划分成4个亚群。以GLM分析, 发现12个与株高、穗长、穗粒数和分蘖数相关联的标记, 对表型变异的解释率分别为11.5%~17.6%、19.4%~45.4%、15.4%~22.1%和29.2%; 以MLM分析, 发现8个与株高、分蘖数和小穗数相关的标记, 各标记对表型变异的解释率分别为31.7%~49.8%、28.1%~37.2%、22.7%~32.7%。关联标记分布在基因组全部6个连锁群上。
关键词:青稞; SSR; 遗传多样性; 群体遗传结构; 关联分析
本研究由国家自然科学基金项目(31460347), 甘肃省财政厅科研业务费(035-041047)和国家现代农业产业技术体系建设专项(CARS-05)资助。
This study was supported by the National Natural Science Foundation of China (31460347), Gansu Provincial Department of Finance Research Operating Expenses (035-041047), and the China Agriculture Research System (CARS-05).
第一作者联系方式: E-mail: yxmeng1@163.com; Tel: 13919964113
青稞(Hordeum vulgare L. var. nudum HK. f.)是一种古老栽培作物, 主要分布在中国青藏高原、南美安地斯高原、北非高原及俄罗斯的考克萨斯等山区[1], 具有较高的营养价值, 是酿造工业的重要原料和发展养殖业的优良饲料。在我国青藏高原地区,总人口50%以上的藏族居民都以青稞为主食, 因此青稞育种的突破性进展对藏族地区经济发展和社会稳定具有十分重要意义。和其他作物一样, 青稞也面临着亲本遗传基础狭窄的育种困境[2], 亟需加强遗传资源的引进、创制及其农艺性状特征和遗传基础的了解与掌握。对现有亲本材料进行遗传多样性研究, 可有效减少相似遗传背景的组合, 减轻育种的工作量, 对于亲本的杂交组合配置具有重要的指导意义。同时, 寻找与目标性状相关联的分子标记,可以为复杂数量性状的遗传学研究和分子标记辅助育种奠定重要基础。
分子标记技术的快速发展为作物种质资源的遗传多样性研究提供了便利。其中SSR (simple sequence repeat, SSR)标记具有重复性好、多态性高、数量丰富、呈共显性且广泛分布于基因组等优点, 已被广泛应用于作物遗传多样性和基因发掘等研究[2-5]。通过关联分析对供试群体候选基因进行检测或分子标记扫描, 可以获得丰富的基因位点及其等位基因信息, 在大麦[6]、小麦[7-8]、玉米[9-11]、水稻[12-13]、马铃薯[14]等作物中均有相关报道。Wu等[15]通过对188份西藏野生大麦材料的耐盐性关联分析, 找到2个与耐盐基因HvCBF4相关联的标记。Brantestam等[16]利用22个SSR标记对北欧和波罗的海区域197份春大麦的遗传多样性研究, 共检测出191个等位变异,平均每个标记检测2~23个等位变异, 并且发现, 与早期品种相比, 当前栽培种的遗传多样性发生了明显变化, 一些起源地材料的遗传多样性也发生了少量变异。Sun等[17]利用42个SSR标记对中国西藏野生大麦及世界各起源地的部分大麦材料开展了遗传多样性研究, 同时进行了14个数量性状的关联分析。赖勇等[6]用57个SSR标记对113份大麦材料进行关联分析, 以GLM和MLM两种关联分析模型, 分别获得9个与株高、穗长、芒长、穗粒数和小穗着生密度及6个与株高、芒长和小穗着生密度相关联的标记。Kraakman等[18]将AFLP和SSR标记同148份春大麦栽培种的抽穗期、株高、叶锈病抗性、大麦黄矮病抗性进行连锁不平衡(linkage disequilibrium, LD)作图,找到其相关标记, 证明LD作图是寻找与主效基因或QTL相关联的合适标记的另一种重要途径。Ivandic 等[19-20]利用33个SSR标记对大麦作关联分析, 检测出与开花时间、抗白粉病和耐水分胁迫能力等显著相关的标记。
关联分析利用自然变异, 不需要花费过多的时间和精力去构建作图群体, 可以广泛地检测遗传变异。株高、穗粒数、穗长、芒长和小穗着生密度等农艺性状是传统育种的主要选择标准。利用关联分析寻找与这些性状显著相关的标记, 可以减少专门构建群体进行QTL定位的工作量。而当前SSR标记应用于青稞农艺性状关联分析研究的报道较少。本研究对108份青稞亲本材料进行遗传多样性分析, 确定大麦亲本材料间的遗传背景差异, 同时寻找与株高、穗长、穗粒数和分蘖数显著相关联的分子标记,为后期青稞育种工作中的亲本组配、等位基因发掘和标记辅助选择提供参考。
1 材料与方法
1.1试验材料及其DNA提取
以108份青稞农家品种、野生种和栽培品种(系)构成自然群体(表1), 由中国农业科学院作物科学研究所、甘肃省作物遗传改良与种质创新重点实验室麦类种质创新课题组提供。从每份材料取10粒饱满种子置培养皿中, 在室内避光培养。10 d后采黄化苗, 液氮–80℃速冻。采用CTAB法[21-22]从黄化苗嫩叶中提取各青稞材料基因组DNA, 并用紫外分光光度计法测其质量和浓度, 然后置于–20℃冰箱保存。
1.2农艺性状测定方法
于2013年3月底和2014年3月底, 将所有材料种植于甘肃农业大学黄羊镇育种试验站亲本圃,每份材料种5行, 行长为1.2 m。在成熟收获时随机抽样, 每份材料取10株, 分别测定其株高、穗长、分蘖数、穗粒数、千粒重, 取平均值。
1.3SSR标记检测方法及遗传多样性分析

表1 供试大麦亲本材料Table 1 Hulless barley materials used in this study
(续表1)
选取分布于大麦1H~7H染色体的92个SSR标记进行PCR扩增, 扩增程序为: 95℃ 5 min; 94℃50 s, 64~55℃ (touch-down PCR) 50 s, 72℃ 50 s, 10个循环, 每个循环退火温度降低1℃; 94℃ 50 s, 55 ℃ 50 s, 72℃ 50 s, 30个循环; 72℃ 10 min, 4℃ 保存。扩增产物经8%聚丙烯酰胺凝胶电泳, 银染显色后拍照。扩增结果按相同迁移率下有带记为1, 无带记为0; 同时记录基因型, 以大写字母A、B、C等表示。
应用NTSYS-pc计算遗传相似系数(genetic similarity, GS)并按非加权配对法(UPGMA)和SHAN程序聚类分析。用Popgene32统计基因频率和Shannon指数。
1.4关联分析
将多态性引物检测结果以Structure 2.3.1软件进行群体遗传结构分析, 估计最佳群体组群数K, 其取值范围为2~10, 将参数iterations设为10 000, burnin period设为100 000, 每个K值重复运行6次, 依据似然值最大原则选取合适的K值为群体数目, 计算Q参数, 将其作为协变量, 以SPAGeDi-1.3d处理基因型数据获得个体间亲缘关系Kinship矩阵[23]。采用Tassel 2.1软件一般线性模型(general linear model, GLM)和混合线性模型(mixed linear model, MLM)两种程序进行关联分析。GLM分析中, 以各亲本材料的对应Q值作为协变量, 将SSR标记与株高、穗长、芒长、分蘖数和穗粒数等表型变异进行回归分析, 寻找与之相关联的标记, 并确定其解释率; MLM分析中, 采用Q+K方法分析, 选择计算每个标记的遗传力(calculate heritability for each marker)的方式, 分析方法选择EM。分别运用两年试验表型数据结合分子标记数据和群体结构进行标记-性状关联分析, 确定关联位点(P<0.01), 并计算标记对表型变异的解释率。
2 结果与分析
2.1SSR标记分析
用92对SSR引物对108份青稞材料进行扩增, 其中有48对能够得到清晰、重复性好的条带, 并且扩增结果表现多态性, 共检测出156个等位变异, 变幅为2~6个, 平均每个标记3.2个; 3H染色体上的8对引物检测到的等位变异最多, 共28个; 而1H上的5对引物检测出等位变异数最少, 共15个。SSR引物的Shannon指数变幅为0.6727~1.1368, 平均1.0507 (表2)。可见, 这48对SSR标记的检测效率较高, 参试青稞种质材料群体间的遗传变异较大, 具有较为丰富的遗传多样性。
2.2遗传相似系数及聚类分析
根据48个多态SSR标记的检测结果, 108份青稞亲本材料的GS变异范围为0.2250~1.0000。其中ZDM5163和ZDM5737以及ZDM5199和ZDM5169 的GS最大, 为1.0000, 其次是ZDM6552和ZDM5702, GS值为0.9938, GS最小的是川83-5319与甘青1号, 为0.2250。按UPGMA法进行聚类分析(图1)表明, 在GS值为0.5106水平上聚为3个大类, 分别包含91份、13份和4份材料, 其中由肚里黄、ZDM5740、ZDM5376和ZDM6468四份材料构成第III大类; 第II大类分为两个亚类, 第1亚类由白青稞等9个品种构成, 第2亚类由ZYM2263、ZDM6553、甘青1号和ZDM6171构成。
2.3群体遗传结构分析
利用Structure 2.3.1软件, 基于数学模型分析由108份材料构成的自然群体的遗传结构, 发现样本的等位变异频率特征类型数K呈持续增大趋势, 当K=4时出现峰值, 并呈现明显的拐点(图2-A)。由此将108份青稞亲本分为4个类群(图2-B), 分别包含21、41、3和43份。该自然群体结构比较简单, 能够有效地降低群体结构对关联分析产生的影响。
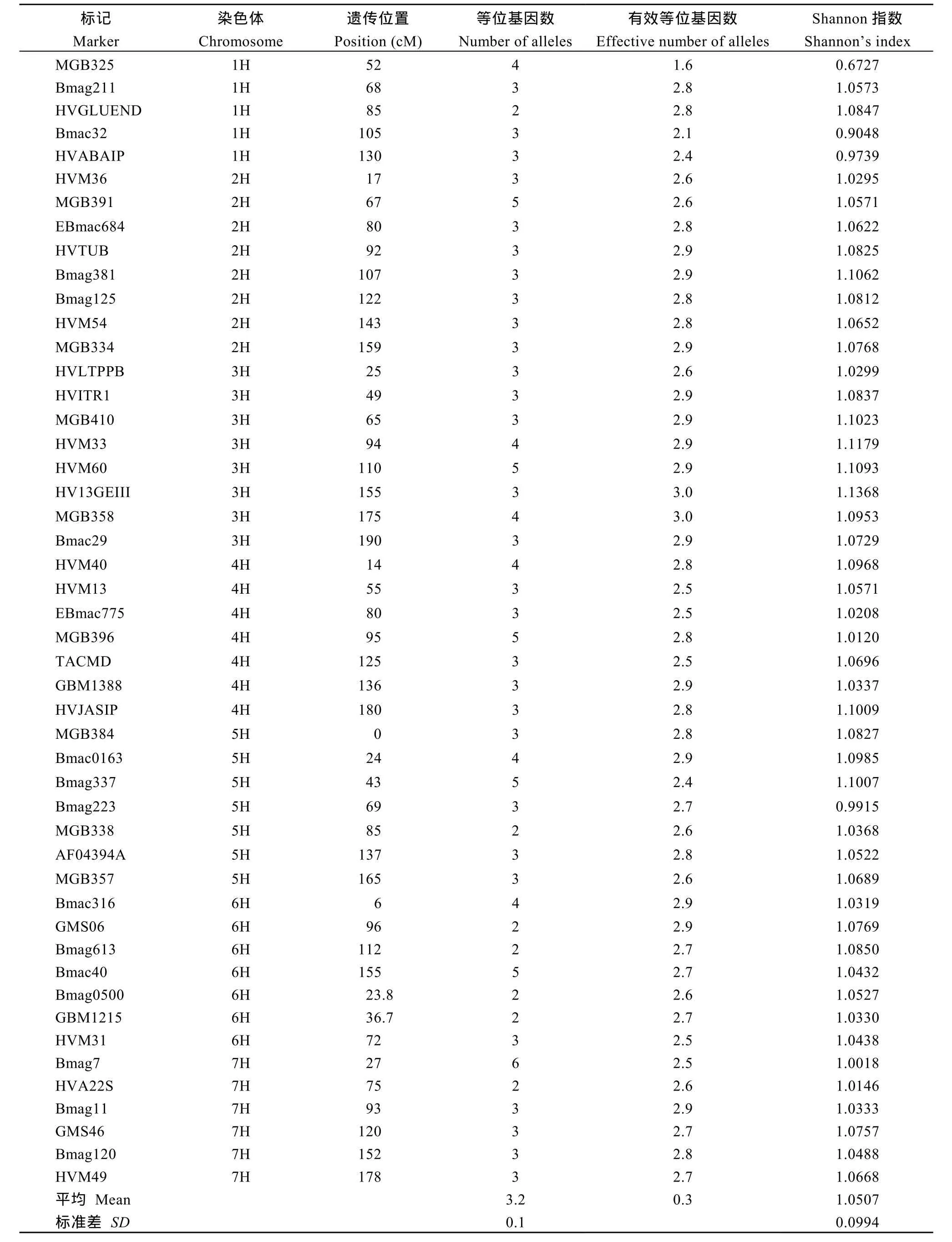
表2 48对SSR引物在108份青稞种质材料中的多态性Table 2 Fourty-eight polymorphics SSRs among 108 accessions of hulless barley

图1 108份青稞亲本材料的聚类图Fig. 1 Dendrogram of 108 accessions of hulless barley based on SSR markers品种编号与表1一致。Codes of varieties consistent with those given in Table 1.
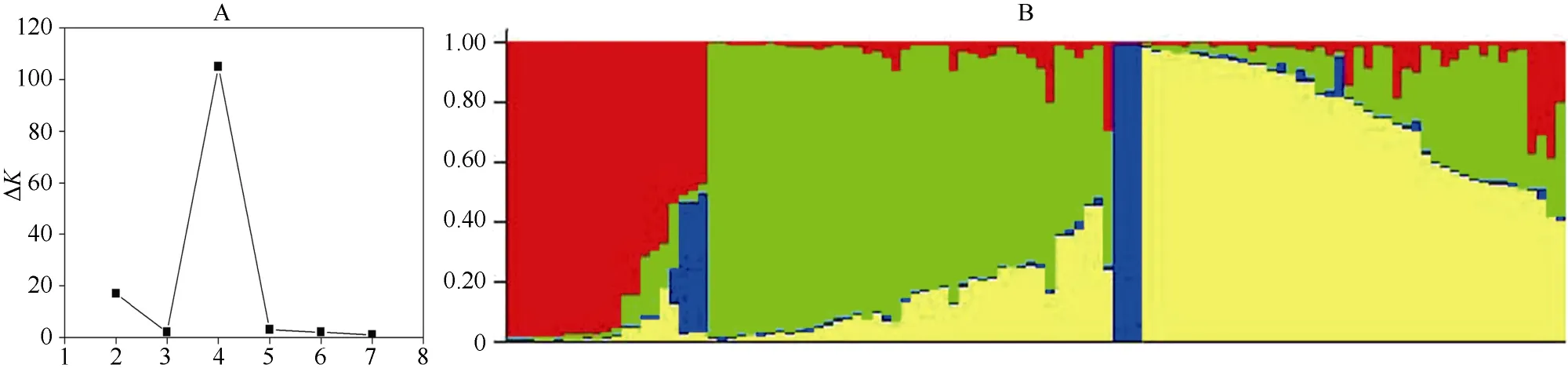
图2 青稞亲本材料的群体遗传分析图Fig. 2 Population genetic analysis of hulless barley accessions
2.4SSR标记与部分农艺性状的关联分析
GLM分析结果显示, 在所检测的48个标记中有12个与穗长、分蘖数、穗粒数显著相关(P<0.01)。其中6个与穗长相关, 1个(EBmac775)与穗长和分蘖数都相关, 3个和穗粒数相关, 2个与株高相关。各标记对表型变异的解释率为11.5%~45.4%, 解释率最大的标记是HVA22S, 为45.4%, 与穗长相关, 解释率最小的标记是Bmag381, 为11.5%, 也与穗长相关。MLM分析结果显示, 在所检测的48个标记中有8个与株高、分蘖数、穗粒数显著相关(P<0.01), 其中2个与株高相关, 3个与分蘖数相关, 3个与穗粒数相关,各标记对表型变异的解释率为22.7%~49.8%, 标记Bmac0163对株高的解释率最大, 为49.8%, 标记Bmac32对穗粒数的解释率最小, 为22.7%。MLM分析检测到与农艺性状相关的SSR比GLM分析结果少4个, 其中有5个标记与GLM 分析得到的相同, 但解释率均有所下降, MLM分析未检测到与穗长相关的标记(表3)。

表3 与农艺性状相关的SSR标记及其表型解释率Table 3 SSR markers associated with agronomic traits and their explained phenotypic variations (%)
3 讨论
3.1青稞亲本材料的性状及遗传多样性分析
种质材料是进行作物育种的基础, 遗传基础狭窄是制约培育出优良品种的主要因素之一。因此分析亲本材料的遗传多样性, 比较材料相互间亲缘关系的远近, 对于开展作物育种工作具有重要的指导意义。本研究选用48对分布于大麦1H~7H染色体的
SSR标记, 基本覆盖全基因组, 能够较全面地分析108份材料的遗传多样性, 比较它们遗传背景的差异, 为亲本的杂交组合合理配置在分子水平上提供依据。国内已开展了SSR标记检测青稞的遗传多样性的研究, 如孟凡磊等[1]结合青稞育种和生产利用的实际, 对我国西藏主要农区青稞品种的遗传多样性进行了研究, 发现供试青稞材料间存在一定的遗传差异, 但品种间的遗传距离较近, 西藏青稞育成品种的基因来源相对较窄。潘志芬等[24]利用SSR标记分析了64份青藏高原栽培青稞的遗传多样性, 认为青藏高原栽培青稞具有丰富的遗传多样性。杨平等[25]利用SRAP分子标记技术, 对25份来自四川高原的青稞育成品种进行了遗传多样性研究, 发现聚类特征与材料来源地有明显相关, 25份材料间的平均遗传距离较小(0.3240), 平均遗传多样性较低(0.5126), 遗传基础较为狭窄。本研究所用的108份青稞种质材料也存在类似的问题, 遗传相似系数(GS)变异范围为0.2250~1.0000, 平均为0.7585, 在GS值为0.5106水平上可将参试材料聚为3个大类群, 各大类群分别包括91、13和4份材料, 说明各材料亲缘关系较近, 需要引入一些新的材料, 拓展亲本遗传基础, 其中品种ZDM5163和ZDM5737以及ZDM5199和ZDM5169之间的遗传相似系数最大, GS为1.0000,表明ZDM5163和ZDM5737以及ZDM5199和ZDM5169遗传背景可能比较相近, 本研究中出现两份材料间GS值为1的情况, 原因可能是品种本身遗传相似性较高, 或者也有可能是本研究所选取的引物数量有限导致, 本研究中只选用了48对引物, 引物的数量有限可能导致了未能完全区分供试材料间差异。川83-5319与甘青1号的GS值最小, 为0.2250,说明它们之间的遗传背景可能比较远, 可以考虑用于杂交组合的选配。
3.2青稞分子标记的关联分析
关联分析通过分析作物种质资源中标记与紧密连锁QTL之间的LD关系来鉴定QTL, 能够直接对基因型变异和表型变异进行分析, 可以确定不同种质资源中所携带的等位基因及其对目标性状的贡献[6]。对种质资源进行遗传多样性及群体遗传结构分析, 是关联作图的前提, 因为群体遗传结构会通过对连锁不平衡位点LD的影响从而对关联分析的准确性产生影响, 而LD水平的高低是关联分析的基础, 在亚群的基础上对是否达到哈德温伯格平衡的数学模型的群体结构分析, 可以计算出相对应的Q值(第i个材料基因组变异源于第k群体的概率), 在群体结构中亚群的混合能使整个群体LD的强度增加, 从而造成伪关联[26-29]。因此, 可以将Q值作为协变量纳入回归分析, 从而消除参试材料的群体结构引起的伪关联,也保证了后续关联分析的准确性。所以, 对现有种质资源进行群体遗传结构分析能更好地体现单个样本趋向各群体的比例, 对今后全面了解资源整体的遗传信息具有重要意义。
Hansen等[30]利用AFLP标记研究野生甜菜生长习性, 这是首次在植物中进行全基因组关联分析的报道。Cockram等[31]利用关联分析对控制春化反应的基因VRN-H1和VRN-H2分析鉴定证明, 运用适当的统计学方法, 校正群体结构复杂程度高的亚群划分后, 可将该群体用于关联分析。株高是最受重视的农艺性状之一, 研究表明, 在大麦1H到7H染色体上均有控制株高的QTL[6,32-34]。GrainGenes2.0网站(http://wheat.pw.usda.gov/GG2/index.shtml)公布的大麦株高相关标记有88个, 其中12个为SSR标记。本研究表明108份青稞可被分成4大类群, 分别包含21、42、3和44份材料。以CLM和MLM两种模型的关联分析, 分别找到12个和8个与株高、穗长、分蘖数、穗粒数相关的标记, 并且MLM中检测到的标记Bmac32、Bmac0163、HVGLUEND、EBmac775和HVM36在GLM中同样被检测到, 但标记的解释率较之要高。本研究与分蘖数相关的3个标记HVGLUEND、HVM13、EBmac775分别位于1H、4H、4H上, 其中标记EBmac775在前人研究中也被检测到, 但与千粒重相关[35], 可能是性状间相关的遗传原因所致。与穗长相关的标记GBM1215、Bmag7、MGB391、Bmag381、EBmac775、GBM1388、HVA22S分别位于6H、7H、2H、2H、4H、4H、7H上, 其中MGB391和Bmag7还曾被报道与株高相关联[6,34]。目前与穗粒数相关标记的文献报道较少, 本研究发现了4个与穗粒数相关的标记, 分别是位于1H、1H、2H 和4H上的MGB325、Bmac32、HVM36和HVJASIP, 其中HVM36与Wang等[36]报道的一致, 落在标记区间15.8~20.9内。
4 结论
108份材料可被分成4个大类群。并通过GLM 和MLM两种关联分析模型, 分别寻找到12个和8个与株高、穗长、穗粒数和分蘖数相关联的标记, 这些标记位于青稞各条染色体上。
References
[1] 孟凡磊, 强小林, 佘奎军, 唐亚伟, 胡银岗. 西藏主要农区青稞品种的遗传多样性分析. 作物学报, 2007, 33: 1910–1914
Meng F L, Qiang X L, She K J, Tang Y W, Hu Y G. Genetic diversity analysis among hulless barley varieties from the major agricultural areas of Tibet. Acta Agron Sin, 2007, 33: 1910–1914 (in Chinese with English abstract)
[2] Tanksley S D, McCouch S R. Seed banks and molecular maps: unlocking genetic potential from the wild. Science, 1997, 277: 1063–1066
[3] Bhagwat A A, Cregan P B, Akkaya M S. Length polymorphisms of simple sequencerepeat DNA in soybean. Genetics, 1992, 132: 1131–1139
[4] Maroof M S, Biyashev R M, Yang G P, Zhang Q, Allard R W. Extraordinarily polymorphic microsatellite DNA in barley: species diversity, chromosomal locations, and population dynamics. Proc Natl Acad Sci USA, 1994, 91: 5466–5470
[5] Wang Z, Weber J L, Zhong G, Tanksley S D. Survey of plant short tandem DNA repeats. Theor Appl Genet, 1994, 88: 1–6
[6] 赖勇, 王鹏喜, 范贵强, 司二静, 王晋, 杨轲, 王化俊. 大麦SSR标记遗传多样性及其与农艺性状关联分析. 中国农业科学, 2012, 46: 233–242
Lai Y, Wang P X, Fan G Q, Si E J, Wang J, Yang K, Wang H J. Genetic diversity and association analysis using SSR markers in barley. Sci Agric Sin, 2013, 46: 233–242 (in Chinese with English abstract)
[7] Maccaferri M, Sanguineti M C, Noli E, Tuberosa R. Population structure and long-range linkage disequilibrium in a durum wheat elite collection. Mol Breed, 2005, 15: 271–290
[8] Liu S B, Yang X P, Zhang D D, Bai G H, Chao S M, Bockus W. Genome-wide association analysis identified SNPs closely linked to a gene resistant to soil-borne wheat mosaic virus. Theor Appl Genet, 2014, 127: 1039–1047
[9] Ducrocq S, Madur D, Veyrieras J B, Camus-Kulandaivelu L, Kloiber-Maitz M, Presterl T, Ouzunova M, Manicacci D, Charcosset A. Key impact of Vgt1 on flowering time adaptation in maize: evidence from association mapping and ecogeographical information. Genetics, 2008, 178: 2433–2437
[10] Kumar B, Abdel-Ghani A H, Pace J, Reyes-Matamoros J, Hochholdinger F, Lübberstedt T. Association analysis of single nucleotide polymorphisms in candidate genes with root traits in maize (Zea mays L.) seedlings. Plant Sci, 2014, 224: 9–19
[11] Wen W W, Li D, Li X, Gao Y Q, Li W Q, Li H H, Liu J, Liu H J, Chen W, Luo J. Metabolome-based genome-wide association study of maize kernel leads to novel biochemical insights. Nat Commun, 2014, 5: 34–38
[12] Eizenga G C, Agrama H A, Lee F N, Yan W, Jia Y. Identifying novel resistance genes in newly introduced blast resistant rice germplasm. Crop Sci, 2006, 46: 1870–1878
[13] Yonemaru J, Mizobuchi R, Kato H, Yamamoto T, Yamamoto E, Matasubara K, Hirabayashi H, Takeuchi Y, Tsunematsu H, Ishii T. Genomic regions involved in yield potential detected by genome-wide association analysis in Japanese high-yielding rice cultivars. BMC Genomics, 2014, 15: 346
[14] D’hoop B B, Keizer P L, Paulo M J, Visser R G, van Eeuwijk F A,van Eck H J. Identification of agronomically important QTL in tetraploid potato cultivars using a marker-trait association analysis. Theor Appl Genet, 2014, 127: 731–748
[15] Wu D Z, Qiu L L, Xu L, Ye L Z, Chen M X, Sun D F, Chen Z H, Zhang H T, Jin X L, Dai F, Zhang G P. Genetic variation of HvCBF genes and their association with salinity tolerance in Tibetan annual wild barley. PLoS One, 2011, 6: e22938
[16] Brantestam A K, Bothmer R, Dayteg C, Rashal I, Tuvesson S, Weibull J. Genetic diversity changes and relationships in spring barley (Hordeum vulgare L.) germplasm of Nordic and Baltic areas as shown by SSR markers. Genet Resour Crop Evol, 2007, 54: 749–758
[17] Sun D F, Ren W B, Sun G L, Peng J H. Molecular diversity and association mapping of quantitative traits in Tibetan wild and worldwide originated barley (Hordeum vulgare L.) germplasm. Euphytica, 2011, 178: 31–43
[18] Kraakman A W, Martnez F, Mussiraliev B, Eeuwijk F A, Niks R E. Linkage disequilibrium mapping of morphological, resistance, and other agronomically relevant traits in modern spring barley cultivars. Mol Breed, 2006, 17: 41–58
[19] Ivandic V, Hackett C A, Nevo E, Keith R, Thomas W T B, Forster B P. Analysis of simple sequence repeats (SSRs) in wild barley from the fertile crescent: associations with ecology, geography and flowering time. Plant Mol Biol, 2002, 48: 511–527
[20] Ivandic V, Thomas W T B, Nevo E, Zhang Z, Forster B P. Associations of simple sequence repeats with quantitative trait variation including biotic and abiotic stress tolerance in Hordeum spontaneum. Plant Breed, 2003, 122: 300–304
[21] Paterson A H, Brubaker C L, Wendel J F. A rapid method for extraction of cotton (Gossypium spp.) genomic DNA suitable for RFLP or PCR analysis. Plant Mol Biol Rep, 1993, 11: 122–127
[22] Porebski S, Bailey L G, Baum B R. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components. Plant Mol Biol Rep, 1997, 15: 8–15
[23] Earl D A. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method. Conserv Genet Resour, 2012, 4: 359–361
[24] 潘志芬, 邹弈星, 邓光兵, 翟旭光, 吴芳, 余懋群. 青藏高原栽培青稞SSR标记遗传多样性研究. 中山大学学报(自然科学版), 2007, 46: 82–86
Pan Z F, Zou Y X, Deng G B, Zhai X G, Wu F, Yu M Q. Genetic diversity of SSR Markers in cultivated hulless barley from Qinghai-Tibet plateau in China. Acta Sci Nat Univ Sunyatseni, 2007, 46: 82–86 (in Chinese with English abstract)
[25] 杨平, 刘仙俊, 刘新春, 李俊, 王希文, 何守朴, 冯宗云. 利用SRAP标记研究四川高原青稞育成品种的遗传多样性. 遗传, 2008, 30: 115–122
Yang P, Liu X J, Liu X C, Li J, Wang X W, He S P, Feng Z Y. Genetic diversity analysis of the developed Qingke (hulless barley) varieties from the plateau regions of Sichuan province in China revealed by SRAP markers. Hereditas (Beijing), 2008, 30: 115–122 (in Chinese with English abstract)
[26] Harris B P, Stokesbury K E. The spatial structure of local surficial sediment characteristics on Georges Bank, USA. Continental Shelf Res, 2010, 30: 1840–1853
[27] Wang M L, Zhu C S, Barkley N A, Chen Z B, Erpelding J E, Murray S C, Tuinstra M R, Tesso T, Pederson G A, Yu J M. Genetic diversity and population structure analysis of accessions in the US historic sweet sorghum collection. Theor Appl Genet, 2009, 120: 13–23
[28] Kline J B, Moore D J, Clevenger C V. Activation and association of the Tec tyrosine kinase with the human prolactin receptor: mapping of a Tec/Vav-receptor binding site. Mol Endocrinol, 2001, 15: 832–841
[29] 武玉国, 吴承来, 秦保平, 王振林, 黄玮, 杨敏, 尹燕枰. 黄淮冬麦区175个小麦品种的遗传多样性及SSR标记与株高和产量相关性状的关联分析. 作物学报, 2012, 38: 1018–1028
Wu Y G, Wu C L, Qin B P, Wang Z L, Huang W, Yang M, Yin Y P. Diversity of 175 wheat varieties from Yellow and Huai River Valleys facultative wheat zone and association of SSR markers with plant height and yield related traits. Acta Agron Sin, 2012, 38: 1018–1028 (in Chinese with English abstract)
[30] Hansen M, Kraft T, Ganestam S, Sall T, Nilsson N O. Linkage disequilibrium mapping of the bolting gene in sea beet using AFLP markers. Genet Res, 2001, 77: 61–66
[31] Cockram J, White J, Leigh F J, Lea V J, Chiapparino E, Laurie D A, Mackay I J, Powell W, O'Sullivan D M. Association mapping of partitioning loci in barley. BMC Genet, 2008, 9: 16
[32] Marquez-Cedillo L A, Hayes P M, Kleinhofs A, Legge W G, Rossnagel B G, Sato K, Ullrich S E, Wesenberg D M. QTL analysis of agronomic traits in barley based on the doubled haploid progeny of two elite North American varieties representing different germplasm groups. Theor Appl Genet, 2001, 103: 625–637
[33]Teulat B, Borries C, This D. New QTLs identified for plant water status, water-soluble carbohydrate and osmotic adjustment in a barley population grown in a growth-chamber under two water regimes. Theor Appl Genet, 2001, 103: 161–170
[34] Korff M, Wang H, Léon J, Pillen K. AB-QTL analysis in spring barley: II. Detection of favourable exotic alleles for agronomic traits introgressed from wild barley (H. vulgare ssp. spontaneum). Theor Appl Genet, 2006, 112: 1221–1231
[35] 司二静, 张宇, 汪军成, 孟亚雄, 李葆春, 马小乐, 尚勋武, 王化俊. 大麦农艺性状与SSR标记的关联分析. 作物学报, 2015, 41: 1064–1072
Si E J, Zhang Y, Wang J C, Meng Y X, Li B C, Ma X L, Shang X W, Wang H J. Association analysis between SSR marker and agronomic traits in barley. Acta Agron Sin, 2015, 41: 1064–1072 (in Chinese with English abstract)
[36] Wang J, Yang J, McNeil D L, Zhou M. Identification and molecular mapping of a dwarfing gene in barley (Hordeum vulgare L.) and its correlation with other agronomic traits. Euphytica, 2010, 175: 331–342
URL: http://www.cnki.net/kcms/detail/11.1809.S.20151207.1041.002.html
Genetic Diversity and Association Analysis of Agronomic Characteristics with SSR Markers in Hulless Barley
MENG Ya-Xiong1,2, MENG Yi-Lin1,2, WANG Jun-Cheng1,2, SI Er-Jing1,2, ZHANG Hai-Juan1,2, REN Pan-Rong1,2, MA Xiao-Le1,2, LI Bao-Chun1,3, YANG Ke1,2, and WANG Hua-Jun1,2,*
1Gansu Provincial Key Laboratory of Aridland Crop Science / Gansu Key Laboratory of Crop Improvement & Germplasm Enhancement, Lanzhou 730070, China;2College of Agronomy, Gansu Agricultural University, Lanzhou 730070, China;3College of Life Sciences and Technology, Gansu Agricultural University, Lanzhou 730070, China
Abstract:The objectives of this study were to find molecular markers associated with yield-related traits and guide parental combination in molecular marker-assisted breeding and hybrid breeding of hulless barley (Hordeum vulgare L. var. nudum HK. f.). A natural hulless barley population composed of 108 parental varieties/lines was screened with 92 SSR markers, in which 48 markers were polymorphic. Population structure was analyzed based on the polymorphic SSR data and association between markers and five agronomic traits were performed in TASSEL GLM (general linear model) and MLM (mixed linear model) programs. A total of 156 alleles were detected in the 108 varieties/lines with 2–6 alleles per locus. The Shannon’s index of the population ranged from 0.6727 to 1.1368 and the genetic similarity between varieties ranged from 0.2250 to 1.0000, with the mean of 0.7585. Structure analysis revealed four genetic subpopulations for the entire materials tested. Based on GLM analysis, 12 SSR markers were found to be associated with plant height, spike length, grain number per spike and tiller number, with phenotypic contributions of 11.5–17.6%, 19.4–45.4%, 15.4–22.1% and 29.2%, respectively. Based on MLM analysis, 8 SSR markers were associated with plant height, awn length, and spikelet compactness, with the phenotypic contributions of 31.7–49.9%, 28.1–37.2%, and 22.7–32.7%, respectively. These associated markers were distributed on 6 chromosomes of the barley genome.
Keywords:Hulless barley; SSR; Genetic diversity; Population structure; Association analysis
收稿日期Received(): 2015-07-03; Accepted(接受日期): 2015-11-20; Published online(网络出版日期): 2015-12-07.
通讯作者*(Corresponding author): 王化俊, E-mail: whuajun@yahoo.com, Tel: 13809315256
DOI:10.3724/SP.J.1006.2016.00180
