过表达转录因子AhAREB1对拟南芥生长素分布的影响
2015-12-14林莹莹李晓云钟钰婷
林莹莹,李晓云 ,刘 帅,钟钰婷,李 玲
(华南师范大学生命科学学院,广东省植物发育生物重点实验室,广州510631)
植物的株型是植物从胚胎发生直至衰老死亡整个生长发育过程中与植物形态、相关器官发生,包括侧枝、株高、分枝、叶片及花器官等形态的发生相关[1].侧枝形成、株高、花的发育、结荚率、种子育性等均与农作物的产量、能源生物量等密切相关.借助基因芯片、测序手段,结合控制单个性状突变体的研究,研究者已获得了一些数量性状与质量性状共同参与植物株型的调控知识.其中,生长素(IAA)与细胞分裂素(CK)参与侧枝形成的调控,对株高的调控主要与赤霉素(GA)和油菜素内酯(BR)的作用有关[1].已报道拟南芥的IAA、CK、GA 和BR 相关基因突变体的株型发生改变,如GA 合成突变体(ga1-3)、GA 信号缺失突变体(gid)植株短小,晚花,雄性不育[2-3];此外,BR 合成途径的关键酶基因DET2、DWF4、CPD、DX、DDWF 及信号途径的br1、bin2、bak1、bes 和bsu1 等突变体[4-5],IAA 相关基因ibr5[6]、tir1[7]、axr1[8]、aux1[9]及iaa16[10]等突变体,以及细胞分裂素氧化酶(AtCKX)过表达植株均出现程度不同的矮化、育性降低等表型,外施ABA 导致植株矮小[11],以上研究表明,植物的株型涉及到多种激素参与的复杂调控.
AREB (ABA responsive element binding protein)/ABFs (ABRE binding factors)转录因子是ABA 信号途径下游的关键调控分子,可启动下游基因的表达[12].前期研究从耐旱花生品种粤油7 号克隆得到AhAREB1(Genbank No. JF766570),通过同源比对,AhAREB1 属于AREB 转录因子家族,与拟南芥 中 ABI5、ABF3/4、ABF2、ABF1 及 大 豆 的GmAREB1、马铃薯SlAREB 等高度同源[13]. AREB/ABFs 转录因子可被磷酸化修饰激活,与相关基因启动子中的DNA 顺式作用元件特异结合,激活干旱胁迫下ABA 依赖基因的表达,提高植物对环境胁迫的适应能力. 在拟南芥的ICK1、RD29B、RAB18、KIN2、KAT2、ADH1、CHS、racS、SUS1 等逆境响应相关基因的启动子区域,都存在ABREs(ACGTGGC)顺式作用元件[14-17]. 拟南芥中过表达该家族的ABF1、ABF2/AREB1、ABF3、ABF4/AREB2 均引起植物生长受抑制,植株矮化[14-17],但机制不详.
课题组在前期研究发现,p35S::AhAREB1(编码全长蛋白)转化拟南芥,增强转基因植株的抗旱和ROS 的清除能力,提高ABA 敏感性[18];拟南芥中异源过表达AhAREB1 转录因子后,植株呈现抽苔后矮小、叶片卷曲、荚果短小等生长素抑制的表型,植株体内IAA 含量显著高于野生型植株,外施IAA 也并不能完全恢复植株的表型[19];芯片表达谱分析发现一些与IAA 调控相关的基因表达发生差异变化,推测过表达AhAREB1 可能影响了植株体内IAA 的分布.pDR5::GUS 转基因植株是携带3个重复IAA诱导化学元件启动子控制GUS 基因表达,GUS 组织化学染色可观察GUS 基因表达强弱,直接表征植株体内IAA 的含量和分布.因此,本文利用GUS 组织化学染色方法探讨过表达AhAREB1 拟南芥植株内IAA 分布情况,初步探讨AhAREB1 转录因子对植株体内生长素分布的影响以及对植物株型的可能作用.
1 材料与方法
1.1 植物材料
植物材料为AhAREB1 过表达拟南芥株系(A38)和pDR5::GUS 拟南芥植株.取种子进行常规消毒后,均匀地点播于1/2 MS 培养基中,置于4℃冰箱春化2 d;将平板置放于光照培养箱内,在光周期为16 h 光照/8 h 黑暗、21 ℃条件下培养,约5 ~7 d后幼苗根长为1 ~2 cm 移至土中,培养条件为16 h 光照/8 h 黑暗,20 ~22 ℃,湿度70% 左右,2 ~3 d 浇水1 次.
1.2 过表达AhAREB1 植株和pDR5::GUS 植株杂交与鉴定
对母本植株(pDR5::GUS)为未开花刚露出白色花瓣的花蕊,用尖镊子或解剖针小心拨开花瓣,去除雄蕊.选择完全盛开的花作为父本(A38),用镊子夹取父本植株中花药在母本花柱头上轻轻擦拭数次,用细线在授粉花花柄处做好标记.2 ~3 d 后柱头膨大则说明杂交成功. 待角果成熟后,收取F1 代种子进行鉴定.
将F1 代的种子铺板并移苗于土中培养,2 周后取叶片进行PCR 检测,检测引物见表1.收取T1 代的种子铺潮霉素抗性板,选取绿苗种于土中,2 周后取叶片提DNA 进行基因组检测和GUS 组织化学染色检测.
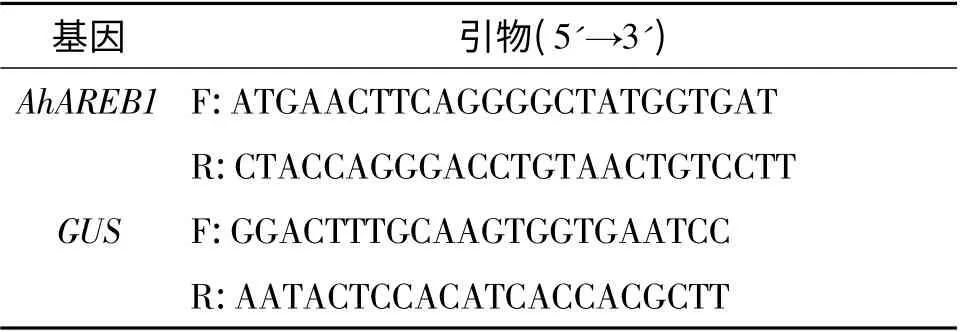
表1 PCR 鉴定引物Table 1 Primers for PCR check of target genes
1.3 GUS 组织化学染色
GUS 染色参照Jefferson(1987)的方法进行.GUS 染色反应液含0.5 g/L X-Gluc(5-溴-4-氯-3-吲哚-β-D-葡萄糖苷酸),50 mmol/L NaH2PO4,pH 7.0,0.001% TritonX-100,10mmol/L EDTA,0.5 mmol/L铁氰化钾和0.5 mmol/L 亚铁氰化钾. 待测样品与GUS 染色反应液于37 ℃保温4 h,然后用脱色液(V(无水乙醇)∶V(冰乙酸)=3∶1)脱色后,将材料的染色结果用扫描仪扫描或显微拍摄系统拍照.
1.4 基因芯片表达谱分析
取1/2MS 平板生长14 d 幼苗各100 μg,使用Trizol (TAKARA)法提取RNA 并进行反转成cDNA(TAKARA),进行Cy5 和Cy3 荧光标记,使用拟南芥的385 k 微阵列芯片(Roche NimbleGen)进行基因表达谱分析.
1.5 基因表达检测
定量荧光PCR 检测根据SYBR® Premix Ex TaqTM(Perfect Real Time)试剂盒说明书进行,每个样品重复3 次反应,取平均值,重复3 次实验. 在每次扩增的退火区间读取荧光强度,获取形成扩增曲线的荧光信号. 两步法PCR 扩增,即95 ℃,30 s;95 ℃,5 s,60 ℃,34 s,40个循环;于60℃收集信号.反应体系:ddH2O:6.8 μL,SYBR® Premix Ex Taq(2 ×)10 μL,PCR forward primer (10 μmol/L)0.4 μL,PCR Reverse primer(10 μmol/L)0.4 μL,ROX reference Dye Ⅱ0.4 μL,DNA 模板2 μL,总体积为20 μL.所用仪器为ABI 7500 PRISM real-time PCR system(Applied Biosystems,San Francisco). 检测基因所用的QPCR 引物见表2.

表2 Q-PCR 鉴定引物Table 2 Primers for Q-PCR detection of target genes
2 结果与分析
2.1 过表达AhAREB1 植株和pDR5::GUS 植株杂交鉴定
将AhAREB1 过表达株系与pDR5::GUS 植株杂交所得的T2 代种子种植培养,提取叶片DNA 进行PCR 鉴定.结果表明,1-2、9-1、16-1 及21-1 株系均可扩增出AhAREB1 全及GUS 基因(图1A),AhAREB1表达载体带有潮霉素抗性,通过潮霉素抗性板筛选,其中9-1、16-1 及21-1 为纯合体;提取幼苗RNA 进行RT-PCR 检测AhAREB1 的表达,结果表明,9-1、16-1 及21-1 株系中AhAREB1 过表达,而对照组WT和pDR5::GUS 植株中未检测到AhAREB1 基因表达(图1B).
2.2 过表达AhAREB1 植株休内生长素的分布

图1 拟南芥植株杂交后代PCR 鉴定Figure 1 PCR identification of filial generations of Arabidopsis plants
与对照相比,14 d 的AhAREB1 过表达幼苗经10 μmol/L ABA 处理5 h,在叶片和根尖GUS 染色较深,表明外源ABA 可能促进IAA 的累积和不均衡分布,增加植株体内IAA 的含量(图2A).5 μmol/L IAA 处理AhAREB1 过表植株5 h 后,与对照组植株相同,IAA 大部分积累于叶片和根尖,值得注意的是,对照组pDR5::GUS 在根的伸长区IAA 累积较多(图2B),推测AhAREB1 过表植株内IAA 的运输可能受阻.
与对照植株(pDR5::GUS)比较,过表达AhAREB1 的成苗植株叶片色较深(图3),表明累积较多的IAA,推测过高浓度的且分布不均衡的IAA抑制了叶片的伸长,导致叶片小而卷曲,AhAREB1转录因子可能参与对IAA 分布的调控.
2.3 过表达AhAREB1 植株内差异表达基因
基因芯片表达谱分析结果表明,在AhAREB1 过表达植株(A38)内,18个IAA 相关的基因表达发生显著变化(表3),其中有9个基因表达显著下调,另9个则显著上调;LAX3 为生长素运输样蛋白基因,AT5G65980 为编码生长素输出蛋白的基因,其它基因则编码生长素响应蛋白.
进一步通过QPCR 验证IAA29、LAX3、ARF9 和ARF2 四个基因表达,结果表明,AhAREB1 过表达植株中ARF9 和ARF2 表达显著上调,而IAA29 和LAX3 显著下调(图4),与芯片结果趋势一致.
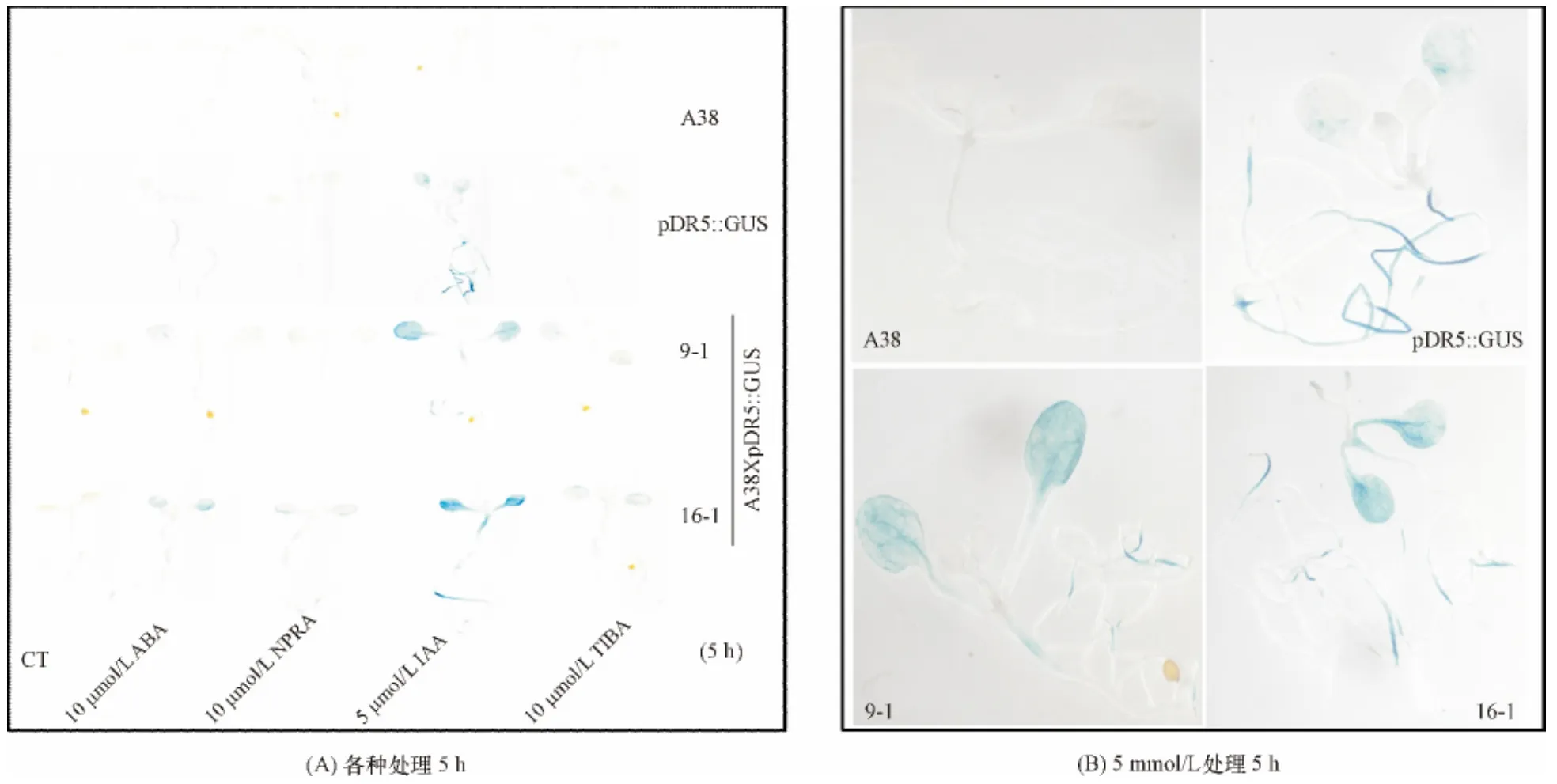
图2 过表达AhAREB1 拟南芥幼苗体内IAA 分布Figure 2 IAA distribution in Arabidopsis seedlings over-expressing AhAREB1

图3 过表达AhAREB1 拟南芥成苗体内IAA 分布Figure 3 IAA distribution in Arabidopsis mature plants over-expressing AhAREB1
3 讨论
植物AREB/ABF 类转录因子通过与ABRE 元件结合,倡导ABA 信号启动下游基因的表达. 拟南芥中AREB/ABF 转录因子共有4个成员,分别是AREB1/ABF2、AREB2/ABF4、ABF1 和ABF3[16,17].AhAREB1 转录因子属于AREB 类转录因子,可通过ABA 信号诱导下游相关基因响应表达;我们发现过表达AhABRE1 基因的拟南芥植株具有较强的抗旱性和ABA 敏感性[18],呈现出生长素过量的表型.本研究通过GUS 组织化学染色方法,直接观察过表达AhABRE1 植株体内IAA 在多处累积,主要集中在根尖,叶片边缘. 已知ABA 信号中的ABI1、ABI2 及ABI3 蛋白参与了IAA 信号的调控,抑制主根的伸长及促进侧根的发育[16-20]. 而AREB 类转录因子位于ABA 信号中ABI1、ABI2 及ABI3 下游. 因此,在AhABRE1 过表植株中,IAA 在叶片边缘累积导致叶片的伸长受阻,在根尖累积,抑制主根的伸长,促进更多侧根发生,有利于植物保水和增强抗旱的能力.

表3 差异表达的生长素相关基因Table 3 Differential expression of auxin related genes
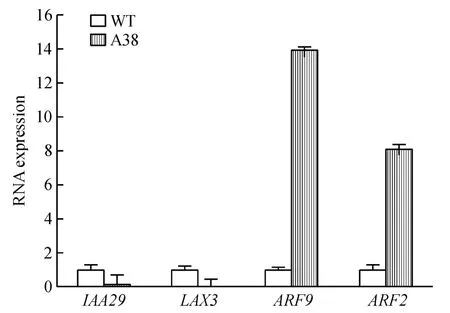
图4 过表达AhAREB1 植株中生长素相关基因表达Figure 4 Expression of auxin related genes in Arabidopsis plants over-expressing AhAREB1
目前认为ABA 信号与IAA 信号在胁迫响应和生长发育调控中存在关联[21-22]. 本研究结果发现,过表达AhABRE1 导致体内IAA 累积和不均衡分布,与部分IAA 的运输基因及响应基因差异表达.其中IAA29 为IAA 响应蛋白基因,在过表达植株体内显著下调.与Akkasaeng 等[23]报导该基因为ABA 诱导显著下调表达相符合. 通过启动子元件分析也发现IAA29 基因启动子有4个ABRE 作用元件,但AhABRE1 转录因子是否直接调控其表达,仍需进一步研究.LAX3 为生长素运输相关基因,为ABA 和干旱显著诱导下调,拟南芥ABF3(AREB 的同源基因)过表达植株表达下调,但经IAA 及ABA 合成抑制剂萘普生处理后诱导期表达上调,表明该基因下调可能阻碍IAA 的运输[24],本文结果进一步表明过表达AhABRE1 转录因子影响IAA 的分布和累积,为发掘植物AREB/ABFs 基因参与内源IAA 分布与调控,为认识AREB/ABFs 转录因子调节株型的研究提供依据.
[1]陈晓亚,薛卫红. 植物生理与分子生物学[M]. 4 版.北京:高等教育出版社,2012:424-435.
[2]Gan Y,Liu C,Yu H,et al. Integration of cytokinin and gibberellin signalling by Arabidopsis transcription factors GIS,ZFP8 and GIS2 in the regulation of epidermal cell fate[J]. Development,2007,134:2073-2081.
[3]Griffiths J,Murase K,Rieu I,et al. Genetic characterization and functional analysis of the GID1 gibberellin receptors in Arabidopsis[J]. Plant Cell,2006,18:3399-3414.
[4]Hong Z,Ueguchi M,Fujioka S,et al. The rice brassinosteroid-deficient dwarf2 mutant,defective in the rice homolog of Arabidopsis DIMINUTO/DWARF1,is rescued by the endogenously accumulated alternative bioactive brassinosteroid,dolichosterone[J]. Plant Cell,2005,17:2243-2254.
[5]Tanabe S,Ashikari M,Fujioka S,et al. A novel cytochrome P450 is implicated in brassinosteroid biosynthesis via the characterization of a rice dwarf mutant,dwarf11,with reduced seed length[J]. Plant Cell,2005,17:776-790.
[6]Monroe-Augustus M,Zolman B K,Bartel B. IBR5,a dual-specificity phosphatase-like protein modulating auxin and abscisic acid responsiveness in Arabidopsis[J].Plant Cell,2003,15:2979-2991.
[7]Strader C,Monroe-Augustus M,Rogers K C,et al. Arabidopsis iba response5 suppressors separate responses to various hormones[J]. Genetics,2008,180:2019-2031.
[8]Strader C,Monroe-Augustus M,Bartel B. The IBR5 phosphatase promotes Arabidopsis auxin responses through a novel mechanism distinct from TIR1-mediated repressor degradation[J]. BMC Plant Biology,2008,18:28-41.
[9]Rinaldi M A,Liu J,Enders A,et al. A gain-of-function mutation in IAA16 confers reduced responses to auxin and abscisic acid and impedes plant growth and fertility[J].Plant Molecula Biology,2012,79(4/5):359-373.
[10]Werner T,Motyka V,Laucou V,et al. Cytokinin-deficient transgenic Arabidopsis plants show multiple developmental alterations indicating opposite functions of cytokinins in the regulation of shoot and root meristem activity[J]. Plant Cell,2003,15:2532-2550.
[11]Finkelstein R. Abscisic acid synthesis and response[J].American Society of Plant Biologists,2013,30:1-36.
[12]刘帅,朱明鲲,刘旭,等. 拟南芥abf3 和abf4 突变体对ABA 和盐胁迫响应[J].华南师范大学学报:自然科学版,2012,44(4):100-104.Liu Shuai,Zhu Mingkun,Liu Xu,et al. Studies on the responses to ABA and Salt Stress in abf3 and abf4 related mutants of Arabidopsis[J]. Journal of South China Nomal University:Natural Science Edition,2012,44(4):100-104.
[13]Hong L,Hu B,Liu X,et al. Molecular cloning and expression analysis of a new stress-related AREB gene from Arachis hypogaea[J]. Biologia Plantarum,2011,57:56-62.
[14]Kim J B,Kang J Y,Kim S Y. Over-expression of a transcription factor regulating ABA-responsive gene expression confers multiple stress tolerance[J]. Plant Biotechnology Journal,2004,2:459-466.
[15]Kim S,Kang J Y,Cho D I,et al. ABF2,an ABREbinding bZIP factor,is an essential component of glucose signaling and its overexpression affects multiple stress tolerance[J]. Plant Journal,2004,40:75-87.
[16]Uno Y,Furihata T,Abe H,et al. Arabidopsis basic leucine zipper transcription factors involved in an abscisic acid-dependent signal transduction pathway under drought and high-salinity conditions[J]. Proceedings of the National Academy of Sciences,2000,97:11632-11637.
[17]Choi H,Hong J,Ha J,et al. ABFs,a family of ABA-responsive element binding factors[J]. The Journal of Biological Chemistry,2000,275:1723-1730.
[18]Li Y,Liu X,Yao Y,et al. Overexpression of arachis hypogaea AREB1 gene enhances drought tolerance by modulating ROS scavenging and maintaining endogenous ABA content[J]. International Journal of Molecular Sciences,2013,14:12827-12842.
[19]李健湄,李逸豪,林莹莹,等. 过表达AhAREB1 通过影响GA 代谢调控拟南芥株型的研究[J]. 植物生理学报,2014,50:925-930.Li J M,Li Y H,Lin Y Y,et al. Overexpression of AhAREB1 affected plant type of Arabidopsis regulated by GA metabolism[J]. Plant Physiology Journal,2014,50(7):925-930.
[20]Brady S M,Sarkar S F,Bonetta D,et al. The ABSCISIC ACID INSENSITIVE 3 (ABI3)gene is modulated by farnesylation and is involved in auxin signaling and lateral root development in Arabidopsis[J]. Plant Journal,2003,34:67-75.
[21]Rock C D,Sun X. Crosstalk between ABA and auxin signaling pathways in roots of Arabidopsis thaliana (L)Heynh[J]. Planta,2005,222:98-106.
[22]Wang H,Qi Q,Schorr P,et al. ICK1,a cyclin-dependent protein kinase inhibitor from Arabidopsis thaliana interacts with both Cdc2a and CycD3,and its expression is induced by abscisic acid[J]. Plant Journal,1998,15:501-510.
[23]Booker J,Auldridge M,Wills S,et al. MAX3/CCD7 is a carotenoid cleavage dioxygenase required for the synthesis of a novel plant signaling molecule[J]. Current Biology,2004,14:1232-1238.
[24]Akkasaeng C,Tantisuwichwong N,Chairam I,et al. Isolation and identification of peanut leaf proteins regulated by water stress[J]. Pakistan Journal of Biological Sciences,2007,10:1611-1617.
