融合多机器学习方法的siRNA在线设计系统
2015-12-12王璿,张法,陈伟
王 璿,张 法,陈 伟
(1.燕山大学信息科学与工程学院,河北秦皇岛066004;2.中国科学院计算技术研究所,北京100083;3.中国环境管理干部学院信息工程系,河北秦皇岛066102)
融合多机器学习方法的siRNA在线设计系统
王 璿1,∗,张 法2,陈 伟1,3
(1.燕山大学信息科学与工程学院,河北秦皇岛066004;2.中国科学院计算技术研究所,北京100083;3.中国环境管理干部学院信息工程系,河北秦皇岛066102)
siRNA设计是RNAi研究中的一个重要部分。由于靶向基因可分割成数以千计的候选siRNA,找到其中最有效的siRNA具有一定的挑战性。本文融合特征分析研究成果和多机器学习方法,设计并实现了一个siRNA在线设计系统。将目标RNA的二级结构作为影响siRNA干扰效率的评分因素,以挑选靶向合适位置的siRNA序列。对于给定的目标基因,系统经过设计得出若干高效siRNA序列的沉默效率及其相关信息。实验测试结果表明,本系统具有较高的siRNA有效性预测精度。
siRNA;有效性预测;机器学习方法;在线设计
0 引言
RNA干扰(RNA interference,RNAi)是生物进化过程中,由双链RNA(Double⁃stranded RNA,dsR⁃NA)诱发,使得同源mRNA特异性降解,转录后水平的基因沉默现象[1]。由于采用RNAi技术能迅速阻断特定基因表达,该方法广泛应用于基因功能分析、药物靶标发现、传染病防治及抗肿瘤研究等领域[2-3]。从作用机制上分析,长的dsRNA通常被切割成19~23 nt的干扰性小RNA(Short interfering RNA,siRNA),由经过设计的 siRNA识别靶向mRNA从而导致特定基因沉默[4⁃5]。针对靶基因不同点位设计的siRNA差别很大,因此有效的siRNA的选取是RNAi是否发挥效用的关键因素。
随着siRNA技术的深入研究,国内外陆续推出一些免费的siRNA设计软件。BIOPREDsi[6]是将机器学习方法引入到siRNA设计中的软件。它采用神经网络进行有效的siRNA预测,其沉默效率用0~1之间的小数表示。siDRM[7]用DRM算法对与siRNA有效性相关的特征进行合并和重组,最终得到一组规则集。SiDirect 2.0[8]将高TM值、转录过程中产生的移位等产生脱靶效应因素考虑到 siRNA设计中,设计出减少脱靶效应的siRNA序列。这 3个软件共同缺点是没有将siRNA分子及目标mRNA靶点的二级结构引入siRNA有效性设计的过程。siRNA[9]是Sfold软件中进行siRNA设计的一个功能模块,提供RNA二级结构预测功能,但是缺乏特异性检查。并且由于二级结构特征的计算耗时,软件限制目标基因序列长度。麻省理工学院的Whitehead[10]能够选择21nt的siRNA来阻断特定目标基因的表达。Dharmacon公司开发的siRNA设计软件采用SM⁃ARTpool[11]形式提供,将4条由SMARTselection技术设计的siRNA按比例混合,确保对任何人、小鼠和大鼠3个物种已知基因有高效沉默性。虽然混合siRNA提高了实验的成功率,但混合siRNA使得无法了解到底哪个siRNA真正有效,同时混合siRNA有引发副反应的可能且难以排除。siRNA Pro2.0[12]是华南理工大学设计的在线分析软件,规则设计中增加了经验、理性等综合因素,以提高siRNA预测准确性。
综合考虑已有的siRNA有效性设计方法,设计并开发了一个siRNA在线软件(siRNA Online)。用户可输入基因访问标识符或基因序列,经过设计分析获得多个高效的、特异的siRNA候选序列。该系统在有效性评分规则中考虑了mRNA二级结构信息,并采用融合BP神经网络和SVM多种机器学习方法实现评分,提高了有效性预测的准确性。
1 在线设计系统工作流程
siRNA在线分析系统根据用户输入的基因信息来设计有效的siRNA。用户可选3种输入形式:Accession访问标识符,GI访问标识符,mRNA序列。选择相关设计选项后系统进入设计阶段,如是否限定siRNA的起始序列;Blast等级选择等。设计结束后显示结果,也可保存或查询历史的设计记录。
siRNA设计阶段是系统的核心功能,设计流程如图1所示。首先从碱基、序列和自由能三方面入手,针对siRNA的19个碱基位制定各自位置的打分规则。剔除含有有害规则的siRNA序列,并提取靶标mRNA的二级结构信息,以挑选位于环区域效率高的 siRNA候选序列。其次,利用SVM特性筛选以及BP神经网络训练出较高精度的预测模型进行siRNA序列的有效性选择。采用BLAST将选择结果与非同源mRNA进行比对,完成特异性分析。最后,获得多个高效的、特异的siRNA候选序列。
2 系统关键技术
2.1 二级结构预测
目前mRNA二级结构预测分为基于序列比较和能量最小化两种方法。前者通过多重序列比对,根据相似序列具有相似结构的原理实现二级结构预测。后者通过能量优化或分析动力学计算评价所有可能配对的能量,以搜索具有最小能量的稳定结构。Zuker开发的MFold采用最近邻能量规则计算一个结构的能量[13]。其原理是考虑到环区内所有碱基均存在相互作用的可能,因此计算环区内所有碱基对的能量。本系统采用MFold实现二级结构预测。确定目标mRNA的单链环环区,根据mFold输出的mRNA的各碱基间配对情况,计算19nt的siRNA结合区域中未配对碱基的百分比,从而确定最佳的靶向位置,选择合适位置的siRNA序列。
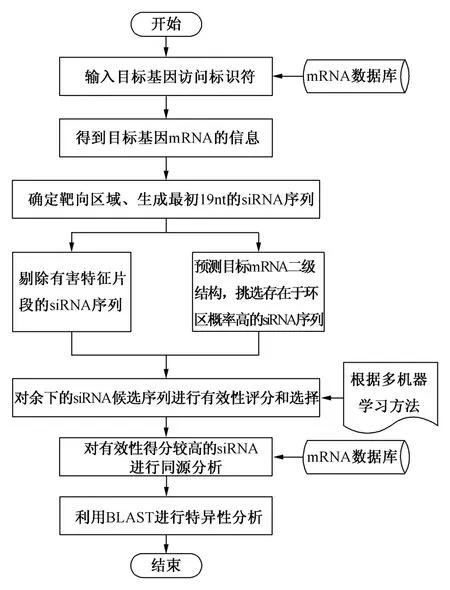
图1 siRNA设计阶段工作流程Fig.1 Workflow of siRNA design phase
2.2 特征分析
为保证设计结果的应用普遍性和设计规则的合理性,在前期工作中搜集和整理了3个siRNA数据集中的有效数据,如表1所示。通过统计学分析的方法进行了特征提取[14]。
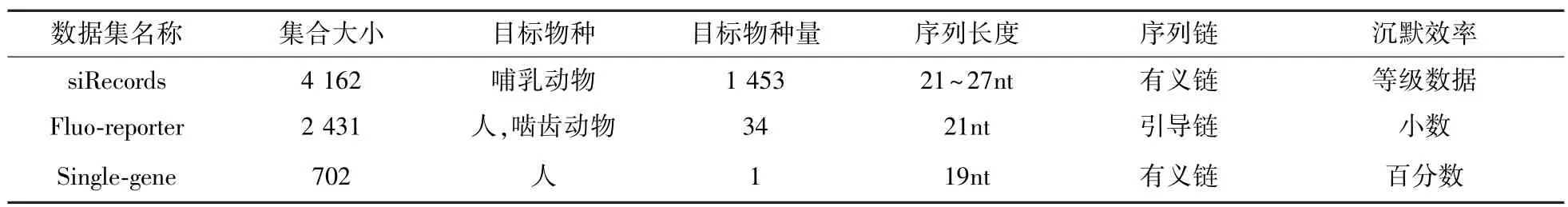
表1 siRNA数据集Tab.1 siRNA dataset
特征提取的具体方案为:1)SR和FR数据集分别划分为两个部分,作为特征分析集和内部测试集。由于SG与特征分析集来自不同的源数据集,SG作为外部测试集。2)由于SG数据集中的数据是靶向一个基因的所有可能的siRNA,没有经过挑选和设计,能够反映自然状态下siRNA沉默效率的整体分布规律。经统计分析得知其整体分布近似于正态分布,按此规律从SR和FR中随机抽取特征分析集的样本数据。3)采用SPSS11.5作为特征分析工具,SR特征集采用列联分析法,FR特征集采用方差分析法,且两组分析独立进行。分析过程中涉及到与siRNA沉默效率相关的已知的所有序列特征,例如特定位置的碱基偏好、特征片段、GC含量、siRNA双链稳定性差异等。当一个特征与siRNA沉默效率相关的显著性检验水平α≤0.05时,判定它是与siRNA的沉默效率相关的特征。对比两组特征得到最终的含有20个特征的相关特征集,并表示成一组siRNA设计规则。4)应用设计规则在内部测试集和外部测试集上对siRNA序列进行打分,根据得分结果评估该方案的可靠性。
2.3 BP神经网络模型
研究发现,各相关特征对siRNA有效性的影响各异。BP神经网络的自学习特性能够挖掘出各特征上的影响因子,从而给出更准确的siRNA有效性评分结果。基于获取的20个特征,构建了三层BP神经网络模型。采用FR数据集作为样本数据集,将反义链转换成19nt的正义链用于BP神经网络的训练。20个特征向量化为20维列向量,每个维度表示 siRNA序列的特征分,作为BP的输入。模型输出一个siRNA序列沉默效率,表示为0和1之间的小数。隐含层节点数目设定为6。利用遗传算法从初始输入中挑选适当的初值,减少训练过程的随机性。
为了有效评估BP神经网络模型,采用十折交叉验证的方法分别在FR和SG两个数据集上进行测试,并对比同等条件下不考虑权值的线性评分结果,如表2所示。测试结果表明,在两个数据集中BP神经网络的预测效果好于线性评分结果。SG数据集上的评分结果略高于FR数据集,由于SG数据集未参与模型训练,表明系统所设计模型具有良好的自学习能力和一定的泛化能力。
2.4 SVM学习方法
SVM采用的二次寻优的方法得到全局最优点,能够解决神经网络方法存在的局部极值问题。将SVM应用于siRNA有效性预测,能够提高结果的泛化性。利用LIBSVM软件包构建SVM模型,输入siRNA候选序列,输出siRNA序列的有效集和无效集。定义沉默效率>50%的为有效siRNA序列,沉默效率<50%的为无效siRNA序列。将SVM模型与 BP神经网络模型线性组合用于siRNA有效性预测。首先,SVM将siRNA候选序列划分为有效和无效两类。然后,选择有效候选序列中的siRNA作为BP神经网络模型的输入,通过BP模型筛选得到siRNA序列最终评分结果。
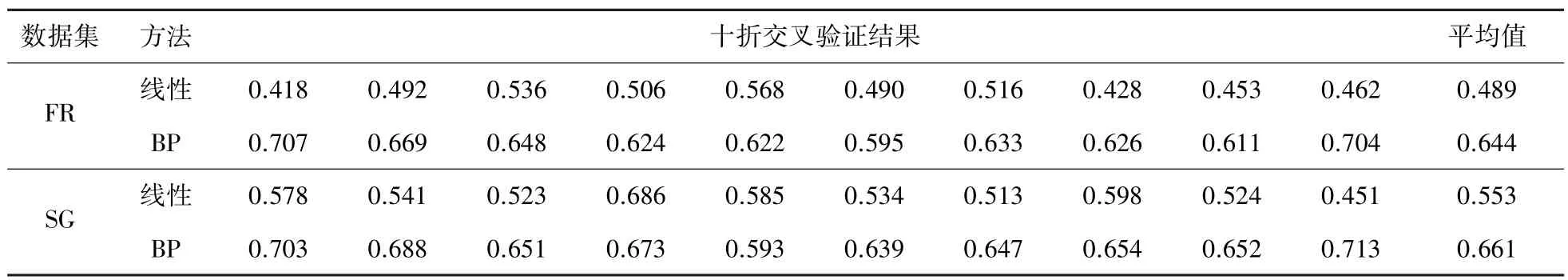
表2 BP神经网络模型与线性评分方法比较结果Tab.2 Comparison of BP neutral network model and linear scored method
实验中选取FR数据集中800条序列作为训练集,SG数据集中400条序列作为测试集,其中有效和无效序列数各占一半。图2对比了BP、SVM和SVM+BP 3种方法在两个数据集上测试结果。其中EP为有效序列的查准率,ER为有效序列的查全率,NR为无效序列的查全率。计算公式如下:EP=α/(α+γ),ER=α/(α+β),NR=δ/(δ+γ),式中α、β、γ、δ分别代表有效序列被预测为有效、有效序列被预测为无效、无效序列被预测为有效、无效序列被预测为无效的数目。由图2可知,在FR训练集中,SVM+BP方法表现出了接近BP方法良好的学习能力。在SG测试集中,SVM+BP表现出了接近SVM的良好的泛化能力。
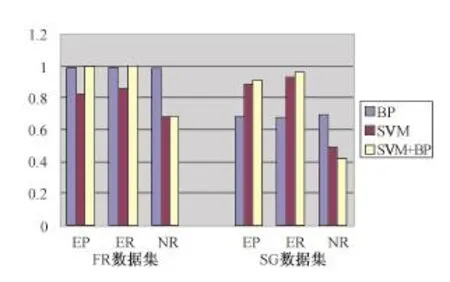
图2 3种机器学习方法的比较Fig.2 Comparison of three machine learning methods
2.5 BLAST特异性分析
为避免siRNA与非目标基因结合而产生脱靶效应,系统从两个方面来进行特异性分析。一方面针对siRNA序列与全基因组数据库内的非目标基因进行比对,剔除匹配过高的 siRNA序列。另一方面针对siRNA的seedregion区域(2nt~9nt)与非目标基因的3’UTR区域进行比对,剔除匹配过高的记录。经过两次匹配性筛选,有效地降低了siRNA与非目标基因结合的概率,减少了脱靶效应。系统中使用BLAST[15]并以mRNA数据库为目标数据库进行分析比对,并将筛选结果中的进行降序排列,取其中前十位作为最后的输出记录。
3 软件测试分析
为了测试siRNA在线设计系统的设计效果,与现有的White⁃head和Dharmacon软件进行了比对。针对P53mRNA,White⁃head和Dharmacon分别设计合成 14条,siRNA在线软件(siRNA Online)设计合成13条siRNA。用设计结果转染Hela细胞,并提取RNA通过荧光定量Real⁃time PCR检测其RNAi效果。3种软件产生干扰效率如表3所示。
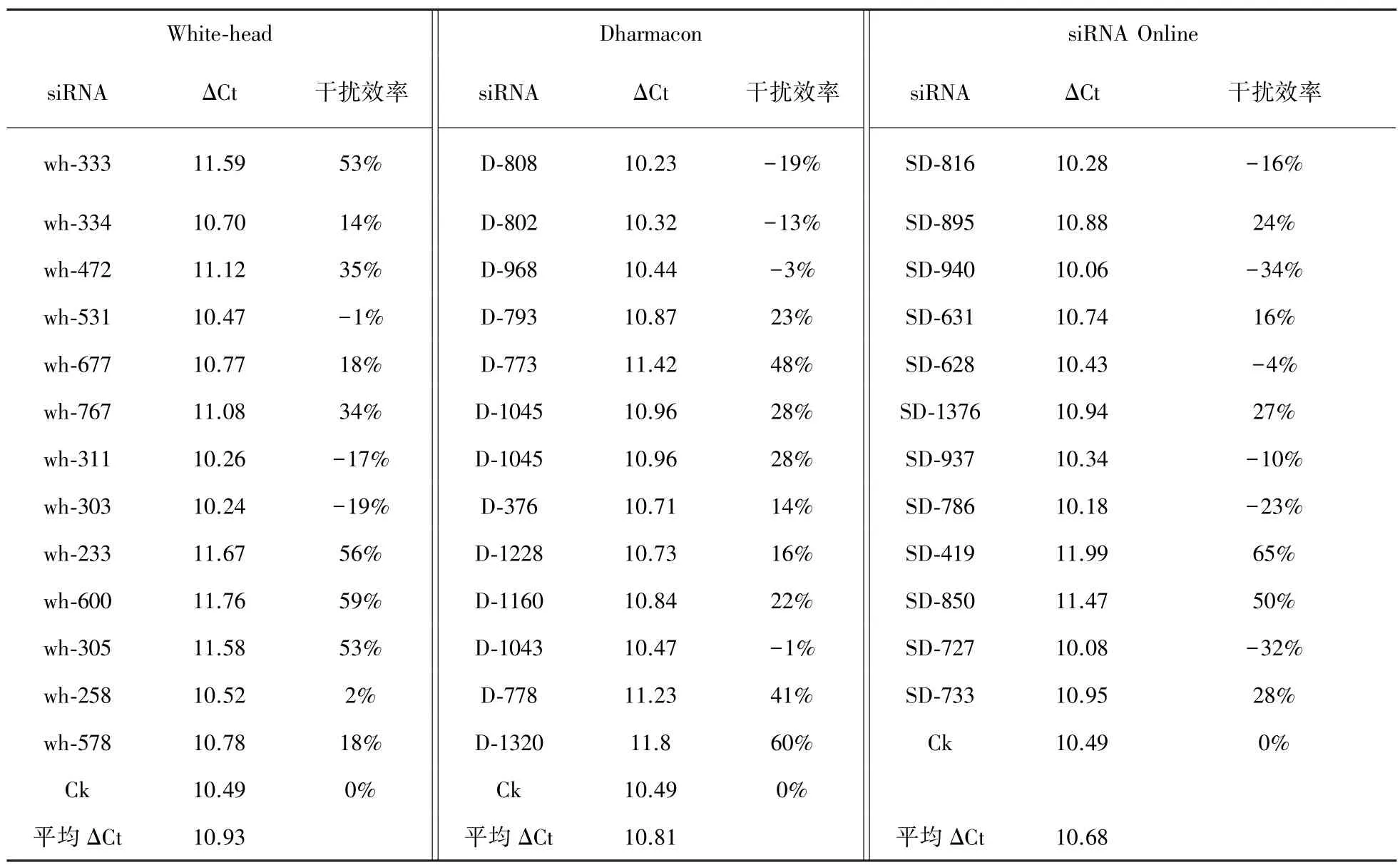
表3 3种软件干扰效率的比较Tab.3 The interference efficiency comparison of three softwares
表3中,ΔCt值表示RNAi实现效果的变量,其值越大则RNAi效果越好。分析各软件设计得到的siRNA分子对应RNAi实验得到的平均ΔCt值:White⁃head(10.93)>Dharmacon(10.81)>siRNA Online(10.68),表明siRNA Online的设计效果与White⁃head和Dharmacon软件设计效果相当。从各软件产生的有效 siRNA的个数分析,White⁃head(4个)>siRNA Online(2个)>Dharmacon(1个),表明siRNA Online系统具有良好的寻找有效siRNA的能力。
此外,siRNA Online还与siRNA Pro2.0软件进行了比较。数据测试采用SR数据集。随机选取14个目标mRNA,针对每个目标mRNA,在SR中有多条沉默效率已知的靶向该mRNA的siRNA分子记录。对于每个目标 mRNA,分别用 siRNA Online和siRNA Pro2.0进行设计,两个软件设计结果如表4中所示。通过与SR数据集中的数据比对,siRNA Online的设计结果比siRNA Pro2.0获得的有效siRNA数量多,且候选siRNA序列的沉默效果多为 Very High和 High。表明 siRNA Online具有较好的发现有效siRNA的能力。

表4 siRNA Online与siRNA Pro2.0设计结果比较Tab.4 Comparison of siRNA Online and siRNA Pro2.0 on design results
4 结论
1)设计并实现了一个siRNA有效性在线分析系统。利用统计分析方法提取了20个siRNA有效性特征用于siRNA有效性预测,并考虑了二级结构对预测结果的影响。采用SVM和BP神经网络两种机器学习方法应用于siRNA有效性预测,提高了预测精度。
2)采用了全序列BLAST比对以及seed region区域与mRNA的3’UTR区域的匹配的特异性分析方法,排除了大部分可能引起脱靶效应的候选siRNA序列,减少了脱靶效应的发生。
[1]Fire A,Xu S,Montgomery M K,et al.Potent and specific genetic interference by double⁃stranded RNA in Caenorhabditis elegans[J].Nature,1998,391(6669):806⁃811.
[2]Hayden C,Erika.RNA interference rebooted[J].Nature,2014,508(7497):443.
[3]Singh S K,Gaur R K.Progress towards therapeutic application of RNA interference for HIV infection[J].BioDrugs,2009,23(5):269⁃276.
[4]Hannon G J.RNA interference[J].Nature,2002,418(6894):244⁃251.
[5]Carthew R,Sontheimer E.Origins and mechanisms of miRNAs and siRNAs[J].Cell,2009,136(4):642⁃655.
[6]Huesken D,Lange J,Mickanin C,et al.Design of a genome⁃wide siRNA library using an artificial neural network[J].Nature Bio⁃technology,2005,23(8):995⁃1001.
[7]Gong W,Ren Y H,Wang Y,et al.siDRM:an effective and gen⁃erally applicable online siRNA design tool[J].Bioinformatics,2008,24(20):2405⁃2406.
[8]Naito Y,Ui⁃Tei K.Designing functional siRNA with reduced off⁃target effect[J].Methods in Molecular Biology,2013,942:57⁃68.[9]Ye D,Yu C C,Lawrence C E.Sfold web server for statistical fold⁃ing and rational design of nucleic acids[J].Nucleic Acids Re⁃search,2004,32(12):W135⁃W141.
[10]Yuan B,Latek R,Hossbach M,et al.siRNA selection server:an automated siRNA oligonucleotide prediction server[J].Nucleic Acids Research.2004,32(1):W130⁃W134.
[11]Montenmuros F D,Parise P.New technologies from siRNA world[J].Minerva Biotecnologica,2008,20(1):3⁃11.
[12]方翔,杜正平,曹以诚,等.siRNA pro 2.0:siRNA理性设计在线程序[J].中国生物化学与分子生物学报,2007,23(9):751⁃756.
[13]Zuker M.Mfold web server for nucleic acid folding and hybridiza⁃tion prediction[J].Nucleic Acids Research,2003,31(13):3406⁃3415.
[14]Wang Dongfang,Chen Xiang,Zhang Fa,et al.A method to im⁃prove the universality of siRNA design rules based on siRNA effi⁃ciency distribution[C]//2008 International Symposium on Infor⁃ mation Science and Engineering,Shanghai,2008:84⁃87.
[15]Altschul S F,Gish W,Miller W,et al.Basic local alignment search tool(BLAST)[J].Journal of Molecular Biology,1990,215(3):403⁃412.
siRNA online design system based on multi⁃machine learning methods
WANG Xuan1,ZHANG Fa2,CHEN Wei1,3
(1.School of Information Science and Engineering,Yanshan University,Qinhuangdao,Hebei 066004,China;2.Institute of Computing Technology,Chinese Academy of Sciences,Beijing 100083,China;3.Department of Information Engineering,Environmental Management College of China,Qinhuangdao,Hebei 066102,China)
siRNA Design has attracted attention as an important issue in RNAi research.As a targeted gene could have thousands of potential siRNAs,finding the most efficient siRNAs among them had a huge challenge.In this paper,a siRNA online design system was developed.It included multiple technologies such as feature analysis,machine learning methods.For filtering siRNA sequences on suitable location,the secondary structure of target RNA was also acted as a scoring factor which effect siRNA interference effi⁃ciency.To given target genes,this system showed effective siRNA sequences and related information sorting by the silencing effi⁃ciency.Compared with existing software and methods,siRNA Online has higher prediction accuracy.
siRNA;efficiency prediction;machine learning method;online design;
TP393;Q811
A
10.3969/j.issn.1007⁃791X.2015.05.013
1007⁃791X(2015)05⁃0458⁃06
2015⁃06⁃28 基金项目:国家自然科学基金资助项目(61232001,61202210);河北省高等学校科学技术研究青年基金资助项目(QN2015133)
∗王璿(1977⁃),女,黑龙江齐齐哈尔人,博士,副教授,主要研究方向为并行计算、生物计算、数据库理论,Email:wangxuan@ysu.edu.cn。
