杆状病毒的DNA聚合酶
2015-11-22杨洪桂李俊凯冯国忠
方 洋,杨洪桂,李俊凯,冯国忠
(1.长江大学农学院,湖北荆州434025;2.中国水稻研究所,水稻生物学国家重点实验室,杭州310006)
1 杆状病毒
杆状病毒 (Baculovirus)是一类感染无脊椎动物的病原微生物,其基因组为闭合双链环状DNA,病毒粒子呈杆状,其基因组大小一般为80-180 kb,编码90-180个基因。杆状病毒分为4个属:α杆状病毒属、β杆状病毒属、γ杆状病毒属与δ杆状病毒属 (King et al.,2012)。杆状病毒在一个病毒复制周期内产生两种不同形态、不同功能的病毒,即芽生型病毒 (budded virus,BVs)和包埋型病毒 (occlusion derived virus,ODV)。芽生型病毒产生在病毒感染早期,它主要负责细胞与细胞之间的传播;而在感染晚期产生包埋型病毒,主要在昆虫之间进行传播。目前杆状病毒广泛应用于生物农药、蛋白质表达和疫苗生产(Moscardi,1999;Szewczyk et al.,2006;Vicente et al.,2011;Cox,2012;Barford et al.,2013;Contreras-Gomez et al.,2014)。
2 杆状病毒DNA的复制
杆状病毒基因组是环状的DNA分子,它携带病毒所有的遗传信息。病毒一旦感染昆虫细胞,病毒粒子便释放DNA进入细胞核基质,病毒DNA在基质内开始复制。DNA复制是病毒生命周期中最重要的一环,也是病毒增殖周期中最重要的事件。杆状病毒DNA的复制是通过顺式作用元件和反式作用因子及其宿主细胞提供的复制因子相互作用来进行的。顺式作用元件指病毒DNA的复制原点,它包括同源重复序列 (homologous region)和非同源重复序列 (non-homologous region)两大类。反式作用因子即病毒DNA复制所需要的病毒表达产物。参与病毒复制的宿主成分目前还不清楚。对于反式作用因子,瞬时表达表明,杆状病毒的6类基因在病毒DNA复制过程中是必需的,包括转录激活因子 ie-1,DNA聚合酶 (DNA polymerase,dnapol),解旋酶基因p143,引物酶lef-1,引物酶辅助因子lef-2和DNA单链结合蛋白lef-3。这些基因都直接参与病毒DNA的复制,其中lef-1,lef-2和 lef-3为晚期表达因子 (Kool et al.,1994;Lu& Miller,1995;Huang& Levin,2001a;Sahdev et al.,2010),在杆状病毒的细菌人工染色体缺失实验中也证明dnapol、p143、ie-1和lef-3是病毒复制必需的 (Bideshi&Federici,2000;Stewart et al.,2005;Yu & Carstens,2010,2012;Feng et al.,2012)。其它基因如p35、ie-2、p38等基因能够刺激DNA的复制。ie-1作为一个早期蛋白,是病毒复制时主要的反式作用因子,在病毒感染的早期和晚期表达 (Stewart et al.,2005)。
3 DNA聚合酶基因的表达与调控
杆状病毒的表达具有时序性,在不同的时间内,所表达的基因不同。按照时间的先后,将杆状病毒的基因分为两大类,即早期基因和晚期基因,早期基因是指那些表达发生在病毒DNA复制之前的基因,它又可细分为极早期基因和滞早期基因;晚期基因是指发生在病毒DNA复制后表达的基因。实验表明,杆状病毒的时序表达是通过时序之前的一组病毒基因产物,直接或间接的反式激活其时序之后的一组病毒基因转录 (Friesen,1997)。
苜蓿丫纹夜蛾核多角体病毒 (Autographa californica multiple nucleopolyhedrovirus,AcMNPV)感染昆虫细胞2 h后,DNA聚合酶的mRNA开始转录,在感染4-6 h后表达量达到最高,感染10 h后mRNA量急剧下降 (Tomalski et al.,1988)。AcMNPV极早期基因ie-1的表达,能够反式激活AcMNPV DNA聚合酶的启动子,显著地增强了DNA聚合酶的转录。由于AcMNPV的DNA聚合酶在IE-1后转录,又在DNA复制之前表达,因此属于杆状病毒的滞早期基因。AcMNPV DNA聚合酶没有传统的杆状病毒早期基因启动子元件TATA盒以及起始位点。对AcMNPV DNA聚合酶ORF上游连续截断分析,发现在起始密码子ATG上游的210至240 bp之间为DNA聚合酶的启动子区域(Ohresser et al.,1994)。在DNA聚合酶的5'末端非编码区鉴定了至少两个转录起始位点,一个位于ATG起始密码子上游-120核苷酸处,另一个位于-212核苷酸处,而DNA聚合酶基因的3'端多腺苷化位点AATAAA与终止密码 (+2952)重叠 (Tomalski et al.,1988)。海灰翅夜蛾核多角体病毒(Spodopteras littoralis multinucleocapsid nucleopolyhedrovirus,SpliNPV)DNA聚合酶也是在感染细胞2 h后转录,但是这个转录可持续到感染后的48 h,且只有一个转录本 (Huang&Levin,2001)。家蚕核多角体病毒 (Bombyx mori nuclear polyhedrosis virus,BmNPV)的DNA聚合酶也没有TATA启动子元件,但是包含一个富含GC的转录起始位点区域。在家蚕感染细胞2 h后,能检测到DNA聚合酶的转录本,到感染后10 h转录本达到最大量,BmNPV DNA聚合酶的转录一直持续到病毒感染后的18 h。与AcMNPV和SpliNPV的DNA聚合酶不同,BmNPV至少有7个转录本,且这些转录本在病毒感染的不同时间表达 (Chaeychomsri et al.,1995)。
4 杆状病毒DNA聚合酶的生化特征
DNA聚合酶作为病毒复制过程中的关键酶,忠实地复制病毒基因组的信息。在DNA复制时,DNA聚合酶通过识别RNA引物的3'-OH,以单链DNA为模板沿5'-3'方向不断延伸,催化DNA的合成,在复制的过程中起聚合作用;而3'-5'核酸外切酶活性主要是校验和纠正DNA合成时发生的错误,删除相反方向合成的DNA,保证DNA准确的复制和先导链的持续合成。
DNA聚合酶作为保守基因之一,存在于所有的杆状病毒之中。AcMNPV DNA聚合酶的复制是在单链结合蛋白 (single-stranded DNA binding protein,SSB)LEF-3的共同作用下,催化链的置换来保持DNA的持续合成能力的 (Hang et al.,1995)。AcMNPV DNA聚合酶拥有3 kb大小的开放阅读框,编码984个氨基酸,分子量大小为114.31 kDa(Tomalski et al.,1988;Lu &Carstens,1991)。早期的生化分析研究表明,AcMNPV DNA聚合酶除了具有重要的聚合活性外,还具有3'-5'外切酶活性,但不具有5'-3'外切酶活性 (Hang&Guarino,1999)。AcMNPV DNA聚合酶与宿主细胞的DNA聚合酶对热的敏感性以及盐的浓度等方面存在较大的差异,AcMNPV DNA聚合酶在宿主细胞中的表达抑制了宿主DNA聚合酶的活性 (Miller et al.,1981)。SpliNPV的DNA聚合酶编码998个氨基酸的阅读框,分子量大小为114.93 kDa。生化分析显示纯化的SpliNPV DNA聚合酶具有聚合酶活性和3'-5'外切酶活性,离体测定证实SpliNPV的DNA聚合酶能够刺激病毒DNA的复制原点 (Huang&Levin,2001)。BmNPV的DNA聚合酶编码988个氨基酸的阅读框,分子量大小为114.65 kDa。纯化的BmNPV DNA聚合酶具有3'-5'外切酶活性,但不具有5'-3'外切酶活性 (Mikhailov et al.,1986)。BmNPV的DNA复制不依赖增殖细胞核抗原 (Proliferating Cell Nuclear Antigen,PCNA)(Liu et al.,2013)。
5 杆状病毒DNA聚合酶基因的结构
DNA聚合酶通过两种不同方式调节DNA复制及其保真性:(1)聚合酶结构域选择正确的核苷酸并将其插入到合成的引物端;(2)核酸外切酶结构域对已配对的碱基进行校正,并删除错配的碱基。杆状病毒DNA聚合酶的功能与氨基酸的结构域相对应,对杆状病毒氨基酸序列比对以及功能预测显示,杆状病毒的DNA聚合酶分为四个结构域,分别是:(1)N末端;(2)外切酶结构域;(3)聚合酶结构域;(4)C末端。其基因结构如图1所示。目前已有一些病毒的DNA聚合酶晶体结构被解析,如单纯性疱疹病毒 (herpes simplex virus,HSV)、噬菌体 RB69的 DNA聚合酶(Franklin et al.,2001;Liu et al.,2006),这些DNA聚合酶与杆状病毒的DNA聚合酶均为family B的DNA聚合酶,这类蛋白在进化上保守,暗示其在功能上具有相似性 (Blanco et al.,1991;Braithwaite& Ito,1993;Huang& Levin,2001b;Matthew et al.,2001;Gilbert,2002;Liu et al.,2006)。这些DNA聚合酶晶体结构的解析为杆状病毒DNA聚合酶的研究提供了便利。
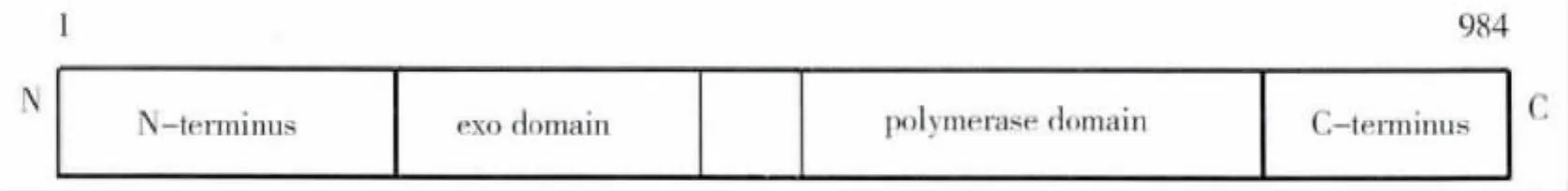
图1 DNA聚合酶的结构域Fig.1 The structural domains of DNA polymerase
5.1 外切酶结构域
杆状病毒DNA聚合酶外切酶结构域具有3'-5'外切酶功能,在DNA复制过程中起校正、编辑和删除错配碱基的作用。对AcMNPV DNA聚合酶氨基酸序列分析显示,具有外切酶活性的结构域靠近DNA聚合酶的N末端,该结构域含有三个极为保守的模序:EXOⅠ(氨基酸189-202)、EXOⅡ (氨基酸271-289)和 EXOⅢ (氨基酸387-401)(Huang& Levin,2001b;Feng& Krell,2014),这三个模序的氨基酸为杆状病毒DNA聚合酶的保守序列,这些保守序列与其它病毒DNA聚合酶的外切酶结构域序列极为相似,如HSV-1、EB病毒 (Epstein-Barr Virus,EBV)等DNA病毒的DNA聚合酶。HSV-1 DNA聚合酶模序ExoIII的位点突变 (Y577H和D581A)导致DNA聚合酶失去了外切酶活性,带有突变的重组病毒转染细胞后,在细胞中检测不到病毒基因组的复制。模序ExoI(D368A和E370A)和ExoIII(Y577F和D581A)的点突变也导致了外切酶活性的丧失,证实这些保守的区域与外切酶活性有关 (Kuhn&Knopf,1996;Hwang et al.,1998;Tian et al.,2009)。另外,HSV-1 DNA聚合酶的外切酶结构域ExoⅢ的突变不仅造成外切酶功能的缺陷,导致了更高的突变率,而且还改变了对药剂的敏感性(Hwang,1997;Hwang et al.,1998)。HSV -1 DNA聚合酶ExoII(D471A)的点突变导致外切酶活性减弱,从而消弱了这些突变重组病毒的复制和感染能力,这些点的突变都发生在外切酶结构域中 (Joyce& Steitz,1994;Hall et al.,1995;Liu et al.,2006)。杆状病毒的外切酶结构域模序与这些病毒DNA聚合酶结构域相似,暗示外切酶结构域中三个保守模序也具有同样的功能。
5.2 聚合酶结构域
聚合酶结构域的功能是选择正确的核苷酸并将其插入到正在合成的引物端。氨基酸序列分析以及功能预测显示杆状病毒的DNA聚合酶结构域由七个保守模序 (motif)组成,其结构顺序依次为:Ⅳ-Ⅱ -Ⅵ -Ⅲ -Ⅰ -Ⅶ -Ⅴ (Hall et al.,1989;Chaeychomsri,1995;Hwang et al.,2003)。在DNA聚合酶中,RegionⅣ并没有与聚合酶结构域连接在一起,而是在外切酶结构域内,其结构如图2所示。因为聚合酶结构域的模序Ⅱ-Ⅵ-Ⅲ-Ⅰ-Ⅶ-Ⅴ晶体几何图形类似于人的手,因此又把它分为palm、fingers和thumb三个亚结构域。其中 RegionⅢ和Ⅵ属于 fingers亚结构域,RegionⅠ、Ⅱ和Ⅶ在palm亚结构域内,thumb亚结构域包括RegionⅤ (Franklin et al.,2001;Liu et al.,2006)。RegionⅠ、Ⅱ、Ⅲ、Ⅴ和Ⅶ的保守区域在底物识别中起着直接或间接作用,根据已经发表HSV-1和噬菌体RB69 DNA聚合酶的晶体结构,这些保守区域参与了dNTPs、引物与模板DNA间的相互作用 (Franklin et al.,2001;Liu et al.,2006)。两个抗病毒药剂 aphidicolin和abacavir诱导了AcMNPV DNA聚合酶在RegionⅡ和RegionⅢ的点突变 (C543R和S611T),而这两个点的突变减少了DNA的复制,也降低了病毒的滴度 (Feng et al.,2012),证实这两个区域直接参与了病毒DNA的复制。HSV-1的DNA聚合酶在RegionⅦ中的点突变 (Y937H)增加了对药剂acyclovir和foscarnet的抗药性,推测regionⅦ可能与核苷酸的识别和结合有关 (Hwang et al.1992),水痘 -带状疱疹病毒 (varicella-zoster virus,VZV)DNA聚合酶在regionⅢ的位点突变 (N779S和G805C)以及在Region I的位点突变 (V855M)增加了对acyclovir(ACV)和phosphonoacetic acid(PAA)的抗药性,暗示这些位点的变化导致了蛋白质构像的变化,同时也预示这些位点参与了核苷酸的识别 (Kamiyama et al.,2001)。HSV-1 RegionⅥ的位点突变 (L774F)增加了DNA复制的效率与精确性 (Tian et al.,2009)。在RegionⅠ和RegionⅥ突变的HSV-1重组病毒在转染细胞后与野生型病毒比较,结果显示突变后的重组病毒对药剂敏感性有所改变,同时也提高了DNA复制的保真性 (Marcy et al.,1990)。这些实验结果证明,聚合酶结构域的各个模序在dNTP的结合、链的延伸上具有重要的功能。
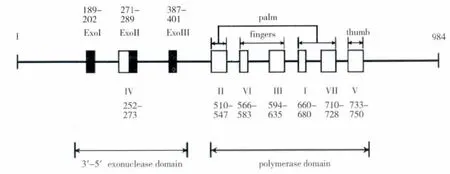
图2 AcMNPV DNA聚合酶结构域的模序Fig.2 The modifs of AcMNPV DNA polymerase structural domains
5.3 DNA聚合酶的C末端和N末端
Family B的类αDNA聚合酶的外切酶结构域和聚合酶结构域序列高度保守,因此在功能上也具有相似性,迄今为止已有大量关于聚合酶结构域和外切酶结构域的研究,但是对DNA聚合酶C末端的报道不多。目前对DNA聚合酶C末端的报道也仅限于 AcMNPV和 HSV-1的 DNA聚合酶(Monahan et al.,1993;Feng et al.,2012)。对AcMNPV DNA聚合酶的研究证实,在其C末端包含一个单分型的核定位信号和一个经典的多分型核定位信号,这两个核定位信号对于DNA聚合酶的入核是至关重要的,缺失掉其中的任何一个,DNA聚合酶都不能进入核内。另外,在AcMNPV DNA聚合酶C末端的972-981位点还包含一个典型的模序 (NNTYKFCLYK),这个模序存在于所有α杆状病毒,对这个区域的位点突变导致病毒无法传播,同时也影响了病毒的复制,暗示这个区域具有重要的功能,对其功能的鉴定有待深入的研究 (Feng et al.,2014)。与AcMNPV DNA聚合酶相似的HSV-1 DNA聚合酶C末端也具有重要的功能,它与能够增加DNA延伸作用的附属蛋白UL42相互作用,从而激活了UL42的活性,证实HSV-1 DNA聚合酶C末端同样也具有重要的功能 (Monahan et al.,1993;Digard et al.,1993)。
目前,除HSV-1 DNA聚合酶N末端外,其他病毒尚未见报道 (Terrell&Coen,2012)。Liu等(2006)根据HSV-1 DNA聚合酶的N末端氨基酸序列比对结果,结合已发表的HSV-1 DNA聚合酶晶体结构,推测N末端第1-140保守序列在病毒的复制过程中或许具有重要功能。通过缺失聚合酶N末端氨基酸1-51位点以及位点突变氨基酸序列44-49并构建重组病毒,发现这些重组病毒转染细胞后,这两个缺失体的病毒DNA在细胞中不能正常合成,证实DNA聚合酶N末端与5'-3'聚合酶活性有关 (Terrell&Coen,2012)。
6 DNA聚合酶的核定位信号
真核细胞的细胞核有核膜包被,核膜上的核孔复合体 (nuclear pore complex,NPC)是细胞核内外进行物质交换的主要通道,蛋白质或多肽自由通过NPC的能力取决于他们的大小:一般分子量小于5 kD的小分子蛋白或多肽可以自由的通过NPC;分子量在5-50 kD的蛋白,通过被动扩散和主动运输的方式进入细胞核;分子量大于50 kD的蛋白只能通过主动运输进入细胞核。通过主动运输进入细胞核的蛋白质必须在其序列上拥有特殊的信号,即核定位信号 (nuclear localization signal,NLS)。NLS被相应的核转运蛋白识别,并与核孔蛋白相互作用,帮助携带NLS信号的蛋白通过核孔到达细胞核 (Quimby&Dasso,2003)。
目前已鉴定的NLS主要有以下类型:(I)单分型NLS(monopartite NLS)。单分型NLS是一段由4-8个氨基酸组成的富含碱性氨基酸的短肽,在其中部一般包含4个或4个以上精氨酸和/或赖氨酸,两端是酸性氨基酸或脯氨酸或甘氨酸(Shields&Yang,1997)。单分型的核定位信号分为5种类型:①KR(K/R)R或K(K/R)RK;② (P/R)XXKR(^DE)(K/R);③KRX(W/F/Y)XXAF;④ (R/P)XXKR(K/R)(^DE);⑤LGKR(K/R)(W/F/Y),其中X代表任何一种氨基酸,^DE为除酸性氨基酸 (天冬氨酸和谷氨酸)之外的氨基酸。(Ⅱ)双分型NLS(bipartite NLS)。双分型NLS中间间隔10-12个氨基酸残基,这类核定位信号的特征是由两簇碱性氨基酸组成,两簇碱性序列之间被10-12个非保守的氨基酸残基间隔,其序列通式可表示为 R/K(X)10-12RRKK,X代表任何一种氨基酸 (Lange et al.,2007;Kosugi et al.,2009)。 (Ⅲ)其它类型的NLS。除了上述单分型和双分型的NLS外,还有一些无一定序列特征的NLS,他们主要存在于能在细胞核和细胞质之间穿梭的蛋白质中,M9、Rev等均具有这种特征 (Bohnlein et al.,1991,Wu et al.,1999)。
核定位信号在杆状病毒蛋白中的存在十分广泛,尤其是与病毒复制和装配相关的杆状病毒蛋白。这些蛋白包含单分型或多分型的NLS,暗示这些蛋白的入核与核转运蛋白有关 (Olson et al.,2002;Chen& Carstens,2005;Wang et al.,2008;Guo et al.,2010)。AcMNPV的DNA聚合酶C末端包含两个核定位信号模序,其中一个在DNA聚合酶的氨基酸位点939-948,其氨基酸序列为CSVKRKRDDD,具有典型的KRKR信号,为单分型①型核定位信号;另一个在DNA聚合酶的氨基酸位点 804-827,其氨基酸序列为DNPGKKRKSTDDNEGPSPKRRVIT,为典型的双分型核定位信号,这两个核定位信号共同介导了DNA聚合酶的入核 (Feng&Krell,2014)。我们也对已发表的63个杆状病毒DNA聚合酶进行了分析,结果显示α杆状病毒的groupⅠ多个DNA聚合酶的C末端包含至少两个核定位信号,其中一个为双分型的核定位信号,另一个为单分型的核定位信号,且这些单分型的核定位信号多为①型或④型。但是对于杆状病毒α属的groupⅡ,β,δ和γ的DNA聚合酶,只有7个能够预测到核定位信号,且这些核定位信号多为单分型①型,仅有斜纹夜蛾核多角体病毒 (Spodoptera litura NPV,SpliNPV)的DNA聚合酶核定位信号为单分②型。其余的37个杆状病毒DNA聚合酶不能预测到核定位信号,暗示这些DNA聚合酶包含新的核定位信号 (Feng& Krell,2014)。
7 DNA聚合酶与病毒结构
杆状病毒在其复制周期中产生两种不同表型的病毒BV和ODV。这两种不同表型的病毒不仅功能不同,而且在结构的组分上也存在差异。它们都具有衣壳和囊膜,且由多种蛋白组成。对AcMNPV多角体病毒蛋白质组学分析显示,包埋型病毒的核衣壳和囊膜涉及约44个病毒编码蛋白,其中DNA聚合酶为包埋型病毒的结构蛋白之一 (Braunagel et al.,2003)。
DNA聚合酶点的突变也影响了病毒粒子的结构和多角体的形成。AcMNPV DNA聚合酶在药剂的影响下产生了单点突变 (C543R)和双点突变(C543R/S611T),这两种点突变的DNA聚合酶补回到缺失DNA聚合酶的细菌人工染色体上,产生了APCrep(C543R)和ABCrep(C543R/S611T)两种补回型病毒。构建的重组病毒转染细胞后发现ABCrep产生单粒包埋的多角体,APCrep既有单粒包埋的多角体,又有多粒包埋的多角体,而野生补回型 (WTrep)基本上是多粒包埋的病毒多角体。另外,ABCrep产生的多角体直径约为 2.3μm,APCrep产生的多角体直径约为1.71μm,这两种突变补回型病毒的多角体显著大于WTrep的1.45μm(Feng et al.,2012)。杆状病毒的DNA聚合酶是怎样影响病毒粒子和多角体的形成有待深入的研究。
8 结语
DNA聚合酶既是病毒复制的重要酶,也是许多抗病毒药剂的靶标。深入地研究病毒DNA聚合酶的结构和功能,对理解病毒的复制机理具有重要意义。杆状病毒的DNA聚合酶与其它DNA病毒DNA聚合酶同属Family B的类α家族,因此在许多方面具有相似性 (Braithwaite&Ito,1993;Franklin et al.,2001)。本文通过总结杆状病毒DNA聚合酶的表达调控与生化特征、基因组结构及其功能,阐述了杆状病毒DNA聚合酶与病毒复制的相互关系。除此之外,杆状病毒DNA聚合酶也在病毒的装配和多角体的形成过程中发挥了重要作用,且在这些过程中担任了不同角色。杆状病毒作为模式DNA病毒之一,其基因结构和功能的研究将推动其它动物病毒的研究与发展。如杆状病毒DNA聚合酶像其它DNA聚合酶一样,对许多抗病毒药剂敏感 (Thumbi et al.,2007),由于杆状病毒只感染无脊椎动物而对人类安全,因此杆状病毒也可作为理想的抗病毒药物筛选体系。
References)
Barford D,Takagi Y,Schultz P,et al.Baculovirus expression:tackling the complexity challenge[J].Curr.Opin.Struct.Biol.,2013,23(3):357-364.
Bideshi DK,Federici BA.The Trichoplusia ni granulovirus helicase is unable to support replication of Autographa californica multicapsid nucleopolyhedrovirus in cells and larvae of T.ni[J].J.Virol.,2000,81(Pt 6):1593-1599.
Blanco L,Bernad A,Blasco MA,et al.A general structure for DNA-dependent DNA polymerases[J].Gene,1991,100:27 -38.
Bohnlein E,Berger J,Hauber J.Functional mapping of the human immunodeficiency virus type 1 Rev RNA binding domain:new insights into the domain structure of Rev and Rex [J].J.Virol.,1991,65(12):7051-7055.
Braithwaite DK, Ito J. Compilation, alignment, and phylogenetic relationships of DNA polymerases[J].Nucleic.Acids Res.,1993,21(4):787-802.
Braunagel SC,Russell WK,Rosas-Acosta G,et al.Determination of the protein composition of the occlusion-derived virus of Autographa californica nucleopolyhedrovirus [J].Proc. Natl.Acad.Sci.USA,2003,100(17):9797-9802.
Chaeychomsri S,Ikeda M,Kobayashi M.Nucleotide sequence and transcriptional analysis of the DNA polymerase gene of Bombyx mori nuclear polyhedrosis virus [J]. J.Virol.,1995,206:435-447.
Chen Z,Carstens EB.Identification of domains in Autographa californica multiple nucleopolyhedrovirus late expression factor 3 required for nuclear transport of P143 [J] .J.Virol.,2005,79(17):10915-10922.
Contreras-Gomez A,Sanchez-Miron A,Garcia-Camacho F,et al.Production production using the baculovirus-insect cell expression systemsystem [J].Biotechnol.Prog.,2014,30(1):1-18.
Cox MM.Recombinant protein vaccines produced in insect cells[J].Vaccine.,2012,30(10):1759-1766.
Digard P,Chow CS,Pirrit L,et al.Functional analysis of the herpes simplex virus UL42 protein[J].J.Virol.,1993,67(3):1159-1168.
Feng G,Krell PJ.Autographa californica multiple nucleopolyhedrovirus DNA polymerase C terminus is required for nuclear localization and viral DNA replication [J].J.Virol.,2014,88(18):10918-10933.
Feng G,Thumbi DK,de Jong J,et al.Selection and characterization of Autographa californica multiple nucleopolyhedrovirus DNA polymerase mutations[J].J.Virol.,2012,86(24):13576-13588.
Franklin MC,Wang J,Steitz TA.Structure of the replicating complex of a pol alpha family DNA polymerase.[J].Cell,2001,105(5):657-667.
Friesen PD.The Baculoviruses[M].New York:Plenum Press,1997,141-166.
Gilbert C,Bestman-Smith J,Boivin G.Resistance of herpesviruses to antiviral drugs:clinical impacts and molecular mechanisms[J].Drug Resistance Updates:Reviewsand Commentariesin Antimicrobial and Anticancer Chemotherapy,2002,5(2):88-114.
Guo ZJ,Wang DX,Yao Q,et al.Identification of a novel functional nuclear localization signal in the protein encoded by open reading frame 47 of Bombyx mori nucleopolyhedrovirus[J].Arch.Virol.,2010,155(12):1943-1950.
Hall D,Wang YS,Pierpont J,et al.Aphidicolin resistance in herpes simplex virus type I reveals features of the DNA polymerase dNTP binding site[J].Nucleic.Acids.Res.,1989,17(22):9231-9244.
Hall JD,Orth KL,Sander KL,et al.Mutations within conserved motifs in the 3'-5'exonuclease domain of herpes simplex virus DNA polymerase[J].J.Gen.Virol.,1995,76:2999-3008.
Hang X,Dong W,Guarino LA.The lef-3 gene of Autographa californica nuclear polyhedrosis virus encodes a single-stranded DNA-binding protein[J].J.Virol.,1995,69(6):3924-3928.
Hang X,Guarino LA. Purification of Autographa californica nucleopolyhedrovirus DNA polymerase from infected insect cells[J].J.Virol.,1999,80(Pt 9):2519-2526.
Huang J,Levin DB.Identification,transcription and sequence analysis of the Spodoptera littoralis nucleopolyhedrovirus(SpliNPV)DNA polymerase gene [J].Arch.Virol.,2001a,146(2):303-326.
Huang J,Levin DB.Expression,purification and characterization of the Spodoptera littoralis nucleopolyhedrovirus (SpliNPV)DNA polymerase and interaction with the SpliNPV non-hr origin of DNA replication [J].J.Gen.Virol.,2001b,82(Pt 7):1767-1776.
Hwang CB,Ruffner KL,Coen DM.A point mutation within a distinct conserved region of the Herpes Simplex Virus DNA Polymerase gene confers drug resistance [J].J.Virol.,1992,66(3):1774-1776.
Hwang YT,Liu BY,Coen DM,et al.Effects of mutations in the Exo III motif of the herpes simplex virus DNA polymerase gene on enzyme activities,viral replication,and replication fidelity[J].J.Virol.,1997,71(10):7791-7798.
Hwang YT,Smith JF,Gao L,et al.Mutations in the Exo III motif of the herpes simplex virus DNA polymerase gene can confer altered drug sensitivities[J].Virology,1998,246(2):298-305.
Hwang YT,Zuccola HJ,Lu Q,et al.A point mutation within conserved region VI of herpes simplex virus type 1 DNA polymerase confers altered drug sensitivity and enhances replication fidelity [J].J.Virol.,2003,78(2):650-657.
Joyce CM,Steitz TA.Function and structure relationships in DNA polymerases[J].Annu.Rev.Biochem.,1994,63:777-822.
Kamiyama T,Kurokawa M,Shiraki K.Characterization of the DNA polymerase gene of varicella-zoster viruses resistant to acyclovir[J].J.Gen.Virol.2001,82(Pt 11):2761-2765.
King AM, Adams, MJ, Lefkowitz EJ, et al. Classification and Nomenclature of Viruses:Ninth Report of the International Committee on Taxonomy of Viruses[M].New York:Academic Press,2012.
Kool M,Ahrens CH,Goldbach RW et al.Identification of genes involved in DNA replication of the Autographa californica baculovirus[J].Proc.Natl.Acad.Sci.USA,1994,91(23):11212-11216.
Kosugi S,Hasebe M,Tomita M,et al.Systematic identification of cell cycle-dependent yeast nucleocytoplasmic shuttling proteins by prediction of composite motifs[J].Proc.Natl.Acad.Sci.USA,2009,106(25):10171-10176.
Kuhn FJ,Knopf CW.Herpes simplex virus type 1 DNA polymerase.Mutational analysis of the 3'-5'-exonuclease domain[J].J.Bio.Chem.,1996,271(46):29245-54.
Lange A,Mills RE,Lang CJ,et al.Classical nuclear localization signals:definition,function,and interaction with importin alpha[J].J.Biol.Chem.,2007,282(8):5101-5105.
Liu L,Song H,Zhang L,et al.Expression,purification,and enzymatic characterization of Bombyx mori nucleopolyhedrovirus DNA polymerase[J].Arch.Virol.,2013,158(12):2453-2463.
Liu S,Knafels JD,Chang JS,et al.Crystal structure of the herpes simplex virus 1 DNA polymerase[J].J.Bio.Chem.,2006,281(26):18193-18200.
Lu A,Carstens EB.Nucleotide sequence of a gene essential for viral DNA replication in the baculovirus Autographa californica nuclear polyhedrosis virus[J].Virology,1991,181(1):336-347.
Lu A,Miller LK.The roles of eighteen baculovirus late expression factor genes in transcription and DNA replication[J].J.Virol.,1995,69(2):975-982.
Marcy AI,Hwang CB and Ruffer KL,et al.Engineered herpes simplex virus DNA polymerase point mutants:the most highly conserved region shared among alpha-like DNA polymerases is involved in substrate recognition [J].J.Virol.,1990,64:5883-5890.
Matthew C,Franklin JW,Steitz TA.Structure of the Replicating Complex of a Polα Family DNA Polymerase [J].Cell,2001,105:657-667.
Mikhailov VS,Marlyev KA,Ataeva JO,et al.Characterization of 3'-5'exonuclease associated with DNA polymerase of silkworm nuclear polyhedrosis virus[J].Nucleic Acids Res.,1986,14(9):3841-3857.
Miller LK,Jewell JE,Browne D.Baculovirus induction of a DNA polymerase[J].J.Virol,1981,40(1):305 -308.
Monahan SJ,Barlam TF,Crumpacker CS,et al.Two regions of the herpes simplex virus type 1 UL42 protein are required for its functional interaction with the viral DNA polymerase [J].J.Virol.,1993,67(10):5922-5931.
Moscardi F.Assessment of the application of baculoviruses for control of Lepidoptera[J].Annu.Rev.Entomol.,1999,44,257-289.
Ohresser M,Morin N,Cerutti M,et al.Temporal regulation of a complex and unconventional promoter by viral products[J].J.Virol.,1994,68(4):2589-2597.
Olson VA,Wetter JA,Friesen PD.Baculovirus transregulator IE1 requires a dimeric nuclear localization element for nuclear import and promoter activation [J].J.Virol.,2002,76(18):9505-9515.
Quimby BB,Dasso M.The small GTPase Ran:interpreting the signs[J].Curr.Opin.Cell Biol.,2003,15(3):338-344.
Sahdev S,Saini KS,Hasnain SE.Baculovirus P35 protein:an overview of its applications across multiple therapeutic and biotechnological arenas[J].Biotechnol.Prog.,2010,26(2):301-312.
Shields JM,Yang VW.Two potent nuclear localization signals in the gut-enriched Kruppel-like factor define a subfamily of closely related Kruppel proteins[J].J.Biol.Chem.,1997,272(29):18504-18507.
Stewart TM,Huijskens I,Willis LG,et al.The Autographa californica multiple nucleopolyhedrovirus ie0-ie1 gene complex is essential for wild-type virus replication,but either IE0 or IE1 can support virus growth[J].J.Virol.,2005,79(8):4619-4629.
Szewczyk B,Hoyos-Carvajal L,Paluszek M,et al.Baculovirusesremerging biopesticides[J].Biotechnol.Adv.,2006,24(2):143-160.
Terrell SL and Coen DM.The pre-NH(2)-terminal domain of the herpes simplex virus 1 DNA polymerase catalytic subunit is required for efficient viral replication [J].J.Virol.,2012,86(20):11057-11065.
Thumbi DK,Arif BM,Krell PJ.Susceptibility of Autographa californica multiple nucleopolyhedrovirus to inhibitors of DNA replication[J].J.Virol.Methods,2007,146(1-2):209-217.
Tian W,Hwang YT,Lu Q,et al.Finger domain mutation affects enzyme activity,DNA replication efficiency,and fidelity of an exonuclease-deficient DNA polymerase of herpes simplex virus type 1 [J].J.Virol.,2009,83(14):7194-7201.
Tomalski MD, Wu JG, Miller LK. The location, sequence,transcription,and regulation of a baculovirus DNA polymerase gene[J].J.Virol.,1988,167(2),591-600.
Vicente T,Roldao A,Peixoto C,et al.Large-scale production and purification of VLP - based vaccines[J].Invertebr Pathol.,2011,107:S42-S48.
Wang Y,Wang Q,Liang C,et al.Autographa californica multiple nucleopolyhedrovirus nucleocapsid protein BV/ODV-C42 mediates the nuclear entry of P78/83 [J].J.Virol.,2008,82(9):4554-4561.
Wu J,Zhou L,Tonissen K,et al.The quaking I-5 protein(QKI-5)has a novel nuclear localization signal and shuttles between the nucleus and the cytoplasm [J]. J.Biol.Chem.,1999,274(41):29202-29210.
Yu M,Carstens EB.Identification of a domain of the baculovirus Autographa californica multiple nucleopolyhedrovirus single-strand DNA-binding protein LEF-3 essential for viral DNA replication[J].J.Virol.,2010,84(12):,6153-6162.
Yu M, Carstens EB. Choristoneura fumiferana multiple nucleopolyhedrovirus LEF-3-P143 complex can complement DNA replication and budded virus in an AcMNPV LEF-3-P143 double knockout bacmid [J].J.Gen.Virol.,2012,93(Pt2):383-388.
