基于生物信息学方法挖掘奶山羊miRNAs研究
2015-11-11
基于生物信息学方法挖掘奶山羊miRNAs研究
曲波,甄贞,仇有文,袁肖寒,王春梅
(东北农业大学生命科学学院,哈尔滨150030)
摘要:microRNAs(miRNAs)是长约22 nt的内源非编码小分子RNA,在转录后基因调控中发挥重要作用。奶山羊是具有重要经济价值的产乳动物,有关奶山羊miRNAs研究相对匮乏,识别和鉴定新的奶山羊miRNA至关重要。文章以与奶山羊高度同源的绵羊基因组为参考数据库,应用生物信息学方法得到101条新的奶山羊miRNAs序列,并对其进行序列特性分析,为今后基因组信息不全物种的miRNAs挖掘与鉴定提供参考。
关键词:miRNAs;奶山羊;绵羊基因组;生物信息学方法
网络出版时间2015-1-12 9:53:07
[URL]http://www.cnki.net/kcms/detail/23.1391.S.20150112.0953.007.html
曲波,甄贞,仇有文,等.基于生物信息学方法挖掘奶山羊miRNAs研究[J].东北农业大学学报, 2015, 46(1): 86-93.
miRNA(microRNA)是一类长约22 nt的内源非编码小分子RNA,通过与靶基因互补发挥转录后水平的负调控作用,广泛参与细胞发育、分化、增殖、凋亡、代谢、肿瘤转移等多种生物学过程[1]。奶山羊是最早被人类驯化的动物之一,但其基因组测序工作却远落后于其他家畜,直到2012年底,山羊基因组序列组装才初步完成,这也使其miRNAs研究工作极大受限。迄今有关奶山羊miR⁃ NAs研究相对较少,miRBase数据库中尚未收录相关miRNAs信息[2]。因此,挖掘新的奶山羊miRNAs具有重要理论和现实意义。
尽管山羊基因组序列信息仍不完整,有关奶山羊miRNAs挖掘工作开展,但都以新一代测序为主,集中于乳腺、睾丸、肌肉等组织特异miRNAs的鉴定。本研究应用生物信息学方法,以与奶山羊高度同源的绵羊基因组为参考比对数据库,挖掘新的奶山羊miRNAs,并对其进行序列特性分析,为进一步研究奶山羊miRNAs及其调控机制和功能提供基础。建立一套适合基因组信息不全的物种miRNAs挖掘及分析方法,对预测、筛选和鉴定其他物种miRNAs具有指导和借鉴意义。
1 材料与方法
1.1相关数据库
1.1.1本研究涉及的相关数据库
NCBI:http://www.ncbi.nlm.nih.gov;miRBase:http://www.mirbase.org/;
UCSC:http://genome.ucsc.edu。
1.1.2构建miRNAs参考数据库
本研究采用已报道的主要哺乳动物成熟miRNAs序列作为参考数据库。首先从miRBase(Release 20)数据库获取绵羊(Ovis)、牛(Bos)、猪(Sus)、狗(Canis)、小鼠(Mus)、大鼠(Rat)和人(Homo)等主要哺乳动物miRNAs成熟序列7 127条,去除重复序列后,共得到5 485条非冗余miRNAs序列,作为miRNAs参考数据库。
1.1.3构建绵羊基因组比对数据库
从UCSC数据库获取绵羊基因组序列(Oar3.1/oviAri3),本地解压缩,构建比对数据库。
1.2相关应用软件
1.2.1 Blast
在线版:http://blast.ncbi.nlm.nih.gov/Blast.cgi;
本地版:ftp://ftp.ncbi.nlm.nih.gov/blast/executa⁃bles/blast+,版本为2.2.28。
1.2.2 RNAFold
在线版:http://rna.tbi.univie.ac.at/cgi-bin/RNA fold. cgi;
本地版: http://rna.tbi.univie.ac.at/,版本为1.8.4。
1.2.3 MiPred
在线版:http://www.bioinf.seu.edu.cn/miRNA/。
1.3奶山羊miRNAs预测流程
根据miRNAs保守性原则,本研究采用同源搜索方法进行奶山羊miRNAs挖掘筛选,主要流程见图1。包括①获取已知主要哺乳动物miRNAs序列,去冗余,构建miRNAs参考数据库;②将miR⁃NAs参考数据库与绵羊基因组进行本地Blastn比对,字长为7,E值为10;③将与已知miRNAs错配数<4的序列提取出来,与非冗余蛋白质数据库进行比对,去除蛋白编码序列;④在绵羊基因组中,将剩余的候选序列匹配区域上下游各延伸200 nt,作为候选pre-miRNAs序列;⑤采用RNAFold 1.8.4分析候选pre-miRNAs二级折叠结构,筛选出具有发夹型二级结构、最小折叠自由能(Minimal fold free energy, MFE)<-15 kcal·mol-1、成熟miRNA序列中至少有16 bp位于前体二级结构中的茎部,且不含有环或缺口的为最终候选pre-miRNAs序列;⑥采用MiPed对已筛选的候选pre-miRNAs进行筛选验证,预测模型为随机森林法(Random forest)。

图1 奶山羊miRNAs预测流程Fig. 1 Overview of in silico detection of potential miRNAs in dairy goat
2 结果与分析
2.1奶山羊miRNAs的预测挖掘
按照图1所示流程,利用miRNA在不同物种间保守性和前体二级结构特点,通过同源片段搜索的方法寻找奶山羊miRNAs,共发现101条与其他物种同源的miRNAs,结果见表1。
2.1.1绵羊基因组与miRNAs参考数据库比对结果
将含有5 485条非冗余miRNAs序列的参考数据库,应用Blast(2.2.28)本地版与绵羊基因组进行比对,得到53 522条与已知miRNAs序列错配碱基数<4、比对长度≥18的候选序列。
2.1.2候选序列中蛋白质编码序列的去除
将候选序列转换为bed格式,在UCSC上调取上下游各200 nt作为miRNA前体二级结构分析序列,将这些序列去冗余后,与蛋白数据库比对,去除编码蛋白序列,得到8 407条候选序列。
2.1.3二级结构预测与MiPed筛选鉴定
采用RNAFold 1.8.4预测候选序列的二级结构,并计算其MFE,共有973条序列满足筛选条件。这些序列经MiPred进行进一步的预测筛选,结果共得到101条新的miRNAs(见表1)。
部分奶山羊预测miRNAs前体的二级结构见图2。
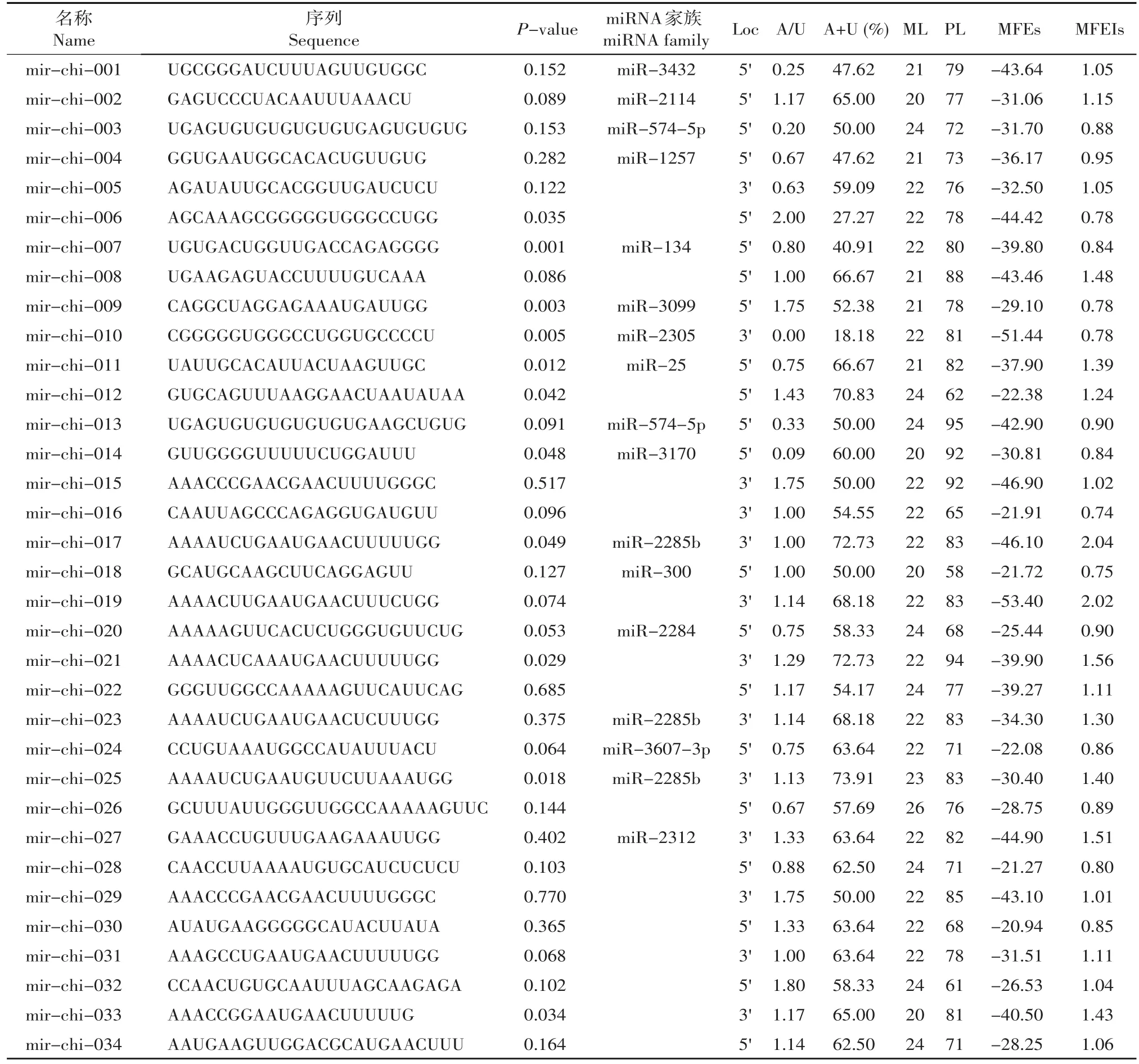
表1 奶山羊乳腺miRNA预测结果Table 1 Potential miRNAs of mammary gland in dairy goat
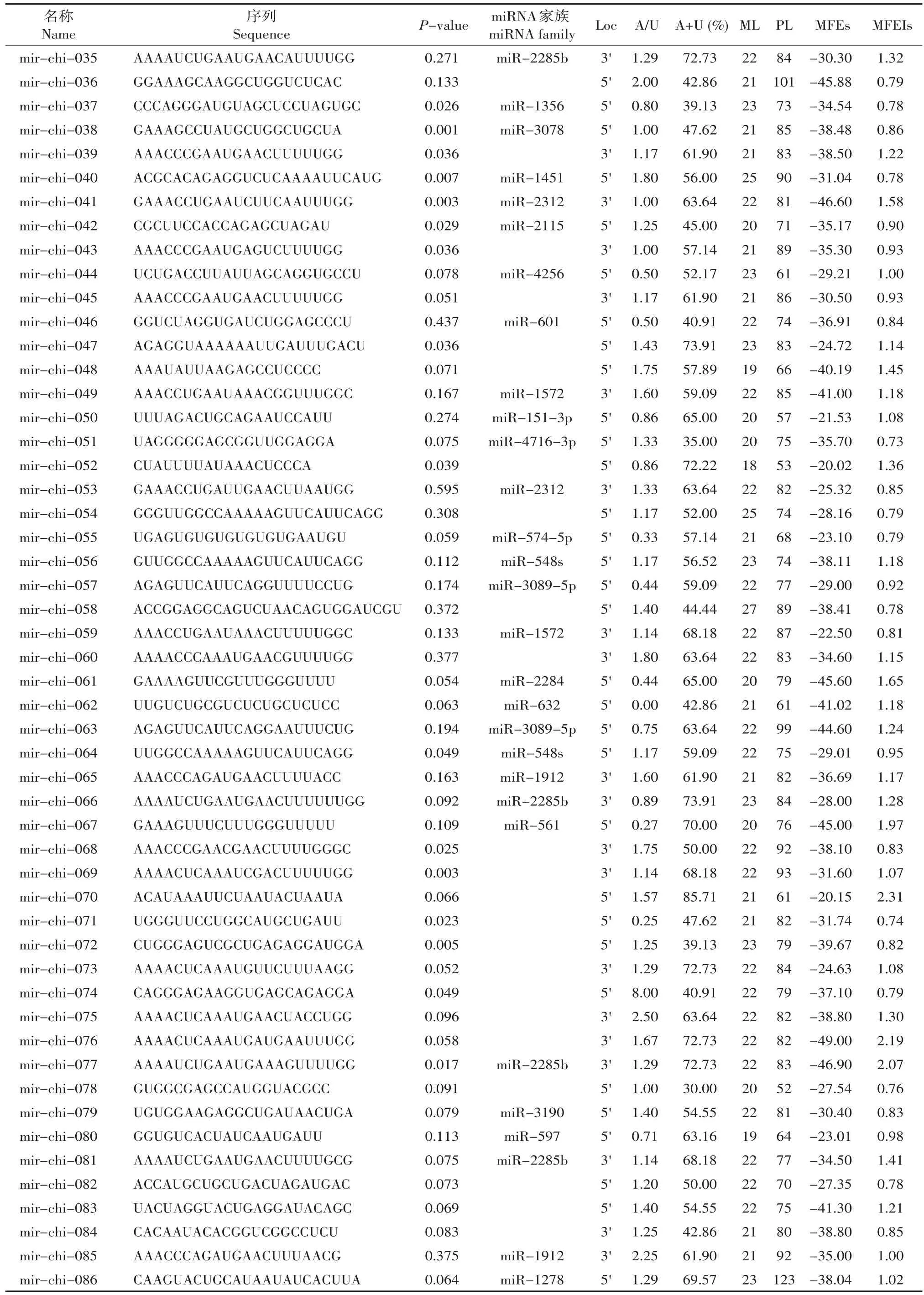
续表

续表

图2 奶山羊miRNAs前体的二级结构Fig. 2 Predicted stem-loop structures of some newly identified pre-miRNAs in dairy goat
2.2奶山羊新miRNAs序列特性分析
近来有关miRNAs挖掘和鉴定的研究中,miRNAs序列特征分析逐渐成为研究重点。本研究也对新预测的奶山羊miRNAs序列特性进行详细分析,包括序列长度、碱基偏好性、MFE及miRNA家族等方面(见表1、2),进一步验证miRNAs预测的正确性。

表2 奶山羊pre-miRNAs主要序列特性Table 2 Major sequence characteristics of the newlyidentified pre-miRNAs in dairy goat
2.2.1序列长度与碱基偏好性
从表1、2可知,新预测的101条miRNAs中,成熟miRNAs序列长度在18~27 nt,平均为(22±2)nt;pre-miRNAs序列长度为52~123 nt,平均为(79±10)nt。成熟miRNAs序列中,长度为22 nt的所占比例最高,达41.58%(见图3A);pre-miRNAs序列中,长度在80~89 nt比例最大,为38.61%(见图3B)。值得注意的是,有59.41%(60个)miRNAs序列位于pre-miRNAs序列5'端,其余40.59%(41个)miRNAs序列定位于3'端。
一般情况下,动物miRNAs前体序列中4种碱基比例不同。由表2可知,本研究中A、U、G、C含量亦如此。含量最高为A(29.22%±10.73%),其次为U(28.65%±9.32%)和G(24.71%±11.73%),最低为C(17.42%±8.23%)。显然,新预测的奶山羊miRNAs前体序列中A+U(57.87%±12.48%)含量大大超过G+C(42.13%±12.48%)。此外,A/U和G/C平均值分别为(1.19±0.86)和(1.41±1.04),表明奶山羊miRNAs前体序列中A和G含量要相对高于U 和C。
成熟miRNAs序列各个位置的碱基偏好性也是miRNAs序列特性分析的主要指标之一。由图4可知,在新预测的奶山羊miRNAs序列中,在首位至第4位,A出现的频率最高,U最低;在第15~20位,U出现的频率最高;在第23位之后,C出现的几率最低。
2.2.2 MFE和MFEI
由表1可知,新预测的奶山羊miRNAs中,MFE在(-20.02~-58.87)kcal·mol-1,平均值为(-34.93± 8.47)kcal·mol-1。MFEI在0.73~2.19,平均(1.12± 0.36)。其中,MFEI>0.85的miRNAs序列比例高达82.18%。
2.2.3 miRNA家族
根据miRNA家族分类原则和种子序列(Seed sequence)鉴定,新预测的101个奶山羊miRNAs中,共有52条miRNAs分属于29个miRNA家族(见表1)。其中,miR-2285b家族数量最多,共有8条新预测的miRNAs。miR-2312家族次之,有5 条miRNAs。

图3 奶山羊成熟miRNAs(A)和pre-miRNAs(B)序列长度分布Fig. 3 Length distribution of novel mature miRNAs (A) and pre-miRNAs (B) in dairy goat

图4 奶山羊成熟miRNAs序列各个位置碱基偏好性分布Fig. 4 Distribution of base composition at each position in mature miRNA sequences of dairy goat
3 讨论
3.1奶山羊新miRNAs的预测挖掘
奶山羊是产乳动物,但miRNAs研究相对滞后。直到2012年底,山羊基因组序列组装才初步完成,该研究整合使用NGS和最新的DNA单分子光学作图技术,成功克服山羊基因组的组装难题,提供首个小型反刍动物参考基因组,目前正在进行基因组注释工作[3]。
miRNAs挖掘鉴定主要有三类方法,①传统克隆方法,精准度相对较高,但不能检测低丰度的miRNAs,且需要相对完整的基因组信息;②新一代测序技术(Next-generation sequencing, NGS),实现高通量筛选新的miRNAs,但费用高,需要处理庞大的数据流,目前已报道的奶山羊乳腺、睾丸和肌肉等特异miRNAs挖掘均采用此方法[4];③生物信息学方法、高效、应用广,并为NGS的高通量筛选提供技术保证,已成为寻找和鉴定miRNAs及靶基因的主要方法[5]。
一般认为,山羊与绵羊具有极高同源性(> 90%),只是由于罗伯逊易位造成它们染色体组型出现细微差异[6]。由于目前山羊基因组信息还不完整,本研究以绵羊基因组为参考序列,采用生物信息学方法进行奶山羊miRNAs挖掘,共获得101条新的奶山羊miRNAs序列。众所周知,生物信息学方法预测结果都有一定的假阳性[7],在后续研究中,将对新预测的miRNAs序列进行生物学试验验证,进一步鉴定出新的奶山羊miRNAs序列。
Dong等研究表明,山羊与牛同源性比绵羊高[3],但考虑到山羊与绵羊同属于羊亚科,亲缘性可能更近,最终选择绵羊作为参考基因组。
3.2奶山羊新miRNAs序列特性分析
目前,miRNAs序列特征分析正成为miRNAs鉴定重要指标之一。本研究中,pre- miRNAs平均序列长度为(79±10)nt,并且93.07%序列长度为60~99 nt;成熟miRNAs平均序列长度为(22±2)nt,73.27%序列长度为20~33 nt,这与miRNAs鉴定结果一致,包括猪[8]、绵羊[9]、马[10]、大豆[11]、玉米[12]等物种。此外,pre-miRNAs前体序列中A+U含量达到57.87%±12.48%,这也使pre-miRNAs二级结构不稳定,更易形成RNA诱导沉默复合体(RNA-induced silencing complex, RISC)[10]。
RNA二级结构的稳定性决定于其最小折叠自由能(MFE)。通常情况下,MFE越小,RNA越稳定。与其他RNA分子相比,pre- miRNAs的MFE值要更小。由于pre-miRNAs的序列长度不同,提出修正的最小折叠自由能(Adjusted minimal fold free energy, AMFE)和MFEI两个指标,修正序列长度对MFE影响,逐渐成为鉴定pre-miRNAs主要标准[11]。本研究MFE平均值为(-34.93±8.47)kcal·mol-1,MFEI平均为(1.12±0.36),且大部分序列MFEI超过0.85,均符合miRNA二级结构稳定性要求,这一结果与Zhou报道一致[8-12]。
miRBase已收录几万条miRNAs序列,普遍存在于从植物、线虫到人类细胞中,miRNAs在不同物种普遍存在,揭示其在生物进化过程中保守趋势,高度保守的miRNA在生命活动中发挥重要调节作用[13]。miRNAs被分成不同miRNA家族。miRNA家族成员都具有相同的种子序列区,即成熟miR⁃NA 5'端第2~7位碱基序列[14]。种子序列区是miR⁃NAs靶基因识别的主要结合位点,种子区内一个碱基变化将导致整个miRNA功能改变,因此对新预测miRNAs进行miRNA家族分类至关重要。本研究应用种子区特征鉴定法,发现共有52条miRNAs分属于29个miRNA家族,具体靶基因及功能有待进一步研究。
4 结论
miRNA生物信息学预测方法根据pre-miRNA独特的序列结构特征及保守性原则,依赖于研究物种的基因组信息,对山羊没有完整参考基因组非模式动物,传统生物信息学方法有一定困难。本研究以与奶山羊高度同源的绵羊基因组为参考数据库,应用生物信息学方法得到101条新的奶山羊miR⁃NAs序列,为今后基因组信息不全物种的miRNAs挖掘与鉴定提供参考。在后续研究中,新miRNAs进行生物学鉴定及靶基因的预测鉴定与功能分析,为奶山羊miRNAs研究提供理论基础。
[参考文献]
[1]Bartel D P. MicroRNAs: Genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281-297.
[2]Kozomara A, Griffiths-Jones S. miRBase: Integrating microRNA annotation and deep-sequencing data[J]. Nucleic Acids Res, 2011, 39: 152-157.
[3]Dong Y, Xie M, Jiang Y, et al. Sequencing and automated wholegenome optical mapping of the genome of a domestic goat (Capra hircus)[J]. Nat Biotechnol, 2013, 31(2): 135-141.
[4]金晓露,杨建香,李真,等.乳腺发育及泌乳相关miRNA研究进展[J].遗传, 2013, 35(6): 695-702.
[5]Huang Y, Zou Q, Wang S P, et al. The discovery approaches and detection methods of microRNAs[J]. Mol Biol Rep, 2011, 38(6): 4125-4135.
[6]Kaftanovskaya H M, Serov O L. High-resolution GTG-banded chromosomes of cattle, sheep, and goat: A comparative study[J]. J Hered, 1994, 85(5): 395-400.
[7]Mendes N D, Freitas A T, Sagot M F. Current tools for the identifi⁃cation of miRNA genes and their targets[J]. Nucleic Acids Res, 2009, 37(8): 2419-2433.
[8]Zhou B, Liu H L. Computational identification of new porcine mi⁃croRNAs and their targets[J]. Anim Sci J, 2010, 81(3): 290-296.
[9]Barozai M Y. The novel 172 sheep (Ovis aries) microRNAs and their targets[J]. Mol Biol Rep, 2012, 39(5): 6259-6266.
[10]Zhou M, Wang Q, Sun J, et al. In silico detection and characteris⁃tics of novel microRNA genes in the Equus caballus[J]. Genomics, 2009, 94(2): 125-131.
[11]Frazier T P, Zhang B. Identification of plant microRNAs using ex⁃pressed sequence tag analysis[J]. Methods Mol Biol, 2011, 678: 13-25.
[12]Zhang B H, Pan X P, Cox S B, et al. Evidence that miRNAs are different from other RNAs[J]. Cell Mol Life Sci, 2006, 63(2): 246-254.
[13]Friedman R C, Farh K K, Burge C B, et al. Most mammalian mRNAs are conserved targets of microRNAs[J]. Genome Res, 2009, 19(1): 92-105.
[14]Lewis B P, Burge C B, Bartel D P. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets[J]. Cell, 2005, 120: 15-20.
Qu Bo, Zhen Zhen, Qiu Youwen, et al. In silico detection and characteristics of miRNAs in dairy goat[J]. Journal of Northeast Agricultural University, 2015, 46(1): 86-93. (in Chinese with English abstract)/QU Bo,
In silico detection and characteristics of miRNAs in dairy goat
ZHEN Zhen, QIU Youwen, YUAN Xiaohan, WANG Chunmei
(School of Life Sciences, Northeast Agricultural University, Harbin 150030, China)
Abstract:microRNAs (miRNAs) are a large class of endogenous non-coding small RNAs that average 22 nucleotides (nt) in length, which play important roles in post-transcriptional gene regulation because they can negatively regulate gene expression. Dairy goat is a milk producing animal with economic importance. The studies on miRNAs in dairy goat are relatively lack and then detecting and identifying the new miRNAs of dairy goat is very important. In this study, 101 miRNAs of dairy goat were obtained using bioinformatics approach based on sheep genome, which was highly homologous with goat. Finally, detailed analysis of sequence characteristics in novel miRNAs of goat were carried out. This study would provide a reference for further identification of miRNAs in animals without complete genome.
Key words:miRNAs; dairy goat; sheep genome; bioinformatics approach
作者简介:曲波(1977-),男,副研究员,博士,研究方向为泌乳生物学与乳腺功能调控。E-mail: qb5172@neau.edu.cn
基金项目:国家自然科学基金项目(31100959);黑龙江省博士后启动基金项目(LBH-Q11169)
收稿日期:2014-06-27
文章编号:1005-9369(2015)01-0086-08
文献标志码:A
中图分类号:Q492.7;S858.2
