植物细胞核雄性不育相关bHLH转录因子研究进展
2015-10-29刘永明张玲周建瑜曹墨菊
刘永明,张玲,周建瑜,曹墨菊
植物细胞核雄性不育相关bHLH转录因子研究进展
刘永明1,张玲1,周建瑜2,曹墨菊1
1. 四川农业大学玉米研究所,教育部作物基因资源与遗传改良重点实验室,农业部西南玉米生物学及遗传育种重点实验室,成都 611130;2. 四川农业大学动物医学院,雅安 625014
雄性不育广泛存在于种子植物中。植物雄性不育不仅是植物生殖发育研究的重要内容,同时也可作为杂种优势利用的有效工具,因而具有重要的理论和应用价值。bHLH转录因子家族是植物中成员最多的转录因子家族,在植株的整个生长发育过程中起着重要的调控作用。本文介绍了拟南芥、水稻、玉米等几种重要模式植物bHLH转录因子调控雄蕊发育的作用机制,并重点阐述其功能异常引起细胞核雄性不育的分子机制,以期为作物育种与理论研究提供参考。
雄性不育;bHLH转录因子;细胞程序性死亡;转录组测序
植物雄性不育是指在有性生殖过程中雄蕊发育异常,不能正常产生有功能花粉的现象,包括由线粒体基因与核基因互作不协调引起的细胞质雄性不育(Cytoplasmic male sterility, CMS)及由核基因单独控制的细胞核不育(Genic male sterility, GMS)。植物细胞质雄性不育应用于种子生产,必须实现三系配套,且大面积推广单一不育胞质配制的杂交种可能导致优势生理小种的出现。细胞核雄性不育具有败育彻底、恢复源广的优势,且不存在不育胞质单一化使用的遗传脆弱性。SPT(Seed production technology)技术的出现有效地解决了隐性核雄性不育系的保持和繁殖的问题,因此细胞核雄性不育在未来不育化制种中将发挥重要作用[1]。植物bHLHs (Basic helix-loop-helix proteins)转录因子家族的部分成员可通过激活或抑制一系列花发育相关基因的时空特异性表达调控植物的雄花发育。目前在拟南芥()、水稻()、玉米()等植物中发现某些雄性不育的发生与特异bHLH转录因子的功能异常有关。因此,了解bHLH转录因子在植物花粉形成过程的作用机制对探讨植物雄性不育的发生机理以及促进作物不育化制种的进程具有重要的理论和现实意义。
1 植物细胞核雄性不育
植物雄性不育在杂种优势利用上具有重要应用价值,因此关于雄性不育的发生机理长期以来一直是人们研究的热点。目前在玉米、水稻、番茄()和大麦()中已分别发现了60、63、55和50个核不育基因[2],这些基因参与植物花粉发育的各个环节,包括减数分裂、胼胝质代谢、绒毡层发育、花粉壁发育及花药开裂等[3],表明在雄蕊发育中任何一个环节的异常可能都会导致雄性不育,因而细胞核雄性不育的发生与小孢子发育相关基因的功能异常紧密相关。此外,最新研究表明植物细胞核雄性不育的发生也可能与非编码RNA的作用有关。Ding等[4]对水稻光敏不育材料农垦58S研究发现,光敏突变体中不育基因发生了G到C的单碱基突变,致使其转录的非编码RNA启动子出现甲基化并导致其转录减少,而足够剂量的RNA转录物对长日照条件下植株花粉产生是必须的,RNA转录量的降低最终导致花药过早地程序化死亡,产生光敏型雄性不育植株。Zhou等[5]对水稻温敏不育体安农S-1的研究表明,高温可破坏水稻花粉母细胞泛素的动态平衡,而非编码RNA可维持细胞内的泛素平衡,其温敏突变体由于小RNA功能丧失导致细胞内泛素平衡被破坏,不可溶的泛素化蛋白增加,诱发花粉母细胞液泡化,最终导致植株败育。
2 bHLH转录因子
转录因子是一类能与真核基因5′端上游特定序列进行专一性结合从而保证目的基因时空特异表达的蛋白质分子。植物中转录因子主要包括bHLH、MYB、WRKY等家族,Jin等[6]利用软件对植物中83个物种的转录因子家族成员进行预测,共得到 11 428个bHLH类转录因子,同时bHLH家族是所有转录因子家族中数量最多的一类,这暗示了其对于植物生长发育具有重要意义。目前对植物bHLH转录因子的研究主要包括两个方面:一是利用高通量测序数据对bHLH转录因子进行鉴定,越来越多物种的bHLH转录因子在全基因组范围内得到鉴定(部分植物的bHLH转录因子数量见表1);二是利用各类突变体对bHLH转录因子在植物发育中的功能进行解析,现已发现bHLH转录因子主要参与种子萌发[7,8]、分枝发育[9]、花器官形成和果实发育[10,11]、激素应答[12,13]和逆境胁迫响应[14,15]等过程。

表1 植物中已鉴定的bHLH转录因子数量
bHLH 基序包含约60个氨基酸,由N端富含碱性氨基酸的DNA结合区和C端的螺旋-环-螺旋组成,其中N端的碱性区域可与下游基因启动子区的E-box(5′-CANNTG-3′)元件结合,并对目的基因的表达进行特异调控,而C端的螺旋-环-螺旋区域负责与其他bHLH转录因子形成同源或者异源二聚体行使功能[31]。bHLH蛋白在生物体内的功能与其空间结构密切相关。Ko等[32]对水稻细胞核雄性不育的两个bHLH转录因子bHLH142、TDR1研究发现,这两个bHLH蛋白单独作用时不能调控下游基因的表达,而只有当编码蛋白与TDR1蛋白发生互作形成二聚体之后才能促进下游靶基因的表达。动物中也发现类似现象,Chang等[33]对人类和果蝇的促进上皮细胞间质转型(Epithelial-mesenchymal transition, EMT)的bHLH蛋白TWIST研究发现,只有当TWIST1与TWIST47形成特异的二聚体结构才能与下游基因的E-box结合进而执行其功能。由此可见,在探究某一bHLH蛋白与其下游靶基因是否具有直接互作时,需要考虑bHLH蛋白须以二聚体行使功能这一特性。同时,bHLH蛋白对下游靶基因的识别也可能受到其他因子的调控,如对bHLH转录因子活性起负调节作用的DNA结合抑制因子(Inhibitor of DNA binding, ID)。ID可与bHLH转录因子结合形成异二聚体,但因其本身不包含与DNA 结合必需的碱性氨基酸序列,致使异二聚体无法与靶基因启动子区域的E-box结合,从而阻止bHLH转录因子对下游基因的转录调控[34,35](图1)。同时,ID蛋白的相互作用对bHLH转录因子的活性起到重要的调控作用,Sharma等[36]发现ID4可与bHLH转录抑制因子ID1、ID2、ID3形成异二聚体来阻止转录抑制因子的功能,并促进bHLH因子的转录激活活性。

图1 ID2对bHLH转录因子的转录激活活性抑制作用(引自文献[35])
A:两个bHLH蛋白形成二聚体,并与基因启动子结合从而促进的表达;B:ID2蛋白与bHLH蛋白形成异二聚体,由于ID2不含有与DNA结合的碱性氨基酸序列,形成的异二聚体不能与下游启动子序列结合,无法促进其转录。
3 bHLH转录因子与植物雄性不育
目前已报道的与花粉形成相关的bHLH转录因子大部分参与花药绒毡层的发育或小孢子发育。花药绒毡层对花粉的生长发育至关重要,除了为小孢子发育提供营养,绒毡层分泌的胼胝质酶能够适时地分解花粉母细胞和四分体的胼胝质壁,以保证小孢子彼此分离;同时绒毡层还能分泌一些物质参与授粉过程中花粉与柱头细胞的识别[37]。已有研究表明,bHLH转录因子功能异常引起的不育植株在败育特征上具有一定的相似性,即雄性不育突变体往往表现出绒毡层细胞提前或延迟降解,致使花粉形成受阻表现败育(表2)。目前已被鉴定出的参与小孢子发育的bHLH转录因子主要集中在拟南芥、水稻、玉米等模式植物中,在其他植物中参与植物小孢子发育的bHLH转录因子的报道较少。对花粉发育相关bHLH蛋白进行同源性分析,发现不同物种间参与小孢子发育的bHLH转录因子具有高度的同源性(图2)。由此可见,分析比较这些bHLH转录因子结构特点与功能特征,对于挖掘其他植物中参与花粉形成的bHLH转录因子的功能具有重要意义。

表2 植物细胞核雄性不育相关的bHLH转录因子
注:bHLH转录因子序号参考文献[18]。

图2 已鉴定的小孢子发育相关bHLH转录因子的进化关系
用NJ法、MEGA5.0软件构建进化树,Bootstrap为1000次重复。
3.1 bHLH转录因子与拟南芥雄性不育
目前在拟南芥全基因组内已经鉴定出167个bHLH转录因子[16]。Dukowic等[38]对拟南芥和玉米的花粉母细胞、成熟花药及种子的转录组进行比较分析,发现14个拟南芥bHLH基因和3个玉米bHLH基因在减数分裂时期表达上调,并且玉米中3个上调表达的bHLH基因在拟南芥中的14个上调表达的bHLH基因中都有对应的同源基因。Yang等[39]利用单细胞测序技术对拟南芥花粉母细胞进行转录组分析,鉴定了在花粉母细胞时期特异表达的转录因子,其中bHLH家族成员占有较大比例。Zhang等[40]对拟南芥花发育早、中、晚3个时期转录组进行分析比较,也发现bHLH转录因子在所有差异表达的转录因子中占有较大比重,同时这些差异表达的bHLH转录因子主要集中在雄蕊发育的花序分生组织(Inflorescent meristem, IM),说明bHLH转录因子对雄蕊早期发育起到重要的调控作用。
Sorensen等[41]首先在由T-DNA插入构建的拟南芥突变体库中鉴定出一个bHLH转录因子功能异常引起的雄性不育突变体,该雄性不育突变体表现出绒毡层发育不正常、小孢子提前降解等特征,进一步分析发现()编码蛋白在花药绒毡层的发育和小孢子母细胞减数分裂中起重要调控作用。Thorstensen等[42]通过酵母双杂交发现AMS与拟南芥花药发育有关的蛋白ASHR3存在互作,并推测它们之间的互作对雄蕊发育具有重要意义。Xu等[43]通过基因表达谱分析并结合细胞生物学、遗传学、生物化学等手段鉴定了花粉形成过程中受AMS蛋白调控的23个基因,这些基因分别参与花粉壁形成中的胼胝质及初生外壁的合成和孢粉素合成、运输、沉积等一系列代谢过程,表明在花粉壁发育中发挥关键作用。Ma等[44]对野生型和不育突变体的未成熟花药进行转录组分析,筛选到参与代谢、转运、泛素化及抗逆等途径的1368个差异表达基因,表明基因还可能在植物发育的其他生理和代谢过程中发挥作用。Zhang等[45]在拟南芥Ds转座子插入突变体库中鉴定得到一个雄性不育突变体,该突变体绒毡层细胞高度液泡化并缺少正常细胞质,虽然其性母细胞能正常进行减数分裂I,但由于没有胼胝质,不能完成减数分裂就解体,最终不能形成花粉而导致不育。DYT1是一个bHLH转录因子,可调控参与花药绒毡层的分化和发育过程中的基因表达。例如,在突变体中很多绒毡层特异表达的基因表达量相对于野生型显著降低[45],而Feng等[46]利用转录组测序发现如、、等很多花药发育基因都受到DYT1的调控。此外,DYT1还可与R2R3 MYB转录因子TDF1互作共同参与拟南芥绒毡层发育及花粉壁的形成[47]。Zhu等[48]鉴定了3个与DYT1存在互作的bHLH转录因子AtbHLH10、AtbHLH89和AtbHLH91,并发现这3个bHLH转录因子对雄花花药的发育存在功能互补现象,单突变株与野生型一致,但双突变及三突变体会表现出雄性不育现象。
bHLH基因编码蛋白还可以通过调节植物信号物质的合成与应答来参与雄蕊发育。茉莉酸(Jasmonic acid, JA)可诱导和协调花药开裂和释放花粉等过程,JA合成或信号转导异常的植株大多也会出现雄性不育或育性降低。Nakata等[13,49]在拟南芥中鉴定出参与茉莉酸信号传导的3个bHLH转录因子JAM1JAM2和JAM3,并分别将3个转录因子进行过表达,转基因植株都表现出雄蕊育性降低,暗示这3个bHLH转录因子在拟南芥雄蕊发育中起负调控作用。Figueroa等[50]发现MYC5(AtbHLH28)能参与雄蕊发育中茉莉酸应答途径,同时为避免转录因子同源家族基因功能的补偿,利用嵌合阻遏基因沉默技术(The chimeric repressor silencing technology, CRES-T)抑制下游靶基因的表达,结果发现转基因植株雄蕊表现出花丝不伸长、花药不开裂以及花粉无活力等败育特征。Xing等[51]在拟南芥中发现1个参与油菜素内酯信号传递的bHLH转录因子BIM1,虽然其突变体与野生型在雄蕊育性表现上基本一致,但与花发育相关基因形成的双突变体相对于半不育的单突变体,表现出花药明显异常、自交结实率明显降低,说明BIM1可与SPL8发生协同作用进而调控拟南芥雄蕊的发育。
3.2 bHLH转录因子与水稻雄性不育
基于水稻全基因测序数据并通过软件预测,目前在水稻全基因组中共鉴定出178个bHLH转录因子[16],其中已报道参与小孢子发育的bHLH转录因子包括UDT1(OsbHLH164)TDR1(OsbHLH5)EAT1/DTD(OsbHLH141)和bHLH142/TIP2(OsbHLH142)等,它们大部分在绒毡层细胞程序性死亡(Programmed cell death, PCD)和花粉形成过程中起关键作用,其功能异常往往会引起雄性不育。同时,研究表明这些bHLH转录因子的相互作用在水稻花粉形成过程中具有重要作用(图3),他们的功能异常所引起的细胞核雄性不育在细胞学水平上也具有一定的相似性(表2)。
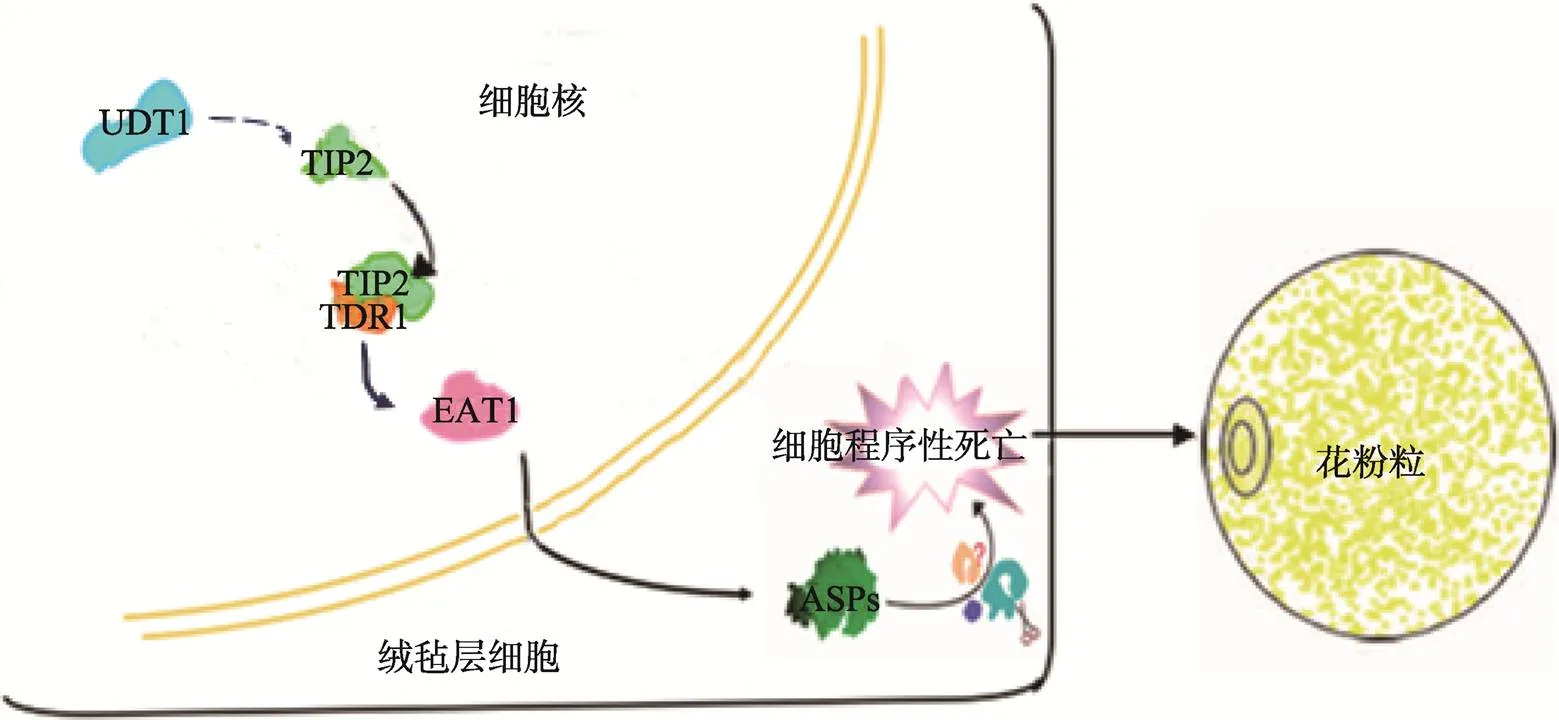
图3 bHLH转录因子在水稻花粉形成过程中调控网络(参考文献[56]修改绘制)
Jung等[52]在水稻中鉴定了1个bHLH转录因子UDT1,其T-DNA插入形成的不育突变体主要表现出:植株雄蕊中周缘细胞异常分化,绒毡层液泡化,中层不能降解,小孢子发育受阻,花粉囊内无法形成花粉粒,最终引起植株不育。水稻中bHLH蛋白TDR1对绒毡层细胞降解和花药发育是必需的,其在花药发育早期的绒毡层细胞中特异积累[53],并可对绒毡层降解过程中的半胱氨酸蛋白酶基因和蛋白酶抑制基因的表达水平进行调控。突变体绒毡层细胞不能及时降解,小孢子由于能量供应不足从四分体释放后即解体,从而表现植株败育[54]。水稻EAT1蛋白可与编码产物形成蛋白复合物,并调控下游诱导绒毡层细胞程序性死亡的和等基因的表达。水稻突变体败育特征与类似,虽然可通过减数分裂形成小孢子,但由于绒毡层细胞PCD延迟,使小孢子能量供应不足,从四分体释放后即被降解,最终无法形成有功能的花粉粒[55,56]。Fu等[57]通过60Co-γ射线进行辐射诱变获得一个水稻雄性不育突变体,Ko等[32]利用T-DNA插入突变技术获得水稻雄性不育突变体,研究发现这两个雄性不育突变体都是由于转录因子bHLH142功能异常引起。突变体花药壁包含未分化的表皮、纤维层、中层,并且绒毡层不能正常解体,最终由于没有正常的小孢子形成导致植株雄性不育。通过DAPI组织染色与基因表达模式分析验证了其主要在花药发育早期花药壁内3层细胞的分裂分化中起关键作用。Fu和Ko两个研究组同时发现bHLH142可调控和两个基因的表达,虽然bHLH142可与TDR1形成蛋白复合体促进绒毡层的细胞分化,但bHLH142并不能直接调控,而需要先与TDR1形成蛋白复合物,进而才能通过调控下游基因的表达以调节绒毡层的程序性死亡过程,进而参与花粉的正常发育。
目前鉴定的参与水稻花粉形成的4个bHLH转录因子均与绒毡层细胞的发育以及程序性死亡有关,其中UDT1居于上游,可调节基因的表达,而bHLH142与TDR1蛋白互作形成的复合体可对下游基因的表达进行调控,基因直接调控绒毡层PCD相关基因的表达保证小孢子正常的发育(图3),可见bHLH转录因子并不都是单独发挥功能,部分bHLH蛋白间的相互协作在花粉形成过程中发挥关键作用。水稻中4个与花粉形成相关bHLH转录因子的功能解析均得益于相关基因突变体的发现,但是针对某一特定或若干未知功能的bHLH基因进行功能解析较为困难,因此Crismani等[58]与Romero[59]等分别通过对水稻花药转录组及蛋白组进行测序鉴定出了一系列参与花粉母细胞减数分裂的bHLH转录因子,这对进一步解析bHLH转录因子与花粉形成之间的关系具有重要参考价值。
3.3 bHLH转录因子与玉米雄性不育
Jiang等[29]通过对玉米全基因组内各个转录因子家族进行预测,发现bHLH转录因子是玉米中数量最多的转录因子家族(8.35%),这暗示bHLH家族在玉米生长发育中的重要性。Zhang等[60]通过对小孢子发育过程中的5个时期进行转录组测序分析,鉴定了在各个时期特异表达的bHLH基因,发现随着小孢子的发育,这些bHLH基因的表达丰度也在不断变化。Dukowic等[61]通过对花粉母细胞前期I的花药、成熟花药及种子进行转录组测序,获得了在减数分裂中差异表达的bHLH基因,其中大部分bHLH基因相对于其他时期都表现为上调表达。这些结果表明bHLH转录因子对植物生殖发育的调控具有严格的时空特异性。
Moon等[62]克隆了玉米细胞核雄性不育基因,该基因编码1个bHLH蛋白,野生型中该基因通过促进绒毡层分化和抑制绒毡层细胞的平周分裂保证花药的正常形成,而其突变体绒毡层分化失败,绒毡层细胞通过多余的平周分裂产生了冗余的细胞层,导致小孢子发育受阻。Ren等[63]克隆了位于细胞核中的玉米C型细胞质雄性不育的育性恢复主效基因,其编码产物含有1个bHLH结构域,且不具有线粒体定位序列,表明作为一个转录因子可能是通过影响下游基因的表达,间接作用于线粒体中的不育基因,从而使C胞质育性恢复。
3.4 bHLH转录因子与其他植物雄性不育
除了上述模式植物,在其他植物中bHLH转录因子与花粉育性的关系也有报道。Liu等[64]在大白菜()中发现一个正向调控花粉形成的bHLH基因,其编码产物同小孢子减数分裂相关的BcSKP1蛋白间的互作参与大白菜雄蕊正常发育。进一步将转化至油菜()中并利用病毒诱导基因沉默技术(Virus-induced gene silencing, VIGS)抑制基因表达,转基因植株绒毡层细胞不能正常降解,小孢子发育受阻,表现雄性不育。Jeong等[65]从番茄中克隆一个在花粉母细胞和绒毡层中高表达bHLH基因Ms10其突变体花粉母细胞减数分裂异常不能形成四分体,绒毡层异常液泡化,最终导致雄性不育。通过转录组测序鉴定了35可能调控的246个基因,这些基因分别参与减数分裂、绒毡层发育、花粉壁形成等过程,表明Ms10在小孢子形成的减数分裂和绒毡层细胞程序性死亡过程中发挥关键调控功能。
4 结语与展望
目前在植物中已鉴定出部分参与植物小孢子形成的bHLH转录因子,这些bHLH转录因子主要调控参与花药绒毡层降解、孢粉素合成等过程相关基因的表达[43]。小孢子发育相关的bHLH转录因子功能异常往往导致植株雄性不育,这些不育突变体败育方式多样,败育程度各异。例如,在拟南芥中,雄性不育突变体没有花粉形成表现为完全败育;不育突变体会出现少量花粉并可自交结实;而AtbHLH10、AtbHLH89和AtbHLH91等转录因子在双突变或三突变时植株才表现败育。这种在败育方式及败育程度上的的差别可能暗示着各个bHLH转录因子在雄蕊发育的不同调控网络或者同一功能网络的不同层次发挥作用,具体机制尚待研究。不同植物中参与小孢子发育的bHLH转录因子氨基酸残基序列的一致性较高。例如,OsUDT1与ZmMS32、OsbHLH142与ZmRf4的氨基酸残基序列一致性分别达到67.52%和66.85%,他们在蛋白质序列上的相似性对于解析物种间bHLH转录因子功能及其调控网络具有一定参考,但目前对不同物种间的同源bHLH转录因子在小孢子发育中的功能异同的相关报道较少。同时,水稻中bHLH转录因子介导的小孢子发育的调控网络在其他植物中是否存在相似功能网络尚不清楚。在拟南芥中发现bHLH转录因子通过介导内源激素的合成与应答参与小孢子发育,在其他植物中是否存在类似机制,及这种机制同参与绒毡层发育的bHLH转录因子作用网络之间的关系仍需深入研究。
目前发现和鉴定参与植物雄花发育的bHLH转录因子的策略主要有两种:一是依赖于各类突变或转基因技术。例如,通过γ射线处理改变了、等基因碱基序列进而获得不育突变体和通过T-DNA插入目的基因区域获得、两个基因的突变体,而和等bHLH基因是通过抑制或过表达获得转基因植株,进而解析其功能。依靠物理、化学或生物技术手段获得目的基因的突变体对于基因功能研究具有重要意义,也是目前研究基因功能的经典方法和主要技术基础。但雄性不育突变体的数量有限,对于基因间的互作及调控网络的解析具有一定局限性。二是依赖于高通量测序技术,解析基因组、转录组以及蛋白组等组学水平上的测序数据,不仅丰富了人们进行基因功能研究的手段,同时也为探究基因互作与基因间的调控网络提供了便利。目前在水稻、拟南芥和玉米等植物中,利用转录组和蛋白组测序技术,已经发掘了许多雄蕊发育关键时期特异表达的bHLH基因,可利用TALEN、CRISPR/Cas等基因编辑技术对这些bHLH转录因子进行功能解析,以便全面的了解bHLH转录因子在植物雄花发育中的关键作用。
参考文献
[1] Wu Y, Fox TW, Trimnell MR, Wang L, Xu RJ, Cigan AM, Huffman GA, Garnaat CW, Hershey H, Albertsen MC. Development of a novel recessive genetic male sterility system for hybrid seed production in maize and other cross-pollinating crops., 2015: 12477.
[2] Hu JG, Rutger JN. Pollen characteristics and genetics of induced and spontaneous genetic male-sterile mutants in rice., 1992, 109(2): 97–107.
[3] 杨莉芳, 刁现民. 植物细胞核雄性不育基因研究进展. 植物遗传资源学报, 2013, 14(6): 1108–1117.
[4] Ding JH, Lu Q, Ouyang YD, Mao HL, Zhang PB, Yao JL, Xu CG, Li XH, Xiao JH, Zhang QF. A long noncoding RNA regulates photoperiod-sensitive male sterility, an essential component of hybrid rice., 2012, 109(7): 2654–2659.
[5] Zhou H, Zhou M, Yang YZ, Li J, Zhu LY, Jiang DG, Dong JF, Liu QJ, Gu LF, Zhou LY, Feng MJ, Qin P, Hu XC, Song CL, Shi JF, Song XW, Ni ED, Wu XJ, Deng QY, Liu ZL, Chen MS, Liu YG, Cao XF, Zhuang CX. RNase ZS1processesL40mRNAs and controls thermosensitive genic male sterility in rice., 2014, 5: 4884.
[6] Jin JP, Zhang H, Kong L, Gao G, Luo JC. PlantTFDB 3.0: a portal for the functional and evolutionary study of plant transcription factors., 2014, 42(Database issue): D1182–D1187.
[7] Shi H, Wang X, Mo XR, Tang C, Zhong SW, Deng XW.DET1 degrades HFR1 but stabilizes PIF1 to precisely regulate seed germination., 2015, 112(12): 3817–3822.
[8] Castelain M, Hir RL, Bellini C. The non--bindingtranscription factoris involved in the regulation of light signaling pathway in., 2012, 145(3): 450–460.
[9] Komatsu M, Maekawa M, Shimamoto K, Kyozuka J. Theandgenes determine the inflorescence architecture of rice by controlling rachis-branch and spikelet development., 2001, 231(2): 364–373.
[10] Ohno S, Deguchi A, Hosokawa M, Tatsuzawa F, Doi M. A basic helix-loop-helix transcription factor DvIVS determines flower color intensity in cyanic dahlia cultivars., 2013, 238(2): 331–343.
[11] Jiang L, Yan SS, Yang WC, Li YQ, Xia MX, Chen ZJ, Wang Q, Yan LY, Song XF, Liu RY, Zhang XL. Transcriptomic analysis reveals the roles of microtubule-related genes and transcription factors in fruit length regulation in cucumber (L.)., 2015, 5: 8031.
[12] Qi TC, Huang H, Wu DW, Yan JB, Qi YJ, Song SS, Xie DX.DELLA and JAZ proteins bind the WD-repeat/bHLH/MYB complex to modulate gibberellin and jasmonate signaling synergy., 2014, 26(3): 1118–1133.
[13] Nakata M, Mitsuda N, Herde M, Koo AJK, Moreno JE, Suzuki K, Howe GA, Ohme-Takagi M. A bHLH-type transcription factor, ABA-INDUCIBLE BHLH-TYPE TRANSCRIPTION FACTOR/JA-ASSOCIATED MYC2-LIKE1, acts as a repressor to negatively regulate jasmonate signaling in., 2013, 25(5): 1641–1656.
[14] Fan M, Bai MY, Kim JG, Wang T, Oh E, Chen L, Park CH, Son SH, Kim SK, Mudgett MB, Wang ZY. The bHLH transcription factor HBI1 mediates the trade-off between growth and pathogen-associated molecular pattern–triggered immunity in., 2014, 26(2): 828–841.
[15] Liu WW, Tai HH, Li SS, Gao W, Zhao M, Xie CX, Li WX.is important for drought and osmotic stress resistance inand in the repression of ABA catabolism., 2014, 201(4): 1192–1204.
[16] Carretero-Paulet L, Galstyan A, Roig-Villanova I, Martinez-Garcia JF, Bilbao-Castro JR, Robertson DL. Genome-wide classification and evolutionary analysis of the bHLH family of transcription factors in, poplar, rice, moss, and algae., 2010, 153(3): 1398–1412.
[17] Li XX, Duan XP, Jiang HX, Sun YJ, Tang YP, Yuan Z, Guo JK, Liang WQ, Chen L, Yin JY, Ma H, Wang J, Zhang DB. Genome-wide analysis of basic/helix-loop-helix transcription factor family in rice and., 2006, 141(4): 1167–1184.
[18] Toledo-Ortiz G, Huq E, Quail PH. Thebasic/helix-loop-helix transcription factor family., 2003, 15(8): 1749–1770.
[19] Heim MA, Jakoby M, Werber M, Martin C, Weisshaar B, Bailey PC. The basic helix-loop-helix transcription factor family in plants: a genome-wide study of protein structure and functional diversity., 2003, 20(5): 735–747.
[20] Bailey PC, Martin C, Toledo-Ortiz G, Quail PH, Huq E, Heim MA, Jakoby M, Werber M, Weisshaar B. Update on the basic helix-loop-helix transcription factor gene family in., 2003, 15(11): 2497– 2502.
[21] Song XM, Huang ZN, Duan WK, Ren J, Liu TK, Li Y, Hou XL. Genome-wide analysis of the bHLH transcription factor family in Chinese cabbage (ssp.)., 2014, 289(1): 77–91
[22] Hudson KA, Hudson ME. A classification of basic helix-loop-helix transcription factors of soybean., 2015, 2015: 603182.
[23] Hudson KA, Hudson ME. The basic helix-loop-helix transcription factor family in the sacred lotus,., 2014, 7(2): 65–70.
[24] Sun H, Fan HJ, Ling HQ. Genome-wide identification and characterization of thegene family in tomato., 2015, 16: 9.
[25] Wang JY, Hu ZZ, Zhao TM, Yang YW, Chen TZ, Yang ML, Yu WG, Zhang BL. Genome-wide analysis of bHLH transcription factor and involvement in the infection by yellow leaf curl virus in tomato ()., 2015, 16: 39.
[26] Chen ZY, Guo XJ, Chen ZX, Chen WY, Liu DC, Zheng YL, Liu YX, Wei YM, Wang JR. Genome-wide characterization of developmental stage- and tissue-specific transcription factors in wheat., 2015, 16: 125.
[27] Jiang Y, Zeng B, Zhao HN, Zhang M, Xie SJ, Lai JS. Genome-wide transcription factor gene prediction and their expressional tissue-specificities in maize., 2012, 54(9): 616–630.
[28] Zhang X, Luo HM, Xu ZC, Zhu YJ, Ji AJ, Song JY, Chen SL. Genome-wide characterisation and analysis of bHLH transcription factors related to tanshinone biosynthesis ina., 2015, 5: 11244.
[29] Kavas M, Baloğlu MC, Atabay ES, Ziplar UT, Daşgan HY, Ünver T. Genome-wide characterization and expression analysis of common bean bHLH transcription factors in response to excess salt concentration., 2015: 1–15.
[30] Chen YY, Li MY, Wu XJ, Huang Y, Ma J, Xiong AS. Genome-wide analysis of basic helix−loop−helix family transcription factors and their role in responses to abiotic stress in carrot., 2015, 35(5), doi: 10.1007/s11032-015-0319-0.
[31] Ma PCM, Rould MA, Weintraub H, Pabo CO. Crystal structure of MyoD bHLH domain-DNA complex: perspectives on DNA recognition and implications for transcriptional activation., 1994, 77(3): 451–459.
[32] Ko SS, Li MJ, Sun-Ben Ku M, Ho YC, Lin YJ, Chuang MH, Hsing HX, Lien YC, Yang HT, Chang HC, Chan MT. The bHLH142 transcription factor coordinates with TDR1 to modulate the expression ofand regulate pollen development in rice., 2014, 26(6): 2486–2504.
[33] Chang AT, Liu YJ, Ayyanathan K, Benner C, Jiang YK, Prokop JW, Paz H, Wang D, Li HR, Fu XD, Rauscher FJ, Yang J. An evolutionarily conserved DNA architecture determines target specificity of the TWIST family bHLH transcription factors., 2015, 29(6): 603–616.
[34] Forrest S, McNamara C. Id family of transcription factors and vascular lesion formation., 2004, 24(11): 2014–2020.
[35] García-Trevijano ER, Torres L, Zaragozá R, Viña JR. The role of Id2 in the regulation of chromatin structure and gene expression., 2013: 54969.
[36] Sharma P, Chinaranagari S, Chaudhary J. Inhibitor of differentiation 4 (ID4) acts as an inhibitor of ID-1, -2 and-3 and promotes basic helix loop helix (bHLH) E47 DNA binding and transcriptional activity., 2015, 112: 139–150.
[37] 张虹, 梁婉琪, 张大兵. 花药绒毡层细胞程序性死亡研究进展. 上海交通大学学报(农业科学版), 2008, 26(1): 86–90.
[38] Dukowic-Schulze S, Harris A, Li JH, Sundararajan A, Mudge J, Retzel EF, Pawlowski WP, Chen CB. Comparative transcriptomics of early meiosis inand maize., 2014, 41(3): 139–152.
[39] Yang HX, Lu PL, Wang YX, Ma H. The transcriptome landscape ofmale meiocytes from high-throughput sequencing: the complexity and evolution of the meiotic process., 2011, 65(4): 503–516.
[40] Zhang LS, Wang L, Yang YL, Cui J, Chang F, Wang YX, Ma H. Analysis offloral transcriptome: detection of new florally expressed genes and expansion of-specific gene families., 2014, 5: 802.
[41] Sorensen AM, Krӧber S, Unte US, Huijser P, Dekker K, Saedler H. Thegene encodes a MYC class transcription factor., 2003, 33(2): 413–423.
[42] Thorstensen T, Grini PE, Mercy IS, Alm V, Erdal S, Aasland R, Aalen RB. The Arabidopsis SET-domain protein ASHR3 is involved in stamen development and interacts with the bHLH transcription factor ABORTED MICROSPORES (AMS)., 2008, 66(1–2): 47–59.
[43] Xu J, Ding ZW, Vizcay-Barrena G, Shi JX, Liang WQ, Yuan Z, Werck-Reichhart D, Schreiber L, Wilson ZA, Zhang DB.acts as a master regulator of pollen wall formation in., 2014, 26(4): 1544–1556.
[44] Ma X, Feng BM, Ma H. AMS-dependent and independent regulation of anther transcriptome and comparison with those affected by otheranther genes., 2012, 12: 23.
[45] Zhang W, Sun YJ, Timofejeva L, Chen CB, Grossniklaus U, Ma H. Regulation oftapetum development and function byencoding a putative bHLH transcription factor., 2006, 133(16): 3085–3095.
[46] Feng BM, Lu DH, Ma X, Peng YB, Sun YJ, Ning G, Ma H. Regulation of theanther transcriptome by DYT1 for pollen development., 2012, 72(4): 612–624.
[47] Gu JN, Zhu J, Yu Y, Teng XD, Lou Y, Xu XF, Liu JL, Yang ZN. DYT1 directly regulates the expression offor tapetum development and pollen wall formation in., 2014, 80(6): 1005–1013.
[48] Zhu EG, You CJ, Wang SS, Cui J, Niu BX, Wang YX, Qi J, Ma H, Chang F. The DYT1-interacting proteins bHLH010, bHLH089 and bHLH091 are redundantly required foranther development and transcriptome., 2015, 83(6): 976–990.
[49] Nakata M, Ohme-Takagi M. Two bHLH-type transcription factors, JA-ASSOCIATED MYC2-LIKE2 and JAM3, are transcriptional repressors and affect male fertility., 2013, 8(12): e26473.
[50] Figueroa P, Browse J. Male sterility ininduced by overexpression of a MYC5-SRDX chimeric repressor., 2015, 81(6): 849–860.
[51] Xing SP, Quodt V, Chandler J, Hӧhmann S, Berndtgen R, Huijser P.acts together with the brassinosteroid-signaling componentin controllingmale fertility., 2013, 2(3): 416–428.
[52] Jung KH, Han MJ, Lee YS, Kim YW, Hwang I, Kim MJ, Kim YK, Nahm BH, An G. Riceis a major regulator of early tapetum development., 2005, 17(10): 2705–2722.
[53] Zhang DS, Liang WQ, Yuan Z, Li N, Shi J, Wang J, Liu YM, Yu WJ, Zhang DB. Tapetum degeneration retardation is critical for aliphatic metabolism and gene regulation during rice pollen development., 2008, 1(4): 599–610.
[54] Li N, Zhang DS, Liu HS, Yin CS, Li XX, Liang WQ, Yuan Z, Xu B, Chu HW, Wang J, Wen TQ, Huang H, Luo D, Ma H, Zhang DB. The ricegene is required for tapetum degradation and anther development., 2006, 18(11): 2999–3014.
[55] Niu NN, Liang WQ, Yang XJ, Jin WL, Wilson ZA, Hu JP, Zhang DB. EAT1 promotes tapetal cell death by regulating aspartic proteases during male reproductive development in rice., 2013, 4(2): 1445.
[56] Ji CH, Li HY, Chen LB, Xie M, Wang FP, Chen YL, Liu YG. A novel rice bHLH transcription factor, DTD, acts coordinately with TDR in controlling tapetum function and pollen development., 2013, 6(5): 1715–1718.
[57] Fu ZZ, Yu J, Cheng XW, Zong X, Xu J, Chen MJ, Li ZY, Zhang DB, Liang WQ. The rice basic helix-loop-helix transcription factor TDR INTERACTING PROTEIN2 is a central switch in early anther development., 2014, 26(4): 1512–1524.
[58] Crismani W, Kapoor S, Able JA. Comparative transcriptomics reveals 129 transcripts that are temporally regulated during anther development and meiotic progression in both bread wheat () and rice ()., 2011: 931898.
[59] Collado-Romero M, Alós E, Prieto P. Unravelling the proteomic profile of rice meiocytes during early meiosis., 2014, 5: 356.
[60] Zhang H, Egger RL, Kelliher T, Morrow D, Fernandes J, Nan GL, Walbot V. Transcriptomes and proteomes define gene expression progression in pre-meiotic maize anthers., 2014, 4(6): 993–1010.
[61] Dukowic-Schulze S, Sundararajan A, Mudge J, Ramaraj T, Farmer AD, Wang MH, Sun Q, Pillardy J, Kianian S, Retzel EF, Pawlowski WP, Chen CB. The transcriptome landscape of early maize meiosis., 2014, 14: 118.
[62] Moon J, Skibbe D, Timofejeva L, Rachel Wang CJ, Kelliher T, Kremling K, Walbot V, Cande WZ. Regulation of cell divisions and differentiation by MALE STERILITY32 is required for anther development in maize., 2013, 76(4): 592–602.
[63] Ren RH, Nagel BA, Kumpatla SP, Zheng PZ, Cutter GL, Greene TW, Thompson SA. Maize cytoplasmic male sterility (cms) c-type restorer rf4 gene, molecular markers and their use: U.S. Patent Application 20120090047. 2013-08-14.
[64] Liu TK, Li Y, Zhang CW, Duan WK, Huang FY, Hou XL. Basic helix-loop-helix transcription factorfunctions as a positive regulator of pollen development in non-heading Chinese cabbage., 2014, 14(4): 731–739.
[65] Jeong HJ, Kang JH, Zhao MA, Kwon JK, Choi HS, Bae JH, Lee HA, Joung YH, Choi D, Kang BC. Tomato Male sterile 1035is essential for pollen development and meiosis in anthers., 2014, 65(22): 6693–6709.
Research progress of the bHLH transcription factors involved in genic male sterility in plants
Yongming Liu1, Ling Zhang1, Jianyu Zhou2, Moju Cao1
Male sterility exists widely in the spermatophytes. It contributes to the study of plant reproductive development and can be used as an effective tool for hybrid seed production in heterosis utilization. Therefore, the study on male sterility is of great value in both theory and application. As one of the largest transcription factor families in plants, basic helix-loop-helix proteins (bHLHs) play a crucial role in regulating plant growth and development. This paper introduces the mechanism of bHLH regulating stamen development in several important model plants. Furthermore, we discuss the molecular mechanisms of genic male sterility resulting from bHLH dysfunction to provide
for crop breeding and theoretical studies.
male sterility; bHLH transcription factor; programmed cell death; transcriptome sequencing
2015-05-19;
2015-07-19
国家高技术研究发展计划项目(863计划)(编号:2011AA10A103)资助
刘永明,博士研究生,专业方向:玉米生物技术育种。E-mail: liuluckforever@163.com
曹墨菊,教授,研究方向:玉米雄性不育。E-mail: caomj@sicau.edu.cn
10.16288/j.yczz.15-229
网络出版时间: 2015-10-15 9:35:48
URL: http://www.cnki.net/kcms/detail/11.1913.R.20151015.0935.001.html
(责任编委: 张宪省)
