黑皮冬瓜LINE逆转座子RT序列的克隆与特征分析
2015-04-29赵芹等
赵芹等

摘 要 根据LINE逆转座子逆转录酶保守序列设计简并引物,PCR扩增黑皮冬瓜“B98K”基因组DNA,获得580 bp左右目的条带。将PCR产物回收、克隆并测序,获得23条逆转酶序列,利用生物信息学软件分析其长度变异、碱基变化、相似性及系统进化关系。结果表明:这些序列长度在557~593 bp区间变异,同源比对核苷酸序列相似性为39.0%~99.3%,存在高度异质性,主要表现为缺失突变、移码突变与终止密码子突变,核苷酸序列聚类分析分为4个家族,家族1与家族2分别包含15和5个成员,占总序列数的65.22%和21.74%。对推导的氨基酸序列分析发现,第12位氨基酸残基处存在一保守的苯丙氨酸(Phe),多处位置存在半保守氨基酸残基;氨基酸序列相似性在20.0%~99.5%之间;其中8条序列可能具有转录活性,8条与15条序列分别发生移码与终止密码子突变。与已知物种构建系统发育进化树,发现冬瓜LINE逆转座子RT序列较保守,且与拟南芥、李、油菜等有较近的亲缘关系。研究结果为后续利用分子标记研究冬瓜种质遗传变异及基因组进化奠定基础。
关键词 黑皮冬瓜;LINE逆转座子;逆转录酶;序列分析
中图分类号 S642.3 文献标识码 A
Abstract Degenerate primers were designed according to the RT conserved motifs of LINE retrotransposons to amplify genomic DNA of black wax gourd“B98K”for investigating and understanding genetic relationship, characteristics and evolution of LINE retrotransposon families from black wax gourd. The PCR products of 580 bp were obtained and cloned to pMD18-T vector, and the positive clones were screened and sequenced. The full-length variation, base composition, homology and phylogenetic relationship of twenty-three different sequences were analyzed by bio-informatics softwares. The results indicated that length of RT sequences concentrated on the region of 557-593 bp, and sequence similarities varied from 39.0%-99.3%. These sequences showed high heterogeneity which was mainly characterized by deletion, frameshift and termination codon mutation. Four families were divided by alignment analyses of nucleotide sequences, and family1 and family2 contained most members of black wax gourd LINE retrotransposons and accounted for 65.22% and 21.74%, respectively. The analysis results of deduced amino acid sequences showed that there is a conservative phenylalanine(phe)in the 12th amino acid residue position of the translated sequences and lots of semi-conservative points appeared in many positions. Eight sequences were found to have transcriptional activity, moreover eight and fifteen sequences presented termination codon and frameshift mutations respectively, and amino acid sequence similarities ranged from 20.0% to 99.5%. The phylogenic tree constructed by these sequences and other RT sequences from GenBank showed that RT sequences of LINE retrotransposons in black wax gourd were conservative, and shared high homology with other species like Arabidopsis, plum and rape, etc. This work offers the basis for developing molecular markers to analyze the genetic variability and research the origin and evolution pathways of black wax gourd.
Key words Black wax gourd; LINE retrotransposons; Reverse transcriptase; Sequence analysis
doi 10.3969/j.issn.1000-2561.2015.03.004
冬瓜(Benincasa hispida Cogn.)为冬瓜属一年生蔓性植物,原产中国与印度,现广泛分布于亚洲的热带、亚热带和温带地区,在中国种植历史悠久,南北地区分布广泛[1]。冬瓜易栽培,产量高,耐贮运,保健药用价值高[2-3],是华南地区北运和出口创汇的重要蔬菜品种,对调节蔬菜淡季、保证蔬菜周年供应起着非常重要的作用[4]。冬瓜种质资源丰富,但目前对其种质资源分类评价研究较少,前人主要依据生态学性状或RAPD与ISSR标记开展此类分析[5-7],而不同种形成与进化之间的关系尚不清楚。源于植物逆转座子的分子标记已广泛应用于种质鉴定、图谱构建和系统进化研究等方面[8-10],为开展冬瓜种质研究提供了新的视野。此外,冬瓜花色、花型与瓜型及抗性性状差异很大,但对这些性状的形成机制尚不明确。据报道逆转座子易受外界因素激活转座导致基因突变、基因组扩增及重排等,造成植物遗传变异[11-14],在葡萄果皮颜色[15]和菜豆花色形成[16]等方面发挥重要作用,因此明确逆转座子与冬瓜生物学性状及遗传进化间关系具有重要意义。
根据DNA结构特征,逆转座子可分为长末端重复序列(Long terminal repeat,LTR)和非长末端重复序列(Non-long terminal repeat,non-LTR)两类。它以RNA为中间产物在寄主基因组内不断转座增殖,影响寄主基因组的大小、结构、功能及进化等[17],大部分逆转座子在生物及非生物因素胁迫下可被激活[18],其序列的克隆与分析对解析植物基因组组成、进化及表达调控具有重要意义[19-21]。non-LTR类逆转座子已在多种植物中进行研究,且开发出不同的分子标记技术[20,22-24]。目前尚未见冬瓜LINE逆转座子RT序列分离及进一步转录活性研究的报道。因此,本研究拟克隆冬瓜LINE类逆转座子RT片段,利用生物信息学软件分析其变化特点及与其他物种序列的相似性,以期为冬瓜基因组起源与遗传进化、种质资源评价提供依据。
1 材料与方法
1.1 材料
供试材料为黑皮冬瓜“B98K”由广东省农科院蔬菜所瓜类研究一室保存;冬瓜种子播种至温室营养盘内,待幼苗长至2片真叶时用于实验。
1.2 方法
1.2.1 DNA的提取及逆转录酶序列PCR扩增 采用改良CTAB法提取冬瓜基因组总DNA,1%琼脂糖凝胶电泳及纳米紫外分光光度计检测DNA浓度及纯度。
参照Hill等[22]设计扩增LINE逆转座子RT序列的引物。引物序列为LINE-F: 5′-GGG ATCCNG
GNCCNGAYGGNWT-3′;LINE-R: 5′-SWNARNGG
RTCNCCYTG-3′,其中R=A/G,Y=C/T,S=C/G,W=A/T,N=A/C/G/T。PCR反应体系25 μL,包括DNA 100 ng,10×Ex buffer(含MgCl2)2.5 μL,dNTPs 0.20 mmol/L,1 U Ex Taq DNA聚合酶,引物终浓度为1.0 μmol/L。扩增程序为:95 ℃ 3 min;95 ℃ 1 min,50 ℃ 1 min,72 ℃ 1 min,35个循环;72 ℃ 10 min。扩增产物在1.2%琼脂糖凝胶中进行电泳分析。
1.2.2 PCR产物克隆及序列测定 回收纯化PCR产物,与pMD19-T vector连接,转化大肠杆菌DH5ɑ感受态细胞过夜培养,Amp抗生素筛选,挑取白斑利用M13通用引物菌落PCR鉴定阳性克隆,送与广州英骏生物技术有限公司测序。
1.2.3 逆转录酶序列分析 测序序列利用NCBI vecscreen程序去除载体部分,利用NCBI Blastx推导比对获得RT基因片段及推导的氨基酸序列,应用DNAStar软件对RT序列长度、碱基组成、G+C含量、氨基酸序列、多重比对等进行分析,结合GenBank报道的其他物种序列构建系统发育进化树。
2 结果与分析
2.1 冬瓜LINE类逆转座子RT序列的扩增与序列测定
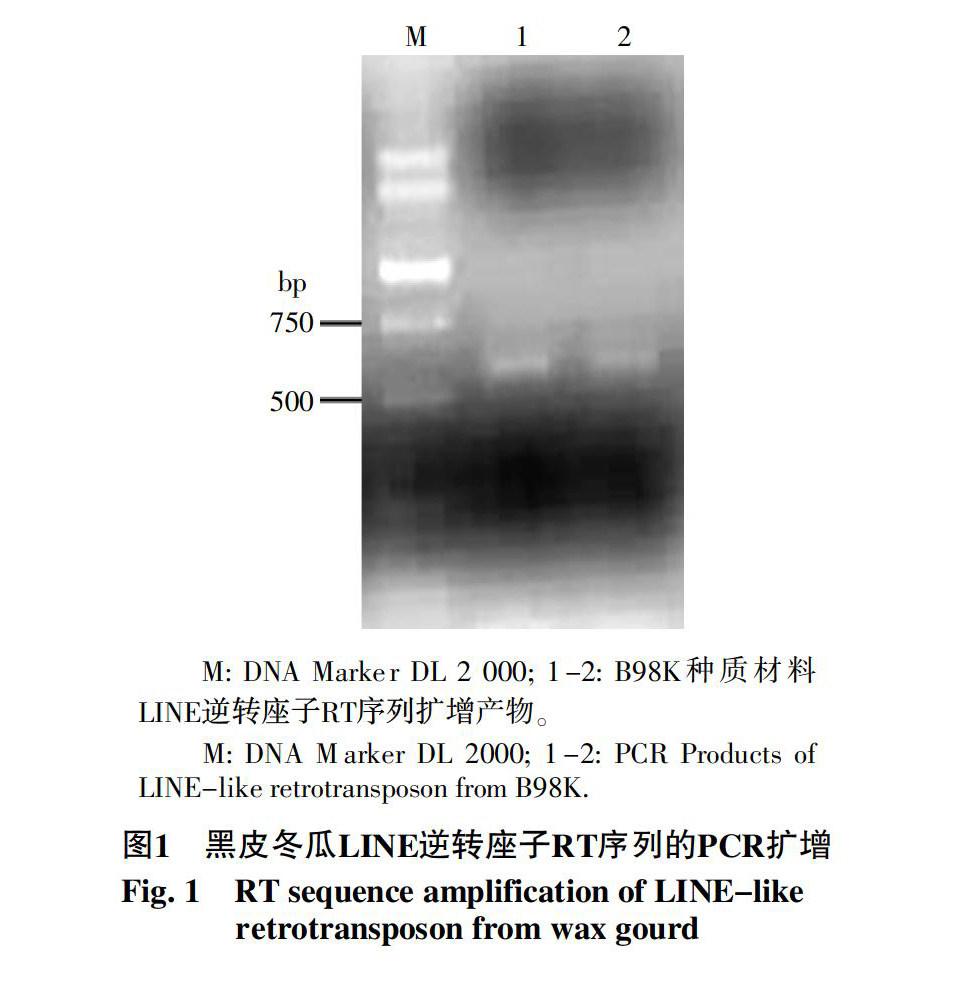
利用简并引物在黑皮冬瓜“B98K”中检测到约580 bp长度的特异片段(图1),预期大小与其他植物LINE逆转座子RT序列基本一致[25],说明该类逆转座子在冬瓜中普遍存在。
将PCR产物回收克隆,随机挑取24个阳性克隆测序。经NCBI blastx程序同源搜索发现,23条序列与其它物种non-LTR逆转座子RT序列具有较高同源性,除BhRT17外,其他序列均含有RT保守域,说明本研究克隆到了冬瓜LINE逆转座子RT序列,依次命名为BhRT1~BhRT23(表1)。
2.2 冬瓜LINE逆转座子RT序列同源性分析
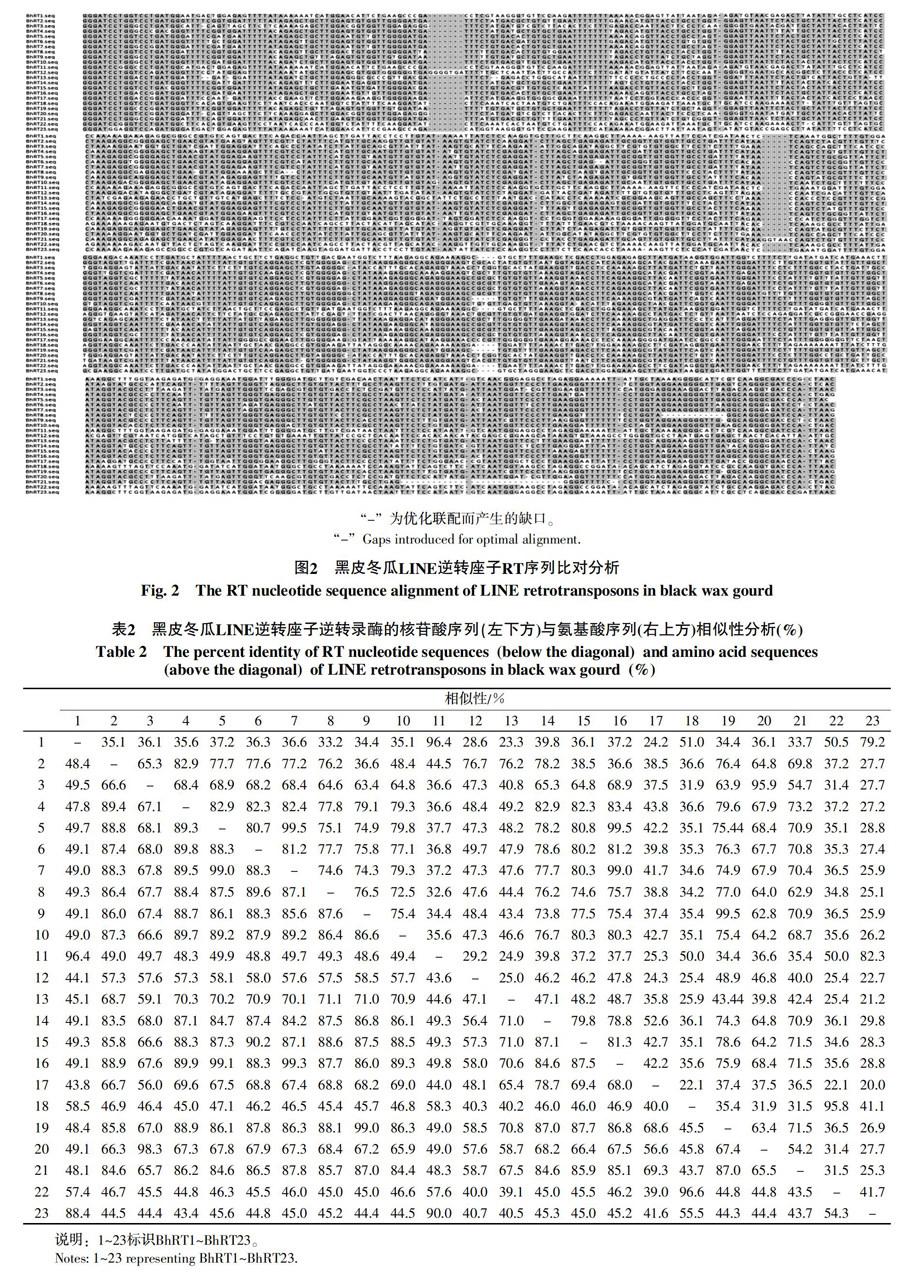
DNAStar软件分析表明,23条RT序列长度并不完全一致,变化范围为557~593 bp,BhRT12最长,BhRT21最短,其余序列长度在570~583 bp之间(表1与图2);碱基分析发现,所有序列富含碱基AT,A、T、C、G数目变化范围分别为110~198、159~217、117~171和81~111,AT与GC比例为1.26~1.46。由表2可以看出,冬瓜RT序列间相似性在39.0%~99.3%之间,其中BhRT16与BhRT3相似性最高为99.3%,BhRT17与BhRT22相似性最低为39.0%(表2)。由此可见,同一简并引物扩增所得的逆转座子RT序列并不一致,在序列长度、碱基变化及序列相似性等方面存在较大差异,表明同一种质内同一类群逆转座子存在高度异质性,甜菜、牡丹等作物也表现类似特点[25-27]。
2.3 冬瓜LINE逆转座子RT序列聚类分析
分析23条RT序列构建的系统发育进化树(图3)发现,RT序列根据遗传距离分成4个家族(family 1~4),每组代表由同一祖先而来或亲缘关系较近的遗传家系。其中family 4仅包含一个序列BhRT12,为长度最长的一个序列(593 bp),与其它家族的遗传距离较远,单独列为一族,可能是由于核苷酸序列间的缺失突变造成的;family 1~3分别包括15个、5个和2个序列,family 1与family 2分别占克隆总数的65.22%与21.74%,说明这2个家族是克隆获得冬瓜逆转座子RT序列的主要成分;family 1包含的15条RT序列相似性在72.3%~ 99.3%之间,核苷酸序列长度除BhRT8与BhRT21差异比较大外,其余序列表现比较一致,也表现出一定的差异性,主要是由不同程度的点突变、碱基替换及缺失突变造成的;family 1可分为3个亚家族,分别包含9个、3个与3个RT序列,亚家族相似性分别在71.3%~99.3%、64.3%~78.5%与87.0%~99.0%之间,其中BhRT16与BhRT7相似性达99.3%,这可能在进化过程中是由一类逆转座子发生突变等造成的。family 2分为2个亚家族,分别含有BhRT23、BhRT11与BhRT1以及BhRT18与BhRT22,序列相似性在88.3%~96.4%与96.6%,同样表现缺失突变、碱基替换与点突变。family 3的BhRT3与BhRT20 RT序列相似性为67.9%。由此可见,碱基替换、点突变或缺失突变都可能是同一家族逆转座子产生多拷贝群的原因。同时在不同家族包含克隆数的差异,反映了各家族转座子转座过程的差异。
2.4 冬瓜LINE逆转座子逆转录酶的氨基酸序列分析
将各RT序列通过NCBI Blastp程序同源搜索,发现与其他物种逆转录酶具有较高相似性,据此将23条序列推导翻译成氨基酸序列,其中BhRT3、BhRT17与BhRT20从第2个核苷酸起始翻译,其它序列从第3个核苷酸起始;进一步分析表明,第12位氨基酸残基处存在一保守苯丙氨酸(Phe),推测此位点可能是LINE逆转座子RT序列中非常保守的位点,另外多处位置存在半保守氨基酸残基(图3);有8条序列(BhRT6、BhRT8、BhRT9、BhRT10、BhRT12、BhRT13、BhRT19与BhRT21)发生移码突变;15条序列发生不同程度终止密码子突变,其中8条序列存在3个终止密码子突变,3条序列存在2个终止密码子突变,4条序列存在1个终止密码子突变;另外,8条序列存在转录活性(BhRT1、BhRT5、BhRT11、BhRT14、BhRT16、BhRT18、BhRT20与BhRT22),占34.78%,所以移框突变和终止密码子突变可能是逆转座子发生突变与不能表达,导致逆转座子异质性的重要原因(图4)。
经DNAStar软件Clustal W同源比对,23条RT氨基酸序列相似性为20.0%~99.5%(表2),BhRT23与BhRT17相似性最低,BhRT19与BhRT9及BhRT16与BhRT5相似性最高。核苷酸序列相似性最低的BhRT13与BhRT18(40.2%),其氨基酸序列相似性并非最低(25.9%),这主要是由于密码子的简并性造成的,也充分说明同一来源的逆转座子异质性较高。对各家族氨基酸序列分析发现,family 1中15条RT序列的氨基酸序列相似性为36.6%~99.5%;famlily 2中5条RT序列相似性为41.1%~95.8%;family 3中2条序列相似性为95.9%;各家族所包含的克隆数越多,序列相似性越高,发生转座时间越近,具有转座活性的可能性亦越大。family1~family3包含全部具有转座活性的逆转座子,氨基酸序列翻译也证实了该结论。
2.5 冬瓜LINE类逆转座子RT序列系统进化树分析
结合GenBank的13份物种RT序列构建系统发育进化树(图5),结果分析表明,来源冬瓜的23条RT序列比较复杂,与李、拟南芥、油菜、可可、甜菜、甜橙、黄瓜等有较高相似性;它们在聚类图中主要分为5组,18条冬瓜RT序列与黄瓜(Cucumis sativus,XP_004172985)、葡萄(Vitis vinifera,CAN66430)、甜橙(Citrus sinensis,XP_006490008)、湿地松(Pinus elliottii,CAA12932)、水稻(Oryza sativa,EEC80620)等12份植物序列相似性较高,划分在第1组,第1组约占冬瓜克隆RT序列总数的78.26%,表明冬瓜逆转座子RT序列具一定保守性;冬瓜RT序列BhRT21单独形成第2组;第3组包括3条冬瓜RT序列BhRT9、BhRT19与BhRT8;冬瓜序列BhRT2单独形成第4组,与其它组遗传距离较远;而可可(Theobroma cacao,XP_007008704)与其他植物亲缘关系最远,单独为一分支,为第5组,这4条冬瓜RT序列单独列为4组,说明冬瓜逆转座子RT序列具有较高异质性与遗传变异率。逆转座子在植物界广泛分布,作为基因组的组成成分之一,在世代间不仅可以纵向传递,还可以横向传递。上述分析表明不同物种中存在着起源相同或相近的逆转座子,不同种属间有时存在比种属内相似性更高的逆转座子。由此可见,在冬瓜进化史中与上述物种的LINE逆转座子RT序列间可能存在横向传递现象。
3 讨论与结论
冬瓜在中国栽培广泛,类型品种丰富,对种质资源的鉴评及系统进化研究是加速品种改良和杂种优势利用的前提。不同分子标记方法开展种质遗传多样性研究,各有其特异性与优势弊端[5-7]。利用逆转座子RT序列开发分子标记的方法为冬瓜遗传多样性分析提供了新的视野。本研究利用LINE逆转座子RT序列简并引物,首次从黑皮冬瓜种质中扩增得到目的片段,说明LINE逆转座子在冬瓜中广泛存在,为后续研究奠定基础。
逆转录转座子是植物基因组的重要组成部分,对基因功能与基因组结构有重要影响[28-29],其序列的克隆和分析对植物基因组学、系统发育学及转录组学具有重要研究意义。由于逆转座子自身转录增殖的碱基错配率高,及长期进化过程中的同源序列重组与生物体防御的互作关系,形成了其高度异质性群体[30]。本研究扩增得到冬瓜LINE逆转座子RT序列长度差为36 bp,小于牡丹的46 bp,在碱基序列与GC含量方面差异也较大,表现很高的异质性。推断冬瓜RT序列中存在的不同程度缺失突变、终止密码子突变与移码突变,可能是造成冬瓜LINE逆转座子异质性的主要原因,这与牡丹、甜菜等作物研究结果一致[25-26]。冬瓜LINE逆转座子RT氨基酸序列存在许多保守与半保守的氨基酸位点,而其他多处的氨基酸位点表现很大的差异性,这可能为进化过程中发生氨基酸突变而形成的。这些都表明冬瓜基因组内LINE逆转座子是一类较古老元件,其异质性是后来伴随寄主基因组的进化,在转座与世代传递过程中逐渐积累突变的。
正常的生长发育过程中,植物体内的逆转座子通常处于静止状态,而在受到生物和非生物胁迫后,转录活性将被激活[31-35]。多数冬瓜RT序列存在碱基缺失与终止密码子突变,无终止密码子的8条序列可能具有转录活性,因此推测外部因素激活了冬瓜逆转座子,发生转座事件,插入位点发生突变的逆转座子,以无活性形式存在于基因组中,而活性转座子可能在其它胁迫下发生遗传学效应。因此在各胁迫条件下处理冬瓜材料研究逆转座子的转座活性,进一步发掘冬瓜各性状变异与逆转座子的关系,将对基因组组成进化及优良基因发掘具有重要意义。
逆转座子不仅可进行世代纵向传递,还可进行物种间横向传递,藉此推测逆转座子的起源、物种的形成及其与物种间的相互关系[36-37]。前人研究表明同一植物内的同一类型逆转座子序列变异较大,而同一植物类型的逆转座子保守性较强,有时种内异质性差异大于种属间[38]。在随机克隆的23条冬瓜RT序列中,BhRT22与BhRT17一致性只有39.0%,有的甚至与葡萄、拟南芥、湿地松等具有更高相似性,例如BhRT1与李(Prunus persica,XP_
007212886)一致性高达58%,说明这些物种的LINE逆转座子也表现上述特点,可能是不同物种间逆转座子横向传递的结果,进一步说明了生物界有共同的起源。这为研究冬瓜系统进化提供了依据。逆转座子散布在基因组中成为进化的种子,与植物进化关系密切[37]。冬瓜23条RT序列经系统聚类分析,结果将其分为4个家族,不同家族里含有的RT序列数量不同,反映了不同家族逆转座子转录过程存在差异,其存在的历史地位也可能不同,包含克隆数愈多的家族历史愈久远,对冬瓜基因组进化作用也可能各异。
本研究首次从黑皮冬瓜中成功克隆了LINE逆转座子RT基因序列,为后续转录活性研究及开发分子标记鉴评冬瓜种质奠定基础。
参考文献
[1] 中国农业科学院蔬菜花卉研究所. 中国蔬菜品种志[M]. 北京: 中国农业科技出版社, 2001.
[2] 尹晴红, 董敏玉. 冬瓜功能食品对肾系疾患的治疗保健作用[J]. 南京农专学报, 2000, 16(4): 40.
[3] Diez M J, Pico B, Nuez F. Cucurbit genetic resources in Europe[C]. Ad hoc meeting, Adana, Turkey. 2002.
[4] 谢大森, 何晓明, 赫新洲, 等. 冬瓜主要农艺性状的杂种优势初步分析[J]. 上海农业学报, 2003, 19(2): 35-37.
[5] Pandey S, Kumar S, Mishra U, et al. Genetic diversity in Indian ash gourd accessions as revealed by quantitative traits and RAPD markers[J]. Scientia Horticulturae, 2008, 118: 80-86.
[6] 张建军, 刘世贵, 余愚群, 等. 100份中国冬瓜种质资源农艺性状与遗传多样性研究[J]. 四川大学学报(自然科学版), 2009, 46(6): 1 855-1 861.
[7] 宋世威, 李 珍, 刘厚诚, 等. 冬瓜和节瓜种质资源遗传多样性的RAPD分析[J]. 中国蔬菜, 2010, 22: 47-53.
[8] Ruiz C, Asins M J. Comparison between Poncirus and Citrus genetic linkage maps[J]. Theoretical and Applied Genetics., 2003, 106: 826-836.
[9] Antonius-Klemola K, Kalendar R, Schulman A H. TRIM retrotransposons occur in apple and are polymorphic between varieties but not sports[J]. Theoretical and Applied Genetics, 2002, 112: 999-1 008.
[10] Venturi S, Dondini L, Donini P, et al. Retrotransposon characterisation and fingerprinting of apple clones by SSAP markers[J]. Theoretical and Applied Genetics, 2005, 112: 440-444.
[11] Mcclintock B. The significance of responses of the genome to challenge[J]. Science, 1984, 226: 792-801.
[12] Grandbastien M A, Lueas H, Morel J B, et al. The expression of the tobacco Tntl retrotransposon is linked to plant defense responses[J]. Genetica,1997, l00(1-3): 241-252.
[13] Mhiri C, De Wilt P J, Grandbastien M A. Activation of the promoter of the Tntl retrotransposon in tomato after inoculation with the fungi pathogen clados poriumfiilvum molecular[J]. Plant-Microbe Interactions, 1999, 12(7): 592-603.
[14] Beguiristain T, Grandbastien M A, Puigdomeneeh P, et al. Three Tntl sub-families show different stress associated patterns of expression in tobacco; consequences for retrotransposon, control and evolution in plants[J]. Plant Physiology, 2001, 127(1): 212-221.
[15] Carrier G, Le Cunff L, Dereeper A, et al. Transposable elements are a major cause of somatic polymorphism in Vitis vinifera L[J]. PloS One, 2012, 7(3): e32973.
[16] Erdmann P M, Lee R K, Bassett M J, et al. A molecular marker tightly linked to P, a gene required for flower and seedcoat color in common bean(Phaseolus vulgaris L.), contains the Ty3-gypsy retrotransposon Tpv3g[J]. Genome, 2002, 45(4): 728-736.
[17] FlavellA J, Smith D, KumarA. Extreme heterogeneity of Ty1-copia group retrotransposons in plants[J]. Molecular and General Genetics, 1992, 231: 233-242.
[18] Mhiri C, De Wit P J G M and Grandbastien M A. Activation of the promoter of the Tnt1 retrotransposon in tomato after inoculation with the fungal pathogen Clodosporium. fulvum[J]. Molecular Plant Mcrobe Interactions, 1999, 12(7): 592-603.
[19] Biswas M K, Baig M N R, Cheng Yun-jiang, et al. Retrotransposon based genetic similarity within the genus Citrus and its relatives[J]. Genetic Resources and Crop Evolution, 2010, 57: 963-972.
[20] Kalendar R, Flavell A J, Ellis T H N, et al. Analysis of plant diversity with retrotransposon-based molecular markers[J]. Heredity, 2010, 106: 520-530.
[21] Ma Yue, He Ping, Sun Hai-yue, et al. Isolation and characterization of transcriptionally active Ty1-copia retrotransposons in Fragaria × ananassa[J]. Agricultural Sciences in China, 2010, 9(3): 337-345.
[22] Hill P, Burford D, Martin D M, et al. Retrotransposon populations of Vicia species with varying genome size[J]. Molecular Genetics and Genomics, 2005, 273(5): 371-381.
[23] Martin S L. Nucleic acid chaperone properties of ORF1p from the non-LTR retrotransposon, LINE-1[J]. RNA Biology, 2010, 7(6): 706-711.
[24] Seibt K M, Wenke T, Wollrab C, et al. Development and application of SINE-based markers for genotyping of potato varieties[J]. Theoretical and Applied Genetics, 2012, 125(1): 185-196.
[25] 宋程威, 郭大龙, 张 曦, 等. 牡丹LINE类反转录转座子RT序列的克隆及分析[J]. 园艺学报, 2014, 41(1): 157-164.
[26] Kubis S E, Heslop-Harrison J S, Desel C, et al. The genomic organization of non-LTR retrotransposons(LINEs)from three Beta species and five other angiosperms[J]. Plant Molecular Biology, 1998, 36(6): 821-831.
[27] Noma K, Ohtsubo E, Ohtsubo H. Non-LTR retrotransposons (LINEs)as ubiquitous components of plant genomes[J]. Molecular and General Genetics, 1999, 261(1): 71-79.
[28] 孙 俊, 房经贵, 高 兵, 等. 苹果中Ty1-copia型逆转座子逆转录酶序列的克隆及分析[J]. 果树学报, 2005, 22(3): 193-197.
[29] Tikhonov A P, SanMiguel P J, Nakajima Y, et al. Colinearity and its exceptions in orthologous adh regions of maize and sorghum[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(13): 7 409-7 414.
[30] Steinhauer D A, Holland J J. Direct method for quantitation of extreme polymerase error frequencies at selected single base sites in viral RNA[J]. Journal of Virology, 1986, l57: 219-228.
[31] Kalendar R, Tanskanen J, Immonen S, et al. Genome evolution of wild barley(Hordeum spontaneum)by BARE-1 retrotransposon dynamics in response to sharp microcli-matic divergence[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97: 6 603-6 607.
[32] Ellis T H N, Poyser S J, Knox M R, et al. Polymorphism of insertion sites of Ty1-copia class retrotransposons and its use for linkage and diversity analysis in pea[J]. Molecular and General Genetics, 1998, 260: 9-19.
[33] Takeda S, Sugimoto K, Otsuki H. Transcriptional activation of the tobacco retrotransposon Tto1 by wounding and methyl jasmonate[J]. Plant Molecular Biology, 1998, 36: 365-376.
[34] Pearce S R, Harrison G, Li D, et al. The Ty1-copia group retrotransposons in Vicia species: Copy number, sequence heterogeneity and chromosomal localisation[J]. Molecular and General Genetics, 1996, 250(3): 305-315.
[35] 胡志昂, 姜国强, 邓 馨, 等. 野大豆种群转座子和转录因子的多样性和分子适应[J]. 植物生态学报, 2007, 31(5): 952-959.
[36] Kumar A. The evolution of plant retroviruses: moving to green pasture[J]. Trends in Plant Science, 1998, 3: 371-374.
[37] Grandbastien M. Retroelements in high Plants[J]. Trends In Genetics, 1992, 8(3): 100-108.
[38] Voytas D F, Cummings M, Koniczny A, et al. Copia-like retrotransposon are ubiquitous among plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(15): 7 124-7 128.
