AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶序列的多样性
2022-06-09刘俊仙刘菁唐荣华阳太亿韩柱强唐秀梅贺梁琼钟瑞春蒋菁黄志鹏吴海宁韦荣昌熊发前
刘俊仙 刘菁 唐荣华 阳太亿 韩柱强 唐秀梅 贺梁琼 钟瑞春 蒋菁 黄志鹏 吴海宁 韦荣昌 熊发前


摘要:以2份AA染色体组野生种花生为试验材料,扩增分离Ty3-gypsy类逆转座子逆转录酶序列,分析其序列特征、多样性及进化关系。利用根据Ty3-gypsy类逆转座子逆转录酶的保守区设计的简并引物进行PCR扩增,对目的条带进行回收、克隆和测序后,对逆转录酶序列进行生物信息学分析。目的条带大小均约为430 bp,分别从2份AA染色体组野生种花生材料中分离到32、33条逆转录酶序列,对于65条逆转录酶序列,其长度变化范围为 397~440 bp,A+T所占比例范围为56.48%~68.14%,(A+T)/(G+C)为1.3~2.13,核苷酸序列间相似性范围为 62.6%~97.9%;65条逆转录酶序列被划分为9个家族,其中家族Ⅰ为主要成分;翻译成氨基酸后,序列间相似性范围为12.4%~98.6%,相比核苷酸序列,氨基酸序列表现出更高的异质性;65条逆转录酶序列中有26条发生了无义突变,2份野生种花生材料的无义突变发生率相当;序列间保守基序大体一致,但也发生了一定的变异,呈现出一定的异质性;9个家族所选代表序列的蛋白质结构总体类似,但也在螺旋结构数、折叠结构数、转角数、氢键数、α-螺旋数、β-折叠数上存在差別;系统进化树显示,所有逆转录酶序列被分为13类,A类和B类中包含大部分逆转录酶序列,E类中的5条AA染色体组野生种花生与其他物种植物的逆转录酶序列具有较高相似性,表明这些序列在进化过程中有可能发生了横向传递;通过比对花生EST数据库,发现3条来自Archis duranensis(PI262133)和2条来自A.duranensis(PI219823)的Ty3-gypsy类逆转座子具有转录活性,本研究为下一步分离Ty3-gypsy类逆转座子全长序列、研究其转录转座活性和功能提供序列基础,也为基于Ty3-gypsy类逆转座子的花生属分子标记开发奠定基础。
关键词:野生种花生;Ty3-gypsy类逆转座子;逆转录酶;多样性
中图分类号:S565.201 文献标志码: A
文章编号:1002-1302(2022)09-0043-12
花生被誉为“长生果”,是世界上最重要的油料作物和经济作物之一,也是最重要的植物食用油来源之一[1]。我国是世界花生生产第一大国,种植面积达500万hm2/年以上,约占全球的20%;我国花生产量达1 700万t/年,约占全球的40%,居全球首位,花生占我国油料作物总产(不包括大豆)的46%;花生油约占国产植物油的25%,仅次于菜籽油,是国产植物油的第二大来源[2]。
转座子是指能从同一条染色体或不同染色体上的一个位点转移到另外一个位点的可移动DNA序列[3]。转座子分为逆转座子和DNA转座子,反转录转座子分为长末端重复序列(LTR)逆转座子和非LTR逆转座子,Ty1-copia和Ty3-gypsy是LTR逆转座子的主要两大类型[4-6],这两大类LTR逆转座子的逆转录酶序列可以利用简并PCR技术扩增出来。LTR逆转座子非常适合用来开发成分子标记,序列特异扩增多态性(S-SAP)、逆转座子位点间扩增多态性(IRAP)和逆转座子-微卫星扩增多态性(REMAP)是目前基于LTR逆转座子的主要分子标记技术[7-8]。
MITEs转座子已在花生上被较广泛地研究和应用[9-23],而花生LTR逆转座子的研究报道较少[24-25]。Nielen等先后分离出花生Ty3-gypsy类逆转座子的FIDEL和Ty1-copia类逆转座子的Matita,对其特性和作用进行分析[24-25]。
笔者曾系统对花生LTR逆转座子和MITE转座子的分离及其应用的研究进展进行了归纳[1],也对四倍体野生种花生(Arachis monticola)的Ty1-copia类逆转座子逆转录酶基因进行克隆与分析[26],但尚未见对AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶序列进行分离和多样性分析的报道。
本研究拟分离AA染色体组野生种花生A.duranensis的Ty3-gypsy类逆转座子逆转录酶序列,分析其序列特征和多样性,弄清其序列组成和变异模式及其与其他物种植物之间的系统发育进化关系,为进一步分离其全长序列、研究其转录转座活性和功能提供序列基础,也为基于Ty3-gypsy类逆转座子的花生属分子标记开发和应用奠定基础。
1 材料与方法
1.1 试验材料
2份AA染色体组野生种花生材料分别为A.duranensis(PI262133)和A.duranensis(PI219823)。
1.2 方法
1.2.1 DNA提取 采用改良过的CTAB法[27]提取高质量花生基因组DNA。
1.2.2 逆转录酶序列的PCR扩增 Ty3-gypsy类逆转座子逆转录酶序列的PCR扩增采用前人设计的简并引物,上下游引物序列分别为:Gyrt1,5′-AGMGRTATGTGYGTSGAYTAT-3′;Gyrt2,5′-CAMCCMRAAMWCACAMTT-3′。其中,R=A/G,Y=C/T,M=A/C,S=C/G,W=A/T,N=A/T/C/G[27]。PCR扩增反应体系、扩增程序以及PCR产物的分离检测参考文献[26]。
1.2.3 目的条带的克隆和测序 具体操作参考文献[26]。
1.2.4 逆转录酶序列分析 序列相似性检索、序列统计分析、序列图及Logo图生成、蛋白质的二级结构和三级结构预测、蛋白质三级结构的转角数和氢键数统计、保守基序预测等参考文献[26]。运用MEGA 6.0软件的邻接法(No. of differences模型)构建系统进化树,自展值设置为1 000。与花生EST数据库比对,鉴定具有转录活性的Ty3-gypsy类反转录转座子。B5F2FA2E-8CD4-41A0-93B1-4A7CB397A425
2 结果与分析
2.1 逆转录酶序列的PCR扩增及测序
对2份AA染色体组野生种花生材料的基因组DNA进行PCR扩增,扩增结果(图1)显示,2份材料均扩增出了1条约430 bp大小的目的條带。对目的条带进行回收、克隆和测序,从A.duranensis(PI262133)和A.duranensis(PI219823)中均获得了34条序列。去除重复序列和非目标序列后,分别从A.duranensis(PI262133)和A.duranensis(PI219823)中获得了32、33条逆转录酶序列(分别命名为AdRT3-X和AdRT4-X),对65条序列进行多重比对分析并生成序列logo(图2、图3)。
2.2 逆转录酶序列分析
所有序列长度都在397~440 bp之间。在A.duranensis(PI262133)的32条序列中,AdRT3-23的序列长度最短,为397 bp,AdRT3-27的序列长度最长,为440 bp,有26条序列的长度均为432 bp,占所克隆序列的81.25%(表1);A、T、C、G数量变化范围分别为109~140、107~153、56~86、71~114个,A+T所占比例范围为56.48%~65.97%,A+T与G+C比例为1.3~1.94(表1);核苷酸序列间相似性范围为63.7%~99.3%,其中AdRT3-15与AdRT3-24以及AdRT3-20与AdRT3-30之间的相似性最高,达99.3%,AdRT3-12 与AdRT3-14之间的相似性最低,为63.7%;氨基酸序列间相似性范围为10.9%~99.3%(表2)。在A.duranensis(PI219823)的33条序列中,AdRT4-27的序列长度最短,为428 bp,AdRT4-28的序列长度最长,为440 bp,有27条序列的长度均为432 bp,占所克隆序列的81.82%(1);A、T、C、G数量变化范围分别为117~139、108~157、51~85、77~114个,A+T所占比例范围为56.48%~68.14%,(A+T)/(G+C)为1.3~2.13(表1);核苷酸序列间相似性范围为62.8%~99.3%,其中,AdRT4-5与AdRT4-28之间的相似性最高,达99.3%,AdRT4-1 与AdRT4-23之间的相似性最低,为62.8%,氨基酸序列间相似性范围为31.5%~99.3%(表2);将2份花生材料的65条序列合并进行分析,核苷酸序列间相似性范围为62.6%~97.9%,氨基酸序列间相似性范围为12.4%~98.6%。
2.3 逆转录酶序列聚类分析
遗传进化树(图4)显示,65条序列被划分为9个家族。家族Ⅰ包含20条序列,占总序列数的30.80%,该家族只有2条序列来自A.duranensis(PI262133);家族Ⅱ包含7条序列,只有1条来自A.duranensis(PI219823);家族Ⅲ和家族Ⅳ均只包含1条来自A.duranensis(PI262133)的序列,这2条序列与家族Ⅰ亲缘关系较远而单独聚为一类;家族Ⅴ包含7条序列,有1条来自A.duranensis(PI219823);家族Ⅵ包含9条序列,有2条来自A.duranensis(PI262133);家族Ⅶ包含7条来自A.duranensis(PI262133)和1条来自A.duranensis(PI219823)的序列;家族Ⅷ包含5条序列;家族Ⅸ包含7条序列;除家族Ⅲ和家族Ⅳ外,其他每个家族中都存在来自这2份野生种花生材料的序列。
2.4 逆转录酶氨基酸序列分析
65条序列中有26条存在无义突变,其中有12条序列来自A.duranensis(PI262133),占该材料序列总数的37.5%,有14条序列来自A.duranensis(PI219823),占该材料序列总数的42.42%(图5)。无义突变在2份野生种花生材料序列中的具体表现为AdRT3-7突变数最多,存在14个,分别在第36、第38、第49、第51、第63、第76、第87、第95、第103、第109、第113、第128、第136、第138个氨基酸处;AdRT4-23存在12个无义突变,分别在第32、第59、第72、第77、第87、第88、第96、第100、第108、第121、第128、第138个氨基酸处;AdRT3-3存在9个无义突变,分别在第30、第77、第78、第88、第100、第104、第108、第121、第128个氨基酸处;AdRT4-27存在9个无义突变,分别在第32、第38、第42、第99、第103、第113、第120、第122、第127个氨基酸处;AdRT4-11存在8个无义突变,分别在第8、第77、第88、第101、第104、第108、第121、第128个氨基酸处;AdRT4-30存在8个无义突变,分别在第8、第77、第88、第101、第104、第108、第121、第128个氨基酸处;AdRT3-26存在7个无义突变,分别在第第52、第54、第80、第91、第111、第124、第131个氨基酸处;AdRT3-33存在5个无义突变,分别在第96、第101、第104、第121、第128个氨基酸处;AdRT3-23存在4个无义突变,分别在第86、第93、第102、第127个氨基酸处;AdRT3-6存在4个无义突变,分别在第98、第105、第114、第139个氨基酸处;AdRT4-5(第106、第112、第136个氨基酸处)、AdRT4-17(第62、第105、第106个氨基酸处)和AdRT4-28(第106、第112、第136个氨基酸处)各存在3个无义突变;AdRT3-14(第9、第115个氨基酸处)和AdRT4-14(第32、第129个氨基酸处)各存在2个无义突变;AdRT3-11(第48个氨基酸处)、AdRT3-13(第43个氨基酸处)、AdRT3-17(第130个氨基酸处)、AdRT3-21(第32个氨基酸处)、AdRT3-32(第30个氨基酸处)、AdRT4-12(第131个氨基酸处)、AdRT4-15(第30个氨基酸处)、AdRT4-16(第48个氨基酸处)、AdRT4-18(第9个氨基酸处)、AdRT4-29(第43个氨基酸处)、AdRT4-36(第45个氨基酸处)各存在1个无义突变;部分序列存在连续无义突变的现象。无义突变导致逆转座子失去转录活性。B5F2FA2E-8CD4-41A0-93B1-4A7CB397A425
2.5 逆转录酶的蛋白结构预测
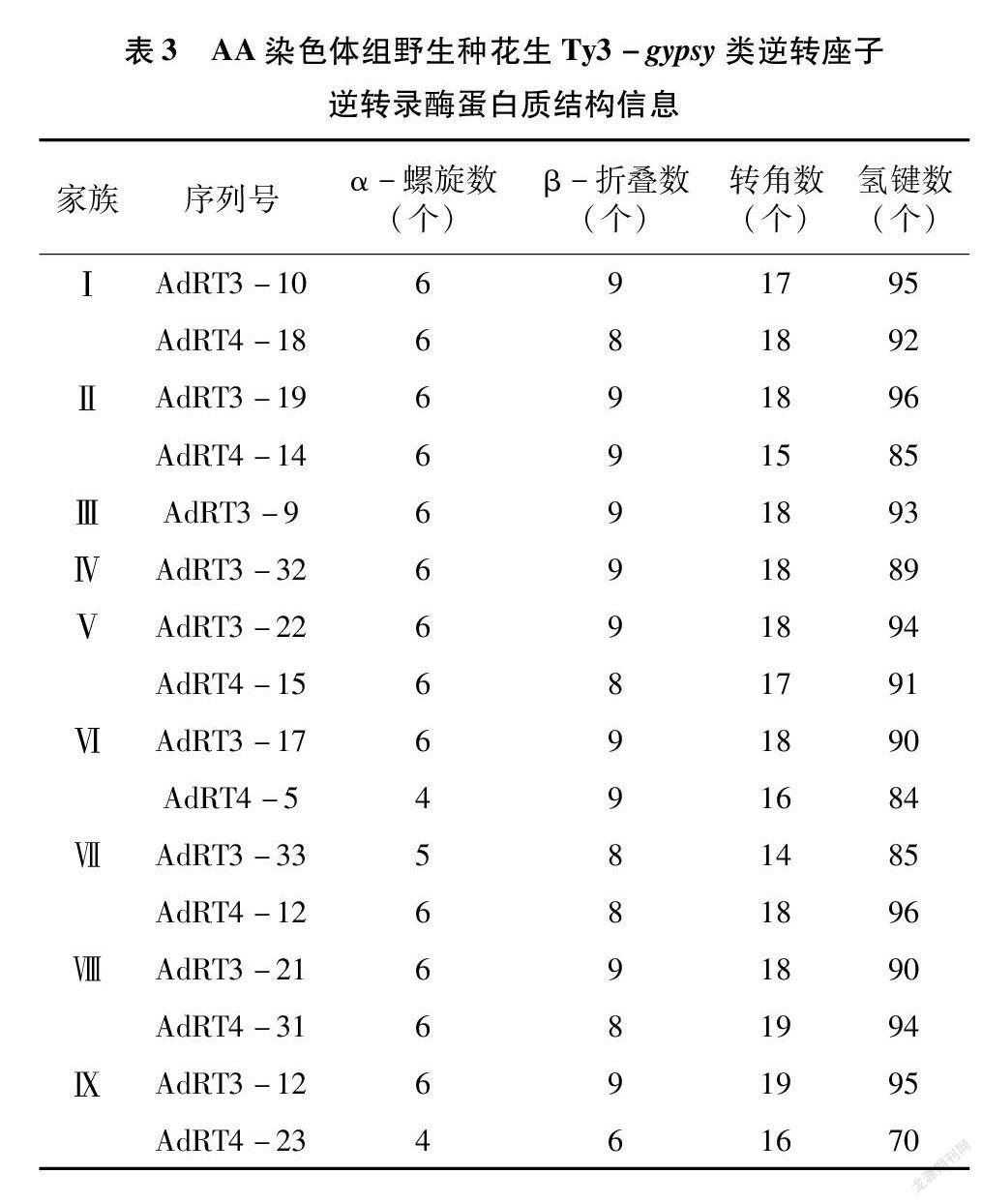
翻译成氨基酸后,根据核苷酸聚类结果,选择2份AA染色体组野生种花生材料中每个家族中的代表序列各1条,家族Ⅲ和家族Ⅳ均只包含1条序列,其选择均来自A.duranensis(PI262133)。利用在线软件Phyre2预测AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶蛋白质的二级结构和三级结构,代表序列蛋白三级结构匹配覆盖度最高的模板为c2opqA、c5dmqA、d2zd1b1和c3kk1B,置信度均为100,都属于逆转录酶家族。二级结构包含4~6个α-螺旋和6~9个β-折叠;三级结构包含 14~19个转角、70~96个氢键,还存在6个明显的螺旋结构和7个明显的折叠结构(表3)。其中,家族Ⅰ代表序列AdRT3-10和AdRT4-18,其蛋白质二级结构和三级结构见图6。
2.6 逆转录酶保守基序预测
65条序列共存在10种保守基序(图7),其中有57条序列包含motif 1,占序列总数的87.69%;除AdRT3-26外,剩余的64条序列均包含motif 2,占序列总数的98.46%;52条序列包含motif 3,占序列总数的80%;62条序列包含motif 4,占序列总数的95.38%;4条序列包含motif 5,5条序列包含motif 6,4条序列包含motif 7,3条序列包含motif 8,7条序列包含motif 9,3条序列包含motif 10。AdRT3-26序列存在7个终止密码子突变,只包含motif 5和motif 9,该序列上游部分无保守基序且与其他序列差异较大,在系统进化树中与其他序列遗传距离最远,单独归为一类;AdRT3-3、AdRT4-11和AdRT4-30同时包含motif 5和motif 7,在系统进化树中为同一类;AdRT3-6、AdRT3-7、AdRT3-23同时包含motif 6和motif 8,在系统进化树中为同一类。
2.7 逆转录酶序列系统进化树构建
从NCBI数据库中下载已登录的来源于其他物种植物的Ty3-gypsy类逆转座子逆转录酶氨基酸序列(表4),与本研究中克隆所获得的65条逆转录酶序列一起构建系统进化树,分析物种以及序列之间的系统进化关系(图8)。进化树显示,所有逆转录酶序列可分为13类。A类和B类中包含大部分AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶序列,不包含来自其他物种植物的逆转录酶序列;C类只有AdRT3-17,D类也只有AdRT4-7。E类包含5条来自A.duranensis(PI262133)和1条来自A.duranensis(PI219823)的逆轉录酶序列,其与来自绿豆(Vigna radiata,AAT85841.1)、黄瓜(Cucumis sativus,ADD83121.1)、山桑(Morus bombycis,BAB40830.1)、落叶松(Larix gmelinii,BAQ22332.1)、牡丹(Paeonia suffruticosa,AFQ94056.1)、火龙果(Hylocereus undatus,AOS58468.1)、李子(Prunus salicina,AGX45501.1)、油棕(Elaeis guineensis,CAD45567.1)、拟南芥(Arabidopsis thaliana,BAB40828.1)、枣(Ziziphus jujuba,AFR43612.1)、白皮松(Phelipanche bungeana,ABD43118.1)、多花黑麦草(Lolium multiflorum,BAB40827.1)、荸荠(Eleocharis uniglumis,ADF46121.1)、苹果(Malus domestica,ABS11067.1)和银杏(Ginkgo biloba,CAA12930.1)等15个物种植物的逆转录酶序列之间具有较高的相似性,亲缘关系较近。Ⅰ类包含来自大豆和菠菜的2条逆转录酶序列,除Ⅰ类外,F类~M类中均是来自2份AA染色体组野生种花生的逆转录酶序列。
2.8 逆转座子转录活性分析
将65条AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶序列提交到NCBI与花生EST数据库进行比对, 以检测AA染色体组野生种花生Ty3-gypsy类逆转座子的转录活性。结果显示,当查询覆盖度都为97%时,AdRT3-7、AdRT3-13、AdRT3-36与花生EST数据库中GO261148.1之间的一致性分别为85.48%、86.26%、85.99%;当查询覆盖度分别为96%和93%时,AdRT4-7、AdRT4-28与花生EST数据库中GO266033.1之间的一致性分别为88.22%和85.68%(表5)。说明GO261148.1和GO266033.1这2条序列为AA染色体组野生种花生Ty3-gypsy类逆转座子的部分转录序列。
3 讨论与结论
本研究首次PCR扩增AA染色体组野生种花生中Ty3-gypsy类逆转座子逆转录酶序列,结果从2份AA染色体组野生种花生材料中均扩增出大小约430 bp的目的条带,这与前人的研究结果[28-33]一致,最终也从2份AA染色体组野生种花生材料中分别获得了32、33条逆转录酶序列。以上说明 Ty3-gypsy 类逆转座子广泛存在于本研究AA染色体组野生种花生材料基因组中,当然也证实了采用简并引物从本研究AA染色体组野生种花生材料中扩增和克隆Ty3-gypsy类逆转座子逆转录酶序列的策略是行之有效的[28]。
所有逆转录酶序列长度在397~440 bp之间,存在缺失或插入突变;2份AA染色体组野生种花生逆转录酶序列的A+T所占比例范围分别为56.48%~65.97%和56.48%~68.14%,(A+T)/(G+C)分别为1.3~1.94和1.3~2.13,均富含 A+T 碱基,A/T碱基含量的增加使序列呈现较高异质性;2份AA染色体组野生种花生逆转录酶核苷酸序列间相似性范围分别为63.7%~99.3%和62.8%~99.3%,呈现较低异质性;翻译后65条逆转录酶序列中有26条发生了无义突变,其中有12条来自A.duranensis(PI262133),有14条来自A.duranensis(PI219823),2份材料的无义突变发生率相当,无义突变导致产生较高的异质性,无义突变也会使基因功能发生改变或丧失;2份AA染色体组野生种花生所有逆转录酶氨基酸序列间相似性范围分别为10.9%~99.3%和31.5%~99.3%,呈现出高度的异质性,相比核苷酸序列,氨基酸序列表现出更高的异质性。B5F2FA2E-8CD4-41A0-93B1-4A7CB397A425
9个家族所选代表序列的蛋白质结构总体类似,但也在螺旋结构数、折叠结构数、转角数、氢键数、α-螺旋数、β-折叠数上存在着差别,如家族Ⅵ中AdRT4-5和Ⅸ中AdRT4-23的α-螺旋数、β- 折叠数和氢键数少于其他代表序列,这些差异可能会影响AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶的转录活性、转座效率及拷贝数,也是AA染色体组野生种花生Ty3-gypsy类逆转座子存在较高异质性和多态性的体现。AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶序列的主要保守基序是4种保守基序(motif 1~motif 4),motif 5~motif 10这6种保守基序在所克隆序列中出现的频率较低,可能是这些序列在进化过程中发生了突变,各序列间保守基序的差异反映了AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶序列具有较高异质性和多态性。
根据聚类分析,所有逆转录酶核苷酸序列被划分为9个家族,其中构成AA染色体组野生种花生Ty3-gypsy类逆转座子的主要成分是家族Ⅰ,表明AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶序列具有较高的保守性与相似性。系统进化树显示,E类中的5条AA染色体组野生种花生逆转录酶序列与绿豆、黄瓜、山桑、落叶松、牡丹、火龙果、李子、油棕、拟南芥、枣、白皮松、多花黑麦草、荸荠、苹果和银杏的逆转录酶序列之间具有较高的相似性,亲缘关系较近,表明这些逆转录酶序列在进化过程中有可能发生了横向传递。A类和B类中包含大部分AA染色体组野生种花生Ty3-gypsy类逆转座子逆转录酶序列,不包含来自其他物种植物的逆转录酶序列,C类只有AdRT3-17,D类也只有AdRT4-7,这4类可能较为特异,有可能只存在于AA染色体组野生种花生中。F类~M类中的逆转录酶序列均来自AA染色体组野生种花生,这些逆转录酶序列不仅与其他物种植物的同源序列遗传距离较远,也与本研究中AA染色体组野生种花生材料的逆转录酶序列遗传距离较远,表明这些逆转录酶序列在起源和进化上可能较为古老,特异性比较强,有可能是本研究AA染色体组野生种花生材料所特有。
通过比对花生EST数据库,发现3条来自A.duranensis(PI262133)的逆转录酶序列与GO261148.1一致性高,发现2条来自A.duranensis(PI219823)的逆转录酶序列与GO266033.1一致性高,表明AA染色体组野生种花生材料中存在可轉录的Ty3-gypsy类逆转座子。结合核苷酸序列的聚类分析,发现这2种来源的序列分别属于家族Ⅴ和家族Ⅵ。
参考文献:
[1]熊发前,刘俊仙,贺梁琼,等. 花生LTR和MITE转座子及其分子标记开发利用研究进展[J]. 分子植物育种,2017,15(2):640-647.
[2]Zhuang W,Chen H,Yang M,et al. The genome of cultivated peanut provides insight into legume karyotypes,polyploid evolution and crop domestication[J]. Nature Genetics,2019,51(5):865-876.
[3]Kumar A,Pearce S R,McLean K,et al. The Ty1-copia group of retrotransposons in plants:genomic organisation,evolution,and use as molecular markers[J]. Genetica,1997,100(1/2/3):205-217.
[4]Kumar A,Bennetzen J L. Plant retrotransposons[J]. Annual Review of Genetics,1999,33:479-532.
[5]Feschotte C,Jiang N,Wessler S R. Plant transposable elements:where genetics meets genomics[J]. Nature Reviews Genetics,2002,3(5):329-341.
[6]Bonchev G,Parisod C. Transposable elements and microevolutionary changes in natural populations[J]. Molecular Ecology Resources,2013,13(5):765-775.
[7]Waugh R,McLean K,Flavell A J,et al. Genetic distribution of Bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (S-SAP)[J]. Molecular & General Genetics,1997,253(6):687-694.
[8]Kalendar R,Grob T,Regina M,et al. IRAP and REMAP:two new retrotransposon-based DNA fingerprinting techniques[J]. Theoretical and Applied Genetics,1999,98(5):704-711.
[9]Patel M,Jung S,Moore K,et al. High-oleate peanut mutants result from a MITE insertion into the FAD2 gene[J]. Theoretical and Applied Genetics,2004,108(8):1492-1502.B5F2FA2E-8CD4-41A0-93B1-4A7CB397A425
[10]Gowda M V C,Bhat R S,Motagi B N,et al. Association of high-frequency origin of late leaf spot resistant mutants with AhMITE1 transposition in peanut[J]. Plant Breeding,2010,129(5):567-569.
[11]Gowda M V C,Bhat R S,Sujay V,et al. Characterization of AhMITE1 transposition and its association with the mutational and evolutionary origin of botanical types in peanut (Arachis spp.)[J]. Plant Systematics and Evolution,2011,291(3/4):153-158.
[12]Shirasawa K,Hirakawa H,Tabata S,et al. Characterization of active miniature inverted-repeat transposable elements in the peanut genome[J]. Theoretical and Applied Genetics,2012,124(8):1429-1438.
[13]Shirasawa K,Koilkonda P,Aoki K,et al. In silico polymorphism analysis for the development of simple sequence repeat and transposon markers and construction of linkage map in cultivated peanut[J]. BMC Plant Biology,2012,12:80.
[14]Shirasawa K,Bertioli D J,Varshney R K,et al. Integrated consensus map of cultivated peanut and wild relatives reveals structures of the A and B genomes of Arachis and divergence of the legume genomes[J]. DNA Research,2013,20(2):173-184.
[15]Mondal S,Hande P,Badigannavar A M.Identification of transposable element markers for a rust (Puccinia arachidis Speg.) resistance gene in cultivated peanut[J]. Journal of Phytopathology,2014,162(7/8):548-552.
[16]Hake A A,Shirasawa K,Yadawad A,et al. Mapping of important taxonomic and productivity traits using genic and non-genic transposable element markers in peanut (Arachis hypogaea L.)[J]. PLoS One,2017,12(10):e0186113.
[17]Gayathri M,Shirasawa K,Varshney R K,et al. Development of AhMITE1 markers through genome-wide analysis in peanut (Arachis hypogaea L.)[J]. BMC Research Notes,2018,11(1):10.
[18]王 洁,李双铃,王 辉,等. 利用AhMITE1转座子分子标记鉴定花生F 1代杂种[J]. 花生学报,2012,41(2):8-12.
[19]王 辉,李双铃,任 艳,等. 利用AhMITE转座子分子标记研究花生栽培种及高世代材料的亲缘关系[J]. 農业生物技术学报,2013,21(10):1176-1184.
[20]尹 亮,任 艳,石延茂,等. 利用AhMITE1转座子分子标记鉴定栽培花生杂交F 1代种子真伪[J]. 山东农业科学,2015,47(12):1-5.
[21]吴 琪,曹广英,尹 亮,等. 利用AhMITE转座子分子标记鉴定花生杂交F 1代真假杂种[J]. 花生学报,2017,46(3):48-53,60.
[22]刘 婷,王传堂,唐月异,等. 利用近红外技术和转座子标记鉴定花生杂交F 1真杂种[J]. 分子植物育种,2017,15(9):3592-3598.
[23]许梦琦,李双铃,任 艳,等. 花生作图亲本间分子标记的多态性分析[J]. 湖北农业科学,2015,54(11):2763-2766.
[24]Nielen S,Campos-Fonseca F,Leal-Bertioli S,et al. FIDEL-a retrovirus-like retrotransposon and its distinct evolutionary histories in the A-and B-genome components of cultivated peanut[J]. Chromosome Research,2010,18(2):227-246.B5F2FA2E-8CD4-41A0-93B1-4A7CB397A425
[25]Nielen S,Vidigal B S,Leal-Bertioli S C M,et al. Matita,a new retroelement from peanut:characterization and evolutionary context in the light of the Arachis A-B genome divergence[J]. Molecular Genetics and Genomics,2012,287(1):21-38.
[26]阳太亿,刘俊仙,刘 菁,等. 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因的克隆与分析[J]. 山东农业科学,2019,51(9):9-20.
[27]熊发前,刘俊仙,刘 菁,等. 花生DNA的五种改良CTAB提取方法的比较分析及其应用[J]. 分子植物育种,2019,17(7):2207-2216.
[28]Kumekawa N,Ohtsubo E,Ohtsubo H.Identification and phylogenetic analysis of Gypsy-type retrotransposons in the plant kingdom[J]. Genes & Genetic Systems,1999,74(6):299-307.
[29]王慶竹,李慧平,文晓鹏,等. 桂花LTR类反转录转座子RT序列的克隆及分析[J]. 园艺学报,2018,45(2):309-320.
[30]侯小改,郭大龙,黄燕梅,等. 牡丹Ty3-gypsy类反转录转座子反转录酶序列的克隆及分析[J]. 园艺学报,2013,40(1):98-106.
[31]彭 磊,吴 艳,刘小翠,等. 火龙果Ty3-gypsy类反转录转座子反转录酶序列的克隆及分析[J]. 果树学报,2017,34(2):186-195.
[32]张文波,陈 凌,李雪辉,等. 兴安落叶松Ty3-gypsy类逆转座子逆转录酶的多样性分析[J]. 分子植物育种,2016,14(5):1098-1106.
[33]白 杨,林晓飞,张文波.杂交构树Ty3-gypsy类逆转座子逆转录酶序列的多样性分析[J]. 分子植物育种,2018,16(22):7429-7437.B5F2FA2E-8CD4-41A0-93B1-4A7CB397A425
