四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因的克隆与分析
2019-12-09阳太亿刘俊仙刘菁蒋菁韩柱强贺梁琼唐秀梅钟瑞春黄志鹏吴海宁唐荣华熊发前
阳太亿 刘俊仙 刘菁 蒋菁 韩柱强 贺梁琼 唐秀梅 钟瑞春 黄志鹏 吴海宁 唐荣华 熊发前


摘要:克隆與分析四倍体野生种花生的Ty1-copia类逆转座子逆转录酶基因序列,可为研究其转录活性、功能及调控提供序列基础。使用根据保守区设计的简并引物对,利用PCR技术对四倍体野生种花生(Arachis monticola)的基因组DNA进行扩增,经回收、克隆和测序,对目的序列进行生物信息学分析。结果显示,目的条带大小约260 bp,克隆获得了32条逆转录酶序列,序列长度范围为257~269 bp,A+T所占比例范围为54.47%~68.77%,A+T与G+C比例为1.20~2.20。核苷酸序列间相似性范围为46.4%~98.9%,存在较高异质性,表现为缺失突变与点突变;氨基酸序列间相似性范围为6.3%~100%,有12条序列发生了终止密码子突变,呈现高度异质性。绝大部分序列的保守基序一致,但各序列间的保守基序也存在一定差异,呈现一定程度的异质性。32条序列系统聚类为4个家族,家族Ⅰ和家族Ⅲ分别占总序列数的31.25%和37.50%。对四倍体野生种花生与其它物种植物同一类型逆转录酶的氨基酸序列构建系统发育进化树,结果显示所有序列被分为6类,其中,Ⅰ类和Ⅱ类分别包含15条和9条四倍体野生种花生逆转录酶序列,表明四倍体野生种花生逆转录酶序列具有比较高的保守性;同时四倍体野生种花生逆转录酶序列与葡萄、拟南芥、马铃薯、野茶树、辣椒、甜菜、烟草、番茄、绿豆以及欧洲云杉等具有较近的亲缘关系,表明它们之间可能存在横向传递。本研究获得的逆转录酶序列为基于LTR逆转座子的花生属分子标记开发和应用奠定了基础。
关键词:花生;四倍体野生种;Ty1-copia类逆转座子;逆转录酶;异质性
中图分类号:S565.2:Q785文献标识号:A文章编号:1001-4942(2019)09-0009-13
Cloning and Analysis of Reverse Transcriptase of
Ty1-copia-like Retrotransposons in Arachis monticola
Yang Taiyi, Liu Junxian*, Liu Jing, Jiang Jing, Han Zhuqiang, He Liangqiong, Tang Xiumei,
Zhong Ruichun, Huang Zhipeng, Wu Haining, Tang Ronghua, Xiong Faqian
(Institute of Economic Crops, Guangxi Academy of Agricultural Sciences, Nanning 530007, China)
Abstract The reverse transcriptase sequences of Ty1-copia retrotransposons from tetraploid wild peanut species were cloned and analyzed, which could provide sequence basis for studying its transcriptional activity, function and regulation. The genomic DNA of Arachis monticola was amplified by PCR using a pair of degenerate primers designed according to the conservative region of reverse transcriptases of Ty1-copia retrotransposons. Then the target band was recovered, cloned and sequenced and the obtained sequences were analyzed through the bioinformatics method. The results showed that the size of the target band was approximately 260 bp and thirty-two reverse transcriptase sequences were obtained. The length of sequences ranged from 257 bp to 269 bp. The proportion of A+T ranged from 54.47% to 68.77%. The ratio of A+T to G+C was 1.20~2.20. The similarity between nucleotide sequences ranged from 46.4% to 98.9%. These indicated that the nucleotide sequences of reverse transcriptases existed higher heterogeneity in the form of deletion mutation and point mutation. The similarity between amino acid sequences ranged from 6.3% to 100% and twelve sequences had termination codon mutations, which indicated that the amino acid sequences of reverse transcriptase existed high heterogeneity. The conservative motifs of most sequences were identical, but also there were some differences between them, showing a certain degree of heterogeneity. The thirty-two reverse transcriptase sequences were clustered into four families. FamilyⅠand Family Ⅲ accounted for 31.25% and 37.50% of the total sequences, respectively. Phylogenetic tree was constructed according to the amino acid sequences of the same type of reverse transcriptases in tetraploid wild peanut species and some other plant species. The results showed that all sequences were classified into six categories. Among them, ClassⅠand ClassⅡ contained fifteen and nine reverse transcriptase sequences from tetraploid wild peanut species, respectively, which showed that the reverse transcriptase sequences of tetraploid wild peanut species were highly conservative. Meanwhile, the reverse transcriptase sequences of tetraploid wild peanut species had close relationship with grape, Arabidopsis, potato, Camellia sinensis, pepper, sugarbeet, tobacco, tomato, mung bean and Picea abies. It indicated that horizontal transmission might occur between these plant species. The reverse transcriptase sequences obtained in this study laid a foundation for the development and application of molecular markers in Arachis based on LTR retrotransposons.
Keywords Peanut; Tetraploid wild species; Ty1-copia-like retrotransposons; Reverse transcriptase; Heterogeneity
转座子是指基因组中能从一条染色体跳跃到另外一条染色体上或从同一条染色体的某个位点跳跃到另外一个位点的可移动DNA序列[1]。逆转座子和DNA转座子是转座子的两组类型,其中,逆转座子根据5′和3′端是否含有约100~5 000 bp大小的长末端重复序列划分为LTR逆转座子和非LTR逆转座子,LTR逆转座子根据其编码区基因排列次序的不同分为Ty1-copia和Ty3-gypsy[2-4]。根据LTR逆转座子序列中逆转录酶基因的保守区,可以通过简并PCR技术扩增克隆出逆转录酶基因序列[5,6]。LTR逆转座子的插入位点多态性、高拷贝数、高度异质性等特点使其非常适合用来开发分子标记,目前,基于LTR逆转座子的分子标记技术主要有逆转座子位点间扩增多态性(IRAP)、逆转座子-微卫星扩增多态性(REMAP)和序列特异扩增多态性(S-SAP)[7,8]。
近年来逐渐兴起对花生转座子的研究。相比LTR逆转座子,MITEs转座子在花生上最早也最广泛地被研究,其研究主要集中在利用AhMITE1s转座子检测栽培种花生的DNA多态性和鉴定杂交F1代杂种的真实性上[9-23]。另外,也有研究结果表明MITEs转座子分子标记揭示出栽培种花生的DNA多态性比SSR分子标记和其它类型分子标记高[12,13]。
有关花生LTR逆转座子的研究报道较少。在国外,Nielen 等[24,25]先后克隆出花生Ty3-gypsy类全长逆转座子的FIDEL和Ty1-copia类全长逆转座子的Matita,对其拷贝数、染色体分布和系统进化进行了分析并探讨了在花生基因组进化和多样性形成过程中扮演的角色。在国内,只有熊发前等[26]系统对花生LTR逆转座子和MITE转座子的分离及其分子标记开发利用的研究进展进行了总结,尚未见对花生属LTR逆转座子逆转录酶基因序列进行克隆的报道,也未见克隆四倍体野生种Ty1-copia类逆转座子逆转录酶基因序列的报道。
本研究拟克隆四倍体野生种花生A. monticola的Ty1-copia类逆转座子逆转录酶基因序列,并对序列特征及多样性进行分析,调查该类逆转录酶的序列组成和变异模式及其与其它物种植物之间的系统进化关系,为进一步研究其转录转座活性、功能及调控提供序列基础,也为基于LTR逆转座子的花生属分子标记开发和应用奠定基础。
1 材料与方法
1.1 材料
本研究所用试验材料为四倍体野生种花生A. monticola(PI468199)。种子进行田间播种育苗,随机摘取10株健康植株的顶端芯叶,立即放入冰袋中,带回实验室于-70℃超低温冰箱保存备用。
1.2 试验方法
1.2.1 基因组DNA的提取 通过使用建立的改良CTAB法[27]提取四倍体野生种花生基因组DNA,DNA纯度和浓度分别通过1.2%琼脂糖凝胶电泳和微量分光光度计(NanoDrop)检测,最后将DNA工作液浓度统一调至50 ng/μL,-20℃下保存备用。
1.2.2 逆转录酶基因的PCR扩增 采用Kumar等[1]设计的简并引物,进行PCR扩增,上游引物,RTp1:5′-ACNGCNTTYYTNCAYGG-3′;下游引物,RTp2:5′-ARCATRTCRTCNACRTA-3′,其中,N=A/T/C/G,Y=C/T,R=A/G。PCR扩增体系和程序参照文献[27]。PCR结束后,产物中加入4 μL上样缓冲液并充分混匀,取6 μL扩增产物在1.2%琼脂糖凝胶中电泳分离,缓冲液为1×TAE,经EB(溴化乙锭)染色后在凝胶成像系统下拍照并保存。
1.2.3 逆转录酶基因片段的回收、克隆及测序 用琼脂糖凝胶回收试剂盒对目的条带进行回收,回收产物连接到pMD18-T载体上,转化大肠杆菌DH5α感受态细胞,使用氨苄青霉素、半乳糖苷酶和底物X-gal并通过蓝白斑筛选,在平板上37℃培养12 ~ 16 h,挑取单克隆白斑于800 μL的LB液体培养基(含50 mg/L氨苄青霉素)中培养4 ~ 6 h,进行菌液PCR鉴定,将初步鉴定为阳性克隆的菌液送至测序公司进行一代测序[27]。
1.2.4 逆转录酶基因序列分析 运用NCBI中的blastn和blastx对逆转录酶基因序列进行同源性检索。综合运用BioEdit和DNAMAN软件对逆转录酶基因序列进行分析。利用Jalview软件生成序列图和在线工具Weblogo(http://weblogo.threeplusone.com/)獲得序列的Logo图。利用在线程序Phyre2(http://www.sbg.bio.ic.ac.ukphyre2/html/)预测逆转录酶蛋白质的二级结构与三级结构,同时利用RasMol软件统计蛋白质结构的转角数和氢键数。利用MEME(http://meme-suite.org/)在线软件预测逆转录酶氨基酸序列的保守基序。运用MEGA 6.0软件邻接法构建系统发育进化树。
2 结果与分析
2.1 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列的PCR扩增及测序
利用根据Ty1-copia类逆转座子逆转录酶基因序列保守区域设计的简并引物,对四倍体野生种花生A. monticola的基因组DNA进行PCR扩增,扩增出的目的条带大小约为260 bp(图1),表明Ty1-copia类逆转座子存在于四倍体野生种花生A. monticola中。
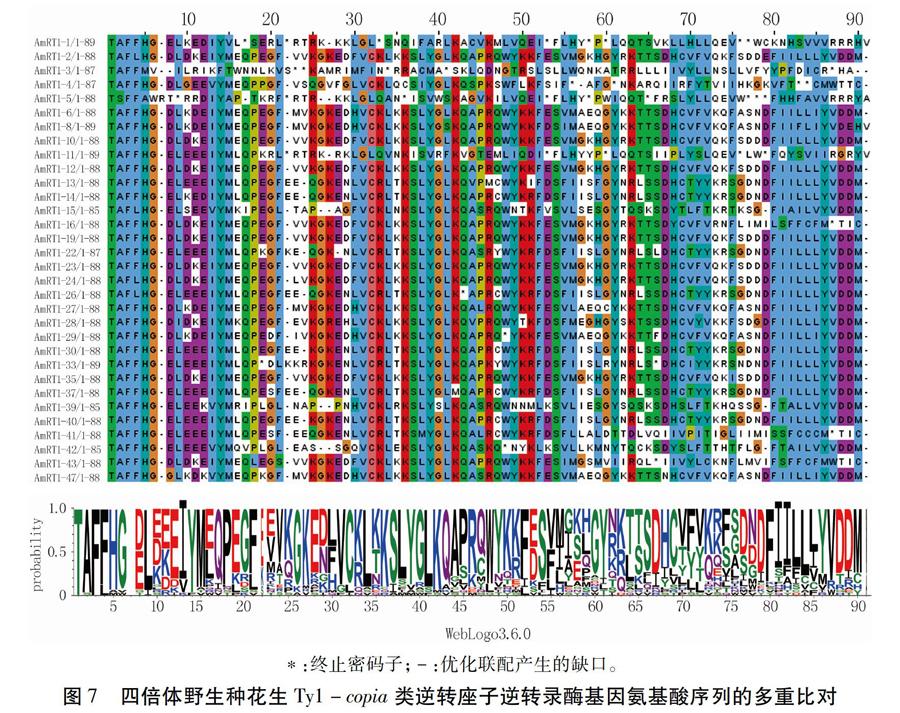
对目的条带进行回收、克隆和测序,共获得46条序列。利用DNAMAN软件等去除46条序列中的重复序列,利用NCBI数据库对序列进行同源性分析去除非目标序列,最后共获得32条非重复的逆转录酶基因序列,将每条序列按照AmRT1-X的形式进行命名,并提交到GenBank(登录号:MK775559 ~ MK775590)。对32条逆转录酶序列进行多重比对(图2),为展示碱基在每个位置上的保守性,利用在线工具Weblogo生成Logo图(图3)。将32条序列递交到NCBI(http://blast.ncbi.nlm.nih.gov/)数据库进行序列比对,结果表明与数据库中其它物种植物Ty1-copia类逆转座子逆转录酶基因序列具有同源性,说明已成功克隆到四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列。
2.2 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列的相似性分析
利用BioEdit软件对四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列信息进行统计,序列长度在257~269 bp之间,其中,AmRT1-33序列最长,AmRT1-15、AmRT1-39和AmRT1-42序列最短,所有序列长度都小于Voytas等[5]报道的273 bp,均存在不同程度的缺失(4~16 bp),说明缺失突变是造成四倍体野生種花生逆转录酶基因序列长度变化的主要原因。32条序列的A、T、C、G数量变化范围分别为67~97、70~94、31~56、44~68,A+T所占比例范围为54.47%~68.77%,G+C所占比例范围为31.23%~45.53%,A+T与G+C比例为1.20~2.20(表1)。
核苷酸序列间相似性范围为46.4%~98.9%,其中AmRT1-10与AmRT1-23之间的相似性最高,为98.9%,AmRT1-4与AmRT1-1之间的相似性最低,为46.4%;氨基酸序列间的相似性范围为6.3%~100%,与核苷酸序列间的相似性并不对应(表2)。由此可知,利用同一对简并引物从同一种质中克隆出来的Ty1-copia类逆转座子逆转录酶基因序列存在较大差异,在序列长度、碱基变化和序列相似性上存在多态性,呈现较高异质性。
2.3 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列的聚类分析
为了阐明四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列之间的亲缘关系,利用MEGA 6.0软件邻接法构建系统发育进化树(图4)。结果显示,32条序列分为4个家族,其中,家族Ⅰ包含10条序列,家族Ⅱ包含6条序列,家族Ⅲ包含12条序列,家族Ⅳ包含4条序列,家族Ⅳ与其它3个家族遗传距离最大,单独聚为一类。家族Ⅰ和家族Ⅲ中的序列数分别占总序列数的31.25%和37.50%。家族内逆转录酶序列长度大部分较一致,差异主要表现为不同程度的点突变与碱基替换;各家族之间的差异较大,差异主要表现为不同程度的缺失突变。总之,各家族内及家族间的差异主要表现为不同程度的点突变、碱基替换和缺失突变,它们很有可能是造成四倍体野生种花生Ty1-copia逆转座子产生较高异质性及拥有多拷贝的重要原因。
2.4 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因的氨基酸序列分析
翻译成氨基酸后,选择核苷酸聚类中各家族具有代表性的氨基酸序列进行分析,利用在线程序Phyre2预测Ty1-copia类逆转座子逆转录酶蛋白质的二级结构(图5)与三级结构(图6)。结果显示,所有序列中的蛋白结构包含2个α-螺旋、4或5个β-折叠及5或6个转角以及25 ~ 32氢键(表3)。在蛋白质三级结构中存在1个明显的螺旋结构和2个不明显的折叠结构,说明螺旋结构是该蛋白的大量结构元件(蓝色端为C端,红色端为N端)。
利用MEGA 6.0软件对32条逆转录酶氨基酸序列进行分析。从图7中可以看出,32条序列中有12条发生了1 ~ 9个终止密码子突变,其中,AmRT1-5的终止密码子突变最多,分别在第9、21、33、50、55、62、74、75、76个氨基酸处;AmRT1-1序列有8个终止密码子突变,分别在第16、21、31、51、56、58、74、75个氨基酸处;AmRT1-3序列有5个终止密码子突变,分别在第21、22、32、39、85个氨基酸处;AmRT1-11在第21、51、58、74、77个氨基酸处;AmRT1-4有4个终止密码子突变,分别在第54、58、80、81个氨基酸处;AmRT1-33在第18、65个氨基酸处发生了2次终止密码子突变;有6条序列发生了1次终止密码子突变,分别是AmRT1-16(第85个氨基酸)、AmRT1-26(第41个氨基酸)、AmRT1-29(第46个氨基酸)、AmRT1-41(第85个氨基酸)、AmRT1-42(第44个氨基酸)、AmRT1-43(第64个氨基酸)。AmRT1-5连续发生了3个终止密码子突变,AmRT1-1、AmRT1-3和AmRT1-4连续发生了2个终止密码子突变。终止密码子突变导致了逆转录转座子失去转录活性,推测终止密码子突变是四倍体野生种花生Ty1-copia类逆转座子产生多拷贝和较高异质性的重要原因。
2.5 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因蛋白保守基序分析
利用在线软件MEME(http://meme-suite.org/)预测四倍体野生种花生Ty1-copia类逆转座子逆转录酶氨基酸序列的保守基序,结果(图8)显示32条序列共存在8种保守基序,其中包含motif 1的序列有28条,占所有序列数的87.50%;含有motif 2 的序列有30条,占所有序列数的93.75%;含motif 3的序列有24条,占所有序列数的75.00%;同时包含motif 1、motif 2和motif 3的序列有24条,占所有序列的75.00%;同时包含motif 5和motif 7的序列只有3条,分别是AmRT1-1、AmRT1-5和AmRT1-11;包含motif 6的序列仅有3条,分别是AmRT1-16、AmRT1-41和AmRT1-43;AmRT1-3和AmRT1-5拥有1个共同的保守基序即motif 8:5′-XRRACMA-3′;AmRT1-3序列只含有一个保守基序motif 8。
2.6 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列的系统发育进化树构建
将四倍体野生种花生与其它物种植物(表4)的逆转录酶氨基酸序列进行多重比对并构建系统发育进化树,分析四倍体野生种花生与其它物种植物Ty1-copia类逆转座子逆转录酶序列间的进化关系。根据进化树(图9)可将所有逆转录酶氨基酸序列分成6类,AmRT1-4单独归为一类;Ⅰ类分别包含15条四倍体野生种花生和15条其它物种植物的逆转录酶序列,说明四倍体野生种花生的15条逆转录酶序列与葡萄(Vitis vinifera, CAN72446.1)、拟南芥(Arabidopsis thaliana, S71291)、马铃薯(Solanum tuberosum, CAA13065.1)、野茶树(Camellia sinensis, CAJ09751.1)、辣椒(Capsicum annuum, AFS89513.1)、甜菜(Beta vulgaris, T14589)、烟草(Nicotania tabacum, AAA03507.1)、番茄(Lycopersicon esculentum, AF072638.1)、绿豆(Vigna radiata, AAT90494.1)以及欧洲云杉(Picea abies, O49918)等之间具有较高相似性,亲缘关系较近;Ⅲ类中有3条四倍体野生种花生逆转录酶序列与铁皮石斛(Dendrobium officinale, AIL54325.1)和草莓(Fragaria × ananassa, ACZ81628.1)之间具有较高相似性,亲缘关系较近;Ⅴ类和Ⅵ类中分别
含来自四倍体野生种花生的1条和4条逆转录酶序列,这两类序列与其它四倍体野生种花生Ty1-copia类逆转座子逆转录酶序列亲缘关系最远。
3 讨论与结论
本研究首次对四倍体野生种花生Ty1-copia类逆转座子逆转录酶序列进行PCR扩增,最终扩增出约260 bp大小的目的条带。回收、克隆和测序目的片段,获得46条序列,经去除非逆转录酶序列和重复序列后得到32条真实的逆转录酶序列。以上说明该类逆转座子存在于四倍体野生种花生基因组中,也证实了采用简并引物从四倍体野生种花生基因组中扩增和克隆Ty1-copia类逆转座子逆转录酶序列的策略是行之有效的[1]。
32条逆转录酶序列的长度变化范围为257~269 bp,所有序列长度都小于Voytas等[5]报道的273 bp,说明缺失突变是造成四倍体野生种花生逆转录酶基因序列长度变化的主要原因。A+T所占比例在54.47%~68.77%之间,A+T与G+C比例为1.20~2.20,高A+T碱基含量导致较高异质性。核苷酸序列间相似性范围为46.4%~98.9%,呈现较高的异质性;相比核苷酸序列,氨基酸序列呈现出更高的异质性。绝大部分序列的保守基序一致,但各序列间的保守基序也存在一定差异。32条序列中有12条序列发生终止密码子突变,最多的一条序列出现9个终止密码子,终止密码子使得基因功能丧失或改变,推测终止密
码子突变可能是造成逆转座子较高异质性的重要原因。逆转座子的异质性产生原因包括以下5点:(1)在转座过程中,逆转座子易发生突变。逆转座子的转座和增殖是以DNA到RNA再到DNA的形式进行,期间需要逆转录酶的辅助,但在增殖过程中逆转录酶缺乏高保真性和错读校对功能[28],跟DNA聚合酶相比,逆转录酶的碱基错配率高10 000倍以上[29],造成突变几率急剧上升,从而导致突变在增殖过程中不断积累;(2)碱基自然突变和同源重组。基因组在整个进化过程中,逆转座子会频繁地发生自然突变,或与其自身及其它物种的基因组发生同源重组,从而导致胞嘧啶的甲基化和胸腺嘧啶含量的增高,提高了DNA可塑性,使得DNA序列变异的几率增加[30];(3)寄主的防御机制导致逆转座子发生变异以适应寄主[31,32];(4)逆转座子的垂直传递。垂直传递是指所有逆转座子都整合分布于染色体中,随着世代的更替,从亲代遗传给子代,随之上述发生的这些突变也被传递到下一代,世代间不断积累[33,34]。(5)逆转座子的水平转移。水平转移也能导致产生异质性[35],水平转移现象曾在水稻和狗尾草属植物之间发生过[36]。
对32条四倍体野生种花生逆转录酶核苷酸序列进行聚类分析,遗传进化树显示,32条序列分成4个家族。家族Ⅰ和家族Ⅲ是构成四倍体野生种花生Ty1-copia类逆转座子的主要成分,表明该物种的逆转录酶序列具有较高保守性。不同家族包含拷贝数成员的不同,反映了不同家族之间转座机制的不同,其存在的历史地位以及对四倍体野生种花生基因组进化的作用也可能不同。各家族内部成员数量越多,序列间相似性越高,其转座发生时间也可能越近,存在有转录活性的逆转座子的可能性也越大[37],从而推测具有转录活性的Ty1-copia类逆转座子很可能存在于家族Ⅰ和家族Ⅲ中。
四倍体野生种花生逆转录酶各家族序列的蛋白质三级结构整体构型基本类似,但在螺旋结构数、折叠结构数、转角数和氢键数上存在细微差别,推测这些不同会影响Ty1-copia类逆转录转座子逆转录酶的转录活性、转座效率以及LTR逆转录转座子的拷贝数。
四倍体野生种花生Ty1-copia类逆转座子逆转录酶氨基酸序列的保守基序预测显示,motif 1、motif 2和motif 3在所有序列中的比例较大,表明这三种保守基序是四倍体野生种花生Ty1-copia类逆转座子逆转录酶氨基酸的主要基序,也表明四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列具有较高保守性。各序列之间的保守基序也存在一定差异,这在一定程度上体现了四倍体野生种花生Ty1-copia逆转座子的多态性及较高异质性。AmRT1-1、AmRT1-3、AmRT1-5和AmRT1-11的保守基序跟其它序列差異较大,其中AmRT1-1、AmRT1-5和AmRT1-11在核苷酸聚类分析中归属家族Ⅲ,在氨基酸系统发育进化树中归属家族Ⅵ。
从构建的四倍体野生种花生和其它物种植物的Ty1-copia类逆转座子逆转录酶氨基酸序列的系统发育进化树中可以看出,Ⅰ类和Ⅱ类分别包含15条和9条四倍体野生种花生逆转录酶序列,分别占总序列数的46.88%和28.13%,表明四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因序列具有较高保守性。Ⅰ类中的15条四倍体野生种花生逆转录酶序列与葡萄、拟南芥等具有较高相似性,亲缘关系较近;Ⅲ类中的3条四倍体野生种花生逆转录酶序列与铁皮石斛和草莓之间具有较高相似性,亲缘关系较近;以上暗示它们可能具有共同的起源,也可能是四倍体野生种花生Ty1-copia类逆转座子在进化过程中与这些物种植物发生过横向传递。Ⅴ类和Ⅵ类中分别包含四倍体野生种花生的1条和4条逆转录酶序列,与其它四倍体野生种花生Ty1-copia类逆转座子逆转录酶序列亲缘关系最远,说明这两类序列在起源和进化上较为古老,特异性较强,极有可能为四倍体野生种花生所特有,也反映了该两类序列在四倍体野生种花生进化过程中有着不同的历史地位。
本研究从四倍体野生种花生中成功分离出Ty1-copia类逆转座子逆转录酶基因序列,为下一步分离其全长序列、研究其转录转座活性和功能提供序列基础,对花生属基于LTR反转录转座子的分子标记开发及花生分子育种具有重要意义。
参 考 文 献:
[1]Kumar A, Pearce S R, Mclean K, et al. The Tyl-copia group of retrotransposons in plants: genomic organisation, evolution and use as molecular markers[J].Genetica, 1997, 100(1/2/3): 205-217.
[2]Kumar A, Bennetzen J L. Plant retrotransposons: annual review of genetics[J].1999, 33: 479-532.
[3]Feschotte C, Jiang N, Wessler S R. Plant transposable elements: where genetics meets genomics[J].Nature Reviews Genetics, 2002, 3(5): 329-341.
[4]Bonchev G, Parisod C. Transposable elements and microevolutionary changes in natural populations[J].Molecular Ecology Resources, 2013, 13(5): 765-775.
[5]Voytas D F, Cummings M P, Konieczny A, et al. Copia-like retrotransposons are ubiquitous among plants[J].Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(15): 7124-7128.
[6]Kumekawa N, Ohtsubo E, Ohtsubo H. Identification and phylogenetic analysis of gypsy-type retrotransposons in the plant kingdom[J].Genes & Genetic Systems, 1999, 74(6): 299-307.
[7]Kalendar R, Grob T, Regina M, et al. IRAP and REMAP: two new retrotransposon-based DNA fingerprinting techniques[J].Theoretical and Applied Genetics, 1999, 98(5): 704-711.
[8]Waugh R, Mclean K, Flavell A J, et al. Genetic distribution of bare-1-like retrotransposable elements in the barley genome revealed by sequence-specific amplification polymorphisms (S-SAP)[J].Molecular and General Genetics, 1997, 253(6): 687-694.
[9]Patel M, Jung S, Moore K, et al. High-oleate peanut mutants result from a MITE insertion into the FAD2 gene[J].Theoretical and Applied Genetics, 2004, 108(8): 1492-1502.
[10]Gowda M V C, Bhat R S, Motagi B N, et al. Association of high-frequency origin of late leaf spot resistance mutants with AhMITE1 transposition in peanut[J].Plant Breeding, 2010, 129(5): 567-569.
[11]Gowda M V C, Bhat R S, Sujay V, et al. Characterization of AhMITE1 transposition and its association with the mutational and evolutionary origin of botanical types in peanut (Arachis spp.)[J].Plant Systematics and Evolution, 2011, 291(3/4): 153-158.
[12]Shirasawa K, Hirakawa H, Tabata S, et al. Characterization of active miniature inverted-repeat transposable elements in the peanut genome[J].Theoretical and Applied Genetics, 2012, 124 (8): 1429-1438.
[13]Shirasawa K, Koilkonda P, Aoki K, et al. In silico polymorphism analysis for the development of simple sequence repeat and transposon markers and construction of linkage map in cultivated peanut[J].BMC Plant Biology, 2012, 12(1): 80-92.
[14]Shirasawa K, Bertioli D J, Varshney R K, et al. Integrated consensus map of cultivated peanut and wild relatives reveals structures of the A and B genomes of arachis and divergence of the legume genomes[J].DNA Research, 2013, 20(2): 173-184.
[15]Mondal S, Hande P, Badigannavar A M. Identification of transposable element markers for a rust (Puccinia arachidis Speg.) resistance gene in cultivated peanut[J].Journal of Phytopathology, 2014, 162(7/8): 548-552.
[16]Hake A A, Shirasawa K, Yadawad A, et al. Mapping of important taxonomic and productivity traits using genic and non-genic transposable element markers in peanut (Arachis hypogaea L.)[J].PLoS ONE, 2017, 12(10): e0186113.
[17]Gayathri M, Shirasawa K, Varshney R K, et al. Development of AhMITE1 markers through genome-wide analysis in peanut (Arachis hypogaea L.)[J].BMC Research Notes, 2018, 11(1): 10-15.
[18]王洁, 李双铃, 王辉, 等. 利用AhMITE1转座子分子标记鉴定花生F1代杂种[J].花生学报, 2012, 41(2): 8-12.
[19]王辉, 李双铃, 任艳, 等. 利用AhMITE转座子分子标记研究花生栽培种及高世代材料的亲缘关系[J].农业生物技术学报, 2013, 21(10): 1176-1184.
[20]尹亮, 任艳, 石延茂, 等. 利用AhMITE1转座子分子标记鉴定栽培花生杂交F1代种子真伪[J].山东农业科学, 2015, 47(12): 1-5.
[21]许梦琦, 李双铃, 任艳, 等. 花生作图亲本间分子标记的多态性分析[J].湖北农业科学, 2015, 54(11): 2763-2766.
[22]吴琪, 曹广英, 尹亮, 等. 利用AhMITE轉座子分子标记鉴定花生杂交F1代真假杂种[J].花生学报, 2017, 46(3): 48-53.
[23]刘婷, 王传堂, 唐月异, 等. 利用近红外技术和转座子标记鉴定花生杂交F1真杂种[J].分子植物育种, 2017, 15(9): 3592-3598.
[24]Nielen S, Campos-Fonseca F, Leal-Bertioli S, et al. FIDEL-a retrovirus-like retrotransposon and its distinct evolutionary histories in the A- and B-genome components of cultivated peanut[J].Chromosome Research, 2010, 18(2): 227-246.
[25]Nielen S, Vidigal B S, Leal-Bertioli S C M, et al. Matita, a new retroelement from peanut: characterization and evolutionary context in the light of the Arachis A-B genome divergence[J].Molecular Genetics and Genomics, 2012, 287(1): 21-38.
[26]熊发前, 刘俊仙, 贺梁琼, 等. 花生LTR和MITE转座子及其分子标记开发利用研究进展[J].分子植物育种, 2017, 15(2): 640-647.
[27]熊发前, 刘俊仙, 刘菁, 等. 花生DNA的五种改良CTAB提取方法的比较分析及其应用[J].分子植物育种, 2019, 17(7): 2207-2216.
[28]Steinhauer D A, Holland J J. Direct method for quantitation of extreme polymerase error frequencies at selected single base sites in viral RNA[J].Journal of Virology, 1986, 57(1): 219-228.
[29]Gabriel A, Willems M, Mules E H, et al. Replication infidelity during a single cycle of Ty1 retrotransposition[J].Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(15): 7767-7771.
[30]Song Y, Ji D, Li S, et al. The dynamic changes of DNA methylation and histone modifications of salt responsive transcription factor genes in soybean[J].PLoS ONE, 2012, 7(7): e41274.
[31]Heslop-Harrison J S, Brandes A, Taketa S, et al. The chromosomal distributions of Ty1-copia group retrotransposable elements in higher plants and their implication for genome evolution[J].Genetica, 1997, 100(1/2/3): 197-204.
[32]Ostertag E M, Madison B B, Kano H. Mutagenesis in rodents using the L1 retrotransposon[J].Genome Biology, 2007, 8 (Supplement 1): S16.
[33]Doolittle R F, Feng D F, Johnson M S, et al. Origins and evolutionary relationships of retroviruses[J].Quarterly Review of Biology, 1989, 64(1): 1-30.
[34]Bennetzen J L, Wang H. The contributions of transposable elements to the structure, function, and evolution of plant genomes[J].Annual Review of Plant Biology, 2014, 65(1): 505-530.
[35]Kumar A. The evolution of plant retroviruses: moving to green pastures[J].Plant Science, 1998, 3(10): 371-374.
[36]Diao X M, Freeling M, Lish D. Horizontal transfer of a plant transposon[J].PLoS Biology, 2006, 4(1): 119-128.
[37]Tang Y M, Ma Y Z, Li L C, et al. Identification and characterization of reverse transcriptase domain of transcriptionally active retrotransposons in wheat genome[J].Journal of Integrative Plant Biology (Formerly Acta Botanica Sinica), 2005, 47(5): 604-612.
收稿日期:2019-09-08
基金項目:国家自然科学基金项目(31660428,31960409,31401415,31240059);广西自然科学基金项目(2018GXNSFDA281027);广西农业科学院科技发展基金项目(桂农科2017JZ13,桂农科2015JZ98,桂农科2018YT12)
作者简介:阳太亿(1995—),男,广西桂林人,本科生,研究方向:花生遗传育种与分子生物学。E-mail:528515743@qq.com
刘俊仙(1982—),女,河南驻马店人,硕士,副研究员,研究方向:植物生物技术与分子生物学。E-mail:liujunxian868@163.com
*同为第一作者。
通讯作者:唐荣华(1965—),男,广西桂林人,博士,研究员,研究方向:花生高产高效栽培。E-mail:tronghua@163.com
熊发前(1983—),男,安徽芜湖人,博士,副研究员,研究方向:花生遗传育种与分子生物学。E-mail:xfq2002@126.com
