宿主免疫系统IL-2、TNF-α 基因启动子多态性与丙型肝炎病毒慢性感染的相关性研究*
2015-04-28许秀雯童绍勇姚宇峰俞建昆孙明波
许秀雯,李 莹,童绍勇,姚宇峰,俞建昆,史 荔,孙明波
(中国医学科学院 北京协和医学院医学生物学研究所 云南省重大传染病疫苗研发重点实验室,云南 昆明 650118)
丙型肝炎是由丙型肝炎病毒(hepatitis C Virus,HCV)感染引起的疾病。HCV 感染后,20%~30%的急性感染者可自发清除HCV,其余感染者都将发展为慢性持续性感染,并最终发展为肝硬化(15%~20%)和肝细胞癌(1%~4%)[1]。研究表明,在HCV 感染后的炎性反应及在抗感染的宿主机体免疫应答过程中,肿瘤坏死因子α(tumor necrosis factor,TNF-α)、白细胞介素(interleukin,IL)-2、IL-10、IL-12 等细胞因子可通过介导不同的免疫反应,导致病毒清除、持续感染或肝脏损伤等不同结果[2-6]。炎性因子及调控细胞因子平衡的个体差异在某种程度上取决于细胞因子基因调控区的等位基因多态性,这些多态性可影响细胞因子的表达,从而在HCV 清除及持续感染中发挥重要作用[7-8]。TNF-α 是由主要组织相容性抗原(MHC)Ⅲ类基因编码的多功能免疫调控因子,它能募集和激活巨噬细胞、NK 细胞和T 细胞,从而产生免疫调节及抗病毒细胞因子,促进肝细胞炎性反应、介导肝细胞损伤及慢性肝炎。研究表明,TNF-α 基因启动子单核苷酸多态性位点(single nucleotide polymorphism sites,SNPs)-238 和-308 与TNF-α 的表达水平相关[9-10]。IL-2 是主要由活化的CD4+T 细胞和CD8+T 细胞产生的具有广泛生物活性的细胞因子,是所有T 细胞亚群的生长因子,并可促进活化B 细胞增殖,为调控免疫应答的重要因子。研究表明,IL-2 的表达水平在中度及重度肝硬化及肝癌中高于正常对照,与丙肝的分级相关,且IL-2 启动子基因-330 SNP 与HCV 的持续存在相关[11-13]。本研究选择IL-2 基因启动子SNP rs2069762、rs2069763 和rs4833248,以及TNF-α 基因启动子SNPrs1800629、rs3093668 和rs3093726,探讨其基因及单倍型与慢性HCV 感染的遗传易感性的关联。
1 材料与方法
1.1 标本来源
选择380 名HCV 慢性感染者作为病例组,按照中华医学会肝病学分会和传染病与寄生虫病学分会2011 年制定的《丙型病毒性肝炎防治指南》确定HCV 感染,实验学检查指标ALT、AST、抗HCV、HCV RNA 等检测阳性,并排除其他肝炎的患者。选择健康个体367 名为对照组。健康对照的纳入标准为HCV 诊断及实验学检查指标ALT、AST、抗HCV、HCVRNA 等正常。病例组年龄(44.22±12.40)岁,对照组(44.75±9.21)岁;病例组男性207(54.47%)例,女性173 例(45.53%);对照组 男 性 187 例(50.95%),女 性 180 例(49.05%)。两组间性别比例和年龄分布差异无统计学意义(P >0.05)。所有参加者均是为居住于云南地区的彼此无血缘关系汉族个体。患者均知情同意。
1.2 方法
1.2.1 样品处理 采集空腹静脉血5 mL,用EDTA 或肝素抗凝,使用AxyPrep 血基因组DNA 小量试剂盒提取DNA。
1.2.2 IL-2 及TNF-α 基因SNP 位点基因分型采用实时荧光定量PCR 法进行SNP 分型。由Applied Biosystems 公司定制合成IL-2 基因启动子SNP rs2069762(-330,A >C)、rs2069763(-34,G >T)和rs4833248(-2811,G >A),TNF-α 基因启动子SNPrs1800629(-308,G >A)、rs3093668(G >C)和rs3093726(T >C)位点的引物及Taq-Man 探针。针对每个SNP 位点所设计的两条探针分别用VIC 和FAM 进行荧光标记。罗氏LightCycler480 实时荧光定量PCR 仪检测SNP 位点,LCS480 1.5.1.62 软件进行基因分型。PCR 反应体积为20 μL,反应条件:95 ℃10 min 预变性,92 ℃15 s 变性,60 ℃1 min 退火,共40 个循环,40 ℃5 min 延伸。以3 个已知基因型(野生纯合子、突变纯合子、杂合子)的标准样品作为对照,采用测序的方法对TaqMan 分型结果进行验证。针对各个SNP 位点,应用DNASTAR 软件设计6 对引物,引物合成及PCR 产物测序在上海生工生物技术有限公司完成。
1.3 统计学分析
Hardy-Weinberg 平衡检验基因型频率的代表性,以各基因型的个体数计算各基因型和等位基因频率,经χ2检验检测健康对照组与HCV 慢性感染组6 个SNP 基因型、等位基因频率差异,P <0.05认为差异有显著性。SHEsis 软件程序计算连锁不平衡,常用D'表示;D'值为零时,连锁完全平衡;D'值为1 时,连锁完全不平衡;D'>0.8 时,即存在强连锁。根据LD 结果构建IL-2 基因和TNF-α 基因启动子区域的各3 个SNP 位点的单倍型[14-15]。
2 结果
2.1 IL-2 和TNF-α 多态性位点的等位基因频率与基因型频率
IL-2 基因启动子SNP rs2069762、rs2069763 和rs4833248,以及TNF-α 基因启动子SNPrs1800629、rs3093668 和rs3093726 基因型分布在病例组和对照组中均符合Hardy-Weinberg 平衡(P >0.05)。IL-2-rs2069762(A >C)CC 频率,病例组高于对照组,差异有统计学意义(P <0.05);IL-2-rs2069763(G >T)等位基因G 频率,病例组中高于对照组,差异有统计学意义(P <0.05);其余SNPs 等位基因和基因型频率在病例和对照组中的分布,差异无统计学意义(P >0.05)。见表1。
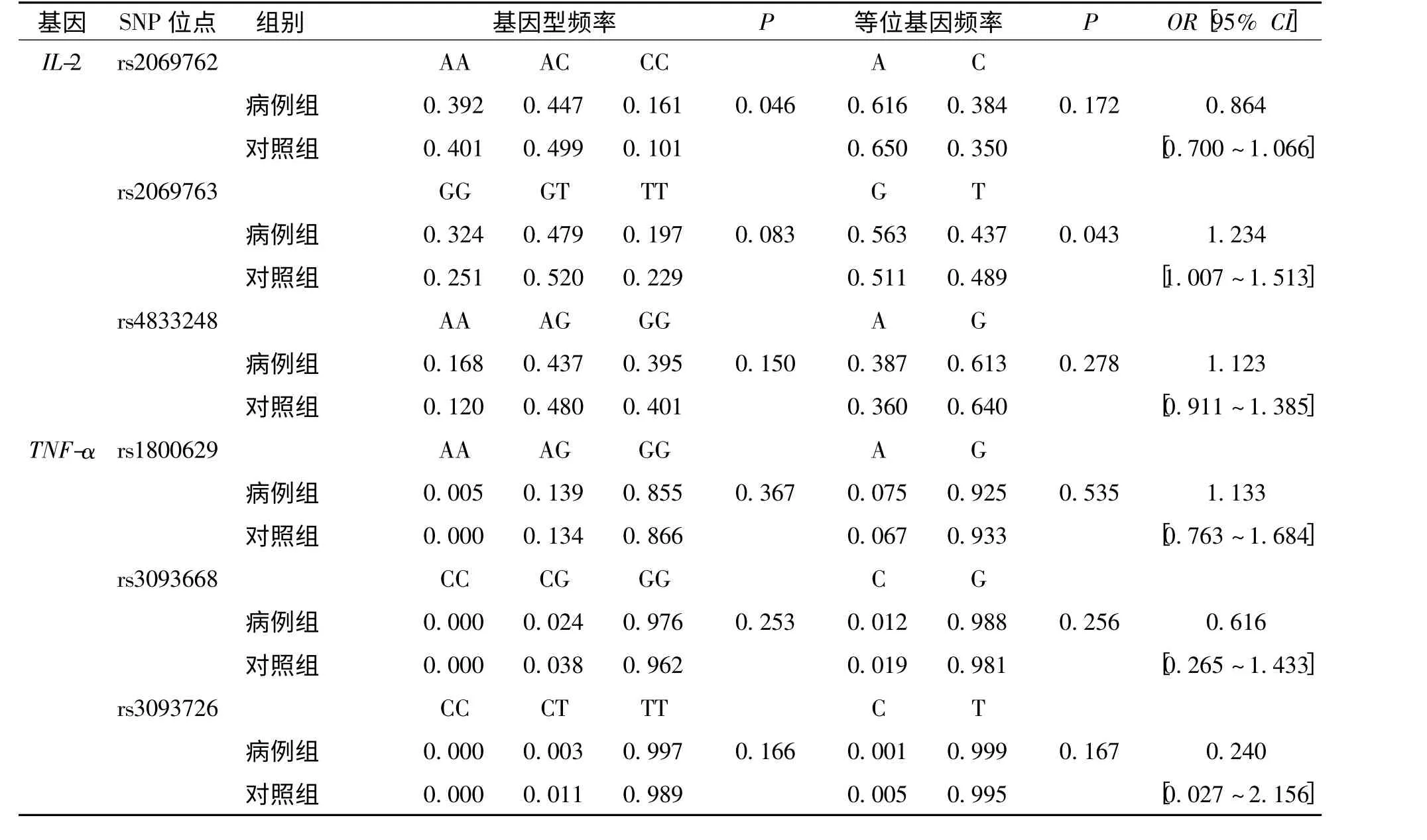
表1 两组被检者IL-2 和TNF-α 基因启动子6 个SNP 位点的基因频率及等位基因频率Tab.1 Gene frequencies and allele frequencies of 6 SNPs in IL-2 and TNF-α gene promoter of objects in the two groups
2.2 IL-2 及TNF-α 多态性位点的连锁不平衡检测结果
IL-2 基因启动子区域3 个多态性位点的连锁不平衡分析显示:rs2069762 与rs2069763 之间的D'值为1.000,rs2069762 和rs4833248 位点间的D'值为0.994,rs2069763 和rs4833248 位点间的D'值为0.958,三位点之间存在强连锁不平衡。TNF-α基因启动子区域3 个多态性位点的连锁不平衡分析显示:rs1800629 与rs3093668 之间的D'值为0.963,rs1800629 和 rs3093726 位 点 间 以 及rs3093668 和rs3093726 位点间的D'值均为1.000,三位点之间存在强连锁不平衡。
2.3 IL-2 及TNF-α 多态性位点单倍型构建及频率
rs2069762/rs2069763/rs4833248-ATG 单倍型频率在病例和对照组中的分布差异具有统计学意义(P <0.05),见表2。TNF-α 基因启动子区域的3 个SNP 位点的单倍型频率在病例和对照组中的分布差异无统计学意义(P >0.05),见表3。

表2 两组被检者IL-2 基因启动子SNP 位点的单倍型频率Tab.2 Haplotype frequencies in IL-2 gene promoter of objects in the two groups
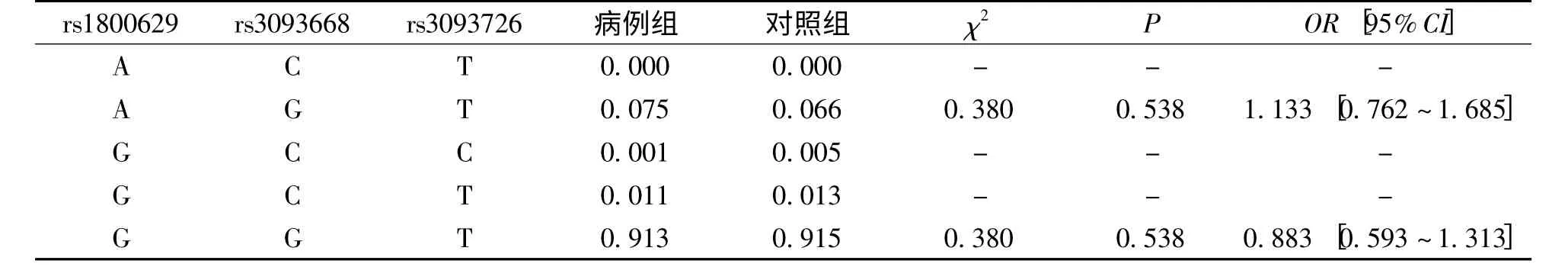
表3 两组被检者TNF-α 基因启动子SNP 位点的单倍型频率Tab.3 Haplotype frequencies in TNF-α gene promoter of objects in the two groups
3 讨论
免疫细胞在抗HCV 感染过程中分泌大量的细胞因子介导不同的免疫反应,从而导致病毒清除、持续感染或肝细胞损伤等不同结果。细胞因子基因,特别是启动子基因多态性影响着细胞因子的表达水平,从而与HCV 慢性感染进程相关。
本研究结果表明,IL-2 基因启动子rs2069762(-330,A >C)CC 基因型在病例组中高于对照组(P <0.05),等位基因型C 在病例组中的频率也高于对照组(P >0.05),提示rs2069762CC 基因型可能是HCV 慢性感染的易感因素。Gao 等[11]研究结果表明,IL-2-330 SNP 与HBV、HCV 以及HBV/HCV 共感染后病毒的持续存在相关。本研究结果显示,与rs2069762 紧密连锁的rs2069763(G >T),等位基因G 的频率在病例组中也高于对照组(P <0.05);rs2069762/rs2069763/rs4833248 位点构建的单倍型ATG 在病例组中的频率低于对照组(P <0.05),提示ATG 单倍型可能降低HCV 慢性感染的风险(OR=0.812,95%CI 0.662 ~0.996)。IL-2能诱导T、B 细胞的增殖与分化,在抗病毒感染的免疫中发挥重要作用[16]。研究发现,IL-2 基因启动子区域-330SNP 与IL-2 的分泌量相关[17]。本研究结果发现,IL-2 启动子区域的2 个SNP 均与HCV 的慢性感染相关,提示IL-2 基因启动子SNP可能是云南汉族人群中HCV 易感的关联因素。
TNF-α 具有诱导靶细胞凋亡,与IL-2 协同促进T 细胞产生IFN-γ,以及增强巨噬细胞的活性和杀伤功能的特性;也能够抑制B 细胞增殖和分泌免疫球蛋白。TNF-α 基因启动子区域含有多个转录因子结合位点,可直接影响TNF-α 的转录调控。多个研究报道TNF-α 基因多态性与HCV 感染等多种感染性疾病相关,其中,报道较多的为TNF-α启动子基因-308G/A 和-238G/A[18-19]。然而,不同研究所报道的TNF-α 启动子基因与HCV 慢性感染的相关性并不一致[20-23]。本研究研究对3 个TNF-α 启动子基因进行分析,但结果未发现与HCV 慢性感染相关的等位基因、基因型及单倍型,与He 等[22]对TNF-α-308G/A 的meta 分析结果一致。另一方面,人群的遗传背景影响着基因多态的分布。在高加索人群中多态性较高的TNF-α 启动子基因SNP 在亚洲人群中的多态性较低,TNF-αrs1800629 A 等位基因频率为6%,rs-3093668 C 等位基因的频率仅为3%,rs-3093726 C 等位基因频率低至1%。因此,在其他人群中报道的与丙肝相关的SNP 与本研究结果并不一致,可能存在其他的SNP,有待于使用全基因组关联分析去发现新的易感位点。
综上所述,在云南汉族群体中,IL-2 基因启动子SNPrs2069762 CC 基因型和rs2069763 G 等位基因可能是HCV 慢性感染的易感因素,rs2069762/rs2069763/rs4833248 单倍型ATG 可能是HCV 慢性感染的保护性因素。TNF-α 基因启动子SNPrs1800629、rs3093668 和rs3093726 与HCV 慢性感染没有相关性。
[1]Seeff LB.Natural history of chronic hepatitis C[J].Hepatology,2002(36):35-46.
[2]Di Bisceglie AM.Natural history of hepatitis C:Its impact on clinical management[J].Hepatology,2000(31):1014-1018.
[3]Fukuda R,Ishimura N,Ishihara S.Intrahepatic expression of pro-inflammatory cytokine mRNAs and interferon efficacy in chronic hepatitis C[J].Liver,1996(16):390-399.
[4]Golden-Mason L,Rosen HR.Natural killer cells:primary target for hepatitis C virus immune evasion strategies[J].Liver Transpl,2006(12):363-372.
[5]Koziel MJ.The role of immune responses in the pathogenesis of hepatitis C virus infection[J].J Viral Hepat,1997(4 Suppl 2):31-41.
[6]Larrea E,Garcia N,Qian C.Tumor necrosis factor alpha gene expression and the response to interferon in chronic hepatitis C[J].Hepatology,1996(23):210-217.
[7]Clark PJ,Thompson AJ.Host genomics and HCV treatment response[J].J Gastroenterol Hepatol,2012(27):212-222.
[8]Ge D,Fellay J,Thompson AJ.Genetic variation in IL28B predicts hepatitis C treatment-induced viral clearance[J].Nature,2009(461):399-401.
[9]Baseggio L,Bienvenu J,Charlot C.Higher LPS-stimulated TNF-alpha mRNA levels in peripheral blood mononuclear cells from non-Hodgkin's lymphoma patients[J].Exp Hematol,2001(29):330-338.
[10]Wilson AG,Symons JA,McDowell TL.Effects of a polymorphism in the human tumor necrosis factor alpha promoter on transcriptional activation[J].Proc Natl Acad Sci USA,1997(94):3195-3199.
[11]Gao QJ,Liu DW,Zhang SY.Polymorphisms of some cytokines and chronic hepatitis B and C virus infection[J].World J Gastroenterol,2009(15):5610-5619.
[12]Napoli J,Bishop GA,McGuinness PH.Progressive liver injury in chronic hepatitis C infection correlates with increased intrahepatic expression of Th1-associated cytokines[J].Hepatology,1996(24):759-765.
[13]Pawlowska M,Halota W,Smukalska E.Serum IL-2 and sIL-2R concentration in children with chronic hepatitis[J].Pol Merkur Lekarski,2005(18):33-35.
[14]Li Z,Zhang Z,He Z.A partition-ligation-combinationsubdivision EM algorithm for haplotype inference with multiallelic markers:update of the SHEsis(http://analysis.bio-x.cn)[J].Cell Res,2009(19):519-523.
[15]Shi YY,He L.SHEsis,a powerful software platform for analyses of linkage disequilibrium,haplotype construction,and genetic association at polymorphism loci[J].cell research,2005(15):97-98.
[16]Gearing A,Thorpe R,Bird C.Human B cell proliferation is stimulated by interleukin 2[J].Immunol Lett,1985(9):105-108.
[17]Haralambieva IH,Ovsyannikova IG,Kennedy RB.Associations between single nucleotide polymorphisms and haplotypes in cytokine and cytokine receptor genes and immunity to measles vaccination[J].Vaccine,2011(29):7883-7895.
[18]Rosen HR,McHutchison JG,Conrad AJ.Tumor necrosis factor genetic polymorphisms and response to antiviral therapy in patients with chronic hepatitis C[J].Am J Gastroenterol,2002(97):714-720.
[19]Yee LJ,Tang J,Herrera J.Tumor necrosis factor gene polymorphisms in patients with cirrhosis from chronic hepatitis C virus infection[J].Genes Immun,2000(1):386-390.
[20]Corchado S,Marquez M,Montes de Oca M.Influence of genetic polymorphisms of tumor necrosis factor alpha and interleukin 10 genes on the risk of liver cirrhosis in HIVHCV coinfected patients[J].PLoS One,2013(8):e66619.
[21]Dogra G,Chakravarti A,Kar P.Polymorphism of tumor necrosis factor-alpha and interleukin-10 gene promoter region in chronic hepatitis C virus patients and their effect on pegylated interferon-alpha therapy response[J].Hum Immunol,2011(72):935-939.
[22]He J,Pei X,Xu W.The relationship between tumor necrosis factor-alpha polymorphisms and hepatitis C virus infection:a systematic review and meta-analysis[J].Ren Fail,2011(33):915-922.
[23]Tarrago AM,da Costa AG,Pimentel JP.Combined impact of hepatitis C virus genotype 1 and interleukin-6 and tumor necrosis factor-alpha polymorphisms on serum levels of pro-inflammatory cytokines in Brazilian HCV-infected patients[J].Hum Immunol,2014(75):1075-1083.
