水华鱼腥藻类菌胞素氨基酸的分子鉴定和化学检测
2015-02-27洋高合意李效宇李仁辉
刘 洋高合意李效宇李仁辉
(1. 河南师范大学生命科学学院, 新乡 453007; 2. 中国科学院水生生物研究所, 武汉 430072)
水华鱼腥藻类菌胞素氨基酸的分子鉴定和化学检测
刘 洋1高合意2李效宇1李仁辉2
(1. 河南师范大学生命科学学院, 新乡 453007; 2. 中国科学院水生生物研究所, 武汉 430072)
类菌胞素氨基酸 (MAAs) 是一类具有吸收紫外线能力的物质, 蓝藻MAAs的生物合成及其分子机制的揭示为 MAAs基因的快速检测提供了可能。研究采用分子生物学方法扩增了一株分离自太湖的水华鱼腥藻(Dolichospermum flos-aquae CHAB1629)编码脱氢醌合成酶(DHQS)基因的部分片段, 系统树分析发现其与报道的Anabaena sp. 90核酸序列相似度达99%, 而与Anabaena variabilis ATCC 29413仅为53.6%; 同时运用HPLC检测发现, 该株水华鱼腥藻MAAs的类型为shinorine。研究结果可为后续浮游类鱼腥藻MAAs的分子鉴定及野外适应性研究提供依据。
蓝藻; 水华鱼腥藻; 类菌胞素氨基酸; 高效液相色谱
类菌胞素氨基酸(Mycosporine-Like Amino Acids, MAAs)是一大类以环己烯酮为基本骨架、与不同类型氨基酸缩合形成的水溶性物质, 共轭双键的结构特征以及侧链的活性基团决定了其具有强紫外(310—360 nm)吸收的能力(图1)[1,2]。MAAs在自然界分布广泛, 从早期在真菌中发现至今(Leach 1965), 已在多种浮游植物类群中发现, 如蓝藻、红藻等。同时MAAs的化学结构多样, 目前已发现了近30种, 2013年, Nazifi等[3]和Miyamoto等[4]报道在地木耳(Nostoc commune)中新发现了两种不同类型的MAAs。研究表明, MAAs还具有光保护和抗氧化、防止珊瑚漂白、调节细胞渗透压、调节休眠体细胞形成及萌发、增强细胞耐受干旱和高温等多种作用, 其主要的抗紫外作用已被应用于相关护肤品的商业开发[1,5]。虽然MAAs较早就被发现, 然而其生物合成机制的研究较为滞后, 1999年Shick[6]对产MAAs珊瑚的草甘膦抑制实验首次证实了 MAAs的莽草酸合成途径; 随后Balskus 和 Walsh[1]揭示了蓝藻 MAAs合成的分子机制, 他们在Anabaena variabilis ATCC 29413中确定了shinorine 合成酶基因簇全长(6.5 kb), 包含了4个 ORFs, 分别负责编码脱氢醌合成酶(DHQS),甲基转移酶(O-MT), ATP-grasp和NRPS-like酶, 其中 DHQS是合成途径的关键酶, 负责催化 3-脱氧-D-阿拉伯庚酮糖酸-7-磷酸生成3-Dehydroquinate[7,8]。MAAs合成分子机制的揭示为 MAAs基因快速检测提供了可能。
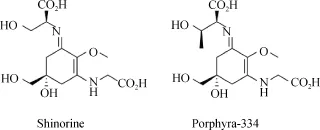
图1 两种代表性MAAs (shinorine and porphyra-334) 的结构图Fig. 1 Structures of representative MAAs (shinorine and porphyra-334)
目前已报道含有MAAs的蓝藻有20多个属, 虽然它们分布广泛, 但不同种类的蓝藻MAAs差异较大[5,9]。Singh对 A. variabilis PCC 7937和 Synechocystis sp. PCC 6803研究发现, 前者能产生MAAs, 而后者却不产[7]。尽管首先在 A. variabilisATCC 29413中揭示了MAAs合成酶的基因簇全长,但通过对 GenBank的数据分析发现, 鱼腥藻相关MAAs数据较少, 这也许是导致鱼腥藻属内编码DHQS的基因mysA差异较大的原因。不同种属间含有的MAAs类型也不尽相同。Sinha等[10]对3株丝状固氮蓝藻进行了研究, 发现它们仅仅含有shinorine这一种类型的MAAs物质; 我国学者对微囊藻属的铜绿微囊藻调查发现, 其含有两种类型的MAAs, 即shinorine和porphyra-334[11], 但对于我国常见鱼腥藻水华种类所含有 MAAs情况还鲜有报道。水华鱼腥藻(新近的分类学研究将浮游类鱼腥藻属名Anabaena改为Dolichospermum[12])是水华蓝藻中常见种类, 分布广泛, 我国三湖均有分布。本研究通过对水华鱼腥藻MAAs合成中编码关键酶DHQS的mysA基因进行特异引物设计, 同时用HPLC检测了其含有MAAs的类型, 为水华鱼腥藻的生态适应性研究提供数据支持。
1 材料与方法
1.1 藻株及培养
实验所用藻株为分离自太湖的水华鱼腥藻, 保存于中国科学院水生生物研究所有害藻类学科组藻种库(CHAB), 培养条件: CT培养基, 光照强度为25 µE/(m2·s), 光周期为 12h∶12h (L∶D), 温度为(25±1)℃。
1.2 藻株DNA提取、引物设计与mysA基因扩增
藻株基因组DNA提取方法参考Li 等[13]的方法。通过对GenBank中已报道的Anabaena sp. 90、A. variabilis ATCC 29413、Nostoc punctiforme PCC 73102、Nostoc sp. PCC 7524中DHQS同源蛋白进行Blast, 定位其核酸序列, 运用 Primer 5.0软件进行mysA基因引物设计, 正向引物为mysaF (5′-TAACC AATTCATTTCCACAG-3′), 反向引物为 mysaR (5′-TCTCAACTCAGAAGTCAAGG-3′)。PCR反应体系为50 µL, 包含5—10 ng 模板DNA, 1U Taq DNA聚合酶(Takara, Japan), 10×PCR反应缓冲液 2 μL, 1.5 mmol/L MgCl2, 引物各10 pmol, 200 μmol/L 牛血清蛋白和200 μmol/L dNTPs (deoxyribonucleoside triphosphate), 其余用双蒸水补足。反应体系于 MJ Mini BioRad PCR仪(BioRad, USA)上扩增, 扩增条件为: 94℃预变性3min; 94℃变性30s, 50℃退火30s, 72℃延伸 1min, 35个循环; 72℃延伸10min。经过1.0%琼脂糖凝胶电泳检测后, 将PCR产物进行切胶回收(Biospin胶回收试剂盒), 回收后的DNA连接到载体pMD18-T(Takara, Japan)上, 然后克隆载体转化至感受态细胞E. coli Competent Cells DH5α (Takara, Japan)中, 挑取单克隆菌液送北京华大基因科技有限公司进行测序。
1.3 序列测定及系统分析
测序获得并经人工校正的序列数据与 GenBank获得的外源数整合后利用CLUSTAL X (version 2.0)进行对位排列[14]。然后运用分子进化遗传分析软件MEGA 4 program package基于Kimura two-parameter模型构建邻接法分子系统关系树(Neighbor-Joining tree, 简称NJ tree), 步展值(Bootstrap value)设置为1000, 空位或缺失位点均当作配对删除(Pairwise deletion)处理[15]。
1.4 藻株MAAs的化学测定
取5 mL藻液, 富集后, 用25%甲醇在45℃条件下萃取3次, 取2 mL样品的甲醇萃取液, 在45℃条件下干燥, 溶入200 µL蒸馏水, 通过0.2 µm滤膜后进行反相高效液相色谱分析(SSI HPLC 1500, USA),色谱柱 Grace Prevail C18规格 5 µm, 4.6 mm× 250 mm。流动相为25%的甲醇加0.2%乙酸, 检测波长 250—450 nm。标样为中国科学院水生生物研究所有害藻类实验室制备, 提取藻株为 Microcystis aeruginosa PCC 7806。
2 结果
2.1 水华鱼腥藻mysA基因扩增及系统树分析
通过PCR检测我们得到了一株含有mysA基因片段的藻株(D. flos-aquae CHAB1629), 克隆测序后得到约 859 bp的目的片段, 通过在 GenBank中对DHQS酶同源蛋白分析, 进一步确定了其为编码此蛋白的 mysA基因片段。目前, 数据库中蓝藻相关mysA数据较少, 本研究对GenBank和EMBL两大数据库中完成全基因组测序的 12株念珠藻科藻株进行mysA数据挖掘, 以M. aeruginosa PCC 7806为外类群构建了NJ系统树(图2), 结果表明, mysA基因在念珠藻科内部变异较大, Cylindrospermum stagnale PCC 7417、Nodularia spumigena CCY9414、Nostoc punctiforme PCC 73102、Nostoc sp. PCC 7524、A. variabilis ATCC 29413、Anabaena sp. 90这5株藻mysA基因最大相似度仅为82%, 而同为鱼腥藻属的A. variabilis ATCC 29413和Anabaena sp. 90其mysA基因最大相似度仅为53.6%。本研究分离的D. flos-aquae CHAB1629与 Anabaena sp. 90及Aphanizomenon flos-aquae NIES-81间的最大相似度分别为99%和97.2%。
2.2 水华鱼腥藻中含有MAAs物质的HPLC检测
对含有 mysA基因片段的藻株 D. flos-aquae CHAB1629进行了HPLC化学检测, 通过与标样的的保留时间比较分析, 鉴定了本藻株含有的 MAAs的类型为shinorine(图3)。
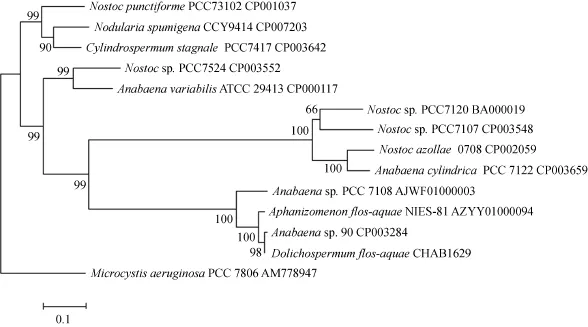
图2 基于mysA 基因序列的NJ分子系统树 (节点处的数字代表1000重复步展值的百分数, 低于50% 的未显示)Fig. 2 The phylogenetic tree (NJ) based on mysA sequences (The numbers at each node represented the percentage of bootstrap setting as 1000, only showing those above 50%)
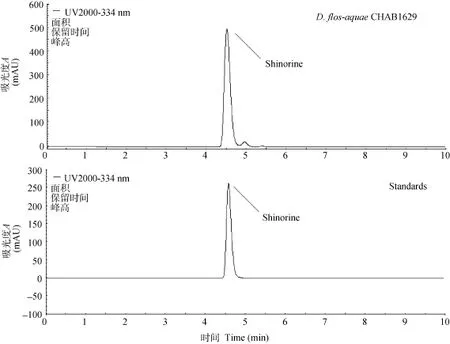
图3 MAAs的HPLC检测Fig. 3 MAAs detection by HPLC
3 讨论
在长期的自然进化与选择过程中, 生物有机体形成了多种应对环境变化的保护机制, 如部分生物在体内合成和积累MAAs来抵抗紫外线的损伤, 便是这些机制中较为重要的一种。蓝藻作为地球上较早出现的原核生物, 同样具有合成 MAAs的能力,不仅如此, 蓝藻体内具有紫外吸收的物质还有伪枝藻素(Scytonemin)、类胡萝卜素(Carotenoids)、糖苷蝶呤(Biopterin glucoside)等[9,16]。虽然MAAs的化学结构和抗紫外特性很早就被认识, 然而其生物合成及其分子机制直到最近才被揭示。通过对A. variabilis ATCC 29413研究, 发现了编码MAAs的4个基因(mysA、B、C、D), 分别编码 DHQS、O-MT、ATP-grasp和NRPS-like 4个酶, 并且发现A. variabilis ATCC 29413与Nostoc punctiforme ATCC 29133中MAAs合成基因中的前3个较为保守, 而第4个基因编码的酶为 NRPS-like或者 D-Ala D-Ala ligase-like, 并且这两种蓝藻含有的 MAAs均为shinorine。然而并非所有蓝藻均含有相同的MAAs、Nodularia baltica、Nodularia harveyana和Nodularia spumigena 3种节球藻所含 MAAs为 shinorine和porphyra-334[16,17], 有些蓝藻却不含有 MAAs, 如Synechocystis sp. PCC 6803 和 Synechococcus sp. PCC 6301。有关微囊藻中 MAAs的检测已有报道,而对于另一种蓝藻水华群体—水华鱼腥藻的研究较少。本研究针对水华鱼腥藻群体, 设计了检测其mysA基因的引物, 并且对其片段进行扩增和系统分析, 发现水华鱼腥藻 mysA基因片段与预想情况并不相同, 其编码区域差异较大, 这也许跟数据量有关。通过对GenBank和EMBL中报道的念珠藻科的12株全基因组进行分析, 构建系统树, 发现我国的水华鱼腥藻D. flos-aquae CHAB1629与Anabaena sp. 90较为相近, 而与 A. variabilis ATCC 29413、Anabaena cylindrica PCC 7122和Anabaena sp. PCC 7108差异较大。推测可能的原因有两种: 一是在鱼腥藻属内部基于 mysA基因确实分为几个类群, 二是由于序列数据较少, 并且是通过同源蛋白比对后预测片段, 仅有少数藻株被化学验证可产生 MAAs,而其他藻株是否能够产生MAAs, 还需要化学验证。因此通过基因的初步筛查和化学检测, 才能较为准确的判定藻株含有MAAs情况。同时, 结合化学方法, 本研究检测了 D. flos-aquae CHAB1629含有MAAs的类型(shinorine), 这与先前报道的我国铜绿微囊藻所含有的MAAs类型较为相似。但推测本藻株含有的类型不止一种(可能含有 porphyra-334)这需要获取标准品进一步研究。由于本研究中的藻株较少, 还不能代表众多浮游鱼腥藻类群的情况, 因此需要大量分离我国野外水体中不同种类的鱼腥藻进行深入研究, 评估其产MAAs能力及其对环境的适应性。本研究结果可为后续浮游类鱼腥藻 MAAs的分子鉴定提供参考。
[1] Balskus E P, Walsh C T. The genetic and molecular basis for sunscreen biosynthesis in cyanobacteria [J]. Science, 2010, 329(5999): 1653—1656
[2] Rosic N N. Phylogenetic analysis of genes involved in mycosporine-like amino acid biosynthesis in symbiotic dinoflagellates [J]. Applied Microbiology and Biotechnology, 2012, 94(1): 29—37
[3] Nazifi E, Wada N, Yamaba M, et al. Glycosylated porphyra-334 and palythine-threonine from the terrestrial cyanobacterium Nostoc commune [J]. Marine Drugs, 2013, 11(9): 3124—3154
[4] Miyamoto K T, Komatsu M, Ikeda H. Discovery of gene cluster for mycosporine-like amino acid biosynthesis from Actinomycetales microorganisms and production of a novel mycosporine-like amino acid by heterologous expression [J]. Applied and Environmental Microbiology, 2014, 80(16): 5028—5036
[5] Chen X L, Deng G B, Liu K Q, et al. UV-protective metabolites in aquatic organisms——mycosporine-like amino acids [J]. Chinese Bulletin of Botany, 2006, 23(1): 78—86 [陈小兰, 邓国宾, 刘开庆, 等. 水生生物的紫外光防护剂——类菌胞素氨基酸. 植物学通报, 2006, 23(1): 78—86]
[6] Shick J M, Romaine-Lioud S, Ferrier-Pagès C, et al. Ultraviolet-B radiation stimulates shikimate pathway dependent accumaltion of mycosporine-like amino acids in the coral Stylophora pistillata despite decreases in its population of symbiotic dinoflagellates [J]. Limnology and Oceanography, 1999, 44(7): 667—1682
[7] Singh S P, Klisch M, Sinha R P, et al. Genome mining of mycosporine-like amino acid (MAA) synthesizing and non-synthesizing cyanobacteria: A bioinformatics study [J]. Genomics, 2010, 95(2): 120—128
[8] Singh S P, Klisch M, Sinha R P, et al. Sulfur deficiency changes mycosporine-like amino acid (MAA) composition of Anabaena variabilis PCC 7937: A possible role of sulfur in MAA bioconversion [J]. Photochemistry and Photobiology, 2010, 86(4): 862—870
[9] Gao H Y. The studies on UV-absorbing compounds MAAs in Microcystis strains from China [D]. Thesis for Master of Science. Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan. 2014 [高合意. 微囊藻中紫外吸收物质类菌胞素氨基酸(MAAs)的研究. 硕士学位论文, 中国科学院水生生物研究所, 武汉. 2014]
[10] Sinha R P, Klisch M, Walter Helbling E, et al. Induction of mycosporine-like amino acids (MAAs) in cyanobacteria by solar ultraviolet-B radiation [J]. Journal of Photochemistry and Photobiology B: Biology, 2001, 60(2—3): 129—135
[11] Liu Z W, Zhong P, Han B P. UV Protective compounds mycosporine-like amino acids (MAAs) and bloom forming mechanism in Microcystis aeruginosa [J]. Journal of Lake Sciences, 2003, 15(4): 359—363 [刘正文, 钟萍, 韩博平.铜绿微囊藻中的紫外保护物质类菌孢素氨基酸(MAAs)与水华形成机制探讨. 湖泊科学, 2003, 15(4): 359—363]
[12] Wacklin P, Hoffmann L, Komárek J. Nomenclatural validation of the genetically revised cyanobacterial genus Anabaena (Ralfs ex Bornet et Flahault) comb. nova [J]. Fottea, 2009, 9(1): 59—64
[13] Li R H, Wilhelm S W, Carmichael W W, et al. Polyphasic characterization of water bloom forming Raphidiopsis species (cyanobacteria) from central China [J]. Harmful Algae, 2008, 7(2): 146—153
[14] Larkin M, Blackshields G, Brown N, et al. Clustal W and Clustal X version 2.0 [J]. Bioinformatics, 2007, 23(21): 2947—2948
[15] Posada D, Crandall K. Modeltest: testing the model of DNA substitution [J]. Bioinformatics, 1998, 14(9): 817—818
[16] Hu C L. Development of a rapid method for microcystin analysis and construction of Microcystis aeruginosa PCC 7806 mutants bearing deficiency in the biosynthesis of mycosporine-like amino acids [D]. Thesis for Doctor of Science. Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan. 2011 [胡臣林. 微囊藻毒素快速分析方法建立和铜绿微囊藻PCC 7806类菌孢素氨基酸生物合成缺陷突变体的构建. 博士学位论文, 中国科学院水生生物研究所, 武汉. 2011]
[17] Sinha R P, Ambasht N K, Sinha J P, et al. UV-B-induced synthes is of mycosporine-like amino acids in three strains of Nodularia (cyanobacteria) [J]. Journal of Photochemistry and Photobiology B: Biology, 2003, 71(1): 51—58
IDENTIFICATION AND DETERMINATION OF MYCOSPORINE-LIKE AMINO ACIDS (MAAS) IN DOLICHOSPERMUM FLOS-AQUAE
LIU Yang1, GAO He-Yi2, LI Xiao-Yu1and LI Ren-Hui2
(1. College of Life Science, Henan Normal University, Xinxiang 453007, China; 2. Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China)
Mycosporine-like aminoacids (MAAs) exist in cyanobacteria, fungi, and eukaryotic micro- and macro-algae. They absorb ultraviolet (UV) and hence protect these organisms from the damages caused by the environmental UV radiation. The understanding of the biosynthetic pathway and the genetic properties of MAAs is fundamental for the fast-detection of MAAs genes. In the present study, we amplified the partial coding sequence of the gene encoding DHQS enzyme in Dolichospermum flos-aquae CHAB1629 that was isolated from the Taihu Lake. The nucleotide similarity between D. flos-aquae CHAB1629 and Anabaena sp. 90 was 99%, and it was only 53.6% between D. flos-aquae CHAB1629 and Anabaena variabilis ATCC 29413. High-performance liquid chromatography (HPLC) assays confirmed that MAAs in D. flos-aquae CHAB1629 were shinorine.
Cyanobacteria; Dolichospermum flos-aquae; MAAs; HPLC
Q943.2
A
1000-3207(2015)03-0549-05
10.7541/2015.72
2014-06-24;
2014-11-25
国家自然科学基金青年基金(31400395); 国家博士后科学基金(2014M552006); 河南省科技厅国际合作项目(144300510046); 河南师范大学博士基金(qd13034)资助
刘洋(1983—), 男, 河南新乡人; 讲师; 主要从事蓝藻分子生态学研究。E-mail: liuyang6368@126.com
李仁辉(1965—), 研究员; 主要从事蓝藻分类系统以及藻类环境生物学研究。E-mail: reli@ihb.ac.cn
