新型卤代醇脱卤酶基因的克隆、序列分析及表达
2015-01-06邬敏辰
冯 峰, 胡 蝶, 余 涛, 曾 妍, 邬敏辰
(1.江南大学 药学院,江苏 无锡 214122;2.江南大学 生物工程学院,江苏 无锡 214122;3.江南大学 无锡医学院,江苏 无锡214122)
新型卤代醇脱卤酶基因的克隆、序列分析及表达
冯 峰1, 胡 蝶2, 余 涛1, 曾 妍1, 邬敏辰*3
(1.江南大学 药学院,江苏 无锡 214122;2.江南大学 生物工程学院,江苏 无锡 214122;3.江南大学 无锡医学院,江苏 无锡214122)
卤代醇脱卤酶可以催化合成具有光学纯的环氧化物及β-取代醇等高价值手性药物中间体。作者所在实验室利用宏基因组技术从无锡新区工业集中地污染土壤中筛选了一段编码基因Y,经测序知其片段包含有765 bp的核苷酸,编码254个氨基酸。经Blast软件分析,其氨基酸序列与来自根癌土壤杆菌 (Agrobacterium tumefaciens)的卤代醇脱卤酶HheC同源性为81%,且含有卤代醇脱卤酶类的保守催化三联体Ser132-Thr145-Arg149。推测其属于卤代醇脱卤酶HheC类,命名为SyHheC。根据大肠杆菌Escherichia coli BL21的密码子偏爱性,对基因Y进行密码子优化和人工合成,命名为SyhheC,构建重组表达质粒pET28a-SyhheC,转化感受态E.coli BL21,表达产物经镍柱亲和层析后获得电泳纯的重组卤代醇脱卤酶reSyHheC。SDS-PAGE分析,reSyHheC的相对分子质量为34 000。以2-溴-1-(4-硝基苯基)-乙醇为底物,reSyHheC催化底物产生2-(4-硝基苯基)环氧乙烷的比活性为5.2 U/mg。
卤代醇脱卤酶;宏基因组技术;序列分析;密码子优化;表达
卤 代 醇 脱 卤 酶 (EC 4.5.1.-,Halohydrin dehalogenase,HHDH)是细菌降解环境中重要污染物有机卤化物的关键酶之一,具有与其它已知脱卤酶不同的脱卤机制[1]。通过分子内亲核取代机制催化邻卤醇转化为环氧化物和卤化氢,是微生物降解重要的环境污染物有机卤化物的关键酶之一。同时该酶在催化环氧化物的开环反应中,可高选择性的催化接受如X-、N3-、NO2-、CN-等亲核基团,生成一系列光学纯的β-取代醇,因而在手性药物合成方面也有广阔的应用前景[2]。
目前,国内外对于产新酶菌株筛选的报道有底物筛选法、基因狩猎法和宏基因组技术等。底物筛选法具有工作量大、盲目性大等缺点;基因狩猎法虽有一定的优势,但却不能对那些不可培养的微生物的基因组进行运用,而这些不可培养的微生物却是新基因及新酶的重要来源;宏基因组技术通过直接从环境中提取全部微生物DNA,为后续的筛选提供更加全面的基因资源,增加了获得新功能基因的机会[3-4]。如Michael Kotika等[5]利用宏基因组技术从土壤中挖掘出环氧化物水解酶 (epoxide hydrolases),因而是一种快速有效的挖掘新酶的方法。
利用宏基因组技术,作者从无锡新区工业集中区污染土壤中筛选出了一段编码基因Y,经送上海生工测序得知其序列,并通过同源比对方法分析其氨基酸序列,推测其为一种新型卤代醇脱卤酶(SyHheC)。对该酶的基因序列进行密码子优化后经人工合成并命名为SyhheC;将优化基因与表达质粒pET28a连接于大肠杆菌 Escherichia coli BL21(DE3)中表达,诱导表达产物经镍离子亲和层析纯化,获得电泳纯的重组卤代醇脱卤酶reSyHheC,以2-溴-1-(4-硝基苯基)-乙醇为底物,测定reSyHheC的酶活力。本研究为该新型卤代醇脱卤酶的深入研究奠定了基础,同时也为其它新酶基因的开发提供了新的策略。
1 材料与方法
1.1 菌株、质粒和培养基
大肠杆菌E.coli JM109、E.coli BL21(DE3)和表达质粒pET28a:Invitrogen公司;质粒pUCm-T:上海Sangon公司;LB培养基:1 g/dL Tryptone、0.5 g/dL Yeast Extract、1 g/dL NaCl(固体培养基添加2 g/dL琼脂);富集培养基的配置参照文献[6]。
1.2 主要试剂和仪器
250 bp DNA Ladder marker、低相对分子质量蛋白质marker、rTaq DNA聚合酶、T4DNA连接酶、各种限制性内切酶:大连TaKaRa公司;2-溴-1-(4-硝基苯基)-乙醇、1,3-二氯-二丙醇 (1,3-DCP)、N,N-二甲基甲酰胺、盐酸半胱氨酸、K2HPO4和KH2PO4等其它试剂:国产或进口分析纯;快速DNA提取检测试剂盒 (KG203):天根生化科技 (北京)有限公司;一站式His标记蛋白质微量纯化套装:北京天恩泽基因科技有限公司;戴安UltiMate-3000高效液相色谱仪:戴安 (中国)有限公司:ZW&A-C18反相色谱柱:科奥美萃生物科技公司。
1.3 引物的设计与合成
用于验证宏基因组中是否含有卤代醇脱卤酶基因,其简并引物GB1,MB1的设计参照汤丽霞[7]等方法。根据已知的来自根癌土壤杆菌(Agrobacterium tumefaciens)的卤代醇脱卤酶HheC(序列号:AF397296)[1],结合生物信息学分析的手段,设计出一对特异性的上下游引物T2-F,T2-R,见表1。
1.4 卤代醇脱卤酶基因的克隆
土壤DNA提取参照Knietsch[7]等的方法进行。分别取2 g样品加入20 mL富集培养基中,30℃、180 r/min培养1 d;1 000 r/min离心10 min,收集上清液,当菌体OD600值为0.2~0.4时,10 000 r/min离心20 min收集菌体;得到的菌体加入20 mL不含酵母提取物的富集培养基中,含有10 mmol/L 1,3-DCP作为惟一碳源,30℃、180 r/min培养4 d,每天检测菌体浓度;收集菌体,用细菌基因组提取试剂盒(KG203)提取DNA,-20℃保存备用。以提取的细菌DNA为模板,GB1,MB2为引物按如下条件进行第一轮PCR扩增:94℃预变性5 min;94℃变性30 s,56℃退火30 s,72℃延伸60 s,30个循环;72℃充分延伸10 min。PCR产物经琼脂糖凝胶电泳检测、割胶回收、与pUCm-T连接,转化E.coli JM109,蓝白斑筛选阳性转化子,送上海Sangon公司测序。以提取的细菌DNA为模板,T2-F,T2-R为引物按如下条件进行第二轮PCR扩增:94℃预变性5 min;94℃变性30 s,56℃退火30 s,72℃延伸90 s,30个循环;72℃充分延伸10 min。PCR产物经琼脂糖凝胶电泳检测、割胶回收、与pUCm-T连接,转化E.coli JM109,蓝白斑筛选阳性转化子,送上海Sangon公司测序,将获得的基因命名为Y。
1.5 基因Y编码的氨基酸序列分析
利用DNAMAN 6.0软件将获得的基因Y翻译成氨基酸序列。用NCBI中的Blast在线工具和DNAMAN 6.0进行氨基酸序列同源性分析[8]。
1.6 卤代醇脱卤酶(SyHheC)基因的密码子优化
根据测序结果对基因Y进行密码子优化和人工合成[9],同时在目的基因两端分别加NcoⅠ和NotⅠ限制性酶切位点,在5'端添加组氨酸标签,并命名为SyhheC。优化后的基因由上海Sangon公司合成并与pUCm-T连接,获得重组质粒pUCm-TSyhheC。
1.7 卤代醇脱卤酶重组表达质粒的构建
分别用限制性内切酶NcoⅠ和NotⅠ双酶酶切质粒pUCm-T-SyhheC和表达载体pET28a,回收目的基因片段和载体片段,以T4DNA连接酶于16℃连接16 h,将连接产物转化感受态E.coli BL21,接种于含卡那霉素(100 μg/mL)的LB培养板上,于37℃培养过夜,挑取单菌落,接种于液体LB培养基中扩增,利用T7引物进行菌液PCR鉴定,挑选阳性重组子送上海Sangon公司测序。测序正确后命名该重组表达质粒为pET28a-SyhheC。
1.8 卤代醇脱卤酶的诱导表达
挑取阳性菌株E.coli BL21/pET28a-SyhheC接种于含卡那霉素的2 mL LB培养基中,37℃、220 r/min培养12 h。取300 μL培养液转接于30 mL含有100 μg/mL卡那霉素的LB液体培养基中,于37℃、220 r/min振荡培养至OD600为0.6~0.8时,向培养物中添加终浓度为1.0 mmol/L的IPTG,于25℃、220 r/min振荡培养12 h后,在4℃、8 000 r/min离心收集菌体[10]。以pET28a空质粒转化的E.coli BL21菌株在同样条件培养作为阴性对照,表达产物用SDS-PAGE进行检测分析。
1.9 重组卤代醇脱卤酶(reSyHheC)的纯化
将离心收集的重组菌 E.coli BL21/pET28a-SyhheC悬浮于0.1 mmol/L K2HPO4-KH2PO4缓冲液(pH 7.0)中进行超声破碎。破碎条件:工作4 s,间隔6 s,工作时间10 min。超声破碎后16 000 r/min离心20 min,上清液即为粗酶液。将粗酶液经微孔滤膜 (0.45 μm)过滤后上样至预先用100 mmol/L K2HPO4-KH2PO4缓冲液 (pH 7.0)平衡的1 mL镍离子亲合层析柱,结合 30 min后用 100 mmol/L K2HPO4-KH2PO4缓冲液(pH 7.0)配置成200 mmol/L的咪唑液洗脱,获得目的重组卤代醇脱卤酶(reSyHheC),全部纯化步骤在4℃下进行。纯化reSyHheC经SDS-PAGE检测,蛋白质浓度测定使用Bradford法,以牛血清蛋白作为标准蛋白质。
1.10 reSyHheC酶活力的测定
采用高效液相色谱法测定reSyHheC酶活力[11]。取100 μL、100 mmol/L的2-溴-1-(4-硝基苯基)-乙醇 (DMF配置)于1.5 mL EP管中,再加入800 μL、pH 7.0的柠檬酸-Na2HPO4缓冲液。于40℃预热5 min,加入100 μL适当稀释的酶液,40℃下反应8 min。吸取200 μL反应液加入800 μL甲醇终止反应,立即混匀进行高效液相色谱 (HPLC)分析。在上述反应条件下,每分钟产生1 μmol 2-(4-硝基苯基)环氧乙烷所需的酶量定义为1个酶活性单位(U)。
2 结果与讨论
2.1 卤代醇脱卤酶基因的克隆
以提取的细菌DNA为模板,GB1、MB2为引物进行PCR扩增,经1 g/dL琼脂糖凝胶电泳检测,在约520 bp处有一条明显的条带,见图1(泳道1),这与理论上简并引物GB1,MB2对来自根癌土壤杆菌(A.tumefaciens)的卤代醇脱卤酶 (HheC)基因PCR产物长度相符,割胶回收产物连接至pUCm-T,经过蓝白斑筛选和菌液PCR检测后进行测序。测序获得512 bp DNA序列,经Blast同源性分析,该序列与根癌土壤杆菌的卤代醇脱卤酶 (HheC)基因相似性为70%,表明该基因可能为一种卤代醇脱卤酶(HheC)基因。以提取的细菌DNA为模板,以T2-F,T2-R为特异性引物进行PCR扩增,经1 g/dL琼脂糖凝胶电泳检测PCR产物,结果显示,在约760 bp处有一条特异性的条带 (泳道2),PCR产物长度与预期相符,割胶回收产物连接至pUCm-T,经蓝白斑筛选和菌液PCR检测后进行测序。测序结果显示获得的Y基因片段含765 bp的核苷酸,包含以简便引物扩增获得的序列,该基因共编码254个氨基酸。
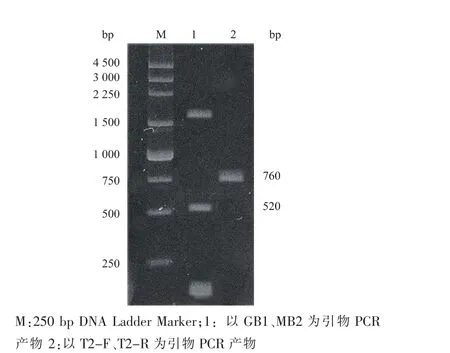
图1 PCR扩增产物的核酸电泳分析Fig.1 Nucleic acid electrophoresis of PCR amplifications
2.2 基因Y编码的氨基酸序列分析
利用NCBI数据库的BLAST软件,以Y基因的氨基酸序列为模板进行同源性序列比对,结果见图2。其氨基酸序列与已知的卤代醇脱卤酶A.tumefaciens HheC(PDB:1PWX_A)、A.tumefaciens HheB(AAD34609.1)和Arthrobacter sp.AD2 HheA(PDB:1ZMO_A)的相似性分别为 81%、75%和35%。另外,SyHheC还含有保守的卤代醇脱卤酶催化三联体Ser132Thr145-Arg149,故推测SyHheC属于卤代醇脱卤酶HheC类。

图2 不同卤代醇脱卤酶氨基酸的序列比对Fig.2 Amino acid sequence alignment of different halogenated alcohol dehalogenation enzymes
2.3卤代醇脱卤酶(SyHheC)重组表达质粒的构建
根据大肠杆菌密码子偏好性,将克隆获得的Y基因进行密码子优化,优化后的基因序列SyhheC已提交GenBank数据库,登录号为 (KF853591.1),人工合成SyhheC与原Y基因序列对比见图3。

图3 人工合成SyhheC序列和筛选Y基因序列对比Fig.3 Compared with designed synthetic gene sequence SyhheC and the screening gene sequence Y
通过密码子优化后获得重组质粒pUCm-TSyhheC,经NcoⅠ和NotⅠ双酶切,与同样双酶切的pET28a进行连接,转化E.coli BL21。经T7通用引物PCR鉴定,可见约1 100 bp的条带,表明重组表达质粒pET28a-SyhheC成功转入E.coli BL21。测序结果表明,pET28a-SyhheC中的SyhheC序列与预期相符,见图4。
2.4 reSyHheC表达及纯化
重组菌E.coli BL21/pET28a-SyhheC和阴性菌E.coli BL21/pET28a,经1.0 mmol/L的IPTG于25℃诱导12 h后,SDS-PAGE结果见图5。与对照E.coli BL21/pET28a相比 (图5中泳道2),重组菌E.coli BL21/pET28a-SyhheC的表达产物在表观相对分子质量34 000处有明显的蛋白质条带 (图5中泳道3)。重组菌经超声破碎、离心获得粗酶液,粗酶液经镍离子亲和层析纯化,SDS-PAGE结果显示,蛋白质为单一条带 (图5中泳道1)。以底物2-溴-1-(4-硝基苯基)-乙醇为底物,测得reSyHheC比活性为5 U/mg,而E.coli BL21/pET28a中并未检测到卤代醇脱卤酶活性。结果表明,SyhheC基因在大肠杆菌中实现了高效表达。
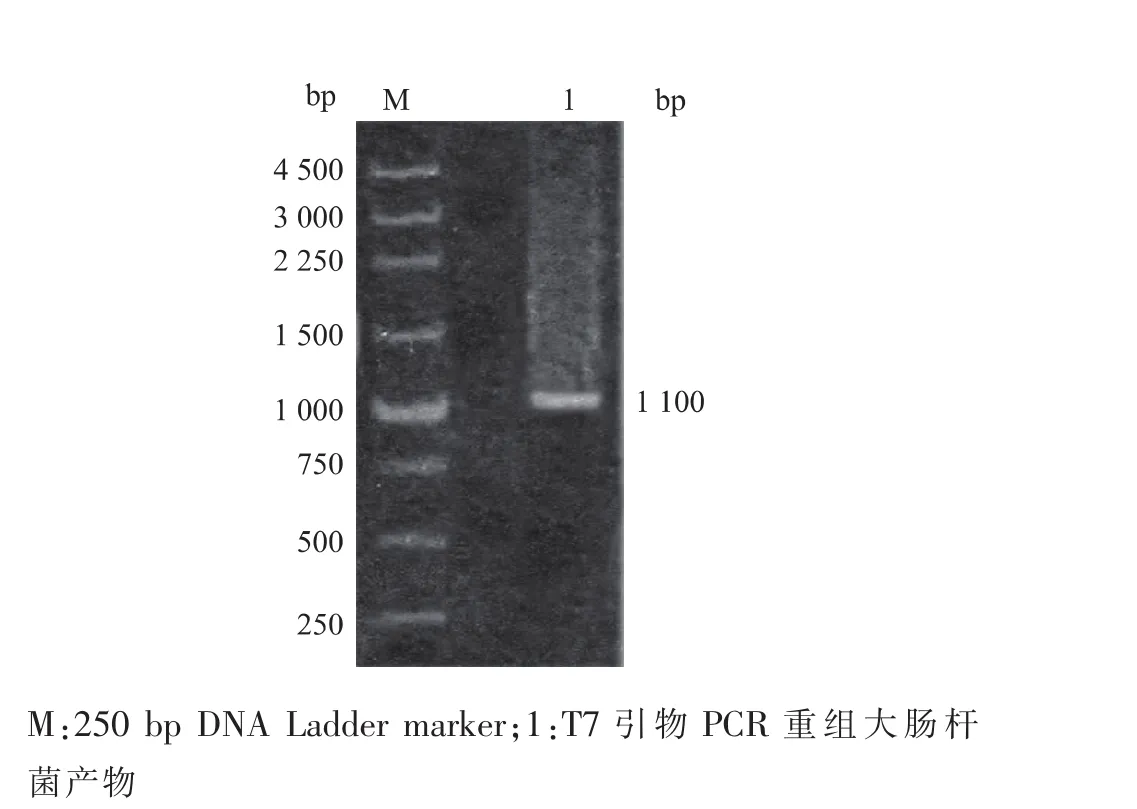
图4 重组表达质粒的鉴定Fig.4 Identification of recombinant expression plasmid
3 结语
近年来,关于卤代醇脱卤酶的研究报道逐年增多。传统的通过基因或cDNA文库开发新基因的方法工作量较大,而且不易操作[12]。随着生物信息学技术的发展,挖掘新基因的手段也越来越多[13]。利用生物信息学和基因组学相结合的方法从土壤中挖掘具有优良性状的新型酶是一条获取优良酶的有效途径。
作者成功地利用宏基因组技术从土壤中筛选出一段编码基因Y,其编码的氨基酸序列与已知的来自根癌土壤杆菌 (A.tumefaciens)的卤代醇脱卤酶(HheC)的同源性为81%。并含有保守的卤代醇脱卤酶催化三联体Ser132-Thr145-Arg149,推测其为一种新型卤代醇脱卤酶。根据大肠杆菌密码子偏好性对Y基因进行密码子优化并人工合成,与pET28a质粒连接后实现该基因在大肠杆菌E.coli BL21的高效表达。经镍离子亲和层析获得纯化表达蛋白质,通过SDS-PAGE鉴定出reSyHheC的相对分子质量在约34 000。以2-溴-1-(4-硝基苯基)-乙醇为底物,reSyHheC酶催化底物产生2-(4-硝基苯基)环氧乙烷,测定其比酶活为5 U/mg。本研究挖掘了一种新型的HheC基因,为卤代醇脱卤酶的深入研究奠定了理论基础,也为其它新酶基因的开发提供了新的策略。
[1]DeJong R M,Tiesinga J J,Rozeboom H J,et al.Structure and mechanism of a bacterial haloalcohol dehalogenase:a new variation of the short-chain dehydrogenase/reductase fold without an NAD (P)H binding site[J].The EMBO Journal,2003,22(19):4933-4944.
[2]郑楷,汤丽霞.一种新型微生物卤醇脱卤酶的研究进展[J].生物技术通讯,2010,21(21):121-125.
ZHENG Kai,TANG Lixia.Current research advances in bacterial halohydrin dehalogenases[J].Letters in Biotechnology,2010,21(21):121-125.(in Chinese)
[3]王魁,汪思迪,黄睿,等.宏基因组学挖掘新型生物催化剂的研究进展[J].生物工程学报,2012,28(4):420-431.
WANG Kui,WANG Sidi,HUANG Rui,et al.Advances of metagenomics in discovering novel biocatalysts[J].Chin J Biotech,2012,28(4):420-431.(in Chinese)
[4]Teeling H,Glockner F O.Current opportunities and challenges in microbial metagenome analysis-bioinformatic perspective[J]. Briefings in Bioinformatics,2012,13(6):728-742.
[5]Kotik M,Stepanek V,Grulich M,et al.Access to enantiopure aromatic epoxides and diols using epoxide hydrolases derived from total biofilter DNA[J].Journal of Molecular Catalysis B:Enzymatic,2010,65:41-48.
[6]聂洪丽,汤丽霞,张诗海,等.富集培养及高质量DNA提取有利于从土壤宏基因组中获取新卤醇脱卤酶基因[J].生物技术通讯,2010,21(6):846-850.
NIE Hongli,TANG Lixia,ZHANG Shihai,et al.Enrichment cultivation and high quality DNA extraction represent an available strategy for isolation new halohydrin dehalogenase genes from soil metagenome[J].Letters in Biotechnology,2010,21(6):846-850.(in Chinese)
[7]Knietsch A,Waschkowitz T,Bowien S,et al.Construction and screening of metagenomic libraries derived from enrichment cultures:generation of a gene bank for genes conferring alcohol oxidoreductase activity on Escherichia coli[J].Applied& Environmental Microbiology,2003,69(3):1408-1416.
[8]张慧敏,李剑芳,邬敏辰.圆弧青霉脂肪酶基因序列的生物信息学分析[J].食品与生物技术学报,2010,4(29):602-608.
ZHANG Huimin,LI Jianfang,WU Minchen.Bioinformatics Analysis on the sequence of lipase gene from Penicillium cyclopium [J].Journal of Food Science and Biotechnology,2010,29(4):602-608.(in Chinese)
[9]金光泽,段作营,张莲芬,等.重组融合人血清白蛋白一人白介素一2 C125A突变体在毕赤酵母中的表达[J].食品与生物技术学报,2010,29(4):595-601.
JIN Guangze,DUAN Zuoying,ZHANG Lianfen,et al.Expression of the fusion protein human serum album mutanhuman interleukin 2 C125A in Pichia pastoris[J].Journal of Food Science and Biotechnology,2010,29(4):595-601.(in Chinese)
[10]李阳,汤丽霞,郑楷,等.重组卤醇脱卤酶的表达优化及纯化[J].化工学报,2010,61(12):3212-3219.
LI Yang,TANG Lixia,ZHENG Kai,et al.Optimizing expression and halohydrin dehalogenase purification of recombinant from A.radiobacter AD1[J].CIESC Journal,2010,61(12):3212-3219.(in Chinese)
[11]Jin H X,Hu Z C,Liu Z Q,et al.Nitrite-mediated synthesis of chiral epichlorohydrin using halohydrin dehalogenase from Agrobacterium radiobacter AD1[J].Biotechnology and Applied Biochemistry,2012,59:170-177.
[12]Wang J Q,Zhang H M,Wu M C,et al.Cloning and sequence analysis of a novel xylanase gene,Auxyn10A,from Aspergillus usamii[J].Biotechnology Letters,2011,33(5):1029-1038.
[13]Shi H L,Yin X,Wu M C,et al.Cloning and bioinformatics analysis of an endoglucanase gene(Aucel12A)from Aspergillus usamii and its functional expression in Pichia pastoris[J].Journal of Industrial Microbiology and Biotechnology,2012,39(2):347-357.
Gene Cloning,Sequence Analyais and Gene Expression of A New Halohydrin Dehalogenase
FENG Feng1, HU Die2, YU Tao1, ZENG Yan1, WU Minchen*3
(1.School of Pharmaceutical Science,Jiangnan University,Wuxi 214122,China;2.School of Biotechnology,Jiangnan University,Wuxi 214122,China;3.Wuxi Medical School,Jiangnan University,Wuxi 214122,China)
Halohydrin dehalogenase catalyzes the synthesis ofoptically pure epoxides,β-substituted alcohol and other similar high-value chiral drug intermediates.Our laboratory screened an encoding gene Y from contaminated soil in Wuxi new district industrial park using metagenomic technology.The gene Y contains 765 bp nucleotides and encodes 254 amino acids. According to the Blast analysis,the amino acids sequence shares 81%homology with HheC from Agrobacterium tumefaciens and also has Ser132-Thr145-Arg149catalytic triad which is the same with the known Halohydrin dehalogenases.Thus,we speculated it belongs to HheC.According to Escherichiacoli BL21 codon preference,the optimized gene was synthesized and named as SyhheC.The constructed expression plasmid pET28a-SyhheC was transformed into E.coli BL21.The expressed enzyme with an apparent molecular weight of 34 000 by SDS-PAGE analysis was purified to homogeneity using Ni-NTA affinity chromatography and characterized using the substrate of 2-bromo-1-(4-nitrophenyl)ethanol.The activity of purified reSyHheC was 5.2 U/mg.This study provides a new strategy to excavate new enzymes with excellent characters from the soil.
halohydrin dehalogenase,metagenomic technology,sequence analysis,codon optimization,expression
Q 785
A
1673—1689(2015)05—0494—07
2014-02-24
国家自然科学基金项目(31271811)。
*通信作者:邬敏辰 (1962—),男,江苏无锡人,理学博士,教授,博士研究生导师,主要从事酶工程与基因工程研究。
E-mail:biowmc@126.com
