基因芯片快速鉴定分枝杆菌菌种方法的建立
2015-01-05唐曙明李爱敏陈海霞杨自华
唐曙明,李爱敏,陈海霞,杨自华
(深圳市人民医院检验科,广东深圳 518000)
基因芯片快速鉴定分枝杆菌菌种方法的建立
唐曙明,李爱敏,陈海霞,杨自华△
(深圳市人民医院检验科,广东深圳 518000)
目的 利用基因芯片技术,建立一种能快速、准确鉴定分枝杆菌菌种的方法,并验证其临床应用价值。方法根据23种分枝杆菌基因序列设计特异性探针并制作基因芯片,通过PCR-反向点杂交鉴定23种分枝杆菌标准菌株、9 种非分枝杆菌和103株分枝杆菌临床分离株的菌种。结果应用基因芯片技术检测23 种分枝杆菌标准菌株和9种非分枝杆菌菌株,特异性为100%。103株临床分离株经鉴定87株为结核杆菌复合群(MTC);16株为非结核分枝杆菌(NTM),其中脓肿分枝杆菌5株、胞内分枝杆菌3株、鸟分枝杆菌3株、偶发分枝杆菌2株,以及堪萨斯分枝杆菌、海分枝杆菌、戈登分枝杆菌各1株。103株临床分离株鉴定结果与测序法完全一致,该方法最低检出限为103copy/mL。结论采用基因芯片技术能快速鉴定分枝杆菌菌种,并区分MTC和NTM,具有简便快速及准确性、特异性、灵敏度高的优点。
分枝杆菌属;菌种鉴定;寡核苷酸序列分析
近年来,中国分枝杆菌疫情不断上升,尤其是非结核分枝杆菌(non-tuberculous mycobacteria,NTM)感染增多趋势明显。2010年第五次全国结核病流行病学抽样调查结果显示,NTM在分枝杆菌分离菌株中占比高达22.9%,相比1990年的4.9%,涨幅超过360%。NTM病和结核病有着相似的临床表现,但对药物的敏感性完全不同,若当成结核病用药,其耐药率为95.9%;耐多药率为83.7%,往往导致治疗失败[1-3]。因此,建立一种快速、简便的分枝杆菌菌种鉴定方法以及时、准确地鉴定分枝杆菌菌种,对诊断、控制和治疗分枝杆菌感染引起的疾病具有重要的意义。随着分子生物学技术的不断发展,基因芯片检测技术得到了广泛应用。基因芯片技术因其高通量、平行性、快速灵敏、样品用量少等优点为病原菌的分子诊断提供了有力的技术平台。将基因芯片技术应用于分枝杆菌菌种鉴定中,可实现对多种分枝杆菌的平行海量检测,从而提高病原菌的检出效率。本研究建立基于基因芯片技术的检测方法,对临床分枝杆菌分离株进行了检测,并以测序法为“金标准”,探讨其临床应用价值。
1 材料与方法
1.1材料
1.1.1标本来源 23种分枝杆菌标准株购于中国生物制品检定所,具体菌株见表1。同时还购买了9种非分枝杆菌标准株(大肠埃希菌、肺炎链球菌、卡他布兰汉菌、红色红球菌、星形奴卡菌、绿脓假单胞菌、肺炎克雷伯菌、表皮葡萄球菌、草绿色链球菌)。103株分枝杆菌临床分离株来自本院2012年2~8月门诊和住院患者。
1.1.2仪器与试剂 PCR仪购于美国ABI公司;电泳仪购于北京六一仪器厂;核酸/蛋白凝胶图像管理系统型号为GSG-2000;分子杂交箱购于韩国FINE PCR公司。
1.1.3探针设计 根据23种常见致病性分枝杆菌的基因序列,在其16S~23S rRNA基因间隔区设计PCR引物和特异性探针。每种菌株均设计多组探针,经比较分析后,最终选择的探针序列见表1。另设一条保守区序列作为内控点(内对照),以监测整个实验过程的有效性。
1.2方法
1.2.1基因芯片制备 尼龙纤维膜浸入5% EDAC 20 min,纯水洗膜,晾干。探针稀释液(5 μm/L)点样于膜条上,晾干后浸于0.1 mol/L NaOH 3 min,纯水洗膜,晾干,室温储藏备用。
1.2.2分枝杆菌核酸制备 分枝杆菌标准株和临床分离株、非分枝杆菌标准株菌悬液直接离心,痰液样本经消化、离心后收集沉淀,将沉淀进行裂解、煮沸、离心,取上清液备用。
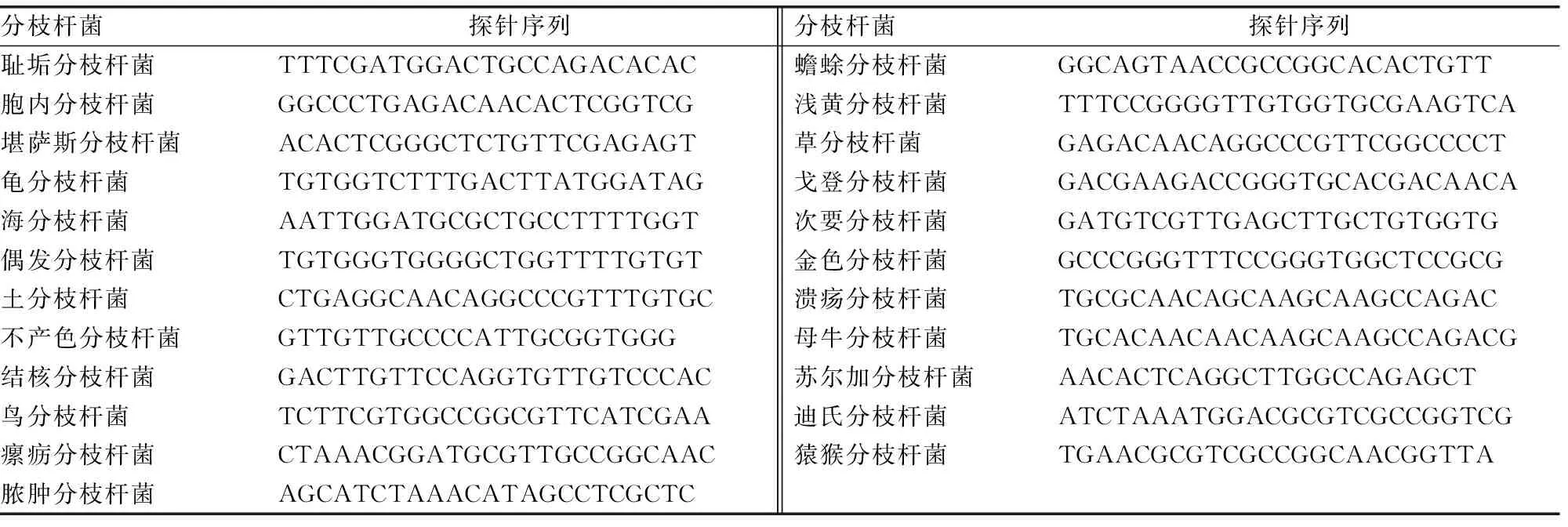
表1 23种分枝杆菌特异性探针序列
1.2.3引物设计及PCR扩增 将Genebank中的分枝杆菌核酸序列进行比对分析,设计可以特异性扩增23种常见致病性分枝杆菌的特定片段作为引物对,引物的5′进行生物素标记。在25 μL 反应体系中,dNTP 混合物的终浓度为0.2 mmol/L,引物的终浓度均为0.4 μmol/L,DNA 模板的终浓度为20 ng,Taq DNA 聚合酶2.5 U。置于PCR仪上扩增,然后在2% 琼脂糖凝胶中电泳检测扩增产物。
1.2.4杂交与显色 将制备好的膜条编号后放入15 mL杂交管中,加入扩增产物和6 mL杂交缓冲液(2×SSC、0.1%SDS)置于沸水浴10 min,放入已预热的杂交箱中,57 ℃杂交1.5 h。取出膜条,移至装有预热至57 ℃的洗液(0.5×SSC、0.1%SDS)50 mL管中,于57 ℃轻摇洗涤15 min。取出膜条,按杂交缓冲液:POD=2 000∶1配制孵育液,室温轻摇孵育30 min,弃去POD溶液。用杂交缓冲液室温轻摇洗2次,每次5 min。用柠檬酸缓冲液(0.1 mol/L)室温洗膜2 min,同时配制显色液(19 mL 0.1 mol/L柠檬酸钠、 1 mL 2 mg/mL TMB、10 μL 3% H2O2),将膜条浸泡于显色液中避光显色10~15 min,观察结果。
1.2.5结果分析 膜条在内控点(CC)位置出现蓝色斑点提示本次检测过程正常;其他出现的蓝色斑点表示为相应的分枝杆菌菌种。
1.2.6确定最低检出限 23种分枝杆菌标准菌株先用结核分枝杆菌核酸检测试剂盒(凯杰生物,careTM TB PCR ASSAY)定量后进行系列稀释,分别制成104、103、102、101copy/mL的菌液,按1.2.2~1.2.5步骤检测,以确定新建方法的最低检出限。
1.2.7测序分析 经芯片鉴定为NTM的临床分离株进一步送上海英骏生物技术有限公司进行测序,以检测PCR扩增的特异性和芯片鉴定结果的准确性。
2 结 果
2.1基因芯片检测结果 用基因芯片对103株分枝杆菌临床分离株进行检测,分别检出87株结核杆菌复合群(MTC)和16株NTM。在16株NTM中,分别检出5株脓肿分枝杆菌、3株胞内分枝杆菌、3株鸟分枝杆菌、2株偶发分枝杆菌、1株堪萨斯分枝杆菌、1株海分枝杆菌、1株戈登分枝杆菌。
2.2测序鉴定结果 将16株经基因芯片鉴定为NTM的临床分离株送上海英骏生物技术有限公司进行DNA 测序。测序结果与GeneBank中已报道的序列进行比对分析,芯片鉴定结果和测序结果完全一致。部分NTM测序结果,见图1。

A:脓肿分枝杆菌;B:胞内分枝杆菌;C:鸟分枝杆菌;D:偶发分枝杆菌。
图1 部分NTM测序结果
2.3基因芯片的准确性与特异性 基因芯片与23 种分枝杆菌标准菌株的扩增产物均可进行杂交反应,检测准确性为100%;9种非分枝杆菌标准菌株扩增产物与基因芯片上的探针均不杂交,检测特异性为100%。
2.4基因芯片最低检出限的确定 对23种分枝杆菌标准菌株系列稀释菌液进行检测,各菌株浓度在103copy/mL时检测结果仍为阳性;当菌液稀释至102copy/mL时,MTC、鸟分枝杆菌、偶发分枝杆菌、堪萨斯分枝杆菌等19种菌株的阳性显色已经明显变弱,而海分枝杆菌、戈登分枝杆菌、脓肿分枝杆菌、胞内分枝杆菌的检测结果为阴性;当菌液浓度稀释至101copy/mL时,所有膜条未见显色,结果见表2。
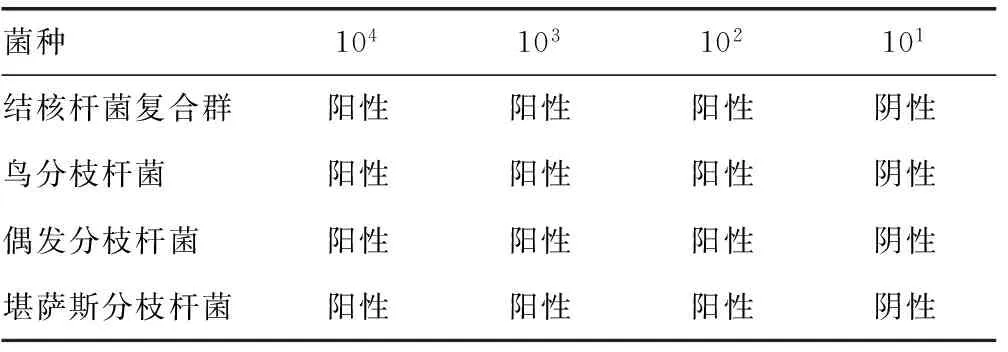
表2 部分分枝杆菌标准株不同浓度的检出结果(copy/mL)

续表2 部分分枝杆菌标准株不同浓度的检出结果(copy/mL)
3 讨 论
NTM引起的感染日益增多,特别是由其引发的医院感染出现频率逐渐增高,已越来越受到人们的关注[4-6]。快速、准确、灵敏、特异地对致病性分枝杆菌做出诊断,是有效预防分枝杆菌病发生和控制其迅速传播的前提和关键,对治疗和疫情控制具有重要意义。传统的分枝杆菌筛查方法主要有涂片抗酸染色法、罗氏培养基培养法和结核抗体金标法,其中抗酸染色涂片法具有简便、快捷、价格低廉的优点,但其敏感性太低;而罗氏培养基培养法报告时间较长(2~8周);结核抗体金标法则只能检测结核分枝杆菌,对NTM检测无效。后期发展的BACTEC快速菌种鉴定法可初步鉴别MTC和NTM,但其培养周期仍较长(2~6 d),有研究对BACTECTB460检测出来的NTM进行菌种鉴定,发现有7.7%的菌株为结核分枝杆菌[7]。随着分子生物学技术的飞速发展,PCR-单链构象多态性方法也被应用于分枝杆菌菌种鉴定,该方法通过分析分枝杆菌16S rRNA,根据其电泳图谱与标准株的相似性进行菌种鉴定,但结果显示对结核分枝杆菌、牛结核分枝杆菌及堪萨斯分枝杆菌、胃分支杆菌之间不能特异性区分[8]。前述方法敏感性或特异性不强,很难满足临床诊断的需要。
基因芯片技术是伴随人类基因组计划的实施而发展起来的一门新兴技术,该技术将成千上万个DNA或寡核苷酸片段固定在玻璃、尼龙膜或硅片等载体上,与标记的样品杂交,分析样品中基因表达、基因序列、基因突变和多态性变化等情况。该技术将PCR技术的高灵敏度和反向点杂交技术的高特异性相结合,真正达到了快速检测的要求,其结果更加准确可靠。本研究以测序法为对照,采用本院与亚能生物技术(深圳)有限公司联合研发的分枝杆菌菌种鉴定试剂盒,利用膜芯片技术,在膜上固定一系列的特异性探针,根据分枝杆菌16S~23S rRNA基因间隔区序列设计引物,利用PCR体外扩增各菌株的DNA,与固定在膜上的基因型特异探针杂交,再通过显色判断分枝杆菌菌种。实验结果显示,本研究中基因芯片上的探针只与23种分枝杆菌的扩增产物杂交,与9种非分枝杆菌标准株扩增产物均不杂交,为进一步验证本芯片的准确性,将16株临床NTM进行DNA测序,证实与GenBank对应序列相一致,其检测特异性达到了100%。据报道,基因芯片检测灵敏度比PCR-电泳法高10倍[9-10],从本研究对23种类型的分枝杆菌标准菌株的系列稀释检测结果可以看出,该方法的最低检出限达到了103copy/mL,解决了传统方法对临床标本中低浓度分枝杆菌不能检出的难题,提高了检测灵敏度。
上述实验结果显示,本研究所建立的基因芯片法具备高通量、高特异性、高灵敏度、操作简便、结果直观的优点,且能在6~8 h内直接测定痰液标本中的MTC和其他22种NTM,具有很好的临床应用价值,是一项值得推广的技术。
[1]王黎霞,成诗明,陈明亭,等.2010年全国第五次结核病流行病学抽样调查报告[J].中国防痨杂志,2012,34(8):485-507.
[2]Wu X,Yang Y,Zhang J,et al.Comparison of antibody responses to seventeen antigens from Mycobacterium tuberculosis[J].Clin Chim Acta,2010,411(19):1520-1528.
[3]张翊,卢建平,叶淼.四种结核分枝杆菌检测方法的临床应用评价[J].中国防痨杂志,2006,28(1):14-17.
[4]Somoskovi A,Mester J,Hale YM,et al.Laboratory diagnosis of nontuberculous mycobacteria[J].Clin Chest Med,2002,23(6):585-597.
[5]朱晓娥,袁耿彪.基因芯片技术在基因突变诊断中的应用及其前景[J].重庆医学,2010,39(22):3128-3131.
[6]Sumi S,Radhakrishnan VV.Diagnostic significance of humoral immune responses to recombinant antigens of Mycobacterium tuberculosis in patients with pleural tuberculosis [J].J Clin Lab Anal,2010,24(5):283-288.
[7]赵亭,潘欣.应用BACTEC 460TB系统区分出的非结核分枝杆菌进一步菌种鉴定意义的探讨[J].中国防痨杂志,1998,20(1):29-31.
[8]吴雪琼,张俊仙,刘佳文,等.PCR-SSCP分枝杆菌菌种初步鉴定方法的建立及其应用[J].中国现代医学杂志,2000,10(7):25-26.
[9]翟俊辉,宋亚军,杜宗敏,等.通用基因芯片检测感染性细菌方法的研究[J].中国公共卫生,2003,19(4):430-431.
[10]Jeda A,Witek A,Janikowska G,et al.Expression profile of genes associated with the histaminergic system estimated by oligonucleotide microarray analysis HG-U133A in women with endometrial adenocarcinoma [J].Ginekol Pol,2014,85(3):172-179.
Establishment of a rapid and accurate gene microarray for identifying Mycobacterium species*
TangShuming,LiAimin,ChenHaixia,YangZihua△
(DepartmentofClinicalLaboratory,ShenzhenPeople′sHospital,Shenzhen,Guangdong518000,China)
Objective To establish a rapid and accurate method for the identification of Mycobacterium species by the gene microarray and to verify its clinical application value.Methods According to the gene sequence of 23 species of Mycobacteria,the specific probes were designed and the gene chips were prepared.23 Mycobacterial standard strains,9 non-mycobacterial strains,103 clinically isolated mycobacterial strains were detected by PCR-based reverse blot hybridization assay in the gene chip.Results 23 mycobacterial standard strains,9 non-mycobacterial strains were detected by gene chip,the results showed that the specificity was 100%.Of 103 mycobacterial clinically isolated strains,87 strains were identified as Mycobacterium tuberculosis compounds (MTC) and 16 strains as non-tuberculosis mycobacteria (NTM) including 5 strains of M.abscessus,3 strains of M.intracellulare,3 strains of M.avium,2 strains of M.fortuitum,1 strain of M.kansas,1 strain of M.marinum and 1 strain of M.gordonae.The identification results of 103 clinically isolated strains were completely consistent with the sequencing results.The lowest detection limit by this method was 103copies/mL.Conclusion The gene microarray technique for rapidly identifying Mycobacteria and differentiate MTC and NTM has the advantages of simpleness,rapidness,high accuracy,high specificity and high sensitivity.
mycobacterium;species identification;oligonucleotide array sequence analysis
术与方法·
10.3969/j.issn.1671-8348.2015.11.026
深圳市科技局资助项目(201102151)。
唐曙明(1975-),副主任技师,硕士研究生,主要从事分子诊断技术研究。
△通讯作者,Tel:13590265712;E-mail:tangshuming@126.com。
R446.5
A
1671-8348(2015)11-1516-03
2014-10-28
2015-01-20)
