花生叶全长cDNA文库的构建和部分表达序列标签分析
2014-12-24蔡铁城熊发前唐荣华周双彪庄伟建
陈 华,蔡铁城,张 冲,邓 烨,熊发前,唐荣华,周双彪,庄伟建
(1.福建省作物分子与细胞生物学重点实验室,福建福州350002;2.广西农业科学院经济作物研究所,广西南宁510225)
花生起源于南美洲热带、亚热带地区,是我国乃至世界上重要的油料经济作物.叶片是花生进行光合作用合成有机营养并贮藏养分的主要器官,但花生叶部病害是花生生产的重要问题,如早斑病、晚斑病、锈病、网斑病以及近年在我国发生的一种新病害——花生叶部菌核病等[1].花生叶部病害的种类繁多,甚至不同病害会同时发生,严重时,叶柄、托叶、茎秆等部位也会发病造成花生大幅减产[2],单靠作物轮作、化学控制等措施并不能有效控制危害[3].目前生产上尚缺乏对多种叶部病害均具有较强抗性的花生品种.因此,选育具有广谱抗性的花生品种十分必要.近年来,利用基因工程手段培育作物新品种已成为一种有效途径.因此,克隆花生抗性基因可为花生分子育种提供良好的基因资源.
cDNA文库的构建和筛选是基因克隆的有效方法之一.全长cDNA文库则是大规模获得基因信息的有效途径,尤其对基因组庞大,暂时尚未获得全基因组序列的生物,更是开展功能基因组研究的重要途径[4].SMART技术由Clontech公司创建,主要利用PrimeScript RT反转录酶和SfiⅠ的特性,能够在原始RNA用量较少的情况下,快速、简单的构建全长cDNA文库.目前,SMART已被广泛应用于各种生物全长cDNA文库的构建.利用SMART技术已有多个花生全长cDNA文库的报道[5-10],但是利用SMART技术构建花生叶全长cDNA文库尚未见报道.本研究利用SMART技术在成功构建了多个花生器官全长cDNA文库的基础上,构建了花生叶全长cDNA文库,以期为进一步分离、克隆花生重要基因提供基础.
1 材料与方法
1.1 材料
花生品种‘闽花6号’为福建农林大学油料作物研究所提供.于大田种植,在苗期、花针期、结荚期、饱果成熟期取叶片,经液氮速冻后置-80℃冰箱保存.
1.2 主要试剂
Primer Script逆转录酶,RNase Inhibitor,SfiⅠ,Ex Taq,dNTP、DL2000 和 DL15000 分子标准等购自大连宝生物公司(TAKARA),连接酶购于NEB,电转化感受态细胞DH5α由本实验室制备,胶回收试剂盒购自杭州博日科技有限公司,其他试剂采用国产分析纯.
1.3 方法
1.3.1 花生叶总RNA的纯化 将采集的不同生育时期的花生叶片等量混合,于液氮中研磨成粉末,参照陈华等[7]的CTAB法提取花生叶总RNA,用1.1%琼脂糖凝胶电泳检测其完整性.用ND-2000型分光光度计(美国Thermo公司)检测总RNA的浓度和纯度.
1.3.2 cDNA第一链的合成 在0.2 mL的 DEPC处理过的 PCR 管中,依次加入2 μg的 RNA,1 μL 3'PCR Primer 引物(5'-AAGCAGTGGTATCAACGCAGAGTGGCCGAGGCGGCCGA-d(T)30N-1N-3'),1 μL SMART IV Oligonucleotide引物(5'-AAGCAGTGGTATCAACGCAGAGTGGCCATTACGGCCGGG-3'),瞬间离心,置于65℃的水浴锅反应5 min,迅速置于冰上2 min,瞬间离心数秒.依次加入以下试剂:4 μL 5×Prime Script Buffer,1 μL dNTPs(10 mm)混合液,20 units RNase 抑制剂,1 μL PrimeScript反转录酶,加 ddH2O 补足至15 μL,轻轻混匀,瞬间离心后于PCR仪上42℃反应1 h后,70℃加热15 min,最后冰上冷却.
1.3.3 LD-PCR 法合成双链 cDNA 在0.2 mL 的 PCR 管中依次加入以下试剂:80 μL ddH2O,10 μL 10 ×Ex Taq Buffer,2 μL dNTP Mixture(10 mM),4 μL 5'PCR 引物(5'-AAGCAGTGGTATCAACGCAGAGT-3'),0.5 μL Ex Taq 酶,2μL cDNA 第一链.PCR 反应条件为95 ℃ 1 min;95 ℃ 15 s,64 ℃ 30 s,72 ℃ 6 min,15个循环.取2 μL的产物,用1%的琼脂糖凝胶电泳进行检测.
1.3.4 全长cDNA文库的构建 取100 μL双链cDNA,加入4 μL蛋白酶K(20g·L-1),45℃温浴反应20 min,去除蛋白后,溶于79 μL去离子水.加入15 μL SfiⅠ酶于50℃酶切2 h.将上述酶切产物经1.5%琼脂糖凝胶电泳,回收0.5-5.0 kb之间的片段,浓缩沉淀后溶于7 μL去离子水中.酶切产物在T4 DNA连接酶作用下,与质粒载体pDNR-LIB于16℃下连接过夜,连接体系为5 μL.连接产物经电击转化入45 μL大肠杆菌DH5a感受态细胞后迅速加入950 μL LB培养基,于37℃恒温震荡培养1 h,即得原始文库.
1.3.5 文库质量鉴定 以M13通用引物(5'-GTAAAACGACGGCCAG-3'、5'-CAGGAAACAGCTATGAC-3')对随机选取的单菌落进行PCR检测,反应结束后取5 μL PCR产物进行检测,确定文库插入片段的大小和重组率.
1.3.6 EST测序及分析 随机挑取50个单克隆分别接种于加氯霉素的LB液体培养基上,于37℃摇床250 r·min-1培养过夜后加甘油保存菌种,将菌液送至上海国家人类基因组南方研究中心进行测序.将测序得到的序列去除载体序列后进行生物信息学分析.
2 结果与分析
2.1 总RNA提取
提取的总RNA经过凝胶电泳分析表明28S和18S条带清晰,无拖尾(图1),两者亮度比约为2∶1,说明所提取的RNA完好无降解.紫外分光光度计测定D260nm/D280nm值为1.92,表明获得的RNA纯度较高,可以用来进行文库构建.
2.2 双链cDNA扩增
以总RNA为模板进行单链cDNA合成并进行LD-PCR合成双链cDNA.经1%琼脂糖凝胶电泳检测,dscDNA弥散分布于500-5000 bp之间(图2),表明双链合成质量很高,可以满足全长文库构建的需要.
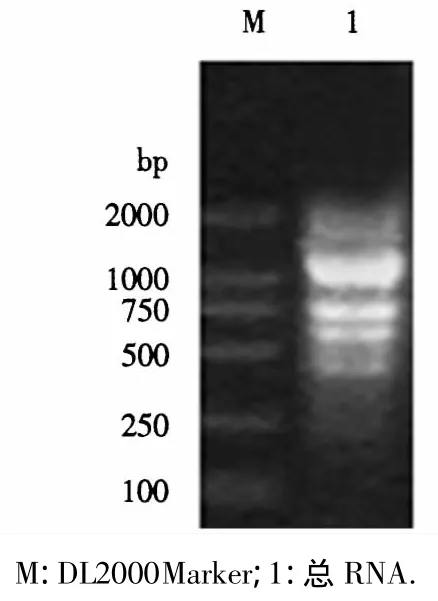
图1 花生叶总RNA电泳图Fig.1 Gel electrophoresis of total RNA isolated from peanut leaf
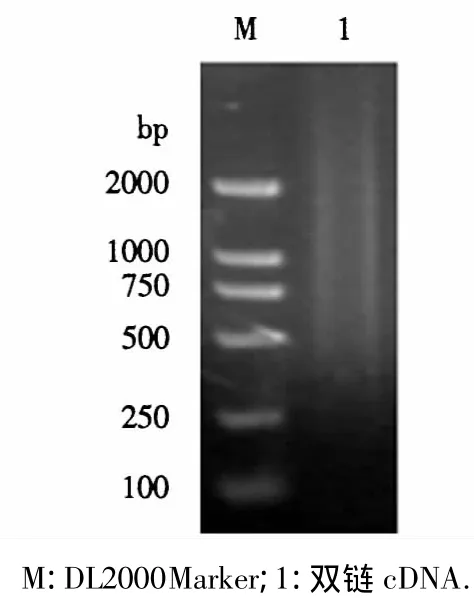
图2 花生叶双链cDNA电泳图Fig.2 Gel electrophoresis of dscDNA of peanut leaf
2.3 文库质量
对文库随机挑取的单菌落进行菌落PCR,如图3所示,凝胶电泳检测结果显示外源片段大小主要分布在500-2000 bp,外源片段插入率为100%,平均长度约为1000 bp.对文库涂板后菌落数量的计算得到原始库容为2.25×106cfu·mL-1,达到文库的质量要求.
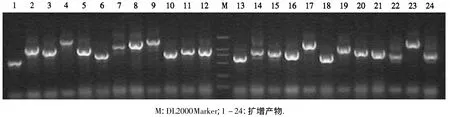
图3 菌液PCR验证Fig.3 Colony PCR validation
2.4 文库测序与分析
随机挑取的50个克隆进行5'端测序后得到50条高质量的EST序列,去除载体序列及引物序列后用NCBI中的BlastX 程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行分析,比对结果显示,有17 条序列与大豆相关基因具有较高的同源性,而与花生仅有1条相匹配,说明NCBI数据库中花生的基因信息还很不全面,相比大豆等豆科植物而言,花生功能基因组学方面的研究比较薄弱.
BlastX结果显示,有11条(22%)序列没有找到与之相匹配的序列,可能为花生新的EST序列.有39条序列(78%)与GenBank中已公布的序列有较高同源性,功能未知的序列有14条,功能已知的序列有25条(表1).其中,Leaf48 编码果糖-1,6-二磷酸酶(fructose-1,6-bisphosphatase,FBPase),该酶调控卡尔文循环,参与光合作用暗反应,虽然含量极其微弱,但是对光合速率极为重要.该酶在油茶[11]、甘蔗[12]等植物上已有报道.Leaf32编码磷酸腺苷酰基转移酶(GPAT),该酶在植物膜系统对温度响应过程中起重要作用.颜坤等研究结果证实转甜椒正义甘油-3-磷酸酰基转移酶基因(GPAT)可提高烟草在高温胁迫下PSⅡ稳定性,缓解PSⅡ光抑制[13].Leaf14编码UDP-糖基转移酶,该酶催化糖基转移反应,将糖基从活化的供体分子转移到受体分子上,从而调节受体分子在细胞和机体内的活性[14],由此可见,本研究所构建的花生叶片全长cDNA文库中可以获得与光合作用及多种代谢相关的重要基因,该文库的构建为今后克隆花生叶片发育过程中的重要基因奠定了基础.
将50条序列利用Blast2go软件[15]进行功能注释,结果显示在生物过程方面包含参与代谢过程、胁迫响应、细胞过程、单一生物过程以及生物调节相关基因;在分子功能方面,主要包含具有催化活性与结合活性的相关基因;在细胞组分方面,所获得的基因主要在细胞与细胞器上表达(图4).
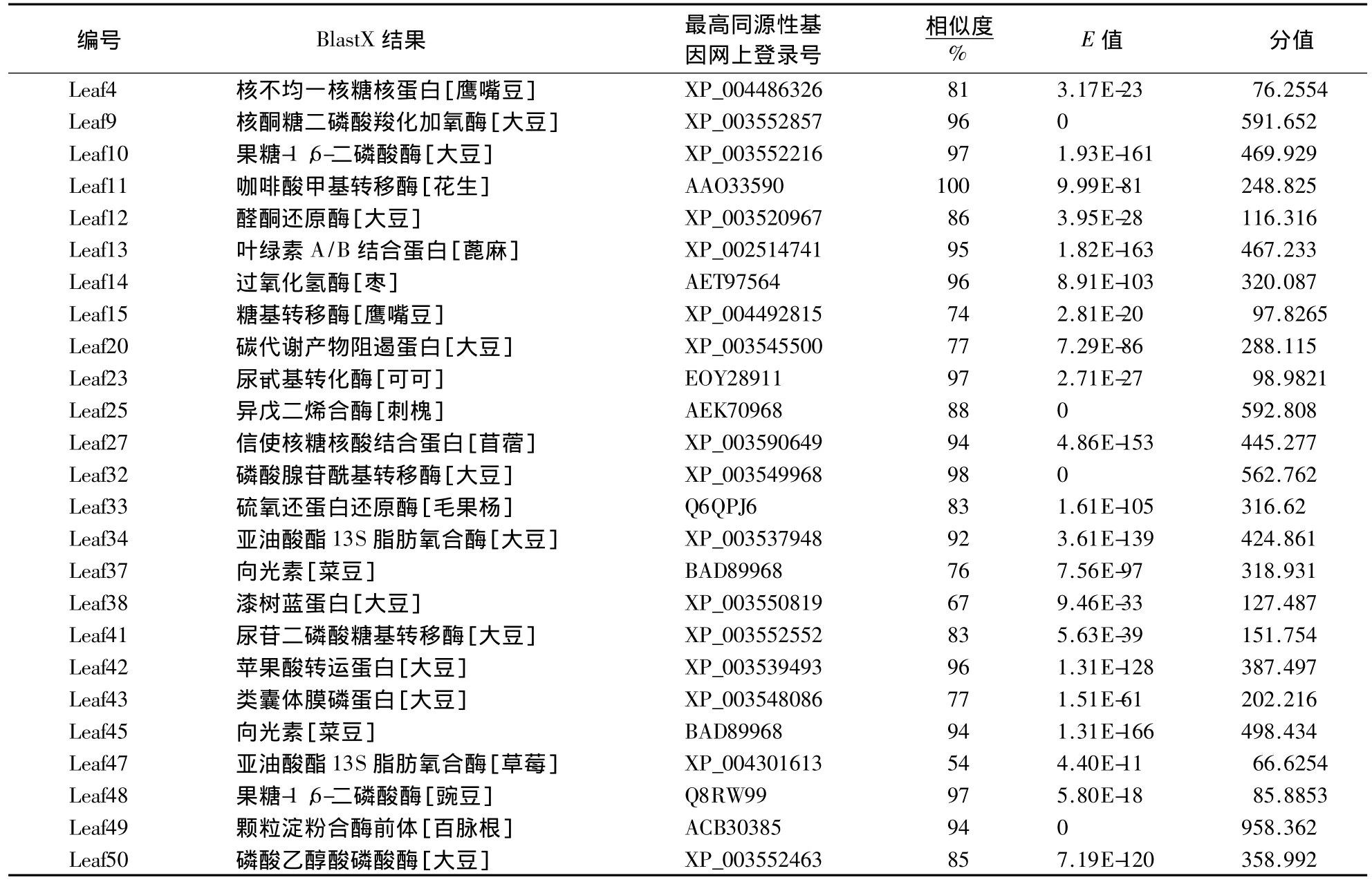
表1 部分功能已知ESTs比对结果Table 1 Partial Blastp results for comparison of homology with function genes in GenBank
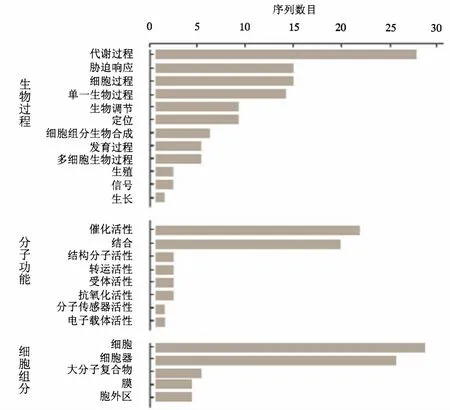
图4 注释分析Fig.4 GO distribution analysis
KEGG代谢途径分析表明,随机测序所获得的序列中主要包括光合作用碳固定、果糖和甘露糖代谢、糖酵解/糖异生以及磷酸戊糖途径等生物过程,其他代谢途径见表2.其中,Leaf8与毛果杨丙酮酸脱氢酶(XP_002324031.1)具有62%的相似性,该酶参与光合作用碳固定、丙酮酸代谢、柠檬酸循环、磷酸戊糖途径等多个重要的代谢反应.Leaf10、Leaf26和Leaf48均编码果糖-1,6-二磷酸酶,参与光合作用碳固定、果糖和甘露糖代谢、磷酸戊糖途径以及甲烷代谢等过程.Leaf12编码的蛋白与大豆醛酮还原酶(XP_003520967)具有95%的同源性,该酶主要参与果糖和甘露糖代谢、丙酮酸代谢、半乳糖代谢、甘油酯代谢等过程.由KEGG代谢途径可以看出,文库中包含了参与多种代谢途径的重要基因.
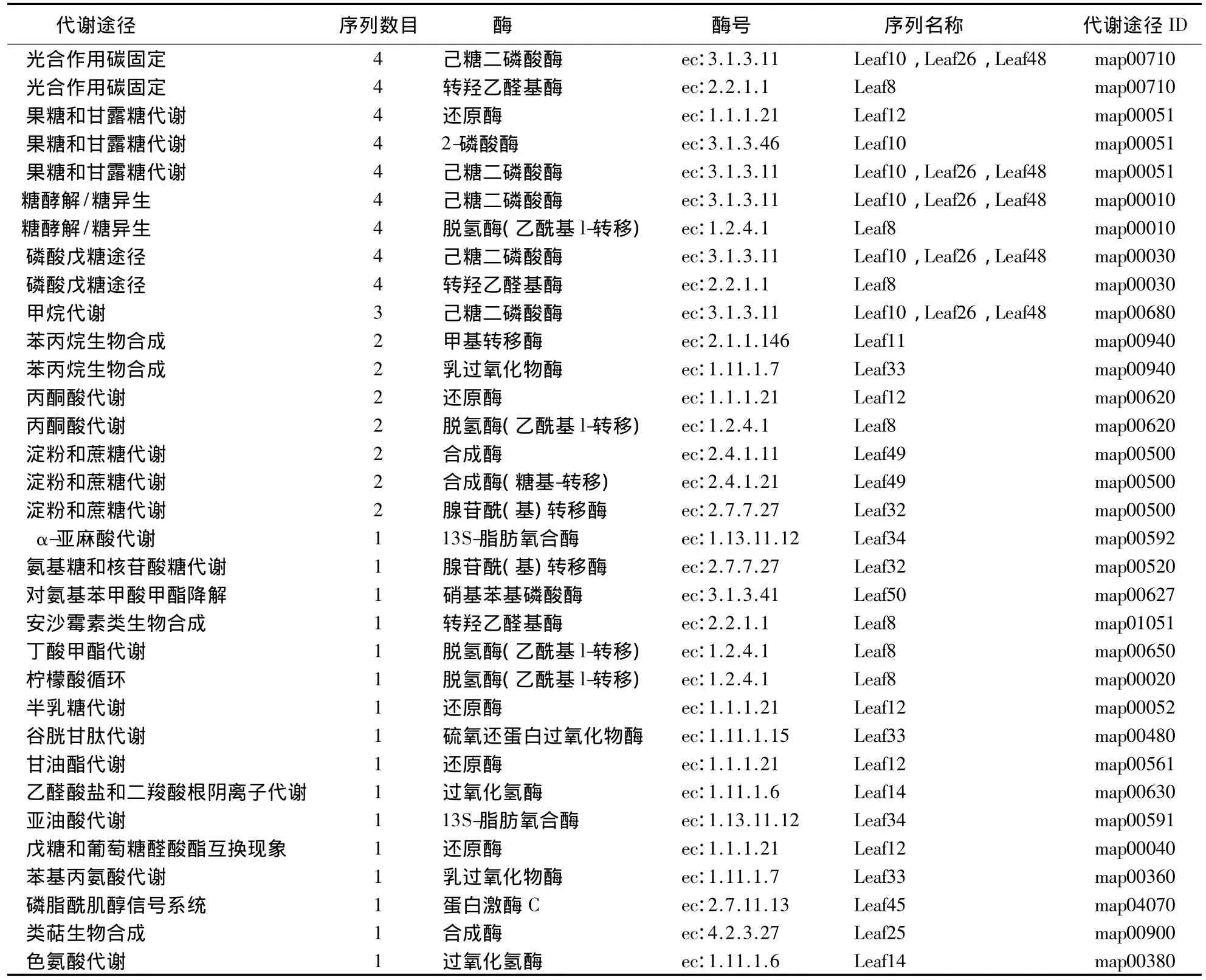
表2 KEGG代谢途径Table 2 KEGG pathway
3 讨论
目前,已有多个植物叶片 cDNA 文库的报道,如水稻[16]、小麦[17]、银杏[18]、人参[19,20]等.本研究利用SMART技术成功构建了花生叶片的全长cDNA文库.
衡量文库质量的重要指标包括文库的滴度、重组率、插入片段的大小.一般来说,文库的滴度达到105cfu·mL-1以上即为有效的文库[21],但为满足筛选到低丰度mRNA的要求,一般cDNA文库构建要求其滴度不少于1×106cfu·mL-1[22].本研究采用SMART技术成功构建了花生叶的全长cDNA文库,原始文库滴度达2.25×106cfu·mL-1,文库重组率接近100%.通过随机挑取50个单菌落 进行PCR扩增检测,cDNA插入片段长度范围为0.5-2 kb,平均长度为1.2 kb.综上说明本研究构建的花生叶片全长cDNA文库已经达到高质量cDNA文库的要求.
栽培花生为异源四倍体,基因组十分庞大,目前野生种和栽培种的基因组测序虽已完成,但序列拼接仍在进行中.截止2013年12月26日NCBI网站上已释放的花生EST序列仅247966条,而大豆已有1530042条,由此可见,花生相对于大豆等其他豆科植物而言,已知的基因信息还较少,花生功能基因组学方面的研究也相对比较薄弱.对文库随机测序的结果表明,本研究所构建的文库中含有大部分花生上尚未报道的新基因,有近50%的基因功能尚未被揭示,由此也可以进一步说明目前花生功能基因组学的研究还处于起始阶段,许多重要的功能基因还有待进一步挖掘.
本研究所构建的文库中含有多个与代谢和胁迫相关的基因,参与光合作用碳固定(Leaf10,Leaf26,Leaf48、Leaf8)、果糖和甘露糖代谢(Leaf12、Leaf10、Leaf10、Leaf26、Leaf48)、糖酵解/糖异生(Leaf10,Leaf26,Leaf48、Leaf8)、磷酸戊糖途径(Leaf10,Leaf26,Leaf48、Leaf8)以及淀粉和蔗糖代谢(Leaf49、Leaf49、Leaf32)等多种代谢途径,在植物的光合作用、抗氧化胁迫、抗各种生物与非生物胁迫等方面发挥着重要作用.本研究结果为进一步开展花生叶功能基因组学研究,深入挖掘花生重要功能基因并进行基因功能鉴定奠定了良好的基础.
[1]鄢洪海,赵志强,卢钰,等.花生叶部菌核病流行规律及生物防治初报[J].花生学报,2006,35(3):28-31.
[2]颜景波,吴占鹏,苏君伟,等.花生叶部病害防治技术研究[J].农业科技通讯,2003(3):123-125.
[3]董炜博.美国花生叶部病害的化学治理[J].花生科技,1998(3):10-13.
[4]毛新国,景蕊莲,孔秀英,等.几种全长cDNA文库构建方法比较[J].遗传,2006,28(7):865-873.
[5]陈华,姜宝杰,邓烨,等.高质量花生花序全长cDNA文库的构建和鉴定[J].豆科基因组学与遗传学,2011(2):14-19.
[6]陈华,姜宝杰,张冲,等.利用改进的SMART法构建花生种皮全长cDNA文库[J].福建农业学报,2012,27(11):1151-1155.
[7]陈华,姜宝杰,张冲,等.花生果皮全长cDNA文库的构建及初步分析[J].福建农林大学学报:自然科学版,2013,42(1):57-62.
[8]姜宝杰,陈华,邓烨,等.利用SMART法构建花生果针全长cDNA文库[J].基因组学与应用生物学,2011(30):1155-1161.
[9]姜宝杰,陈华,张冲,等.环境胁迫诱导的花生全长cDNA文库的构建及序列分析[J].核农学报2013,27(5):545-551.
[10]蔡宁波,黄湘文,庄伟建.花生种子全长cDNA文库的构建和鉴定[J].花生学报,2007,36(2):1-5.
[11]谭晓风,曹彦妮,郭静怡,等.油茶FBPase基因的全长cDNA克隆及序列分析[J].江西农业大学学报,2011,33(3):514-520.
[12]叶冰莹,黄金存,邵武华,等.甘蔗果糖-1,6-二磷酸酯酶的cDNA片段克隆及序列分析[J].中国糖料,2009(3):1-4.
[13]颜坤,陈娜,曲妍妍,等.高温胁迫下转甜椒正义甘油-3-磷酸酰基转移酶基因(GPAT)烟草缓解PSⅡ光抑制[J].中国农业科学,2007,40(11):2468 -2473.
[14]于惠敏,王艳文,任天莹,等.毛白杨糖基转移酶基因PtGT3的克隆与遗传转化.植物生理学报,2013,49(7):682-688.
[15]CONESA A,GOTZ S,GARCIA GOMEZ J M,et al.Blast2GO:a universal tool for annotation,visualization and analysis in functional genomic sresearch[J].Bioinformatics,2005,21(18):3674 -3676.
[16]王峰,王金发.水稻幼叶及幼穗cDNA文库的构建及初步分析[J].中山大学学报:自然科学版,2005,44(2):74-77.
[17]吴耀荣,赵双宜,夏光敏.小麦幼叶cDNA文库的构建[J].山东大学学报:理学版,2003,38(2):101-104.
[18]王义强,谭晓风,陈介南,等.银杏雌树成熟叶cDNA文库的构建[J].中南林业科技大学学报,2009,29(1):6-9,31.
[19]王昆,王颖,鲍永利,等.人参叶全长CDNA文库的构建及部分克隆的序列分析[J].分子科学学报,2006,22(1):58-62.
[20]YANG C J,WANG J,YANG C P,et al.Construction and analysis of a cDNA library from yellow-fruit ginseng(Panax ginseng C.A.Meyer.)leaf tissue[J].African Journal of Biotechnology,2009,22(8):6082 -6088.
[21]李太武,相建海,刘瑞春.中国对虾cDNA文库的构建[J].动物学报,1998,44(2):237-238.
[22]高小丽,李芳,岳鹏,等.欧李叶片全长cDNA文库的构建和部分克隆的序列分析[J].农业生物技术学报,2010,18(1):156-162.
