基于文献的毕赤酵母研究趋势分析*
2014-12-16张建国傅梦月
张建国,傅梦月
(上海理工大学医疗器械与食品学院,上海,200093)
毕赤酵母是一种能利用甲醇为碳源的甲基营养型微生物[1]。经过几十年的研究,它已经是药物蛋白、酶及积累代谢中间体的热门来源之一[2-5]。毕赤酵母高密度发酵后细胞干重达到130 g/L[6],适用于单细胞蛋白的生产。此外,它富含甲硫氨酸、色氨酸等高附加值营养物质。毕赤酵母以过氧化物酶体中3个酶(醇氧化酶、二羟丙酮合成酶、甲醛脱氢酶)代谢甲醇,毕赤酵母过氧化物酶体中含有2个醇氧化酶(AOX1、AOX2)。由于醇氧化酶与甲醇结合的亲和力弱,所以醇氧化酶在毕赤酵母中含量可达35%[7]。将外源基因导入醇氧化酶表达机制将大幅度提高外源蛋白质的生产效率。研究人员通过对毕赤酵母生化特性的研究,阐明了代谢甲醇的多个酶的基因和蛋白质结构,建立了较为全面的培养方法和基因操作方法。毕赤酵母的培养基和培养方法简单,没有Crabtree效应,具有高底物利用效率[8]。随着毕赤酵母的基因组测序完成,很多学者试图建立代谢网络,优化表达外源蛋白的代谢控制。截至目前已有超过600多种不同来源的外源基因在毕赤酵母中成功表达,多种外源蛋白作为工业用酶和蛋白药物已经商业化[9]。本文从文献的角度叙述了毕赤酵母的研究趋势,展示了我国毕赤酵母研究所处的位置,分析了研究动向。
1 研究数据来源
本文以Web of Knowledge数据库为数据来源,以pastoris为关键词搜索相关文献,再将文献下载到Endnote文献管理软件后进行分析,手工剔除数据库中Capsella bursa pastoris等文献及数据库中重复收录的文献。检索的时间截至2014年3月7日,共得到相关文献3 270篇。其中没有收录最早发现毕赤酵母可以利用甲醇为唯一碳源的文献[1]。
国内文献研究以维普中文数据库为数据来源。以“毕赤酵母”为关键词搜索后进行手工剔除,共得到近10年文献1 437篇。
2 文献检索结果与分析
2.1 研究毕赤酵母的文献随时间的变化
Web of Knowledge数据库中最早收录的毕赤酵母文献是1975年Hazeu报道对毕赤酵母代谢甲醇细胞器的观察。其他相关文献在1985年Cregg将毕赤酵母用于外源基因的表达后数量逐步增加,尤其是1994年以后文献数目呈直线上升趋势(图1)。2013年发表的文献达到293篇,说明近年来有关毕赤酵母的研究正在扩张。
2.2 文献种类的组成
从文献类型的组成来看(图2),数量最多的文献是研究论文,占79.20%。说明关于毕赤酵母技术和应用的研究仍占多数。然后依次是专利(11.99%)、会议论文(2.69%)、综述论文(1.56%)、书籍的章节(0.12%),其他文献占4.43%。除研究论文外,专利是数量最多的文献,说明研究人员认为有关毕赤酵母的研究内容具有很大应用潜力。
2.3 文献量前10名的国家及其每篇文献平均引用数

图1 关于毕赤酵母的研究论文数随时间的变化Fig.1 The change of the number of references on Pichia pastoris(P.pastoris)with time
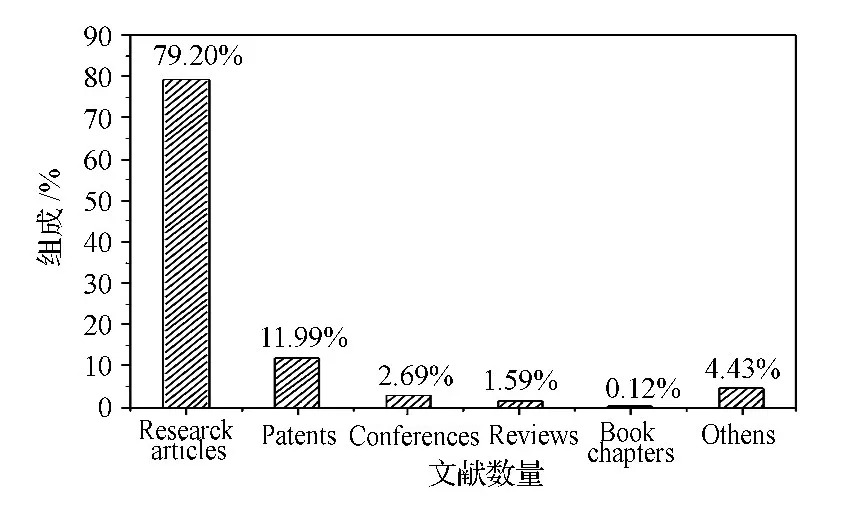
图2 毕赤酵母文献类型的组成Fig.2 The composition of reference types of P.pastoris

图3 毕赤酵母文献来源的前十个国家Fig.3 The countries which are the top 10 references sources of P.pastoris
毕赤酵母研究的文献共来自71个国家和地区。发表文献总数前10的国家见图3。其文献总数为1 899篇,占58.07%。排名前10的国家依次是中国、美国、德国、日本、西班牙、奥地利、韩国、法国、英国、印度,其中中国发表文献634篇,占总文献量的19.39%。图4是来自前10名国家的每篇文献平均引用数。美国的每篇文献平均引用数最多,为21.88。后面是法国(20.13)、英国(18.35)、德国(18.05)。中国的每篇文献平均引用数最少,为7.12。这说明来自中国的有关毕赤酵母的文献量最多,受到的关注度却并不高。
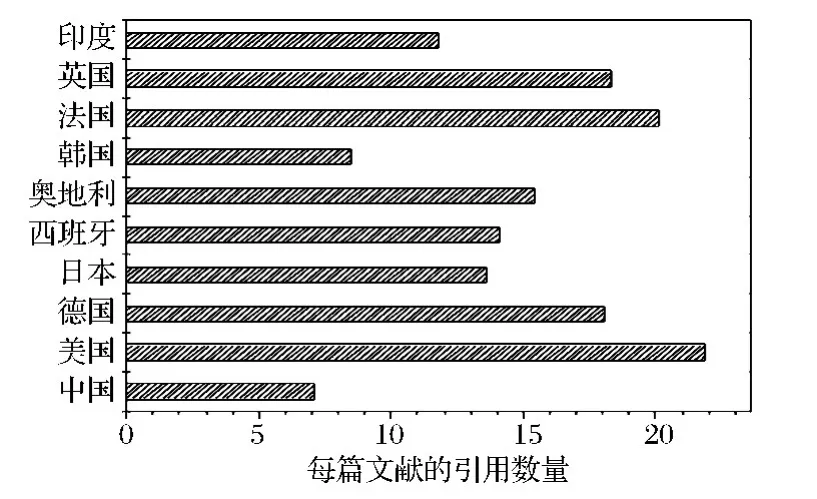
图4 文献总数前十的国家的每篇文献的引用数Fig.4 The number of references from different countries which have the top 10 total number of references
2.4 总文献数前五的国家在近十年内的文献数量
对发表文献总数前5名国家的文献时间进行分析,可以看出中国的文献增长趋势较为明显(图5)。中国文献数从2004一直增长到2009年,经2010年文献数有所降低后持续增长。2012年来自中国的文献为93篇,是美国发表文献最多年份文献数量(2011年35篇)的2.66倍。来自美国的文献数目出现高低的波动,总体上呈2个钟形曲线。其文献数在2004年至2006年期间增加,之后下降到2009年的22篇,2010年和2012年出现短暂增加后又出现下降的趋势。来自德国、日本、西班牙的文献在各个年份基本持平,这说明这些国家有关毕赤酵母的研究处于较稳定状态。自2006年后,每年来自中国的文献数量远高于其他国家的文献量,说明我国是研究毕赤酵母的活跃地区。
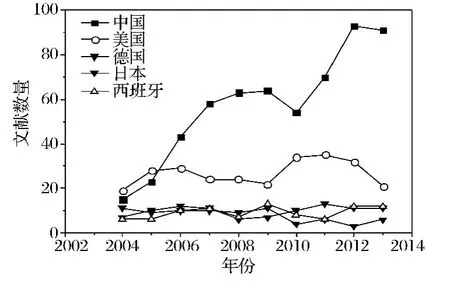
图5 发表文献数前5的国家发表文献数随时间的变化Fig.5 The change of references number with time in countries which have the top 5 references numbers
2.5 基于维普的国内文献数量变化
近十年来,国内研究毕赤酵母的文献数自2004年以来逐渐增加,直到2006年文献数量达到150篇后有较大波动,但总体文献数处于持平状态(图6)。每年在国内发表的文献数量高于在国际刊物上发表的文献数量,说明我国研究人员更偏向于发表国内期刊。
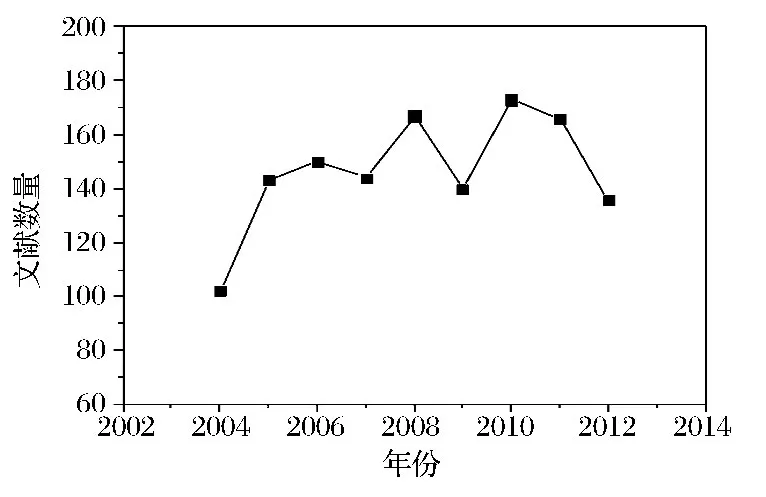
图6 国内发表文献数随时间的变化Fig.6 The change of domestic references number with time
3 研究展望
3.1 基因表达技术
为了简化毕赤酵母重组蛋白的纯化步骤,毕赤酵母实现了分泌表达多种外源蛋白。但是分泌蛋白表达量相比于胞内蛋白表达量较低。这是由于外源基因的密码子在毕赤酵母中的转录、翻译效率低。增加密码子偏好性可以得到适合毕赤酵母的基因序列,这是一个有效提高外源蛋白表达量的方法[10]。提高目的基因的拷贝量也是一种选择。虽然外源蛋白的表达量与其基因拷贝量不是线性正相关[11],但是提高拷贝数可明显提高外源蛋白的表达量。另外,提高外源基因在毕赤酵母中表达量的方法还有开发效率高的质粒[12],提高重组毕赤酵母的二羟丙酮合成酶、己糖转移酶、甲醛脱氢酶活力,提高毕赤酵母比生长速率、比底物利用速率、比产物生产速率[13]等。其他种类蛋白作为分子伴侣也对提高外源蛋白的表达量有着促进作用[14]。毕赤酵母有成熟的基因敲除技术开发不同表型的宿主[15]和代谢控制研究[16]。
毕赤酵母表达系统是目前最成功的接近人源蛋白糖基化的表达系统[17](图7)。所以科学家们力图从发现糖基化转移酶,糖基化酶基因敲除,导入糖水解酶,蛋白质基因序列突变等方式使毕赤酵母能够表达具有人源糖基化的蛋白质。图7中毕赤酵母表达蛋白质的糖基化结构中甘露糖的数目为8~14,其中黑色框内是具有8个甘露糖和2个 N-乙酰葡萄糖胺(Man8NacGlu2)的基本结构。由于人源蛋白质糖基化过程是先在内质网中形成Man8NacGlu2,然后糖结构转移到高尔基体,在 α-甘露糖酶的催化下形成Man5NacGlu2,进而在N-乙酰氨基葡萄糖转移酶I的催化下形成GlcNAcMan5GlcNAc,最后经甘露糖苷酶Ⅱ和N-乙酰氨基葡萄糖转移酶Ⅱ形成GlcNAc2Man3Glc-NAc2[18]。Hamilton等构建唾液酸相关基因和II型膜蛋白定位序列,得到 NANA2Gal2GlcNAc2Man3GlcNAc2。这个糖链结构虽然与人源蛋白的糖基化还有差异,但是与哺乳动物细胞表达的蛋白质具有一样的糖基化结构[19]。目前已有研究成功将毕赤酵母糖基化途径改造后表达适于人的重组蛋白[20]。毕赤酵母来源的蛋白质O-糖基化组成主要是甘露糖,而人源蛋白的O-糖基化的糖组分可能是N-乙酰葡萄糖胺、半乳糖、唾液酸等多种组分。目前关于O-糖基化的研究远没有N-糖基化充分。但是最近Nett阐明了毕赤酵母O-糖基化酶基因,为减少毕赤酵母O-糖基化的甘露糖变为 N-乙酰葡萄糖胺、半乳糖、唾液酸提供了基础[21]。

图7 人源蛋白的N糖基化和毕赤酵母来源蛋白N糖基化结构示意图Fig.7 The sugar chain structures of gly-protein from human and P.pastoris
毕赤酵母表达外源蛋白的严格调控机制是由于AOX的上游序列。AOX1在毕赤酵母中的量占总AOX的95%,阐明PAOX1启动子的控制序列得到不受葡萄糖、乙醇抑制的AOX1上游序列将克服毕赤酵母甲醇代谢的能量限制,大幅度提高外源蛋白表达量,AOX1上游调控序列的研究是一个热点。Inan博士将AOX1上游序列分为5个片段(A到E)。其中片段B和E对AOX1的表达有促进作用,片段C、D具有抑制作用[22]。毕赤酵母中有与片段C、D结合的抑制因子。但是 Hartner报道片段D也有促进作用[23],这仍需进一步证实。Lin-Cereghino报道了一种结合在AOX1上游-415~-172bp之间(属于片段D),对AOX1有促进作用的蛋白因子(Methanol Expression Regulator 1,Mxr1)[24]。后来 Kranthi利用凝胶迁移实验(Electrophoretic Mobility Shift Assay,EMSA)和足迹法(DNaseI footprinting)证明Mxr1有6个结合位点[25]。基于以上初步研究,Vog等提出了AOX的核心调控序列[26]。但是Staley利用计算机辅助分析发现AOX1的控制序列具有更加复杂的调控机制,且天然的 AOX1上游序列已经是较优的排列[27]。除了 AOX1,AOX2[28]、GAP(Glyceraldehyde-3-phosphate Dehydrogenase Promotor)[29]、二羟丙酮(Dihydroxyacetone Synthase,DHAS)[30]的上游序列也吸引了学者的关注。
3.2 发酵策略
毕赤酵母高密度发酵表达外源蛋白条件的控制已被众多学者总结过[31]。甲醇利用慢型的毕赤酵母在表达外源蛋白方面优于甲醇利用快型的毕赤酵母。已有学者详细研究了毕赤酵母表达外源蛋白的基因、分泌、葡萄糖代谢的网络[32],发现甲醇利用慢型毕赤酵母的瓶颈是不能提供充足的能量供细胞表达大量外源蛋白[33],甚至造成细胞的死亡率较高[34]。调节能量来降低细胞死亡率已成为目前研究毕赤酵母高密度发酵的热点[35]。所以能量代谢是影响毕赤酵母表达外源蛋白的一个重要因素。添加葡萄糖、甘油,牺牲AOX表达活力,补充细胞能量代谢、碳代谢是迫不得已的选择[36]。山梨醇被发现是一种不抑制AOX表达活力的碳源[37]。虽然毕赤酵母利用山梨醇的速率达不到利用甘油的水平。但是添加山梨醇提高了磷酸戊糖途径和三羧酸循环的代谢流量[38],为毕赤酵母提供了更多的能量,提高了多种外源蛋白的表达量[39]。山梨醇和甲醇混合流加可以降低细胞的死亡率[40]。在连续高密度培养过程中,筛选高活性的重组菌株也是一种重要方法[41]。
3.3 过氧化物酶体吞噬
过氧化物酶体吞噬是细胞在某种生理条件下将自身的过氧化物酶体有选择性的吞噬、降解的过程[42]。由于改变碳源就能发生2种不同类型的过氧化物酶体吞噬(微吞噬和宏吞噬)[43],营养单倍体在营养限制时容易发生接合,容易获得过氧化物酶体吞噬突变体,所以很适合作为研究过氧化物酶体吞噬的模式生物[44]。近年来,毕赤酵母过氧化物酶体吞噬的基因和蛋白质的研究取得了快速的发展。已有研究表明有30多个吞噬相关基因(autophagy releated gene,atg)和蛋白质(Atg)共同调控 2 种吞噬[45]。Tuttle和Sakai研究发现毕赤酵母中微吞噬和宏吞噬中有共用的基因[43]。例如,在2种过氧化物酶体的吞噬和液泡接触的初始阶段中,Atg30、Atg11、Atg7都参与微吞噬中液泡伸出臂与过氧化物酶体结合。Atg11、Atg7与Atg30结合也带动宏吞噬中其他蛋白质一同参与吞噬体的形成[46]。微吞噬的液泡膜伸出臂延长和宏吞噬吞噬体形成阶段,Atg8(类泛素蛋白)、Atg9和Atg26参与微吞噬的MIPA和宏吞噬吞噬体形成[47]。Atg24参与2种吞噬中过氧化物酶体在液泡中的降解过程[48]。毕赤酵母过氧化物酶体吞噬中还有多种吞噬相关蛋白起作用,吞噬机理尚未阐述清楚,例如MIPA组分的来源,启动吞噬的机理有待进一步研究证实。
4 结语
有关毕赤酵母的研究逐年增多,说明越来越多的研究人员认识到毕赤酵母在外源蛋白表达、高附加值产物的生产方面的优势。其中我国关于毕赤酵母的文献数量多说明我国这方面的投入大,但是每篇文献的引用数较低也反映出研究水平不高的问题。美国等其他国家文献数量变化不大,但是新研究方向都是他们引领。比较我国人员在国内外发表的文献数说明我国研究人员应将更多的研究成果展示到外国,从而引起国际上科学家或者学者的关注,增加影响力。近年来关于毕赤酵母新的研究方向已经出现,我们不仅应该把握新的研究进展,还应当开拓新的研究方向。
[1] Ogata K,Nishikawa H,Ohsugi M.A yeast capable of utilizing methanol[J].Agricultural and Biological Chemistry,1969,33(10):1 519-1 520.
[2] De Schutter K,Lin YC,Tiels P,et al.Genome sequence of the recombinant protein production host Pichia pastoris[J].Nature Biotechnology,2009,27(6):561-566.
[3] Dalziel M,Crispin M,Scanlan C N,et al.Emerging principles for the therapeutic exploitation of glycosylation [J].Science,2014,343(6166):1235681.
[4] Vogl T,Hartner F S,Glieder A.New opportunities by synthetic biology for biopharmaceutical production in Pichia pastoris[J].Curr Opin Biotechnol,2013,24(6):1 094-1 101.
[5] Gasser B,Prielhofer R,Marx H,et al.Pichia pastoris:protein production host and model organism for biomedical research[J].Future Microbiology,2013,8(2):191-208.
[6] Cereghino J L,Cregg J M.Heterologous protein expression in the methylotrophic yeast Pichia pastoris[J].FEMS Microbiology Reviews,2000,24(1):45-66.
[7] Couderc R,Baratti J.Oxidation of methanol by the yeast,Pichia pastoris:Purification and properties of the alcohol oxidase[J].Agricultural and Biological Chemistry,1980,44(10):2 279-2 289.
[8] Baumann K,Carnicer M,Dragosits M,et al.A multi-level study of recombinant Pichia pastoris in different oxygen conditions[J].BMC Systems Biology,2010,4(1):141.
[9] Martínez J L,Liu L,Petranovic D,et al.Pharmaceutical protein production by yeast:towards production of human blood proteins by microbial fermentation[J].Current O-pinion in Biotechnology,2012,23(6):965-971.
[10] TU Y,WANG Y,WANG G,et al.High-level expression and immunogenicity of a porcine circovirus type 2 capsid protein through codon optimization in Pichia pastoris[J].Applied Microbiology and Biotechnology,2013,97(7):2 867-2 875.
[11] Aw R,Polizzi K M.Can too many copies spoil the broth?[J].Microbial Cell Factories,2013,12(1):128.
[12] Sasagawa T,Matsui M,Kobayashi Y,et al.Highthroughput recombinant gene expression systems in Pichia pastoris using newly developed plasmid vectors[J].Plasmid,2011,65(1):65-69.
[13] Krainer F W,Dietzsch C,Hajek T,et al.Recombinant protein expression in Pichia pastoris strains with an engineered methanol utilization pathway[J].Microb Cell Fact,2012,11(1):22-35.
[14] Samuel P,Prasanna Vadhana A K,Kamatchi R,et al.Effect of molecular chaperones on the expression of Candida antarctica lipase B in Pichia pastoris[J].Microbiological research,2013,168(10):615-620.
[15] Nett J H,Hodel N,Rausch S,et al.Cloning and disruption of the Pichia pastoris ARG1,ARG2,ARG3,HIS1,HIS2,HIS5,HIS6 genes and their use as auxotrophic markers[J].Yeast,2005,22(4):295-304.
[16] P Unrean.Pathway analysis of Pichia pastoris to elucidate methanol metabolism and its regulation for production of recombinant proteins[J].Biotechnology Progress,2014,30(1):28-37.
[17] Bretthauer R K,Castellino F J.Glycosylation of Pichia pastoris-derived proteins[J].Biotechnology and Applied Biochemistry,1999,30(3):193-200.
[18] Hamilton S R,Bobrowicz P,Bobrowicz B,et al.Production of complex human glycoproteins in yeast[J].Science,2003,301(5637):1 244-1 246.
[19] Hamilton S R,Davidson R C,Sethuraman N,et al.Humanization of yeast to produce complex terminally sialylated glycoproteins[J].Science,2006,313(5792):1 441-1 443.
[20] Jacobs P P,Geysens S,Vervecken W,et al.Engineering complex-type N-glycosylation in Pichia pastoris using GlycoSwitch technology[J].Nature protocols,2008,4(1):58-70.
[21] Nett J H,Cook W J,Chen M T,et al.Characterization of the Pichia pastoris protein-O-mannosyltransferase gene family[J].PloS one,2013,8(7):e68325.
[22] Inan M.Studies on the alcohol oxidase(AOX1)promoter of Pichia pastoris[D].Nebraska:The University of Nebraska-Lincdn,2000.
[23] Hartner F S,Ruth C,Langenegger D,et al.Promoter library designed for fine-tuned gene expression in Pichia pastoris[J].Nucleic Acids Research,2008,36(12):e76-e76.
[24] Lin-Cereghino G P,Godfrey L,Bernard J,et al.Mxr1p,a key regulator of the methanol utilization pathway and peroxisomal genes in Pichia pastoris[J].Molecular and Cellular Biology,2006,26(3):883-897.
[25] Kranthi B V,Kumar R,Kumar N V,et al.Identification of key DNA elements involved in promoter recognition by Mxr1p,a master regulator of methanol utilization pathway in Pichia pastoris[J].Biochimica et Biophysica Acta(BBA)-Gene Regulatory Mechanisms,2009,1 789(6):460-468.
[26] Vogl T,Ruth C,Pitzer J,et al.Synthetic core promoters for Pichia pastoris[J].ACS Synthetic Biology,2014,3(3):188-191.
[27] Staley C A,Huang A,Nattestad M,et al.Analysis of the 5'untranslated region(5'UTR)of the alcohol oxidase 1(AOX1)gene in recombinant protein expression in Pichia pastoris[J].Gene,2012,496(2):118-127.
[28] Ohi H,Miura M,Hiramatsu R,et al.The positive and negative cis-acting elements for methanol regulation in the Pichia pastoris AOX2 gene[J].Molecular and General Genetics MGG,1994,243(5):489-499.
[29] QIN X,QIAN J,YAO G,et al.GAP promoter library for fine-tuning of gene expression in Pichia pastoris[J].Applied and environmental microbiology,2011,77(11):3 600-3 608.
[30] Vogl T,Glieder A.Regulation of Pichia pastoris promoters and its consequences for protein production[J].New Biotechnology,2013,30(4):385-404.
[31] Potvin G,Ahmad A,Zhang Z.Bioprocess engineering aspects of heterologous protein production in Pichia pastoris:A review [J].Biochemical Engineering Journal,2012,64:91-105.
[32] Mattanovich D,Graf A,Stadlmann J,et al.Genome,secretome and glucose transport highlight unique features of the protein production host Pichia pastoris[J].Microbial Cell Factories,2009,8(1):29.
[33] Heyland J,Fu J,Blank L M,et al.Carbon metabolism limits recombinant protein production in Pichia pastoris[J].Biotechnology and Bioengineering,2011,108(8):1 942-1 953.
[34] Jahic M,Wallberg F,Bollok M,et al.Temperature limited fed-batch technique for control of proteolysis in Pichia pastoris bioreactor cultures[J].Microbial Cell Factories,2003,2(1):6.
[35] Lo T M,Teo W S,Ling H,et al.Microbial engineering strategies to improve cell viability for biochemical produc-tion[J].Biotechnology Advances,2013,31(6):903-914.
[36] Jordà J,Jouhten P,Cámara E,et al.Metabolic flux profiling of recombinant protein secreting Pichia pastoris growing on glucose:methanol mixtures[J].Microb Cell Fact,2012,11:57.
[37] Inan M,Meagher M M.Non-repressing carbon sources for alcohol oxidase(AOX1)promoter of Pichia pastoris[J].Journal of Bioscience and Bioengineering,2001,92(6):585-589.
[38] Çelik E,ÇalIk P,Oliver S G.Metabolic flux analysis for recombinant protein production by Pichia pastoris using dual carbon sources:Effects of methanol feeding rate[J].Biotechnology and Bioengineering,2010,105(2):317-329.
[39] ÇalIk P,Bozkurt B,Zerze G H,et al.Effect of co-substrate sorbitol different feeding strategies on human growth hormone production by recombinant Pichia pastoris[J].Journal of Chemical Technology and Biotechnology,2013,88(9):1 631-1 640.
[40] WANG Z,WANG Y,ZHANG D,et al.Enhancement of cell viability and alkaline polygalacturonate lyase production by sorbitol co-feeding with methanol in Pichia pastoris fermentation [J].Bioresource Technology,2010,101(4):1 318-1 323.
[41] Panagiotou V,Love K R,Jiang B,et al.Generation and screening of Pichia pastoris Strains with enhanced protein production by use of microengraving[J].Applied and Nvironmental Microbiology,2011,77(9):3 154-3 156.
[42] MAO K,LIU X,FENG Y,et al.The progression of peroxisomal degradation through autophagy requires peroxisomal division[J].Autophagy,2014,10(4):49-50.
[43] Tuttle D L,Dunn W A.Divergent modes of autophagy in the methylotrophic yeast Pichia pastoris[J].Journal of Cell Science,1995,108(1):25-35.
[44] Gould S J,McCollum D,Spong A P,et al.Development of the yeast Pichia pastoris as a model organism for a genetic and molecular analysis of peroxisome assembly[J].Yeast,1992,8(8):613-628.
[45] Till A,Lakhani R,Burnett S F,et al.Pexophagy:the selective degradation of peroxisomes[J].International Journal of Cell Biology,2012,2012:512721.
[46] Farre J C,Manjithaya R,Mathewson R D,et al.PpAtg30 tags peroxisomes for turnover by selective autophagy[J].Developmental cell,2008,14(3):365-376.
[47] Chang T,Schroder L A,Thomson J M,et al.PpATG9 encodes a novel membrane protein that traffics to vacuolar membranes,which sequester peroxisomes during pexophagy in Pichia pastoris[J].Molecular Biology of the Cell,2005,16(10):4 941-4 953.
[48] Ano Y,Hattori T,Oku M,et al.A sorting nexin PpAtg24 regulates vacuolar membrane dynamics during pexophagy via binding to phosphatidylinositol-3-phosphate[J].Molecular Biology of the Cell,2005,16(2):446-457.
