一株分离微拟球藻LAMB0002的鉴定*
2014-12-02朱葆华杨官品潘克厚
梁 鑫,朱葆华,杨官品,潘克厚**
(中国海洋大学1.海水养殖教育部重点实验室,2.海洋生命学院,山东 青岛 266003)
微拟球藻属包括Nannochloropsisoculata(Doop)D.J.Hibberd(Hibberd 1981),NannochloropsissalinaD.J.Hibberd (Hibberd 1981),Nannochloropsis gaditanaLubián (Lubián 1982),Nannochloropsis granulataKarlson & Potter[1],Nannochloropsislimnetica[2]和Nannochloropsisoceanica[3]6个 种,都是单细胞藻,属于真眼点藻纲[4]。这些藻含有多种色素,如叶绿素a、玉米黄素、角黄素等[5]和丰富的长链多不饱和脂肪酸,尤其是二十碳五烯酸(C20∶5,eicosapentenoic acid,EPA),一种人体需要的重要多不饱和脂肪酸[6]。另外,微拟球藻还是水产养殖广泛使用的饵料[7]和生物柴油候选生产藻[8]。微拟球藻是小于5μm的球形细胞[9],形态非常简单,难以用形态学方法进行描述和准确鉴定。因此,微拟球藻的种间鉴定主要依据指示基因序列[1-3,10],但至今尚未有广泛认同的基因序列作为种鉴定的分子条码,选择适用基因或者基因组合进行鉴定目前进行了较多的探索[11-12]。本研究基于形态观察、色素和脂肪酸组成分析、18SrRNA基因和rbcL基因分子系统学分析对一株分离的微拟球藻进行了鉴定,希望建立一种适用单细胞微拟球藻的分类体系,使微拟球藻的分类鉴定更快速、准确。
1 材料及方法
实验材料是一株分离的微拟球藻藻株(Nannochloropsissp.)LAMB0002。
N.sp.LAMB0002经扩大培养后,利用平板涂布法2次分离单细胞,在锥形瓶中扩大培养。全过程在无菌条件下操作。培养条件为:温度(20±1)℃;光照60~80μmol·m-2·s-1,光周期为12h∶12h。光源为白炽灯,每天定时摇瓶3次。
1.1 形态学观察
在光学显微镜 (Nikon 50i)下观察N.sp.LAMB0002。将培养至指数中期的N.sp.LAMB0002离心收集,加入用磷酸缓冲液配制的5%戊二醛,4℃固定过夜。然后参考钟秀容等的方法[13],制作切片。用日立透射电镜(Hitachi H-7000Elctron Microscope)观察。
1.2 生理生化分析
色素分析采用Zapata等的方法[14]。将15mL培养至指数中期的N.sp.LAMB0002在弱真空(<0.03 MPa)微光的条件下过滤到47mm Whatman GF/F玻璃纤维滤膜上,再用甲醇提取色素。使用的色谱设备为Waters Alliance HPLC系统,并配有Waters2996二极管阵列检测器(1.2nm光学分辨率)和 Waters2475多波长荧光检测器。使用的液相色谱固定相为C8色谱柱,流动相:A=甲醇∶乙睛∶吡啶水溶液(50∶25∶25 v/v/v),B=甲醇∶乙睛∶丙酮(20∶60∶20v/v/v)。
按照Lepage和Roy的方法提取脂肪酸[15]。取进入指数生长末期N.sp.LAMB0002,离心收集,冷冻干燥。将十九烷酸(Nonadecanoic acid,SIGMA)作为内标进行相对定量分析。脂肪酸组成分析采用美国Agilent 6890series GC system气相色谱仪、氢火焰离子化检测器和DB-225毛细管柱(30m×320μm×0.15 μm,50%-cyanopropylphenyl-dimethylpolysiloxane)。标准品为Accustandard FAMQ-005。根据内标的量计算各种脂肪酸所占藻粉干重的比值。再用SPSS软件做各藻株之间脂肪酸的差异性分析。
1.3 分子系统学分析
离心收集对数生长期的藻细胞,用去离子水冲洗3次,去掉细胞表面的盐类等物质。用CTAB法提取微拟球藻DNA[16],-20℃贮存备用。通过设计(见表1)的18SrRNA基因和rbcL基因引物对这2个目的片段进行PCR扩增。回收扩增产物,送至上海博尚公司测序。将测序得到的序列在GenBank进行Blast N比对,并下载相关的序列。用 MEGA4.1采用邻接法(Neighbor-Joining Method,NJ)和最大简约法(Maximum porosity,MP)经Consensus程序计算多数一致树。构建系统树,用自举检验(Bootstrap test)估计系统树分支节点的置信度,重复次数1 000。
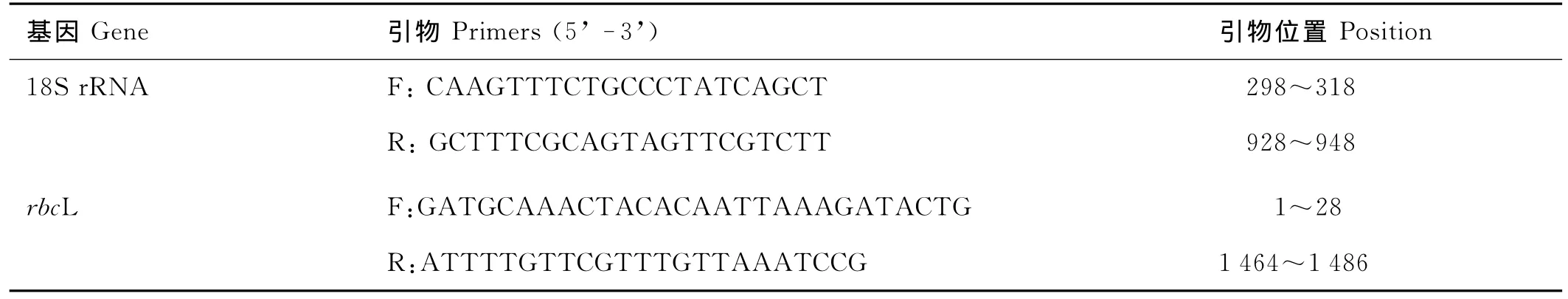
表1 本研究所用引物Table 1 Primers used in this study
2 结果
2.1 形态学观察
在显微镜下观察,发现N.sp.LAMB0002细胞(见图1)单个呈球形、梨形或卵圆形,2~5μm,细胞壁光滑,胞内有一个杯状或侧生块状的黄绿色素体。细胞内常有不动的、颗粒状的内含物。眼点红色,没有蛋白核。这些特征与微拟球藻特征相符[7],将其与本实验室的4株微拟球藻进行比较。结果见图1。
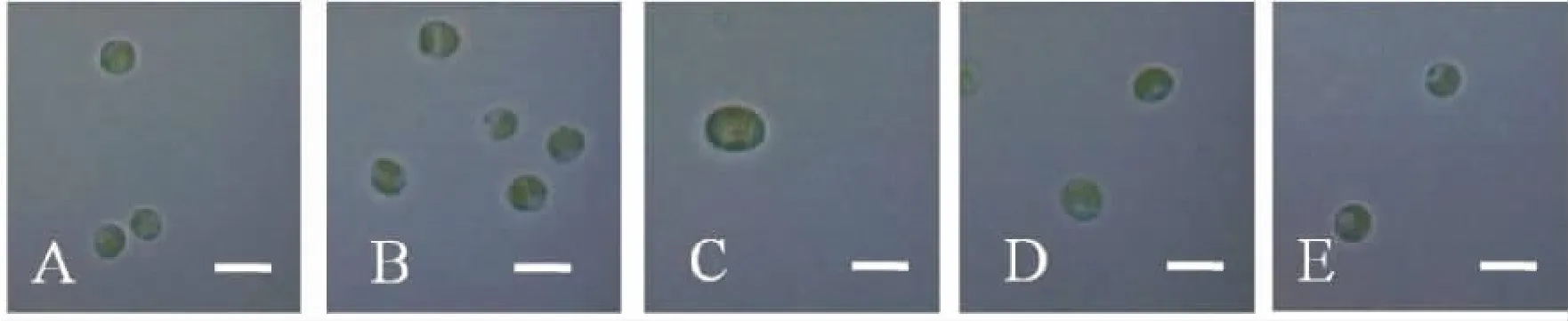
图1 不同微拟球藻藻株的形态Fig.1 Morphology of different Nannochloropsis strains
几株微拟球藻都是不游动的单细胞藻,外形没有明显区别,内含物颗粒状,且都有一个黄绿色色素体。N.oceanic细胞呈球形或卵圆形,色素体杯状或块状,常可见眼点(见图1B);N.salina细胞呈长柱形,相比其他藻株较大,常可见眼点(见图1C);N.oculata细胞呈球形或卵圆形,不常观察到眼点(见图1D、E);N.oculataCS-189(见图1D)和N.oculataCS-192(见图1E)在光学显微镜下差别不明显。光镜下,N.sp.LAMB0002与其他几株微拟球藻十分相似,难以将其鉴定到种。
用透射电镜观察N.sp.LAMB0002,N.salinaCS-190、N.oculataCS-189和N.oculataCS-192。结果如图2所示。
几株微拟球藻都呈现细胞核、叶绿体、线粒体和液泡等结构,叶绿体片层结构十分清晰。N.sp.LAMB0002细胞卵圆形,胞内有2个较大的线粒体和1个明显的液泡(见图2A)。细胞核外膜和叶绿体外膜形成一个连续体(Nucleus-plastid continuum,NPC)[17],核内膜与叶绿体内膜直接接触,将核与叶绿体完全分隔开来形成封闭的分隔[18]。NPC结构出现在整个细胞周期中,跟细胞有丝分裂有关。然而,在NPC中细胞核与叶绿体分裂是如何调适的却是完全未知的[19]。N.oculataCS-189细胞中间有一个较大的脂肪滴(见图2B)。N.oculataCS-192细胞壁上有一个乳突结构(见图2C),这个结构曾出现在N.oculata、N.gaditana和N.salina中,被描述为栓状结构[20],但该结构功能未知[3]。N.salinaCS-190,细胞形状为长卵形(见图2D),与其他几株微拟球藻相比体积较大,这与光镜观察结果相符。几株微拟球藻的超微结构没有明显区别,电镜观察难以将微拟球藻各个种相互区别。
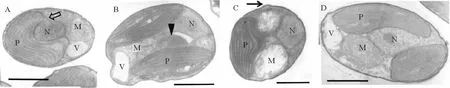
图2 各株微拟球藻超微结构Fig.2 Ultrastructure of different Nannochloropsis strains
2.2 生理生化分析
N.sp.LAMB0002的色素洗脱峰如图3所示。N.sp.LAMB0002的主要色素为紫黄素、无隔藻黄素和叶绿素a,百合黄素、玉米黄素、角黄素和一些未知胡萝卜素含量较少,除此之外还有痕量的β-胡萝卜素。
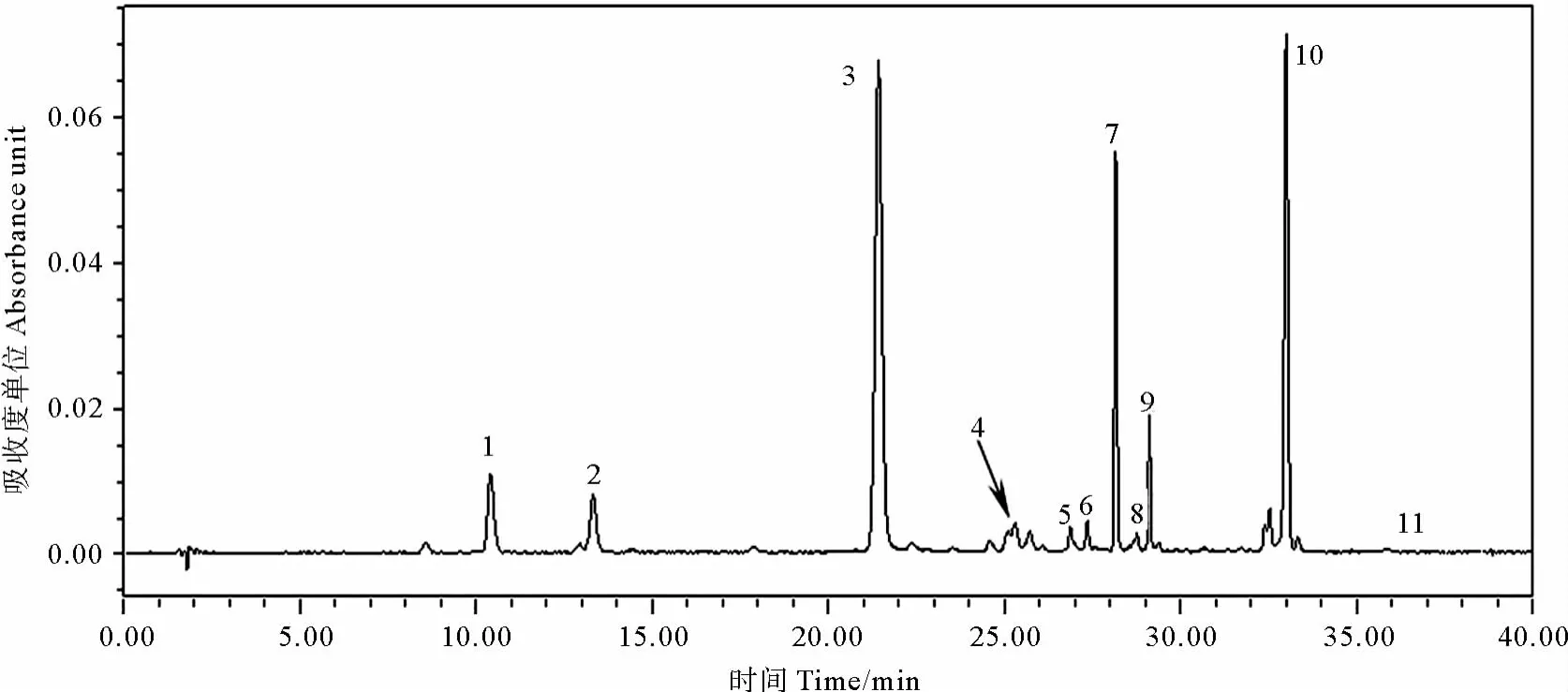
图3 N.sp.LAMB0002高效液相色谱图Fig.3 HPLC profile for N.sp.LAMB0002
微拟球藻属只含叶绿素a,不含叶绿素b和叶绿素c,特征色素为紫黄素和无隔藻黄素[5]。因此,确定N.sp.LAMB0002为微拟球藻。
气象色谱分析N.sp.LAMB0002、N.oceanicLAMB0001、N.salinaCS-190、N.oculataCS-189和N.oculataCS-192的几种主要脂肪酸所占藻粉干重的比值见表2。
N.sp.LAMB0002的主要脂肪酸为C14∶0、C16∶0、C16∶1、C18∶0、C18∶1N9、C18∶2T、C20∶4和C20∶5(EPA),其中含量较高为C16∶0、C16∶1和C20∶5,分别能达到藻粉干重的1.44%、1.61%和1.09%。5株微拟球藻所含的主要脂肪酸种类相同,没有特殊的脂肪酸将其区分。5株中C14∶0、C16∶1、C20∶4和C20∶5的含量并没有显著差异,其中N.oceanicLAMB0001含量最高,N.sp.LAMB0002次之。其余几种主要脂肪酸含量在不同藻株中有显著差异,但差异没有明显规律,不足以将不同种微拟球藻分开。因此,难以用脂肪酸的组成和含量区别微拟球藻各个种。
2.3 分子系统学分析
N.sp.LAMB0002的18SrRNA 基因与N.oceanicaLAMB0001(HQ201714.1)相 似 度 达 到98.7%。N.sp.LAMB0002的rbcL基因部分序列与N.oceanicaLAMB0001(HQ201714.1)相似度达到99.8%。将这2个序列与其他相关序列比对建立系统进化树(见图4和5)。
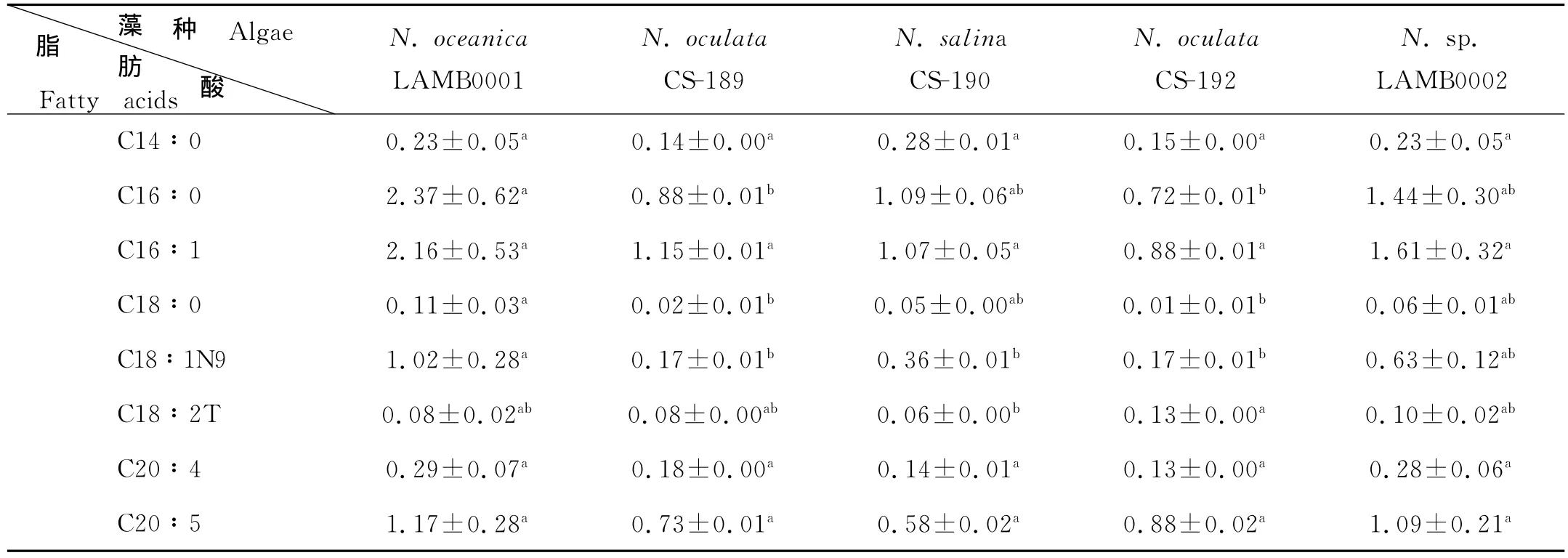
表2 5株微拟球藻主要脂肪酸占干重百分比Table 2 Percentile contents of main fatty acids in dry biomass of 5 Nannochloropsis strains
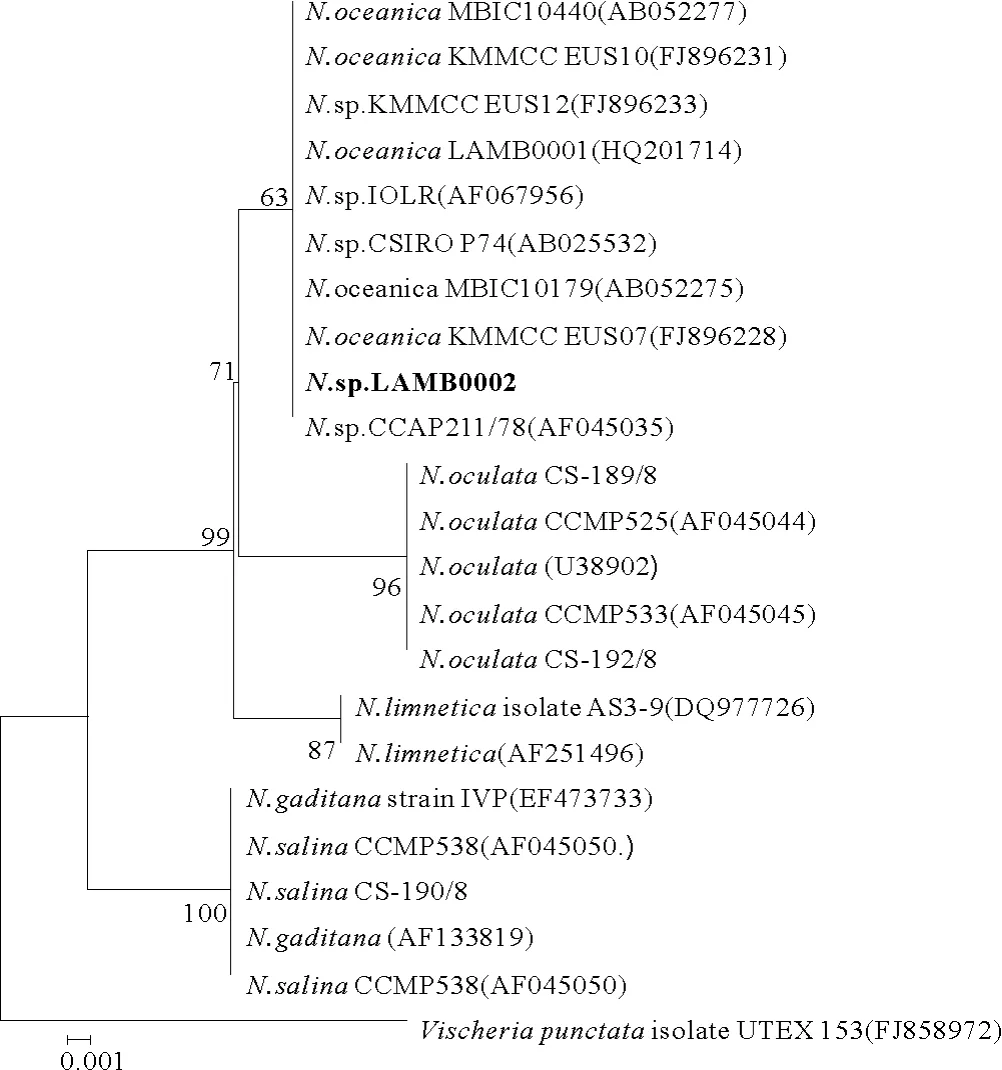
图4 基于18SrRNA基因构建NJ树(Tamura-Nei distances)Fig.4 Neighbor-Joining tree(Tamura-Nei distances)based on 18SrRNA gene sequences
以Vischeriapunctata为外类群基于18SrRNA基因分别构建了NJ树和MP树(未显示)。2种进化树都表明N.sp.LAMB0002与N.oceanica亲缘关系较为接近。以Goniochlorissculpta为外类群基于rbcL基因分别构建了NJ树和MP树(未显示)。2种进化树都将N.sp.LAMB0002分到N.oceanica分枝中。结果表明,N.sp.LAMB0002是N.oceanica。从以上进化树还可以看出rbcL基因与18SrRNA基因相比,能将微拟球藻各种完全分开,更能反映种间的差距。通过形态观察、生理生化分析和分子系统学分析将N.sp.LAMB0002鉴定为N.oceanicLAMB0002。
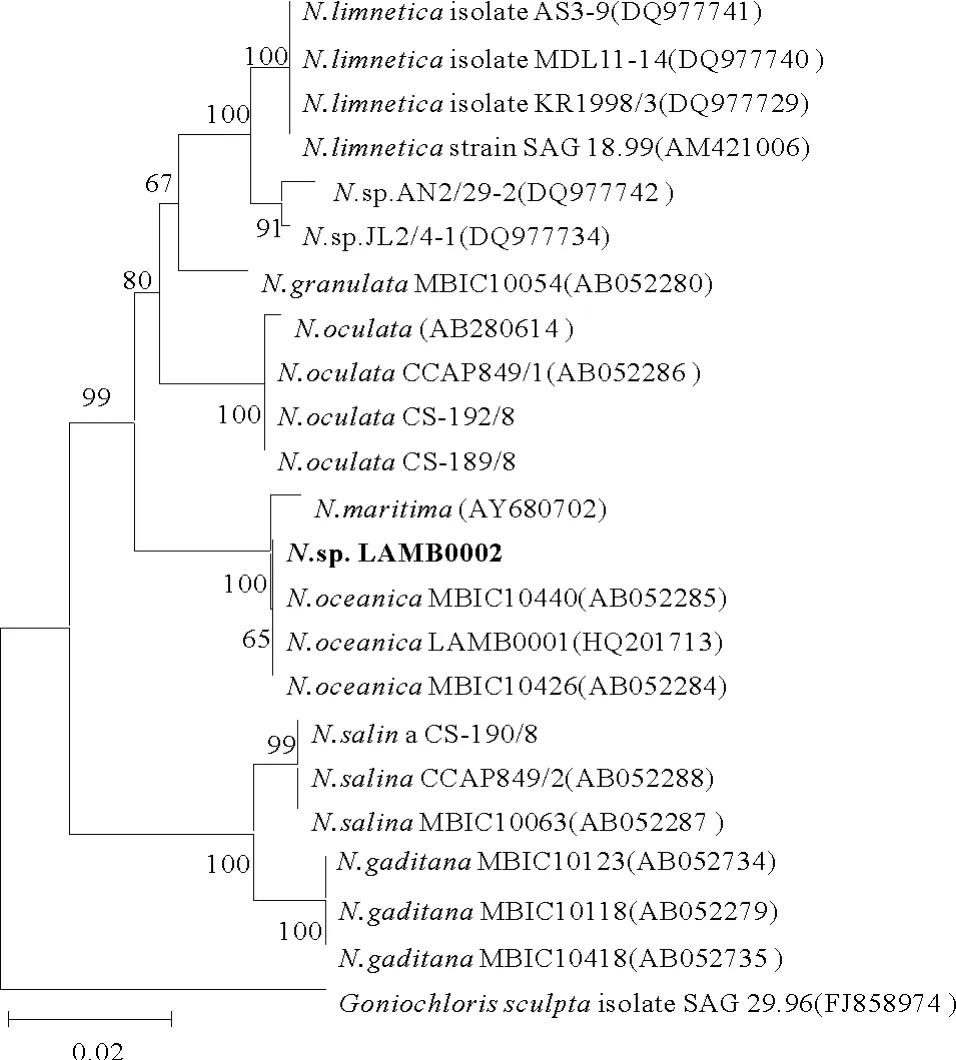
图5 基于rbcL基因构建NJ树(Tamura-Nei distances)Fig.5 Neighbor-Joining tree(Tamura-Nei distances)based on rbcL gene sequences
3 结论
形态学鉴定是鉴定微藻最直接的方法,对于很多种间形态相差较大的藻,形态学描述是最直接明确的手段。但是对于像微拟球藻这样体积微小、结构简单、种间差异微小的单细胞藻,形态学分类鉴定工作难以进行。不同生境条件下的同种微藻形态也有区别,这更加大了形态学鉴定的难度。这项工作对鉴定者的知识背景和经验要求严格,要广泛开展微藻鉴定的工作仅仅依靠形态学是远远不够的。形态学鉴定受人为因素影响大,不同研究者的鉴定结果可能有所偏差,难以标准化。因此,鉴定工作需要更快、更准确的标准化的方法。
不同藻所含有生化成分不同,有些特殊的生化成分可以成为藻类分类鉴定的辅助工具。如微拟球藻含有较高的EPA[6],无隔藻黄素,角黄素,β-胡萝卜素和仅有的叶绿素a(不含叶绿素b和叶绿素c),这些能有效地将微拟球藻与其他微藻区别开来[5]。雨生红球藻含有含量较高的虾青素,被认为是天然虾青素的重要来源[21]。1948年 Millner等[22]提出以脂肪酸含量差异作为标准的化学分类方法;Graeve等[23]认为脂肪酸不仅可以作为分类的标准,还可以作为分析藻类进化关系的工具。至今,这种分类鉴定方法存在较大争议[24-26]。如同其他的生化成分,脂肪酸的组成和含量也会随着不同的培养条件有所变化[26],但是这能否将不同的种和株系分开,这些生化成分是否足够保守,能否阐述不同藻株之间的进化关系,都是需要研究的问题。若利用不同生化成分快速鉴定微藻,必须有一个资料齐备的数据库。这需要各地研究者的长期积累和共同努力。所以,目前用生化成分对微拟球藻鉴定只是一个辅助手段。
DNA条形码技术(DNA barcoding)是现在浮游生物分类鉴定中应用最为广泛的分子手段[27-29]。该技术旨在用一个或几个基因实现快速鉴定。这项技术可以适用于不同生活阶段的物种,生物体的组织部分与培养环境无关,也相对客观。这种技术的应用不需要专业的训练(除了对于PCR相关知识的了解)。目前DNA条形码的数据库主要有Barcode of Life Database(BOLD)、Canadian Centre for DNA Barcoding(CCDB),International Barcode of Life Project(iBOL)和Consortium for the Barcode of Life(CBOL)。除此之外,CBOL还有一个专注于植物陆地植物条形码的组织 Royal Botanic Gardens,Kew(http://www.kew.org/barcoding/)。
在动物中,Cytochrome oxidaseⅠ(COⅠ)基因对于种间的鉴定非常有效,在大多数动物中有很好的分辨力[30]。植物中的线粒体基因组进化较慢,核酸替换率低。因此,排除了COⅠ作为植物条形码的可能性[31]。CBOL推荐的2个质体基因组合rbcL+matK成为现在普遍承认的植物条形码[32]。藻类中还没有通用的条形码,无论是在大型藻还是微藻中作为条形码的候选基因很少。目前的研究都是针对特定种,选择可能成为其条形码的基因或基因组合,且这些研究主要集中在红藻[33-34]、褐藻[35]、硅藻[36-37]和甲藻[28],在其他藻类中的研究很少。
在微拟球藻中,18SrRNA基因是最早提出用于分类鉴定的序列,在N.salina和N.gaditana中18S rRNA基因只有一个碱基的差异[10],微小的差异不足以将微拟球藻中各个种分开。rbcL基因的进化速度较18SrRNA基因快,不仅能分开微拟球藻属的各个种,而且同种间的不同株系之间也有碱基的差异。对于微拟球藻的分类鉴定,rbcL基因比18SrRNA基因更有效[3]。rbcL基因具有通用、易扩增和易比对的特点,并且该基因数据库相对丰富。但能否将rbcL基因作为微拟球藻的候选条形码还需要大量的工作,涵盖更多的微拟球藻种和株系。而该基因是否能作为微藻的候选条形码则需要更多更全面的工作和研究。
该方法中的形态学指标和生理生化指标鉴定未能实现微拟球藻的种间鉴定,且电镜(无论扫描电镜或是透射电镜)的样品制备过程十分繁复,制作切片也需要进行专业的训练,否则会影响鉴定。对于硅藻和甲藻而言,扫描电镜是非常好的鉴定工具。利用扫描电镜能将在光学显微镜下难以观察到的硅壳的花纹细节和甲板的细节放大,这些硅壳和甲板上的细节就是鉴定的依据。但近几年来,硅藻和甲藻的分子系统学分析层出不穷,且该方法不仅能快速准确地实现鉴定,还能省去长时间培养、制作切片等费时费力的步骤。对于微拟球藻而言,光镜或是电镜观察都不能实现快速、准确的鉴定。
依据生理生化指标的鉴定方法所需的藻液量大,所需的培养时间长,而且只能作为辅助的鉴定手段,难以达到大规模、快速鉴定的目的。在本研究中脂肪酸组成和色素组成的分析都无法实现微拟球藻的种间鉴定。因此,依据分子标签序列进行鉴定是实现微拟球藻快速、准确的鉴定的最好方法。
对于野外大规模样品的快速鉴定,可以先用光学显微镜观察的方法初步推断未知藻株的分类地位,再用分子标签序列进行鉴定。该微拟球藻的鉴定方法是否适用于微型海洋微藻的鉴定还需要进行进一步验证。
微藻种类繁多,形态各异,生化组成各不相同,进化速度也有很大差异,所以微藻的鉴定工作仍未形成标准。本研究根据形态学、生化成分和分子手段共同鉴定,准确的将一株未知的微拟球藻鉴定到种,初步形成了准确、快速的微藻分类体系,为今后形成系统的微藻分类体系打下了基础。
[1]Karlson B,Potter D,Kaglenstierna M,et al.Ultrastructure,pigment composition,and 18SrRNA gene sequence forNannochloropsisgranulatasp.nov.(Mondopsidaceae,Eustigmatophyceae),a marine ultraplankter isolated from the Skagerrak,northeast At-lantic Ocean[J].Phycology,1996,35:253-260.
[2]Krienitz L,Hopporle D,Stich H B,et al.Nannochloropsislimnetica(Eustigmatophyceae),a new species of picoplankter from freshwater[J].Phycologia,2000,39:219-227.
[3]Suda S,Atsumi M,Miyashita M.Taxonomic characterization of marineNannochloropsisspecies,N.oceanicsp.nov.(Eustigmatophyceae)[J].Phycologia,2002,41(3):273-279.
[4]Hibberd D J.Notes on taxonomy and nomenclature of algal Eustigmatophyceae and Tribophyceae (synonym Xanthophyceae)[J].Botanical Journal of the Linnean Society,1981,82(2):93-119.
[5]Lubián L M,Montero O,Moreno-Garrido I,et al.Nannochloropsis(Eustigmatophyceae)as a source of commercially valuable pigments[J].J Appl Phycol,2000,12:249-255.
[6]Zou N,Richmond A.Effect of light-path length in outdoor flat plate reactors on output rate of cell mass and EPA inNannochloropsissp.[J].J Biotenchnol,1999,70:351-356.
[7]Apt K E,Behrens P W.Commercial developments in microalgal biotechnology[J].Journal of Phycology,1999,35:215-226.
[8]Gouveia L,Oliverara C.Microaglae as a raw material for biofuels production[J].J Ind Microbiol Biotechnol,2009,36:269-274.
[9]Fawley K P,Fawley M W.Observations on the diversity and ecology of freshwaterNannochloropsis(Eustigmatophyceae),with descriptions of new taxa[J].Protist,2007,158:325-336.
[10]Anderson R A,Brett R W,Potter D.Phylogyeny of the Eustigmatophyceae based upon 18SrRNA gene,with emphasis onNannochloropsis[J].Protist,1998,149:61-74.
[11]Pennisi E.Wanted:A barcode for plants[J].Science,2007,318:190-191.
[12]Kress W J,Erickson D L.DNA barcodes:Genes,genomics,and bioinformatics[J].Proceedings of the National Academy of Science USA,2008,105:2761-2762.
[13]钟秀容,陈连云,陈文列.单细胞海藻的透射电镜样品制备 [J].电子显微学报,2005,24(4):434-434.
[14]Zapata M,Rodríguez F,Garrido J L.Separation of chlorophylls and carotenoids from marine phytoplankton:a new HPLC method using a reversed phase C8:column and pyridine-containing mobile phases[J].Marnie Eeology Progress Series,2000,195:29-45.
[15]Lepage G,Roy C.Improved recovery of fatty acids through direct transesterification without prior extraction or purification[J].J Lipid Res,1984,25:1391-1396.
[16]陈颖,刘根齐,李文彬,等.三种小球藻DNA提取方法的比较[J].植物生理学通报,2001,37(3):242-244.
[17]Hashimoto H.The ultrastructural features and division of secondary plastids[J].J Plant Res,2005,118:163-172.
[18]Van den Hoek C,Mann D G,Jahus H M C.Algae-An Introduction to Phycology[M].New York:Cambridge University Press,1995.
[19]Murakami R,Hashimoto H.Unusual nuclear division inNannochloropsisoculata(Eustigmatophyceae,Heterokonta)which may ensure faithful transmission of secondary plastids[J].Protist,2009,160:41-49.
[20]Santos L M A,Lee Dale G F.Some notes on the ultrastructure of small azoosporic members of the algal class Eustigmatophyceae[J].Nova Hedwigia,1995,60:219-225.
[21]Johnson E A,An G H.Astaxanthin from microbial sources[J].Critical Reviews in Biotechnology,1991,11:297-326.
[22]Millner H W.The fatty acids of Chlorella[J].Biochem,1948,176:813-817.
[23]Graeve M,Kattner G,Wiencke C,et al.Fatty acid composition of arctic and Antarctic marcroalgae:indicator of phylogentic and trophic relationship[J].Marine Ecol Prog Series,2002,231:67-74.
[24]Khotimchenko S U.Fatty acid composition of 12species of Chlorophyceae from the Senegalese coast[J].Phytochemistry,1992,31:2739-2741.
[25]Khotimchenko S V.Fatty acids of brown algal the Russian far east[J].Phytochemistry,1998,49(8):2363-2369.
[26]Carpenter E J,Harvey H R,Fry B,et al.Biogeochemistry tracers of the marineTrichodesmiumcganobacterium[J].Deep-Sea Research PartⅠ,1997,44:27-38.
[27]Bucklin A,Wiebe P H,Smolenack S B,et al.DNA barcodes for species identification of euphausiids(Euphausiacea,Crustacea)[J].J Plantkton Res,2007,29:483-493.
[28]Lin S,Zhang H,Hou Y,et al.High-level diversity of dinoflagellates in the Natural Environment,revealed by assessment of mitochondrial cox1and cob genes for dinoflagellate DNA barcoding[J].Appl Environ Microbiol,2009,75:1279-1290.
[29]Webb K E,Baenes D K A,Clark M S,et al.DNA barcoding:a molecular tool to identify Antarctic marine larvae[J].Deep-Sea Research PartⅡ,2006,53:1053-1060.
[30]Jinbo U,Kato T,Ito M.Current progress in DNA barcoding and future implications for entomology[J].Entomological Science,2011,14:107-124.
[31]Fazekas A J,Burgess K S,Kasanakarti P R,et al.Multiple multilocus DNA barsodes from the plastids genome discriminate plant species equally well[J].PLoS One,2008,3:2802.
[32]CBOL Plant Working Group.A DNA barcode for land plants[J].Proceedings of the National Academy of Science,2009,106:12794-12797.
[33]Clarkston B E,Saunders G W.A comparision of two DNA barcode markers for species discrimination in the red algal family Kallymeniaceae(Gigartinales,Florideophyceae),with a description ofEuthoratrimburtoniisp.nov.[J].Botany,2010,88:119-131.
[34]Saunders G W.Applying DNA barcoding to red macroalgal:a preliminary appraisal holds promise for future applications[J].Phil Trans R Soc B,2005,360:1879-1888.
[35]Lee S R,Oak J H,Keum Y S,et al.Utility ofrbcS gene as a novel target DNA region for brown algal molecular systematic[J].Phycological Resarch,2011,59:34-41.
[36]Hamsher S E,Evans K M,Mann D G,et al.Barcoding Diatoms:Exploring alternatives to COⅠ-5P[J].Protist,2011,162:405-422.
[37]Moniz M B J,Kaczmarska I.Barcoding of Diatoms:Nuclear encoded ITS revisited[J].Protist,2010,161:7-34.
