转座酶的人工改造与修饰
2014-10-31周倩倩周明兵
周倩倩,周明兵
浙江农林大学 亚热带森林培育国家重点实验室培育基地,浙江 临安 311300
转座子 (Transposable elements, TEs)是指在基因组上能从同一条染色体的一个位置转移到另一个位置或者从一条染色体转移到另一条染色体上的一段DNA序列,20世纪40年代美国遗传学家 McClintock[1]首次在玉米中发现。此后科学家们发现了多种类型的转座子,它们广泛存在于细菌、酵母和高等动植物基因组中。转座子依据转座方式的不同分为DNA转座子和反转录转座子两类。DNA转座子以“剪切-粘贴”的方式转座,其转座只是位置的移动,并不增加拷贝数,典型的含有末端反向重复(Inverted terminal repeats, ITRs)和编码转座酶的基因,结构较简单[2];而反转录转座子则以“复制-粘贴”的方式转座,通过 RNA 中间体来实现转座,在转座过程中编码反转录酶,每发生一次转座,即可以增加一个拷贝,反转录转座子结构比较复杂,除了编码基因外,还带有增强子、启动子等调控元件[3]。转座子能够通过它们的转座来塑造物种的基因组[4-5],例如促进基因组失活、调节基因组的表达、诱导不正规的基因组重组、在物种形成和进化的过程中影响基因组的大小。近年来,随着对转座子的研究,转座子作为基因功能分析、转基因、插入突变及突变体库的构建、基因治疗等的工具,已经在多种植物以及酵母Saccharomyces lysate、线虫Caenorhabditis elegans、果蝇Drosophila melanogaster和部分脊椎动物中得到成功应用[6-7]。
转座子在进化过程中以垂直遗传为主,也可通过横向遗传的方式侵入其他物种基因组中,频繁转座,大量扩增自身的拷贝数,从而在宿主基因组的长期进化中保留下来。一个转座子从侵入宿主基因组到在这个宿主基因组稳定下来一般要经历4个阶段:1) 外源转座子的侵入;2) 高频转座以扩增拷贝数;3) 通过物种杂交在群体里广泛扩散,大量转座子积累点突变和插入/缺失突变丧失活性;4) 通过转座子的随机丢失,宿主基因组和转座子达到生态平衡。处在第2) 阶段的转座子活性最强,基因组鉴定的转座子大部分处在3) 或 4) 阶段,积累了或多或少的突变,部分或全部丧失了转座能力,成为低活性或非活性的转座子“化石”[8-10]。但是突变发生是随机的,利用大量克隆的转座子,借助生物信息的技术和手段,可以检测出突变发生的位点和类型,结合分子生物学的技术和手段,构建和再现第2) 阶段的高活性转座子。高活性转座子再通过人工优化组合,构建出自然不存在的超活性转座子。
目前人工优化转座子主要应用在 DNA转座子上,通过蛋白质工程的手段改造DNA转座子的转座酶,获得高催化活性的转座酶是人工构建超高活性的转座子的主要途径。DNA转座酶一般由2部分组成:N端的DNA 结合域 (DNA binding domain, DBD),C端催化作用的功能区 (Catalysis domain)。MLE (Mariner-like element, MLEs)转座酶 (图 1)编码序列长度一般为 900−3 000 bp(动物转座酶一般没有内含子,植物转座酶一般含 2−4个内含子),由 DNA结合域和转座催化域组成。DNA结合域负责识别和结合 TIRs,含有 1−2个螺旋-转角-螺旋结构域(Helix-turnhelix,HTH)。催化域有DD(34/37/39)D保守结构域 (动物为 DD34/37D;植物为 DD39D,在阳离子 Mg2+或 Mn2+协助下,既能催化仅包括TIRs及侧翼序列的短片段DNA转座,也能催化携带有目的基因 (如抗性基因)长片段DNA的转座[11]。
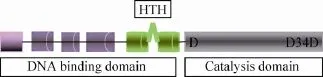
图1 MLE转座酶基因的结构图[11]Fig. 1 Domains of MLE transposase[11].
目前,运用蛋白质工程和生物信息学结合的方法已经成功改造多种转座酶,如 Sleeping Beauty转座酶(SBase)、PiggyBac转座酶(PBase)、Mos1转座酶、Himar1转座酶、Hsmar1转座酶以及玉米中Activator转座酶(AcTPase)、P转座酶等,这里主要介绍上述几种酶的人工构建和改造。
1 活性转座子的人工构建
转座子在生物体内经历了漫长的进化和突变,由于突变积累,大部分失去了转座活性,需要人工构建有活性的转座子。一般采用生物信息学系统分析和蛋白质工程相结合的方法来重新构建活性的转座酶,具体方法有:1) 同源重建:对相关的同源的转座酶进行同源比对分析,找到并分析它们的保守区域,从相关的转座酶中导入小片段氨基酸,把相关转座酶家族的保守氨基酸整合到这个转座酶中;2) PCR定点诱变技术:用蛋白质工程的手段定点突变选定的氨基酸区域和位点,以恢复转座酶的催化活性。SB转座子和 Hsmar1转座子就是利用上述这两种方法重新获得活性[12-13]。
1997年Ivics等[12]用来自8个鱼类品种中的12个失活的鲑鱼亚科家族Tc1类转座酶的序列,基于积累的系统发生数据,利用生物信息学的方法,经过多重序列对比,找到了一个保守序列,并确定了保守区的蛋白序列以及DNA序列。激活转座酶的具体步骤是,首先将两个来源不同的片段互补拼接在一起,恢复一个完整的开放阅读框 (SB1−SB3),由于SB3多肽的序列有24个位置的氨基酸与保守序列不符,因而没有转座能力;随后,通过PCR引入定点突变的方法一步步地改变编码框的序列 (SB4−SB10),直至与保守序列相符,最终完成编码 340个氨基酸的SB10转座酶的基因,使沉睡了1 000多万年的转座子再次被激活而具有转座能力,因此被命名为“睡美人”。SB转座子在包括人在内的脊椎动物的组织中是有活性的,同时在鱼、蛙、小鼠和大鼠中也具有活性,可以用作转基因的载体和基因治疗的工具[14-15]。
Hsmar1是一种古老的Mariner转座子,在5 000万年前进入到灵长类基因谱系中。由于长期的突变积累,Hsmar1不能编码有活性的转座酶。2007年Miskey等[13]利用生物信息学的系统方法来重建古老的Hsmar1转座酶基因,并命名为Hsmar1-Ra,改造方法类似于Sleeping Beauty转座酶。具体步骤是,通过与伞树亚科几种转座酶的氨基酸序列比较,把同源的转座酶的保守区域的氨基酸整合到Hsmar1转座酶基因中;同时依据从黑猩猩体内获得的同源的Hsmar1转座酶基因,利用定点突变技术突变21个氨基酸得到转座酶的共有序列;通过 TBLASTN寻找人类Hsmar1转座酶类似的氨基酸序列,利用最大概率法重建古老的基因序列。将Hsmar1的转座酶中非保守的氨基酸替换成 4个推测的保守的氨基酸 (C53R、P167S、L201V和 A219C),在人Hela细胞、鱼胚胎中进行转座试验,通过琼脂糖凝胶发现了在人Hela细胞和鱼胚胎中转座的片段[16],证明了人工构建的 Hsmar1-Ra可以在包括人在内的脊椎动物细胞内转座。
2 超活性转座子的人工优化
为了进一步提高转座子的转座活性,科学家们优化转座酶的氨基酸组成,人工改造出自然界不存在的超活性的转座子。主要方法有通过专一性位点突变获得高活性的转座酶突变体,利用PCR随机突变技术构建转座酶突变体库筛选出高催化能力的转座酶。
2.1 专一性位点突变
专一性位点突变又称特异性位点突变,主要运用PCR技术产生突变。
Himar1是从黑角蝇属中分离得到的Mariner类转座子,是原核生物基因分析的主要工具之一。而且在人类的细胞中也有活性,但转座效率较低。1999年 Lampe等[17]利用易错PCR获得Himar1的9个单位点突变体,突变区域集中在 HTH 结构和催化作用功能区(DD34D)。有5个位点的突变能增加转座酶的活性,其中3个位点的突变活性较高,H267R能使转座酶的活性提高约10倍,Q131R和E137K分别使转座酶的活性提高约4倍和20倍,把这两个位点结合起来则转座酶的活性提高约为野生型的50倍。基于Q131R和E137K结合的转座酶突变体可以作为基因治疗的有效载体[18]。
Mos1 转座子是从马里塔尼亚果蝇D. mauritiana中发现的Mariner类天然活性转座子,已经开发成埃及伊蚊Aedes aegypti、利什曼虫Leishmania major等动物的转基因工具,为了提高Mos1转座酶的活性,2009年Germon等[19]依据对Himar1突变体位点的研究,在Mos1上选择同一位点或相近位点进行单点突变。突变位点位于二聚作用功能区、转角-螺旋-转角区域、催化作用功能区 (DD34D),Mos1突变了12个位点,用大肠杆菌筛选系统共获得5种高活性的突变类型 (F53Y、Q91R、E137K、T216A和Y237C),分别将这5个突变位点进行两个及两个以上的随机组合,得到活性比野生型至少高20倍的7种组合突变,这些突变组合都包含T216A单点突变。对于 FET (F53Y-E137KT216A)这种突变类型,发现它的活性为野生型的 200–800倍,甚至更高,但是过高的转座活性会产生细胞毒性,导致细胞死亡[19-20]。
PiggyBac转座子是由粉纹夜蛾Trichoplusia ni基因组中分离的转座子家族,在哺乳动物细胞中也能发生转座。PiggyBac转座子作为哺乳动物基因分析的工具,能精确切除而不产生足迹,不影响基因组的整合,广泛应用于插入诱变和基因治疗[21]。依据进化分析和先前序列的重建,替换专一性的位点M282V,其转座切除活性提高到野生型的 2–7倍[22]。P转座子从果蝇中分离得到的,已经开发成果蝇的遗传学分析工具和转基因的载体[23]。P转座酶包含10个同源的磷酸化位点,这10个位点中,有2个位点的取代对转座酶的活性无影响,其中 7个位点的取代会降低转座酶的活性,只有S129A产生超活性的转座活动,转座活性约是野生型的1.72 倍[24]。
Ac/Ds系统因其在众多植物种类中都有转座活性,且优先插入编码序列内部或附近,或插入基因连锁位点使其在植物遗传分析中得到广泛应用[25]。为了提高 Ac/Ds系统转座活性,2012年 Lazarow等[26]通过定点突变技术对AcTPase的氨基酸进行优化,选择了 E249A、E336A、D459A和D545A (丙氨酸不携带有作用的侧链)替换,单个位点的突变切除频率是野生型的 3–5倍。两个位点组合突变 E249A/E336A和 D459A/D545A突变频率是野生型的约 11和 13倍,4个氨基酸残基同时发生替代(即AcTPase4x)则可使 Ds切除活性在酵母细胞中增强 100倍,同时重新插入的频率不变 (57%),在拟南芥中催化 Ds的切除活性为野生型的6倍。
2.2 随机突变获得超活性转座酶
通过对转座酶的随机突变,获得转座酶的突变体库,也是获得超活性转座酶的重要途径。
2009年 Mates等[15]将 SB转座酶通过同源比对,共获得了41个能提高转座酶活性的氨基酸突变位点。用核酸酶把含有这41个氨基酸突变位点的突变体切成约300 bp大小的片段,然后再随机切成30−70 bp的片段。用PCR技术随机重组不重叠的片段,从而得到41突变位点随机组合转座酶突变体库。通过筛选获得的转座酶活性提高至原来100倍以上的 SB100X转座酶,SB100X转座酶是 6个突变位点的组合(K14R、K33A、F115H、214DAVQ、M243H、T314N)。得到的SB100X转座酶在人类的Hela细胞和其他脊椎动物的细胞中进行了转座实验。在人类的Hela细胞中SB100X的转基因效率达到了35%,比野生型SB在动员染色体元件转座效率高至120倍,SB100X比野生型SB产生多3–5倍的插入位点。SB100X转座酶在斑马鱼 Barchydanio rerio胚胎中瞬时转基因的效率达到了原来的 8倍[27]。转座酶人工构建及改造的详细信息归纳在表1中。
3 插入特性的人工改造
通过人工重新构建和优化获得了超活性的转座酶,极大地提高了转座子的转座活性。但转座子插入到基因的编码区,会致使基因突变或基因表达模式的改变,产生细胞毒性。例如,SB100X在斑马鱼胚胎中检测到了相比较原先的 8倍瞬时转基因效率,但同时会形成有缺陷的胚胎,具有转座插入毒性[27],存在潜在的安全问题。因此,通过对转座酶进行改造和修饰,人为地调控转座子插入到非基因区,不影响基因的表达是目前转座子人工改造的另一个重要工作。
3.1 利用 C2H2(Cys2His2)锌指蛋白定位转座子的插入
C2H2 (Cys2His2)型的锌指蛋白是一类真核生物转录因子重要结构域,其主要作用是与DNA 结合,指导酶与模板的正确结合,活化RNA 聚合酶,以便有效地启动转录[28]。利用C2H2型的锌指蛋白与转座酶融合,可以调控转座子的插入位点。
六指锌指-E2C是人工构建,应用广泛的一个 C2H2 (Cys2His2)型的锌指蛋白,可以与人类erbB-2基因(红细胞白血病病毒癌基因同源物2,erythroblastic leukemia viral oncogene homolog 2)启动子域的一个独特的18 bp (GGGGCCGGA GCCGCAGTG)位点结合。锌指域分别被融合到SB转座酶的N端或者C端或者与SB转座酶N端的57个氨基酸的螺旋-转角-螺旋区域 (N57),来定位 SB转座子在人类基因组的插入。在人Hela细胞中,当E2C融合到SB转座酶N端,E2C锌指蛋白引导SB转座子插入到人类基因组erbB-2启动子区域的E2C结合位点上游179 bp处,插入频率为1.44%;当E2C融合到SB转座酶C端和N57区域,E2C锌指蛋白引导SB转座子插入到人类基因组 erbB-2启动子区域的E2C结合位点上游 732 bp处,插入频率为1.45%;但是在E2C结合位点周围20 kb却检测不到任何的插入[29]。
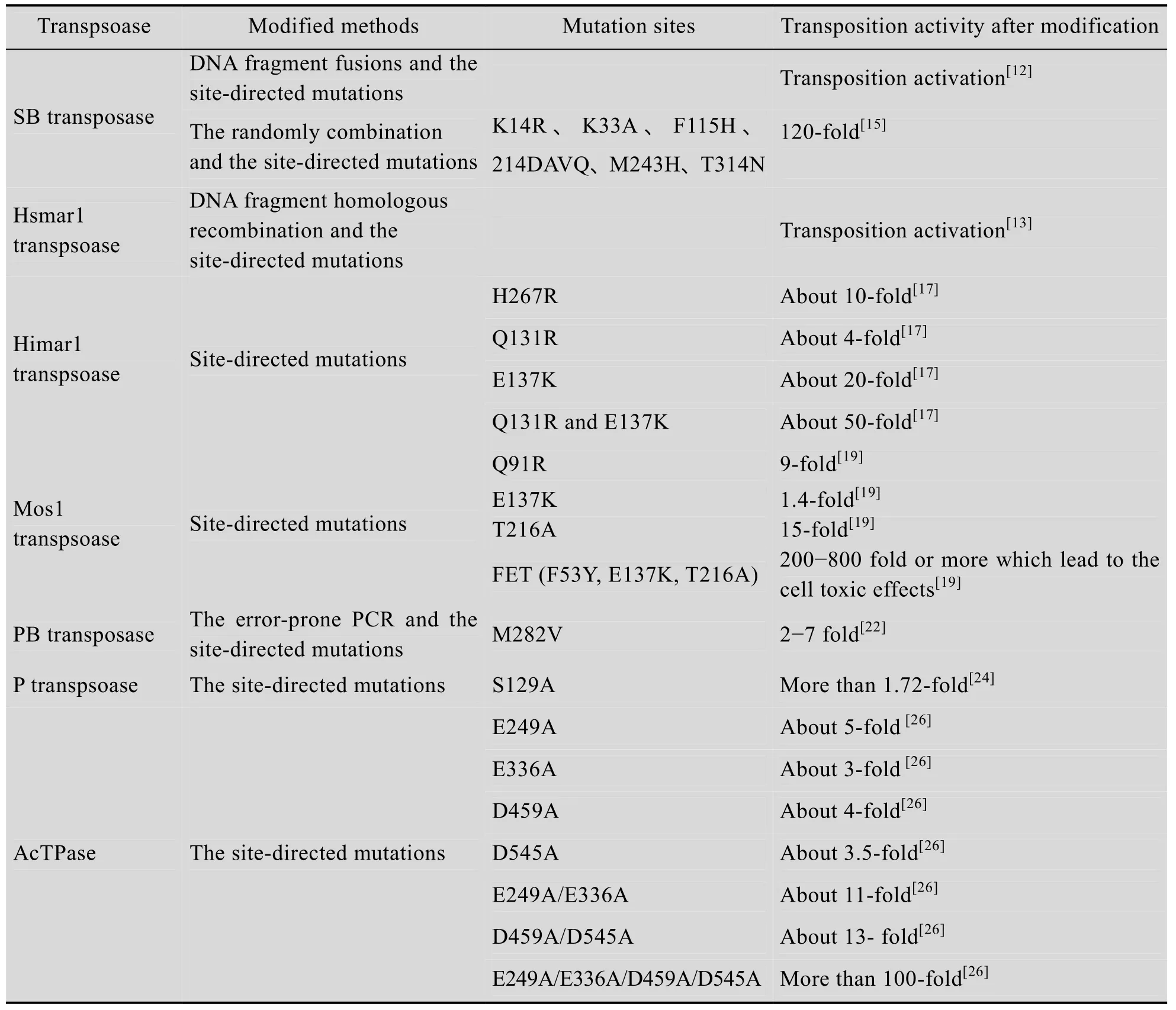
表1 转座酶人工构建及改造Table 1 Artificial reconstruction and modification of transpsoases
锌指结构可能是进行有针对性转座插入的合适转座引导物,但以E2C为基础的转座系统,其转座效率较低,作为替代方案选择人类的内源性反转录转座子LINE1作为潜在的SB定位目标。LINE1在人类基因组中拷贝数高,富含AT,分布在基因贫乏的染色体区域。设计一个新的DNA结合域-锌指蛋白B (Zinc finger B,ZF-B),能识别LINE1反转录转座子3'末端的18 bp序列(GCCATAAAAAATGATGAG)。当ZF-B融合到SB100X转座酶N端,ZF-B引导SB转座子插入到人类的内源性反转录转座子LINE1中ZF-B结合位点周围的400 bp区域,插入频率为1.36%,是未融合的SB100X的转座插入频率的4倍;当ZF-B融合到SB100X转座酶的C端和N57区域,ZF-B引导 SB转座子插入到人类的内源性反转录转座子LINE1中ZF-B结合位点周围的400 bp区域,插入频率为0.82%,是未融合的SB100X的转座插入频率的2.4倍。在ZF-B结合位点周围的20 kb区域,ZF-B融合到SB100X转座酶N端或C端和N57区域的转座插入频率分别为10.69%和 10.05%[29]。
3.2 利用Gal4DBD转座酶嵌合体定位转座子的插入
Gal4是真核细胞普遍存在的转录激活因子,Gal4的DBD能识别基因启动子的上游活性序列 (Upstream activating sequence,UAS)的17 bp长的一段序列,调控靶基因转录[30]。科学家们通过将转座酶与 Gal4 DBD融合,利用GAL4 DBD与UAS序列定点结合来调控转座子的插入。
2012年 Owens等[31]通过双质粒法分析了Gal4 DBD对PiggyBac转座子插入位点的调控,受体质粒含有氯霉素基因和一个含有 5个串联的UAS位点,传递质粒含有PB转座酶。当Gal4 DBD融合到PiggyBac转座酶N端,有87%的N-Gal4 PB插入到UAS下游800 bp区域,其中有 47%的 N-Gal4 PB插入靠近 UAS上下游250bp区域,有39%的N-Gal4 PB插入UAS位点250 bp上下游250 bp以内。当Gal4 DBD融合到PiggyBac转座酶C端,有77%的C-PB Gal4的插入靠近UAS上下游800 bp的位点,其中有32% 的 C-Gal4PB插入在 UAS附近的 250 bp内,而且 C-PB Gal4的插入位点表现出更加均匀的分布。无论N-Gal4 PB还是C-Gal4 PB蛋白都偏向于靠近UAS的位置插入。
3.3 利用病毒蛋白定位转座子插入
基于腺病毒相关病毒(Adeno-associated virus, AAV)的基因治疗的载体系统提供了作为基因运载工具的许多有利条件[32],该病毒显示没有致病性,能够有效地转化各种增殖和非增殖细胞[33]。AAV Report(Rep)蛋白对DNA的Rep识别序列(Report recognition sequences,RRSs)有特殊的结合活性,可以利用其特性来调控定位转座子的插入。
Tol2转座子是 hAT转座子家族中的一员,首次在青鳉鱼Oryzias latipes的基因组内发现,已证实Tol2转座子在斑马鱼、鼠、人等多种动物细胞中都具有转座活性[34]。在人类细胞中的SB、PB和Tol2的整合位点分布图表明,SB转座位点随机分布,PB和Tol2转座位点不是随机分布的,PB优先整合到基因和转录起始位点周围的区域,而Tol2偏好整合到转录单位和转录起始位点附近。2012年Ammar等[32]利用了AAV Rep蛋白特异性结合到RRS 的GAGC重复序列这一性质,来定位PB和Tol2、SB这3个转座子的整合。一个基于质粒转座实验显示,Rep/ SB融和转座酶的转座插入到 RRS下游的 700 bp内,占所有插入的30%,在目标RRS附近插入频率较野生型提高了 15倍。Rep/Tol2在 RRS附近的插入频率是野生型的4倍。但是,Rep/PB转座酶在RRS附近没有明显的转座插入。转座插入位点的人工改造详细信息归纳在表2。

表2 转座插入位点的人工改造Table 2 Artificial regulation of insertion sites based on transposase fusion
4 结语与展望
自 20世纪 40年代美国遗传学家McClintock[1]首次在玉米中发现转座子 (Ac/Ds)以来,科学家们发现了多种类型的转座子,它们广泛存在于细菌、酵母和高等动植物基因组中[2-3]。转座子是动植物基因组的重要组成部分,如在人基因组占45%[35],在玉米Zea mays基因组中比例高达 85%以上[36],在基因组进化及生物多样性形成过程中扮演着重要角色[13,37]。
随着人们在分子水平上对转座子结构和功能认识的不断深入,一些来源植物的转座子已被改造为转座子标签,应用于基因分析,如玉米的 Ac/Ds被应用于多种植物如水稻 Oryza sativa[38]、大麦 Hordeum vulgare[39]、杨树 Populus trichocarpa[40]等植物基因的克隆及突变体库的构建。一些来源动物的转座子已被改造为基因转化和基因治疗的工具,如SB,可以在鱼、小鼠和人等大多数脊椎动物的细胞将所携带的基因在动物体内稳定整合和长期表达[41]。这些使转座子在基因治疗及功能基因组学的研究等领域发挥重要的作用[42-43]。总之,近年来转座子逐渐成为基因克隆、基因表达及其功能、生物多样性及进化研究的重要工具[6-7]。另一方面,由于随机丢失和垂直失活效应[8-10],自然界基因组存在绝大部分转座子丧失了转座活性或转座活性不高,通过生物信息学和蛋白质工程方法改造和优化转座酶,提高其催化转座的能力,人工调控转座子的插入位点,更好地应用于基因标签、基因转化和基因治疗是目前转座子的开发和利用研究的主要方向。
本课题组一直致力于植物 Mariner类转座子开发和利用。近几年研究发现,Mariner类转座子在植物基因组分布也非常广泛[44],具有结构简单、插入位点接近随机和异源转座率高等特点,在开发植物转座标签方面表现出诱人的前景[45-46]。本课题组利用生物信息学和蛋白质工程结合的方法来改造源竹类植物的 Mariner类转座酶,目前在 63个竹种基因组克隆到 79个全长Mariner类转座酶,发现它们结构完整,序列保守,可能来源于进化史上一次横向传递(Horizontal transfer)事件,很可能具有转座活性。取其中一个与79个全长Mariner类转座酶的保守序列同源性最高的转座酶,通过酵母转座检测系统发现该转座酶的确可以催化转座子跳跃[47-48]。利用定点突变技术对该转座酶关键位点的非保守氨基酸残基突变获得相对于野生型 2–3倍高催化活性 (数据未发表)。利用来源竹子基因组高活性的 Mariner类转座酶构建植物首个 Mariner类转座子标签系统的工作正在开展中。
[1]McClintock B. The origin and behavior of mutable loci in maize. Proc Natl Acad Sci USA, 1950,36(6): 3440–3455.
[2]Feschotte C, Pritham EJ. DNA Transposons and the evolution of eukaryotic genomes. Annu Rev Genet,2007, 41: 331–368.
[3]Schulman AH. Retrotransposon replication in plants. Curr Opin Virol, 2013, 3(6): 604–614.
[4]Morgante M, De PE, Radovic S. Transposable elements and the plant pan-genomes. Curr Opin Plant Biol, 2007, 10(2): 149–155.
[5]Rebollo R, Romanish MT, Mager DL.Transposable elements: an abundant and natural source of regulatory sequences for host genes.Annu Rev Genet, 2012, 16(46): 21–42.
[6]Ovcharenko OO, Rudas VA, Kuchuk MV. Plant transposable elements and their application in genetics and biotechnology. Tsitol Genet, 2006,40(4): 68–80.
[7]Feschotte C, Jiang N, Wessler SR. Plant transposable elements: Where genetics meets genomics. Nat Rev Genet, 2002, 3(5): 329–341.
[8]Miskey C, Izsvák Z, Kawakami K, et al. DNA transposons in vertebrate functional genomics. Cell Mol Life Sci, 2005, 62(6): 629–641.
[9]Lampe DJ, Walden KK, Robertson HM. Loss of transposase-DNA interaction may underlie the divergence of mariner family transposable elements and the ability of more than one mariner to occupy the same genome. Mol Biol Evol, 2001,18(6): 954–961.
[10]Gilbert C, Schaack S, Pace JK II, et al. A role for host-parasite interactions in the horizontal transfer of transposons across phyla. Nature, 2010,464(7293): 1347–1350.
[11]Benjamin B, Bigot Y, Corinne AG. Assembly of the Tc1 and mariner transposition initiation complexes depends on the origins of their transposase DNA binding domains. Genetica, 2007,130(2): 105–120.
[12]Ivics Z, Hackett PB, Plasterk RH, et al. Molecular reconstruction of Sleeping Beauty, a Tc1-like transposon from fish, and its transposition in human cells. Cell, 1997, 91(4): 501–510.
[13]Miskey C, Papp B, Mátés L, et al. The ancient mariner sails again: transposition of the human Hsmar1 element by a reconstructed transposase and activities of the SETMAR protein on transposon ends. Mol Cell Biol, 2007, 27(12): 4589–4600.
[14]Zayed H, Izsvak Z, Walisko O, et al. Development of hyperactive Sleeping Beauty transposon vectors by mutational analysis. Mol Ther, 2004, 9(2):292–304.
[15]Mates L, Chuah MK, Belay E, et al. Molecular evolution of a novel hyperactive Sleeping Beauty transposase enables robust stable gene transfer in vertebrates. Nat Genet, 2009, 41(6): 753–761.
[16]Claeys BC, Chalmers R. Transposition of the human Hsmar1 transposon: rate-limiting steps and the importance of the flanking TA dinucleotide in second strand cleavage. Nucleic Acids Res, 2010,38(1): 190–202.
[17]Lampe DJ, Akerley BJ, Rubin EJ, et al.Hyperactive transposase mutants of the Himar1 mariner transposon. Proc Natl Acad Sci USA,1999, 96(20): 11428–11433.
[18]Keravala A, Liu D, Lechman ER, et al. Hyperactive Himar1 transposase mediates transposition in cell culture and enhances gene expression in vivo. Hum Gene Ther, 2006, 17(10): 1006–1018.
[19]Germon S, Bouchet N, Casteret S, et al. Mariner Mos1 transposase optimization by rational mutagenesis. Genetica, 2009, 137(3): 265–276.
[20]Richardson JM, Dawson A, O'Hagan N, et al.Mechanism of Mos1 transposition: insights from structural analysis. EMBO J, 2006, 25(6):1324–1334.
[21]Ding S, Wu X, Li G, et al. Efficient transposition of the piggyBac (PB)transposon in mammalian cells and mice. Cell, 2005, 122(3): 473–483.
[22]Yusa K, Zhou L, Li MA, et al. A hyperactive piggyBac transposase for mammalian applications.Proc Natl Acad Sci USA, 2011, 108(4):1531–1536.
[23]Sonane M, Goyal R, Chowdhuri DK, et al.Enhanced efficiency of P-element mediated transgenesis in Drosophila: microinjection of DNA complexed with nanomaterial. Sci Rep, 2013,3(3408): 1–5.
[24]Beall EL, Mahoney MB, Rio DC. Identification and analysis of a hyperactive mutant form of Drosophila P-element transposase. Genetics, 2002,162(1): 217–227.
[25]Boon GH, Gong Z. Maize Ac/Ds transposon system leads to highly efficient germline transmission of transgenes in medaka (Oryzias latipes). Biochimie,2011, 93(10): 1858–1864.
[26]Lazarow K, Du ML, Weimer R, et al. A hyperactive transposase of the maize transposable element activator (Ac). Genetics, 2012, 191(3):747–756.
[27]Newman M, Lardelli M. A hyperactive sleeping beauty transposase enhances transgenesis in zebrafish embryos. BMC Res Notes, 2010, 3(1):282.
[28]Zhong Q, Zhao SH. The mechanism and application of zinc finger nucleases. HEREDITAS,2011, 33(2): 123–130 (in Chinese).钟强, 赵书红. 锌指蛋白核酸酶的作用原理及其应用. 遗传, 2011, 33(2): 123–130.
[29]Voigt K, Gogol DA, Miskey C, et al. Retargeting sleeping beauty transposon insertions by engineered zinc finger DNA-binding domains. Mol Ther, 2012, 20(10): 1852–1862.
[30]Li CF, Xia QY, Zhou ZY. Study of GAL4/UAS system in transgenic technology. Biotechnology,2006, 1(78): 78–81 (in Chinese).李春峰, 夏庆友, 周泽扬. GAL4/UAS系统在转基因技术中的应用研究进展. 生物技术, 2006,1(78):78–81.
[31]Owens JB, Urschitz J, Stoytchev I, et al. Chimeric piggyBac transposases for genomic targeting in human cells. Nucleic Acids Res, 2012, 40(14):6978–6991.
[32]Ammar I, Gogol DA, Miskey C, et al. Retargeting transposon insertions by the adeno-associated virus Rep protein. Nucleic Acids Res, 2012, 40(14):6693–6712.
[33]Grabundzija I, Irgang M, Mátés L, et al.Comparative analysis of transposable element vector systems in human cells. Mol Ther, 2010,18(6): 1200–1209.
[34]Urasaki A, Morvan G, Kawakami K. Functional dissection of the Tol2 transposable element identified the minimal cis-sequence and a highly repetitive sequence in the subterminal region essential for transposition. Genetics, 2006, 174(2):639–649.
[35]Cordaux R, Batzer MA. The impact of retrotransposons on human genome evolution. Nat Rev Genet, 2009, 10(10): 691–703.
[36]Schnable PS, Ware D, Fulton RS, et al. The B73 maize genome: complexity, diversity and dynamics. Science, 2009, 326(5956): 1112–1115.
[37]Venner S, Feschotte C, Biémont C. Dynamics of transposable elements: towards a community ecology of the genome. Trends Genet, 2009, 25(7):317–323.
[38]Qu S, Desai A, Wing R, et al. A versatile transposon-based activation tag vector system for functional genomics in cereals and other monocot plants. Plant Physiol, 2008, 146(1): 189–199.
[39]Ayliffe MA, Pallotta M, Langridge P, et al. A barley activation tagging system. Plant Mol Biol,2007, 64(3): 329–347.
[40]Fladung M, Polak O. Ac/Ds-transposon activation tagging in poplar: a powerful tool for gene discovery. BMC Genomics, 2012, 13(1): 61.
[41]Davis RP, Nemes C, Varga E, et al. Generation of induced pluripotent stem cells from human foetal fibroblasts using the Sleeping Beauty transposongene delivery system. Differentiation,2013, 86(1/2): 7–30.
[42]Vanden DT, Ivics Z, Izsvák Z, et al. Emerging potential of transposons for gene therapy and generation of induced pluripotent stem cells. Blood,2009, 114(8): 1461–1468.
[43]Ivics Z, Kaufman CD, Zayed H, et al. The Sleeping beauty transposable element: evolution, regulation and genetic applications. Curr Issues Mol Biol,2004, 6(1): 43–55.
[44]Feschotte C, Wessler SR. Mariner-like transposases are widespread and diverse in flowering plants.Proc Natl Acad Sci USA, 2002, 99(1): 280–285.
[45]Yang G, Weil CF, Wessler SR. A rice Tc1/Mariner-like element transposes in yeast. The Plant Cell, 2006, 18(10): 2469–2478.
[46]Yang G, Nagel DH, Feschotte C, et al. Tuned for transposition: molecular determinants underlying the hyperactivity of a Stowaway MITE. Science,2009, 325(5946): 1391–1394.
[47]Zhou MB, Lu JJ, Zhong H, et al. Distribution and polymorphism of mariner-like elements in the Bambusoideae subfamily. Plant Syst Evol, 2010,289: 1–11.
[48]Zhou MB, Zhong H, Tang DQ. Isolation and characterization of seventy-nine full-length mariner-like transposases in the Bambusoideae subfamily. J Plant Res, 2011, 124: 607–617.
