小鼠饲养过程中蛋白质组的整体重稳定性同位素标记
2014-10-31樊锋旭高慧英徐忠伟翟琳辉衣泰龙张涛吴飞林崔春萍徐平1
樊锋旭,高慧英,徐忠伟,翟琳辉,衣泰龙,张涛,吴飞林,崔春萍,徐平1,
1 安徽医科大学,安徽 合肥 230032
2 军事医学科学院放射医学研究所,北京 100850 3 北京蛋白质组研究中心,北京 102206
蛋白质组学是继基因组学后出现的一门在整体水平上研究细胞内蛋白质组成及其活动规律的新兴学科[1-2]。定量蛋白质组学是对细胞、组织或体液等生物样本在某些过程中蛋白质的含量进行比较分析。定量蛋白质组学中最常用的是基于稳定同位素代谢标记 SILAC (Stable isotope labeling with amino acids in cell culture)方法,使用轻、重稳定性同位素合成的氨基酸标记不同来源的体外培养细胞。由于轻、重稳定性同位素标记肽段具有完全一致的物理化学性质,因此在色谱分离过程中能够被共洗脱出来,同时经离子化而进入质谱被检测,但重稳定同位素标记蛋白质的肽段与轻标记肽段的离子在质量上会出现偏移,因此可辨识不同处理的样品来源。而这些成对离子在质谱图中的信号强度与各自的含量相关,因此可用于样品丰度的定量分析[3-4]。SILAC方法具有样本需求量少、定量结果准确、重复性好等诸多优点,已经成为定量蛋白质组学研究中的金标准[5]。然而在体外培养的细胞难于复制机体复杂的内环境,亦无法反映研究模型的生长发育阶段、生理甚至心理状态等要素,使得基于细胞水平的SILAC定量蛋白组学并不能完整阐释生理状态下生物的生长、发育过程和疾病的发生发展过程的蛋白质功能调控及其规律[6]。因此,基于动物水平SILAC标记技术的发展极为重要。
小鼠的生理生化和发育过程与人类相似,并且基因组和人类 90%同源,所以人类疾病的小鼠模型可真实模拟人类疾病的发病过程[7-8]。但由于技术限制,目前国内尚无整体小鼠SILAC标记研究的报道,而国外SILAC小鼠购买价格昂贵,动物源性材料引进的风险等诸多因素和限制,使得我国蛋白质组学科学家难于开展SILAC小鼠的定量蛋白质组学研究发展。本研究旨在通过评价SILAC小鼠制备方法及其标记的心、肝、脾、肺和肾等脏器的标记效率,研究整体小鼠标记过程中蛋白质组水平的标记特性,并通过构建接近完全标记的SILAC小鼠,为肿瘤、心血管、代谢疾病以及老年病等定量蛋白组学研究提供重稳定性同位素标记的蛋白质组学内标样品,促进我国相关疾病发病机制的系统而深入的研究。
1 材料与方法
1.1 材料
1.1.1 小鼠品系基本情况
SPF级C57BL/6J品系雌性小鼠4只,4周龄,12−14 g;雄性小鼠 2只,4周龄,13−15 g,由中国人民解放军军事医学科学院实验动物中心提供,许可证号 SCXK-(军)2012-0004,并按实验动物使用的3R原则给予人道关怀。
1.1.2 小鼠SILAC鼠粮
含有 1%13C6-Lysine (纯度≥98%)重标鼠粮购自于Silantes公司 (Munich,German网址:http://www.silantes.com/)[9-10]。鼠粮在 4 ℃保存。
1.1.3 主要试剂与仪器
色谱级乙腈、甲醇、十二烷基硫酸钠(SDS)、甲叉双丙烯酰胺和丙烯酰胺、碳酸氢铵(NH4HCO3)均购自 Sigma-Aldrich公司;质谱级蛋白质酶 Lys-C (Wako公司);二硫苏糖醇(DTT)和碘乙酰胺 (IAA)为 Amresco公司产品;蛋白酶抑制剂 Cocktail购自 Roche公司;超声破碎仪为宁波新芝生物科技有限公司JY98-IIIDN型号的产品;高精度质谱仪 LTQ Orbitrap Velos为Thermo Fisher Scientific公司产品;超高压纳升级高效液相色谱仪 (Ultra-high Performance Liquid Chromatography)为Waters公司产品。
1.2 方法
1.2.1 小鼠饲养方法
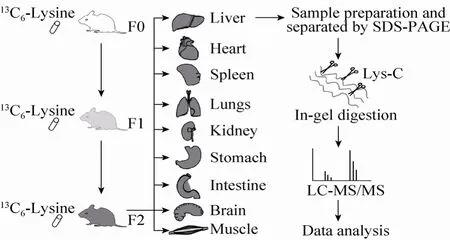
图1 SILAC小鼠代谢标记以及标记效率检测流程图Fig. 1 Overview of the SILAC labeling and labeling efficiency examination workflow.
小鼠于屏障级设施内饲养,二级动物饮水标准,每周换水 2−3次,雌雄小鼠分别喂食含有 1%13C6-Lysine的重稳定性同位素标记的SILAC鼠粮和SPF级繁殖饲料,每周添料3−4次。如图1所示,饲养至8周龄,将1只成熟期雄鼠与2只喂食SILAC鼠粮的标记小鼠 (记为F0代)合笼,隔日以阴道栓检测有无受孕情况。受孕后及时取出雄性小鼠,隔离。雌性受孕小鼠成功受孕后继续饲喂SILAC鼠粮,直到生产仔鼠,为F1代。挑选F1代雌性小鼠,继续以SILAC鼠粮喂养至8周龄,与雄鼠交配后生产,为F2代标记小鼠。
1.2.2 小鼠组织分离及蛋白提取
选取5周龄F2代小鼠,经过眼球摘除法取血,降低血清中高丰度蛋白对于后续实验的影响。如图 1所示,眼科剪分离出大脑、肺脏、心脏、胃、小肠、肝脏、脾脏、肾脏和肌肉 (臀大肌)组织,磷酸盐缓冲液 (Phosphate buffer solution,PBS)清洗残留血液,滤纸吸干后置于液氮中速冻,然后放入–80 ℃冰箱冻存。
取上述9种组织各0.3 g,置于盛有液氮的研钵中速冻,均匀研磨成粉状,加入500 μL蛋白质裂解液 (8 mol/L尿素、50 mmol/L NH4HCO3、蛋白酶抑制剂和5 mmol/L IAA),冰浴条件下进行超声破碎细胞,功率 300 W,工作1 s,间歇5 s,60个循环。细胞破碎后4 ℃低温12 000×g离心10 min,收集上清,分装后置于–80 ℃贮存备用。
1.2.3 蛋白质组样品制备
样品制备参考Zhai等[11]的方法进行,取分装9种组织全蛋白各10 μL,加入10 mmol/L的DTT,50 ℃恒温水浴 5 min,冰浴冷却后加入IAA (终浓度20 mmol/L)避光反应0.5 h。再加入10 μL 2×SDS上样缓冲液,均匀混合后,采用10% SDS-PAGE电泳,待样品入分离胶1 cm时停止电泳。再经考马斯亮蓝G250染色、脱色后,将胶条均分成1 mm3胶粒,脱色、干燥。为确保每个来自 SILAC样品的肽段可用于定量,我们选用对赖氨酸特异的 Lys-C蛋白酶进行酶解、抽提肽段,蒸干后置于–20℃保存备用。
1.2.4 LC-MS/MS分析
肽段样品用高效液相色谱-串联质谱(LC-MS/MS)进行检测。液相色谱柱采用柱内径为75 μm、长15 cm的C18反相色谱柱,C18填料颗粒大小为1.9 μm,孔径200 Å。将样品用适当体积的1%乙腈和1%甲酸的水溶液溶解后,取3 μL上样。流动相A为2%乙腈和0.1%甲酸的水溶液;流动相B为0.1%甲酸的乙腈溶液。使用60 min的梯度洗脱样品:0−3 min,B相由0%升至3%;3−12 min,B相从3%升至12%;20−55 min,B 相从 12%升至 40%;55−58 min,B相从40%升至80%;58−60 min,B相维持80%,流速0.3 μL/min。洗脱组分经纳升级电喷雾离子源 (NSI)接口喷出,进入 LTQ Orbitrap Velos分析。质谱采用一级质谱数据依赖的二级质谱扫描模式 (Data dependent MS/MS scan)碰撞诱导裂解 (CID)模式碎裂一级离子。一级质谱扫描范围 (300−1 600 m/z),分辨率设置为30 000;自动增益控制 (Automatic gain control,AGC)为 106。依次选取一级信号强度最高的 15个离子进行二级碎裂分析,碰撞归一化能量 (NCE)为35%;AGC为5 000;最大离子注射时间为25 ms,动态排除为30 s[12]。
1.2.5 蛋白质的鉴定和定量分析
使用 SorcererTM-SEQUEST®(version 4.0.4 build, Sage-N Research, Inc.)搜索引擎对质谱产出的数据文件 (.raw)进行蛋白质的数据库搜索,数据库采用从NCBI下载的小鼠蛋白序列数据库 (Version:20110718)。搜库参数采用固定修饰为半胱氨酸的烷基化修饰 (+57.021 46 Da),Lys-C 特异性半酶切,13C6-Lysine修饰(+6.020 13 Da);可变修饰为甲硫氨酸氧化修饰(+15.994 92 Da);母离子质量误差20 ppm,子离子质量误差 0.5 Da,允许最大漏切位点数目为2个,肽段长度≥6个氨基酸,肽段最大修饰数目为4种。搜库结果采用Target-decoy策略进行过滤,并设定肽段和蛋白质鉴定FDR均小于1%。数据的定量使用实验室内部编写软件,基本策略是提取肽段离子的提取离子色谱图(Extracted ion currents, XICs),计算非标记轻标和 SILAC标记的重标肽段对 XICs的信噪比(S/N)比值,进行对数转换,即为轻重离子对的定量值[12]。
1.2.6 标记效率分析及拟合
将蛋白质定量值 (Log2Light/Heavy)采取Gauss方程拟合数据分布,正态分布平均值的95%置信区间表示标记效率的范围[13]。SPSS 19.0软件进行统计学分析,以P<0.05为差异具有统计学意义。
标记效率计算公式:
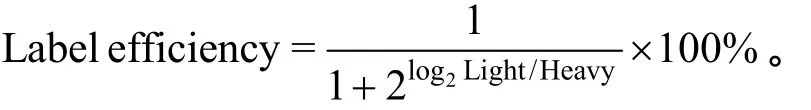
2 结果与分析
2.1 SILAC鼠粮和普通鼠粮饲养小鼠的生长情况比较
喂食SILAC鼠粮的小鼠与SPF级维持鼠粮的对照组小鼠外观上无明显区别。同样表现为体毛光滑,食欲旺盛,活动有力,反应敏捷,粪便正常呈黑色麦粒状。F0代小鼠生长至7周龄时,喂食SILAC小鼠的平均体重为17.55 g,对照组小鼠的体重为17.60 g;标记小鼠培养至F2代,7周龄时体重为15.20 g,小鼠体重有轻微下降的趋势。小鼠合笼交配发现性成熟期延长。通过记录对照组、F0代和F2代SILAC标记小鼠 6、7、10和 11周龄的体重,结果如图2A所示,进行单因素重复测量方差分析,组间的显著性P=0.110,差异不具有统计学意义,表明SILAC鼠粮饲养小鼠对小鼠生长体重的影响不显著。
2.2 小鼠9种器官组织全蛋白检测结果
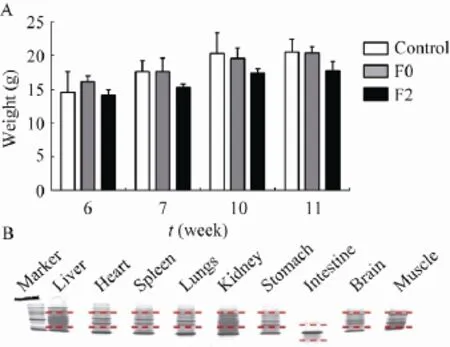
图2 不同鼠粮饲养小鼠体重对比和F2代SILAC不同脏器总蛋白图Fig. 2 Weight variation of control, F0 and F2 generations and separation of proteins extracted from different tissues. (A)Comparison of weight variation of control, F0 and F2 generations at 6, 7, 10 and 11 weeks by repeated measure ANOVA analysis. Every generation has two repeats. (B)The extracted proteins from varied SILAC labeled tissues of F2 generation mouse were separated by SDS-PAGE and stained with Coomassie blue. The gel areas cut and followed by destaining and in-gel digestion were indicated by square marking.
将从9种不同组织提取的全蛋白各取10 μL,用10%的SDS-PAGE电泳检测,经考马斯亮蓝G250染色和脱色。结果如图2B所示,蛋白条带清晰没有弥散现象,说明液氮碾磨裂解和尿素裂解液能较好地防止组织蛋白降解。对比胶图上蛋白条带的深浅,我们发现相同湿重的器官组织,如肝脏、肾脏和肌肉组织等实质性器官的蛋白含量较高;而心脏、脾脏、肺和脑组织等蛋白质的量较低,我们分析认为是由于这些器官含有较多空腔,造成蛋白的提取效率较低;同样,由于胃和肠道组织因含有部分食物残渣,也会造成提取的蛋白质较少。
2.3 蛋白质鉴定和定量数目情况
将 9种不同器官样品的蛋白条带从SDS-PAGE胶上切下,进行胶内蛋白酶Lys-C酶消化后,抽提消化后的肽段,用LC-MS/MS进行蛋白质的鉴定和定量。结果如表 1所示,我们共鉴定1 889个蛋白,有定量结果的蛋白数目1 876个,定量蛋白数占鉴定蛋白数的98 %以上,表明我们检测的蛋白质的信号较强。每个组织样品平均鉴定的蛋白质数目为:(531.6±63.3)个,定量为 (521.8±59.3)个。其中在9种组织样品中被共同鉴定和定量的蛋白质有9个,分别是:ATP 合成酶 β 亚基 (ATP synthase β subunit);延长因子 1α (Elongation factor 1 alpha, eEF1A);蛋白质二硫键异构酶 A3 (Protein disulfide isomerase A3,PDIA3);转酮醇酶(Transketolase);葡萄糖 6磷酸异构酶(Glucose-6-phosphate isomerase,GPI);蛋白质二硫键异构酶 (Protein disulfide isomerase,PDI);泛素-60S核糖体蛋白L40 (Ubiquitin-60S ribosomal protein L40);热休克70蛋白8 (Heat shock 70 kDa protein 8,HSPA8)和 β5微管蛋白(Tubulin beta-5)。
2.4 蛋白质代谢标记分析结果
基于代谢标记的SILAC定量策略是根据对蛋白质同一肽段在一级质谱中的轻和重标离子对的强度进行比较,计算的比值就是相对定量的结果[14]。在本研究中所有组织器官共定量的9种蛋白中,对比Genecard中PaxDb数据库,发现HSPA8蛋白在各种小鼠组织中均高表达 (数据库含有的肝脏、心脏、脾脏、肺脏、肾脏和大脑 6种器官组织数据中,表达丰度均为各器官的蛋白丰度Top 5%),是较好反映不同组织间标记效率差异的蛋白。如图3所示,HSPA8中的一个定量肽段 SINPDEAVAYGAAVQAAILS GDK,根据轻重离子对的比例,计算重离子所占总离子的比例,即被13C6-Lysine标记的肽段占总肽段的比例,就可以计算出代谢标记的蛋白标记效率。分析HSPA8的结果显示肝脏标记效率为98.00%,心脏标记效率为93.63%,脾脏标记效率为96.46%,肺标记效率为96.18%,肾脏标记效率为97.88%,胃标记效率为97.02%,肠标记效率为 96.11%,脑组织标记效率为94.52%,肌肉组织标记效率为95.16%。
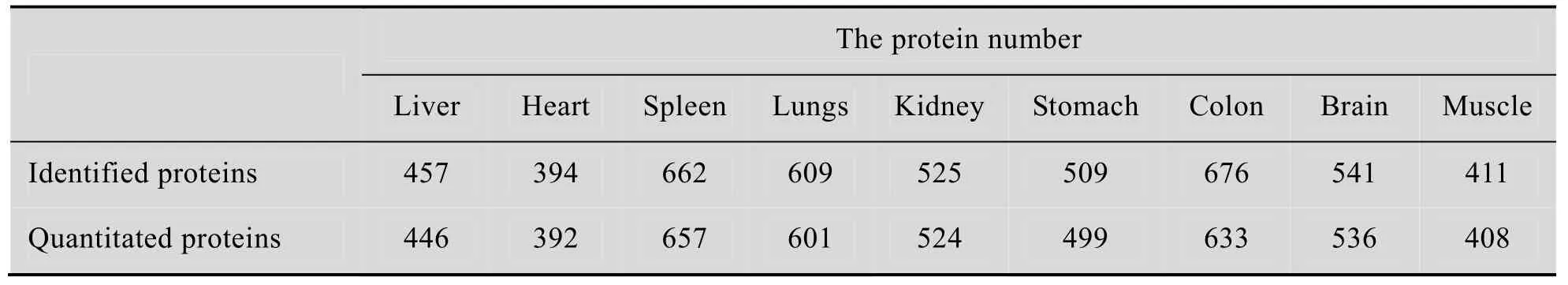
表1 各组织器官鉴定和定量蛋白数量列表Table 1 Protein number of identified and quantified on different tissues
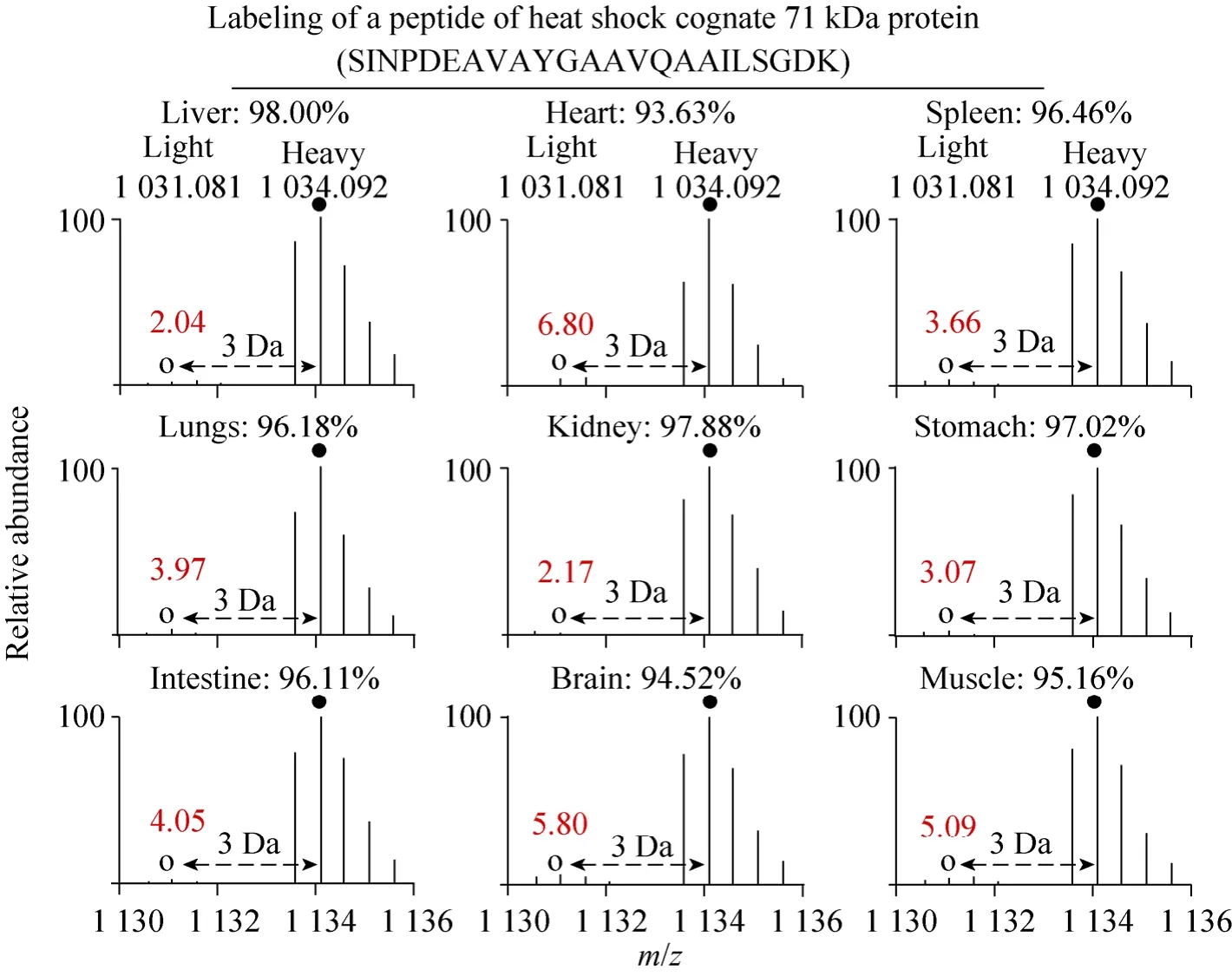
图3 通过计算HSPA8蛋白的特征肽段的轻重离子对的相对峰强度反映不同脏器的标记效率Fig. 3 Analysis of labeling efficiency by comparing the relative peak intensity between the light and heavy ion pairs.Labeling of a representative peptide across different tissues of F2 generation SILAC mouse was selected. Proteins of nine tissues were extracted, in-gel digested, and analyzed by nanoLC-MS/MS to examine labeling efficiency.
进一步对器官组织的所有蛋白的定量信息进行统计学分析可以计算出器官的整体标记效率。为减少因标记导致轻标氨基酸肽段过少或缺失引发的强迫匹配以及由此引起的定量误差,我们对数据进行了过滤,排除了信噪比S/N<10的肽段,并对保留的肽段逐个作了手工检查,最后各组织得到接近200个定量蛋白质。用高斯方程对过滤后蛋白质的定量值数据分布进行正态性拟合,如图 4所示,根据正态分布的均值和标准差,利用标记效率计算公式,计算各种组织的整体标记效率:肝脏为96.34%±0.90%;心脏为94.12%±1.42%;脾脏为95.96%±1.19%;肺脏为95.01%±1.86%;肾脏为95.60%±0.94%;胃脏为 95.71%±1.22%;肠为95.71%±1.98%;大脑为92.62%±1.98%;肌肉为94.16%±1.41%。结果显示标记效率最高的是肝脏 96.34%±0.90%,标记效率最低的是心脏94.12±1.42%和大脑92.62%±1.98%,而小鼠整体的标记效率为95.80%±0.64%。
德国科学家Krüger等研究显示[15],SILAC定量蛋白组学稳定和准确的定量结果的前提是作为内标的蛋白质组样本的整体标记效率应满足≥95.00%,即在器官/组织中蛋白质含有的Lysine至少有95%经过代谢替换成13C6-Lysine。本研究中,F2代的 SILAC小鼠整体标记效率95.80%±0.64%,完全符合以上标准,因此可以成为后续研究中的定量内标。
我们分析的结果如图5所示,F2代的SILAC小鼠肝脏的标记效率高于整体标记水平,差异具有统计学差异 (P<0.05);而小鼠心脏和大脑组织低于整体标记水平,具有统计学差异 (P<0.05)。因此,F2代SILAC小鼠标记效率具有组织特异性。
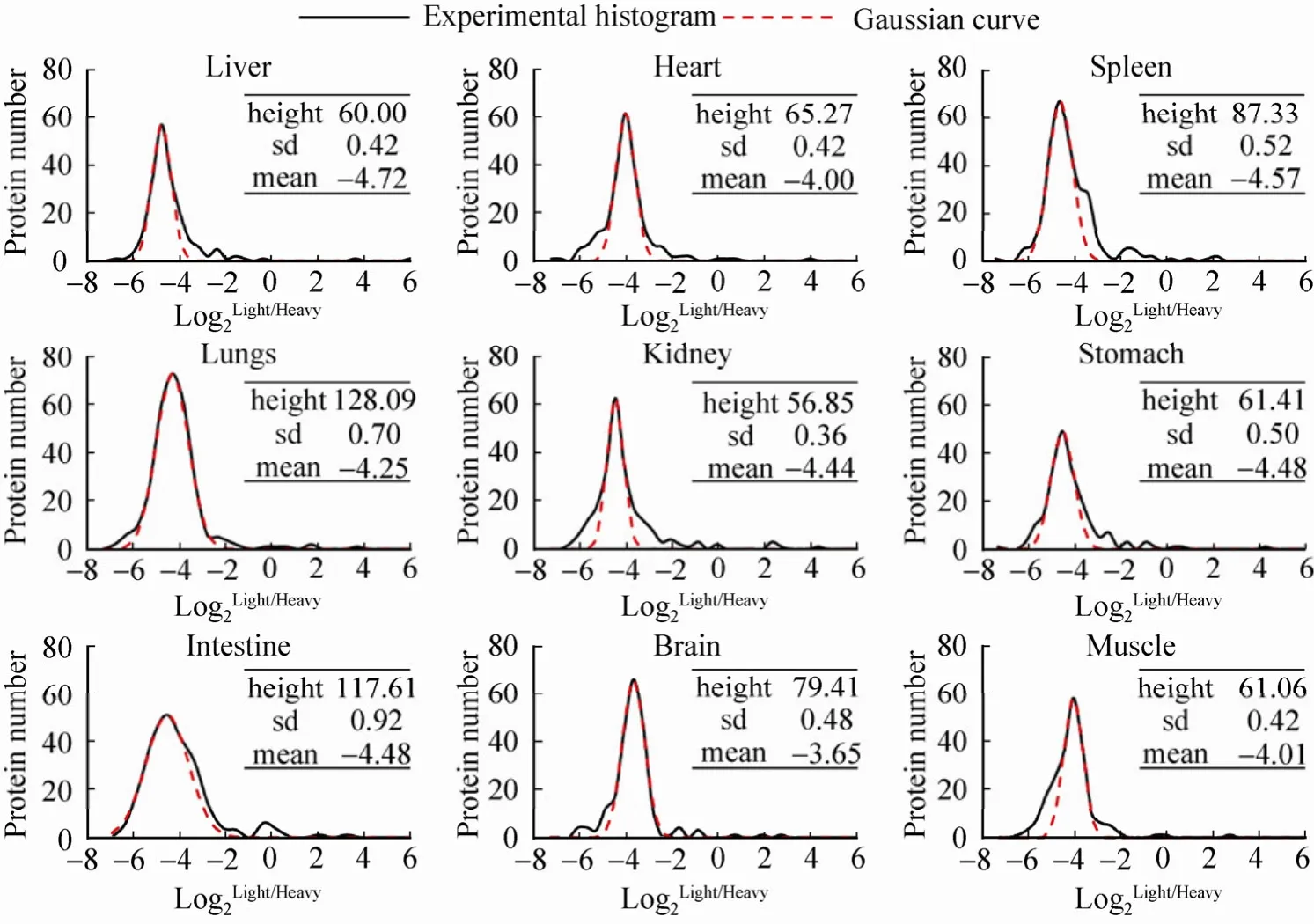
图4 各组织器官定量蛋白的标记效率定量值的高斯拟合图Fig. 4 Histograms of log2Light/Heavy using the quantified proteins from different tissues. Labeling efficiencies were analyzed using the population of proteins quantified by the log-transformed ratio of heavy versus light peptides representing protein labeling efficiency.
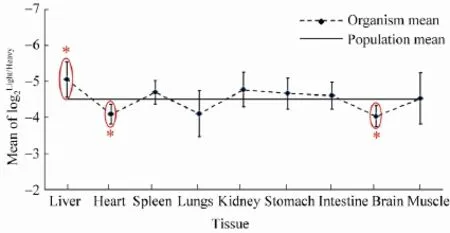
图5 各组织器官整体标记效率Fig. 5 Comparison of SILAC labeling efficiencies in different tissues. The labeling efficiency of liver was higher than whole labeling efficiency of mouse, and the labeling efficiencies of heart and brain were lower. The differences were statistically significant (P <0.05).
3 讨论
目前基于稳定同位素代谢标记的SILAC定量蛋白质组学技术已经成为了蛋白质组学发展的重要方向。SILAC定量方法在不同样品的细胞或者蛋白质层面进行混样,使得后续的生物学处理组和重标对照组样品的处理过程完全保持一致,可有效排除后续样品的纯化、预分离、蛋白质消化、肽段抽提等一系列蛋白质组学样品制备过程各步骤可能引入的误差,有效提高定量蛋白质组学研究的准确性[3]。
由于标记技术的限制和标记成本的昂贵,SILAC技术主要应用在微生物以及细胞等的定量研究中,难以拓展到对动物模型的代谢标记。在诸多的模式生物中,小鼠具有与人类高度同源等优点,是当前研究中的重要动物模型。稳定同位素代谢标记的模式小鼠能为各种疾病小鼠模型、基因敲除小鼠模型和微生物感染小鼠模型的研究提供定量内标。这为研究基因或蛋白质的功能、代谢或信号通路提供了强有力的技术支撑[16-18]。
本研究在国内首次发展了用于小鼠模型的SILAC标记方法,利用含有重稳定性同位素标记赖氨酸 (13C6-Lysine)的 SILAC鼠粮饲养C57BL/6J小鼠,结合LC-MS/MS对不同器官组织进行了定量标记分析,证明F2代小鼠的整体标记效率达到 95%以上。由于不完全标记时作为定量内标的SILAC重标小鼠中掺杂有未被代谢标记的轻标蛋白,在与需要比较的样品混合后,残留的轻标蛋白与样品蛋白难以区分,会对依据轻重标记的离子对定量的结果带来干扰,难以实现高精度的准确定量比较。因此基于目前定量蛋白组学研究中内标应用的标准,95%的标记效率是代谢标记的基本要求[15]。通过不断地方法改进提高标记效率是值得继续探索的课题。然而标记效率不能无限提升至100%,因为标记效率一方面受到稳定同位素的纯度的限制 (目前使用的13C6-Lysine的纯度为98.00%);另一方面跟器官/组织细胞的代谢效率和增殖速度有关。
不同的器官组织存在着不同的标记效率。肝脏是机体代谢的核心器官,拥有肝动脉和门静脉双重血液供应。肠道吸收的13C6-Lysine经过门静脉进入肝脏为其提供了充足的标记物质。肝脏细胞是可再生细胞,这使得肝脏具有良好的自我增殖能力,在应对物质损伤凋亡后能迅速完成修补,因此肝脏的蛋白质更新较快,整体代谢标记效果好。大脑组织作为神经系统与心脏组织的心肌细胞相似,均是终末分化细胞,因此这两个组织的标记效率低于整体水平。这些与 Zanivan等[17]报道结果一致。在Mann等[14]关于小鼠蛋白质代谢动力学的研究中,以MRPL12蛋白 (Mitochondrial 39S ribosomal protein L12)为例,肝脏更新速率为0.205/d,心脏更新速率为 0.065/d。标记一半的MRPL12,肝脏只需要3.4 d,而心脏需要10.6 d。对肝脏和心脏定量的242种蛋白进行分析,肝脏的蛋白质平均更新速率为 0.16/d,心脏的蛋白质平均更新速率为 0.04/d[19]。肝脏蛋白质更新速率明显高于心脏,这使得在相同时间内,肝脏更容易被SILAC标记,而心脏标记效率较低。通过延长饲养时间或者对其子代小鼠进行标记可以提高大脑和心脏等不易于代谢标记组织器官的完全标记。
在研究中,课题组成功地将SILAC方法从微生物和细胞系水平拓展到哺乳动物模型,在国内首先建立了用于各种动物模型研究的蛋白质组学定量内标[11-13-20]。SILAC小鼠能用于各种组织器官的定量蛋白组学或比较蛋白组学研究领域,将研究水平从培养的细胞层面,提升到整体动物的组织/器官层面,并且能够研究某种干预对于机体的不同器官的影响,阐释不同器官间的内在联系。这为定量蛋白组学研究疾病发生、发展机制提供了有力工具。
致 谢:感谢张瑶在英文修改中提供的支持。
[1]Rappsilber J, Mann M. Is mass spectrometry ready for proteome-wide protein expression analysis Genome Biol, 2002, 3(8): comment2008.1–2008.5.
[2]Cox J, Mann M. Is proteomics the new genomics?Cell, 2007, 130(3): 395–398.
[3]Ong SE, Mann M. Mass spectrometry-based proteomics turns quantitative. Nat Chem Biol,2005, 1(5): 252–262.
[4]Robles MS, Cox J, Mann M. In-vivo quantitative proteomics reveals a key contribution of post-transcriptional mechanisms to thecircadian regulation of liver metabolism. PLoS Genet, 2014,10(1): e1004047.
[5]Mann M. Proteomics for biomedicine: a half-completed journey. EMBO Mol Med 2012,4(2): 75–77.
[6]Geiger T, Velic A, Macek B, et al. Initial quantitative proteomic map of 28 mouse tissues using the SILAC mouse. Mol Cell Proteomics,2013, 12(6): 1709–1722.
[7]Rossant J, Nagy A. Genome engineering: the new mouse genetics. Nat Med, 1995, 1(6): 592–594.
[8]Almodovar AJO, Luther RJ, Stonebrook CL, et al.Genomic structure and genetic drift in C57BL/6 congenic metabolic mutant mice. Mol Genet Metab, 2013, 110(3): 396–400.
[9]Zanivan S, Krueger M, Mann M. In vivo quantitative proteomics: the SILAC mouse.Methods Mol Biol. Humana Press, 2012: 435–450.
[10]Krüger M, Moser M, Ussar S, et al. SILAC mouse for quantitative proteomics uncovers kindlin-3 as an essential factor for red blood cell function. Cell,2008, 134(2): 353–364.
[11]Zhai L, Chang C, Li N, et al. Systematic research on the pre‐treatment of peptides for quantitative proteomics using a C18microcolumn. Proteomics,2013, 13(15): 2229–2237.
[12]Xu P, Duong DM, Seyfried NT, et al. Quantitative proteomics reveals the function of unconventional ubiquitin chains in proteasomal degradation. Cell,2009, 137(1): 133–145.
[13]Xu P, Tan H, Duong DM, et al. Stable isotope labeling with amino acids in Drosophila for quantifying proteins and modifications. J Proteome Res, 2012, 11(9): 4403–4412.
[14]Mann M. Functional and quantitative proteomics using SILAC. Nat Rev Mol Cell Biol, 2006, 7(12):952–958.
[15]Krüger M, Moser M, Ussar S, et al. SILAC mouse for quantitative proteomics uncovers kindlin-3 as an essential factor for red blood cell function. Cell,2008, 134(2): 353–364.
[16]Walther DM, Mann M. Accurate quantification of more than 4000 mouse tissue proteins reveals minimal proteome changes during aging. Mol Cell Proteomics, 2011, 10(2): M110. 004523.
[17]Zanivan S, Meves A, Behrendt K, et al. In vivo SILAC-based proteomics reveals phosphoproteome changes during mouse skin carcinogenesis. Cell Rep, 2013, 3(2): 552–566.
[18]Ostasiewicz P, Zielinska DF, Mann M, et al.Proteome, phosphoproteome, and N-glycoproteome are quantitatively preserved in formalin-fixed paraffin-embedded tissue and analyzable by high-resolution mass spectrometry. J Proteome Res, 2010, 9(7): 3688–3700.
[19]Kim TY, Wang D, Kim AK, et al. Metabolic labeling reveals proteome dynamics of mouse mitochondria. Mol Cell Proteomics, 2012, 11(12):1586–1594.
[20]Ping L, Zhang H, Zhai L, et al. Quantitative proteomics reveals significant changes in cell shape and an energy shift after IPTG induction via an optimized SILAC approach for Escherichia coli.J Proteome Res, 2013, 12(12): 5978–5988.
