云南HPV-16型E6、E7基因的突变分析
2014-10-25张晓琳姚月婷李亚东周竞贤姚宇峰俞建昆
张晓琳,刘 峰,姚月婷,李亚东,周竞贤,易 力,姚宇峰,俞建昆*
(1.中国医学科学院&北京协和医学院医学生物学研究所,云南 昆明 650118;2.云南省重大传染病疫苗研发重点实验室,云南 昆明 650118;3.武警云南省总队医院检验科,云南 昆明 650111)
Harald zur Hausen因发现人乳头状瘤病毒(human papillomavirus)为子宫颈癌的成因于2008年获得了诺贝尔奖。1985年 Seedorf等发表了HPV-16型的全基因组序列[1],其NCBI登录号为NC_001526.2。HPV病毒在99.7%宫颈癌患者中都可检测到[2],目前经过对HPV的基因组分离测序已经发现了100多个型[3]。根据HPV不同型致癌危险程度不同,可将HPV分为低危型和高危型2大类,HPV-16型属于高危型,在ICC(invasive cervical carcinoma)和CIN(cervical intraepithelial neoplasia)I-III患者中经常被检测到[4]。E6和E7是其2个致癌蛋白,可以诱导感染病毒的细胞转化成浸润癌。已有的研究结果显示HPV-16型E6和E7基因的序列突变有地域性差别[5],根据E6基因片段的DNA序列可以把HPV-16型分成6个变异亚型:E欧洲型、As亚洲型、AA亚美型、Af1(非洲-1 型)、Af2(非洲-2型)和 NA 北美型[6]。本研究收集了来自中国云南不同地区的HPV-16型病例样本74例,通过对HPV-16型的2个致癌基因E6和E7测序分析,研究该地区E6和E7基因的变异情况和HPV-16型的变异亚型,为研究适合中国人群的宫颈癌疫苗积累流行病学方面的相关资料。
1 材料与方法
1.1 标本来源和HPV分型检测
在知情同意的原则下,本研究随机选取2012年3月至2013年3月来自昆明、曲靖、昭通、红河等地区市县级医院门诊、体检及住院女性。按照上海透景HPV分型试剂盒(26型)使用说明书的要求,用配套宫颈细胞刷采集宫颈上皮细胞后漂洗到细胞保存液中,然后按照试剂盒说明书操作进行DNA制备和分型检测。
1.2 测序
从检测为HPV-16的样本中选取80例进行E6和E7基因的DNA序列检测。扩增HPV-16型E6和E7基因的引物分别为:E6F(nt52~575):5'-CGAAACCGGTTAGTATAA-3',E6R:5'-GTATCT CCATGCATGATT-3';E7F(nt480~985):5'-ATAATATAAGGGGTCGGTGG-3';E7R:5'-CATTTTCGT TCTCGTCATCTG-3'[7]。PCR 反应体积为 25 μL,其中宫颈组织 DNA 样品2 μL(25 ng/μL),TaKa-Ra Ex Taq DNA聚合酶(TaKaRa公司)0.625 U,反应体系参照说明书。PCR循环条件:94℃预变性3 min,35个循环(94 ℃ 30 s,55 ℃ 30 s,72 ℃45 s),72℃延伸5 min。PCR产物条带在2%琼脂糖凝胶电泳上检测观察。阳性结果的样品送上海生工测序。对于发现突变的样本再次用PCR扩增目的片段并测序验证突变。最终有6例样本没有得到测序结果。
1.3 数据分析方法构建系统发生树
采用 Mega5.1的 Neighbor-joining方法对74个样本的E6基因的456 bp(nt104~nt559)DNA片段序列和E7基因的297 bp(nt562~nt858)DNA片段序列分别构建系统发生树(靴值法(Bootstrap method)检验系统发生树,重复数为1000)。HPV-16型全基因组的参比序列及登录号如下[8]:①欧洲变异型:德国变异型(European German):AF_536179;②亚洲变异型:东亚变异型(East Asian):AF_534061,中国甘肃(Gansu):EU_918764,中国新疆(Xinjiang):FJ_006723,泰国(Thailand):FJ_610152;③亚洲美洲变异型:(Asian-American):AF_402678,南美洲亚马逊(Amazonian):HM_057182;④非洲1型变异型:(African type1):AF_472508;⑤非洲2型变异型:(African type2):AF_472509;⑥北美变异型:(North-American):U34116,由于其只含有 E6 基因序列[9],故用E7DNA序列构建的系统发生树不含此变异型。
2 结果与分析
2.1 E6 DNA序列的亚型分类及碱基突变分析
2.1.1 依据E6的DNA序列的亚型分类 采用Mega5.1软件Neighbor-joining方法对74例HPV-16 型样本的 E6的456 bp(nt104~nt559)[10]DNA片段构建进化树。结果显示只有亚洲变异型(49例,66.22%)和欧洲变异型(25 例,33.78%),没有发现非洲1型、非洲2型、亚-美型和北美变异型(图1)。
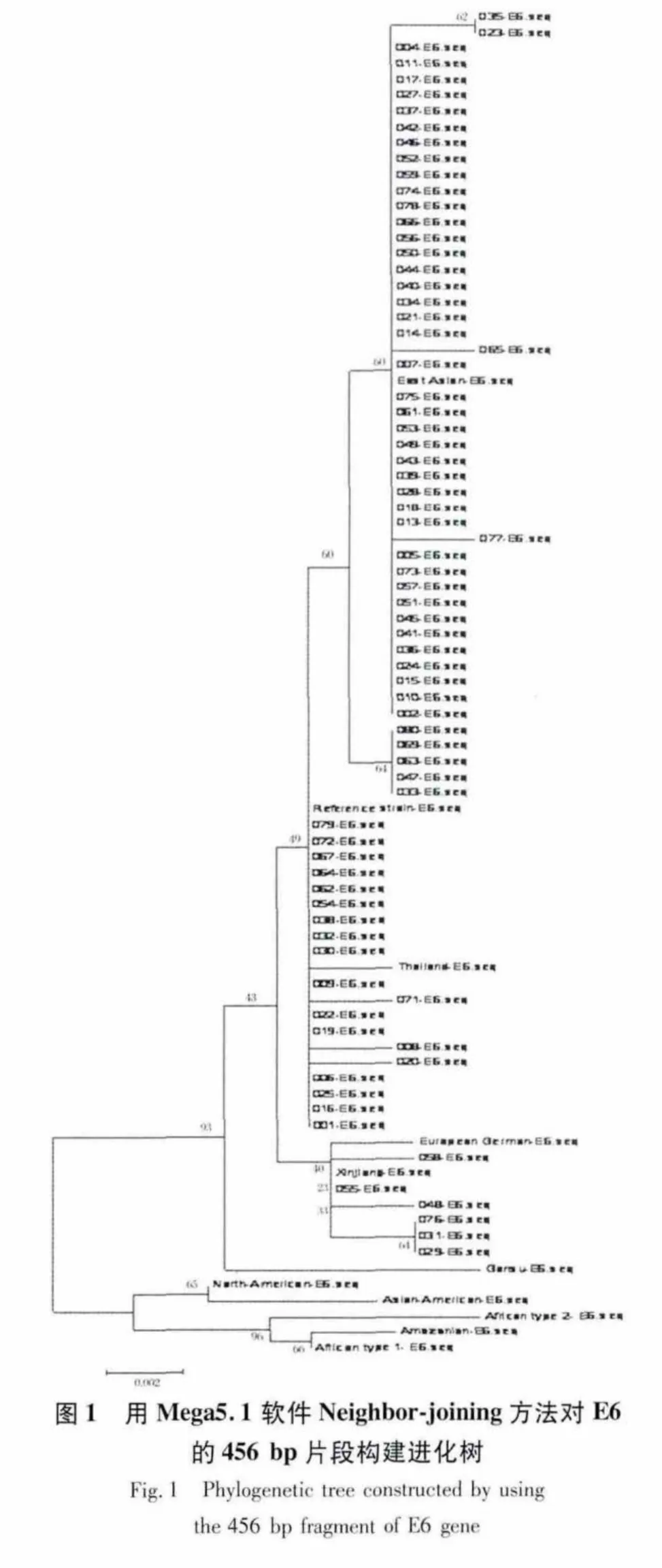
亚洲变异型在178位碱基处含有突变,含有T178G为As-a亚型,含有T178A为As-b亚型,含有C178A为As-c亚型[11]。本研究发现44例Asa亚型,5例As-b亚型,共49例亚洲突变型。欧洲变异型可根据350位点处的突变情况分成ET350亚类、E-G350亚类和E-A350T亚类3个亚类[6];本研究发现有6例E-T350G亚类。
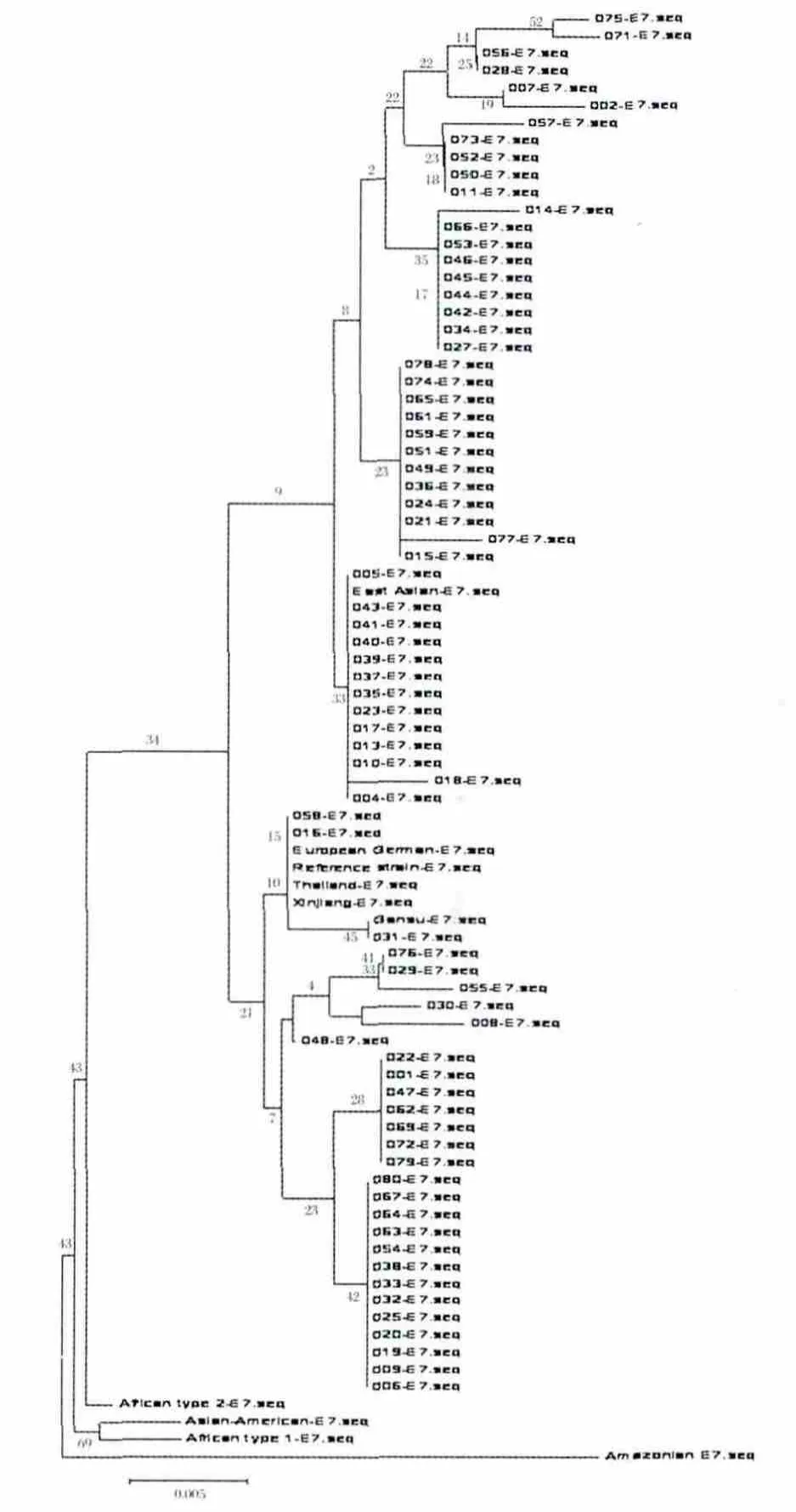
图2 用Mega5.1软件Neighbor-joining方法对E7的297 bp片段构建进化树Fig.2 Phylogenetic tree constructed by using the 297 bp fragment of E7 gene
2.1.2 E6碱基突变分析 对74例样本的E6基因突变分析后共检测到14个碱基位点发生了15种碱基突变,导致了10种氨基酸突变。E6的颠换突变(76.19%)远大于转换突变(23.81%),且错义突变的比重较大(89.29%)(表1)。其中碱基突变概率最高的是T178G(44例,59.46%),导致了44例D25E氨基酸突变,该位点还有5例T178A碱基突变,也导致了D25E氨基酸突变,其突变率最高为 49例,占 66.22%;另外,1例G176A碱基突变导致了1例D25N突变。突变率较高的有:A276G(9例,12.16%)碱基突变导致了N58S氨基酸突变;T350G(6例,8.11%)碱基突变导致了L83V氨基酸突变。导致了氨基酸性质发生改变的突变共有2例:碱基位置Q3E和碱基位置D25N(表2)。
2.2 E7 DNA序列的亚型分类及碱基突变分析
2.2.1 依据E7的DNA序列的亚型分类 采用Mega5.1软件Neighbor-joining方法对74例HPV-16型样本的E7的297 bp(nt562~nt858)片段构建进化树,结果显示只有亚洲变异型(45例,60.81%)和欧洲变异型(29 例,39.19%),没有发现非洲1型、非洲2型、亚-美型和北美变异型(图2);这与E6进化树的结果一致。
2.2.2 E7碱基突变分析 对E7基因突变分析共检测到13个碱基位点发生了13种突变,导致了8种氨基酸突变。E7的转换突变(96.72%)远大于颠换突变(3.28%),且同义突变(50.27%)与错义突变(49.73%)概率基本相同(表1)。碱基突变率最高的是T846C(45例,60.81%),导致了同义突变;其次是 A647G(44例,59.46%),导致了N29S氨基酸突变。另外,有4例样本发生A646C碱基突变,导致了 N29H氨基酸突变;C749T(36例,48.65%)碱基突变导致了S63F氨基酸突变。1例样本有T748G碱基突变,导致了S63A氨基酸突变发生。G666A(21例,28.38%)碱基突变导致了同义突变。此外,1例样本同时发生C625T和C627T碱基突变,导致了1例L22F氨基酸突变。氨基酸性质发生改变的突变有(共46例):N29H(4例)、G43E(1例)、P47S(1例)、S63F(36例)、S63A(1例)和 R71C(3例)(表3)。
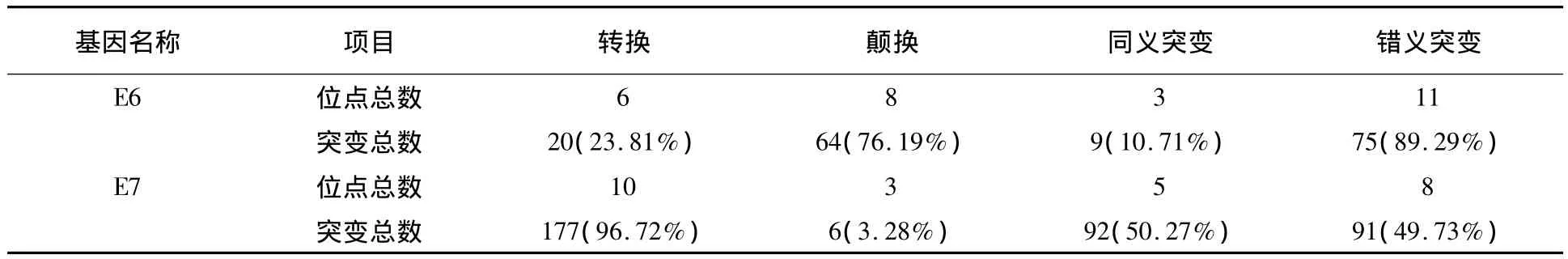
表1 E6、E7突变类型分析Table 1 The types of E6 and E7 mutations
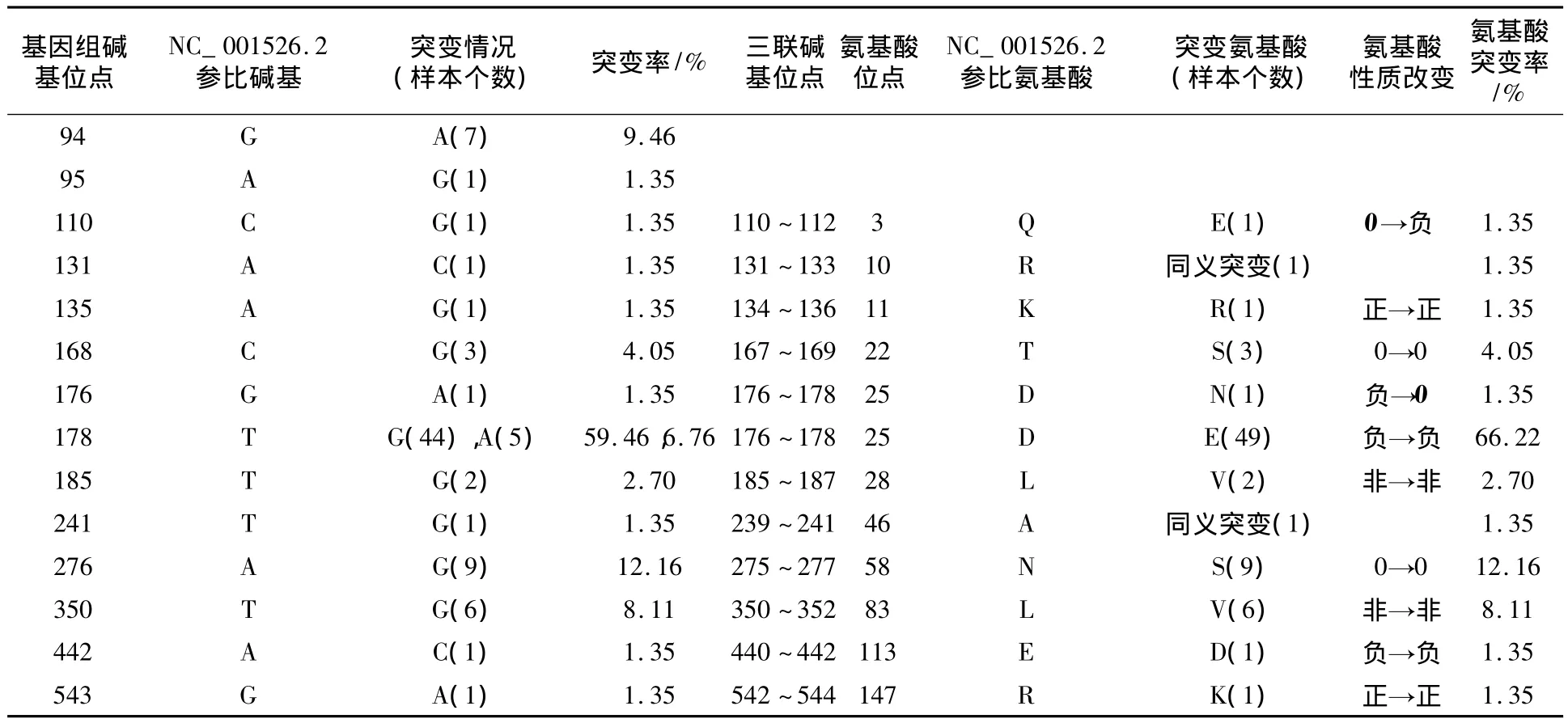
表2 E6突变情况汇总表(n=74)Table 2 The Mutations of E6 Gene(n=74)

表3 E7突变情况汇总表(n=74)Table 3 The Mutations of E7 Gene(n=74)
2.3 参考株、单位点和多位点突变的分析
对74例样本E6和E7基因的DNA序列分析发现与参考株序列相同的病毒株都较少,E6和E7分别为6例占8.1%和2例占2.7%。E6单位点突变(53例,71.6%)超过半数,多位点突变(15例,20.3%)较少;E7的情况则相反,E7单位点突变较少(15例,20.3%),多位点突变(57例,77%)超过半数。对多位点突变的分析发现:74例样本中,44例样本同时含有T178G(共44例)、A647G(共44例)、T846C突变(共45例),只有1例样本只含有T846C突变例外,并且所有含有T843C突变的样本(共16例)都含有 T178G、A647G、T846C突变。此外,所有含有C627T突变(共9例)的样本都含有C749T突变。
3 讨论
本研究以 NCBI登录号为 NC_001526.2的HPV-16型病毒序列为参照,发现E6的野生型(8.1%)和 E7的野生型(2.7%)都较少,这与对泰国31个样本分析发现E6的野生型(13%)较少[5]的结果一致。在检测的74例样本中发现只有亚洲变异型和欧洲变异亚型,未发现非洲1、非洲2、亚-美和北美变异亚型,这与从新疆南部[12]和北京人群中得到的病毒样本[13]的结果一致。此外,本文中发现的突变率较高的G666A(21例,28.38%)碱基突变在东北突变率为38.46%,T846C(45例,60.81%)碱基突变的突变率在北京[13]为45.16%,这提示在中国人群中的HPV-16型上这些位点的突变率都较高。
对于E6癌基因,本研究以HPV基因组碱基104位处的ATG为起始密码子,其共计编码151个氨基酸[14],可分为 E6基因 N端(1~79氨基酸)和E6基因C端(80~151氨基酸)两部分。Q3E、K11R、T22S、D25E、25N 和 L28V 这几种突变发生在E6的N端,且Q3E和D25N氨基酸突变改变了氨基酸的性质,这可能影响到E6*I(编码41个氨基酸)的功能,E6*I是E6的一种剪接异构体可以抑制E6的作用[15]。R147K突变发生在E6的C端,位于保守序列143~151内,该保守序列SRTRRETQL能与PDZ结构域(最早发现有此结构域的PSD95、Dlg1和Zo-1三个蛋白的首字母缩写)相结合[16-17],该突变可能会影响E6蛋白与PDZ结构域相结合,由于E6蛋白可以通过蛋白酶体,将含有PDZ结构域的蛋白降解,从而影响恶性肿瘤的发展进程[18-19]。E6有4个Cysx-x-Cys模块(分别在 30~33、63~66、103~106和136~139氨基酸位置上),即潜在的锌指结构,可以和Zn2+结合[20]。虽然本研究没有在锌指结构处发生突变,但是在锌指结构的链接处发现有N58S、L83V、E113D这3处氨基酸突变。
E7蛋白可分为1区(1~15位氨基酸)、2区(16~37位氨基酸)和比较保守的3区(38~98位氨基酸)3个区域。L22F突变位于E7蛋白2区的LXCXE模序(22~26位氨基酸)内,由于这一模块可以和pRB(Retinoblastoma Protein)第649~772位氨基酸的口袋结构结合[21-22],从而抑制pRB的作用,因此推测可能会影响E7与pRB的相互作用,进而影响细胞周期的调控。而L22F、N29S、N29H突变位于21~34氨基酸处,而该处恰是E7的主要抗原表位所在[23],可能会影响E7蛋白的免疫原性。G43E、P47S、S63F、S63A、R77C突变都发生了氨基酸性质的改变,且位于E7蛋白的比较保守的3区(38~98氨基酸),这些突变也可能会影响E7的功能。3区含有2个Cys-XX-Cys模块(58氨基酸处和91氨基酸处)即锌指结构,可与 Zn2+结合[24-25],在锌指结构的链接处发现S63A、S63F、R77C氨基酸突变,并且这3个氨基酸突变都改变了氨基酸的性质,这有可能会影响E7与Zn2+结合。
本研究发现很多单个病例的共同突变现象,如44例样本同时含有 T178G、A647G、T846C突变,这个结果与熊光武等[13]报道的 A647G和T846C这2种突变都同时存在于同一个样本中一致。Vaeteewoottacharn.K.发现 E6中 90% 的D25E(176~178 nt)突变与E7的N29S(646~648 nt)一同出现[5]。根据这些共突变情况,可以从已知的某个突变,来预测其他有共突变关系的碱基突变的发生。此外,日本的研究结果显示HPV-16型E6基因的D25E突变和人类白细胞抗原HLA II的DRB1*1502等位基因相关,由于HLA II是外源性抗原的递呈分子,因此推测该突变还可能是引发侵润癌和宫颈上皮内瘤的发生最重要的突变[26]。在荷兰,有 L83V突变的癌症病人 HLA DRB1*07 频率也增高(P=0.08)[27],在瑞典、意大利、捷克也发现单倍体型DR04-DQ03与L83V突变相关[28]。因此,推测该突变可能导致癌变的概率增加。所以对HPV-16型病毒的E6、E7基因突变情况的分析显得尤为重要,分析突变热点区域和保守区域可为研究针对中国人群的宫颈癌疫苗提供一定的线索。
[1]Seedorf K,Krammer G,Durst M,et al.Human papillomavirus type 16 DNA sequence[J].Virology,1985,145(1):181-185.
[2]Walboomers J M M,Jacobs M V,Manos M M,et al.Human papillomavirus is a necessary cause of invasive cervical cancer worldwide[J].Journal of Pathology,1999,189(1):12-19.
[3]Bernard H U.The clinical importance of the nomenclature,evolution and taxonomy of human papillomaviruses[J].Journal of Clinical Virology,2005,32:1-6.
[4]Zehbe I,Wilander E,Delius H,et al.Human papillomavirus 16 E6 variants are more prevalent in invasive cervical carcinoma than the prototype[J].Cancer Research,1998,58:829-833.
[5]Vaeteewoottacharn K,Jearanaikoon P,Ponglikitmongkol M.Co-mutation of HPV16 E6 and E7 genes in Thai squamous cervical carcinomas[J].Anticancer Research,2003,23:1927-1932.
[6]Yamada T,Manos M M,Peto J,et al.Human papillomavirus type 16 sequence variation in cervical cancers:a Worldwide Perspective[J].Journal of Virology,1997,71(3):2463-2472.
[7]Zehbe I,Tachezy R,Mytilineos J,et al.Human papillomavirus 16 E6 polymorphisms in cervical lesions from different European populations and their correlation with human leukocyte antigen class II haplotypes[J].International Journal of Cancer,2001,94(5):711-716.
[8]杨丽娟,岳耀斐,陈俊英,等.云南省人乳头状瘤病毒16型E6/E7基因的变异分析[J].病毒学报,2012,28(6):645-651.
[9]Chaudhary D K,Mani I,Singh V.Comparative structural and functional analysis of E6 oncogene of human papillomavirus type 16[J].International Journal of Applied Biology and Pharmaceutical Technology,2012,3(4):439-450.
[10]Cornet I,Gheit T,Franceschi S,et al.Human Papillomavirus type 16 genetic variants:phylogeny and classification based on E6 and LCR[J].Journal of Virology,2012,86(12):6855-6861.
[11]Antonio H S,Diana C.MG,Pablo M,et al.E6 molecular variants of human papillomavirus(HPV)type 16:An updated and unified criterion for clustering and nomenclature[J].Virology,2011,410(1):201-215.
[12]李清阳,玛依努尔·尼亚孜,阿不力孜.新疆南部维吾尔族妇女宫颈癌及癌前病变HPV16 E6/E7基因突变序列分析[J].现代妇产科进展,2009,18(11):851-854.
[13]Xiong G W,Yuan Y,Li M,et al.Human papillomavirus type 16 variant analysis of upstream regulatory region and E6,E7 oncogene from cervical cancer patients in Beijing[J].Hereditas(Beijing),2010,32(4):339-347.
[14]Elliot J A,Nancy L H,John T S,et al.Identification of the HPV-16 E6 protein from transformed mouse cells and human cervical carcinoma cell lines[J].The EMBO Journal,1987,6(4):989-992.
[15]Vaeteewoottacharn K,Chamutpong S,Ponglikitmongkol M,et al.Differential localization of HPV16 E6 splice products with E6-associated protein[J].Virology Journal,2005,2(1):50.
[16]Jaulin-Bastard F,Saito H,Bivic A L,et al.The ERBB2/HER2 receptor differentially interacts with ERBIN and PICK1 PSD-95/DLG/ZO-1 domain proteins[J].Journal of Biological Chemistry,2001,276(18):15256-15263.
[17]Thomas M,Richard L,Karin H,et al.Oncogenic human papillomavirus E6 proteins target the MAGI-2 and MAGI-3 proteins for degradation[J].Oncogene,2002,21:5088-5096.
[18]Massimi P,Gammoh N,Thomas M,et al.HPV E6 specifically targets different cellular pools of its PDZ domain-containing tumour suppressor substrates for proteasome-mediated degradation[J].Oncogene,2004,23(49):8033-8039.
[19]Simonson S J S,Difilippantonio M J,Lambert P F.Two distinct activities contribute to human papillomavirus 16 E6’s oncogenic potential[J].Cancer Research,2005,65(18):8266-8273.
[20]Barbosa M S,Lowy D R,Schiller J T.Papillomavirus polypeptides E6 and E7 are Zinc-binding proteins[J].Journal Of Virology,1989,63(3):1404-1407.
[21]Munger K,Werness B A,Dyson N,et al.Complex formation of human papillomavirus E7 proteins with the retinoblastoma tumor suppressor gene product[J].The EMBO Journal,1989,8(13):4099-4105.
[22]Stirdivant S M,Ahern J D,Oliff A,et al.Retinoblastoma protein binding properties are dependent on 4 cysteine residues in the protein binding pocket[J].The Journal Of Biological Chemistry,1992,267(25):14846-14851.
[23]Steven A J,Yu X P,Janette M V,et al.Characterization of human Antibody-reactive epitopes encoded by human papillomavirus types 16 and 18[J].Journal OF Virology,1991,65(3):1208-1218.
[24]Ohlenschl¨ager O,Seiboth T,Zengerling H,et al.Solution structure of the partially folded high-risk human papilloma virus 45 oncoprotein E7[J].Oncogene,2006,25(44):5953-5959.
[25]William C P,Karl M,Garole L Y.Structure-function analysis of the human papillomavirus type 16 E7 oncoprotein[J].Journal of virology,1992,66(4):2418-2427.
[26]Matsumoto K,Yasugi T,Nakagawa S,et al.Human papillomavirus type 16 E6 variants and HLA class II alleles among Japanese women with cervical cancer[J].International Journal of Cancer,2003,106(6):919-922.
[27]Hetty J B,Mark V D,Tanja D D G.HPV 16 infection and progression of cervical intra-epithelial neoplasia:Analysis of HLA polymorphism and HPV 16 E6 sequence variants[J].International Journal of Cancer,1998,78:166-171.
[28]Ingeborg Z,Ruth T,Joannis M,et al.Human papillomavirus 16 E6 polymorphisms in cervical lesions from different European populations and their correlation with human leukocyte antigen class II haplotypes[J].International Journal of Cancer,2001,94:711-716.
