细菌化学趋向性受体聚集体的相互作用
2014-09-21余大启涂豫海来鲁华
余大启 涂豫海 来鲁华,*
(1北京大学化学与分子工程学院,分子动态与稳态国家重点实验室,北京分子科学国家实验室,北京100871;2北京大学定量生物学中心,北京100871;3IBM T.J.Watson Research Center,Yorktown Heights,NY 10598,USA)
1 引言
细菌可以检测环境中的化学物质从而游向其化学引诱剂和远离其化学排斥物.1这种被称为化学趋向性的现象帮助细菌寻找食物并远离有害物质的伤害.1,2在引诱剂诱导的细菌趋化过程中,细胞膜上的化学趋向性受体通过结合引诱剂分子传导跨膜信号以降低其下游组氨酸激酶CheA的活性.3CheA活性的变化进一步影响到控制马达转动的激酶CheY和去甲基化酶CheB的磷酸化水平,从而改变鞭毛的运动状态以及化学趋向性受体的甲基化状态,最终改变细菌的游动方向并实现对于化学物质的适应性.3,4由于整个控制系统中含有的分子种类比较少,细菌的趋化系统经常被当作刺激-响应二组分调控系统(two component regulatory system)的模型系统进行研究.3,4
根据结合配体的不同,化学趋向性受体分为很多种.大肠杆菌中最主要的有结合丝氨酸(Ser)的受体(Tsr)和结合天冬氨酸(Asp)的受体(Tar).3两个Tsr单体分子或Tar单体分子可以形成稳定的同源二聚体并在界面上提供两个结合口袋以结合引诱剂.3引诱剂结合之后会诱导受体二聚体中第4号跨膜螺旋向下滑动进而传递跨膜信号,5而非引诱剂即使能结合这两个口袋也不能传递跨膜信号.6虽然二聚体通过结合诱导的构象变化已经比较清楚,但是二聚体进一步二聚形成的复杂多聚化结构尚不够清楚.根据Tsr胞质区的X射线晶体结构,7Tsr二聚体的尾部存在进一步三聚的残基-残基相互作用,形成倒金字塔(inverted pyramid)形状的二聚体的三聚体(trimer-of-dimers,TOD),如图1所示.
在CheA和耦合蛋白CheW的帮助下,这些化学趋向性受体在细胞膜上聚集并主要定位在细胞两极,8形成大小不一的受体团簇.这些受体团簇的大小呈指数分布,9较大的团簇能达到含数千个受体的规模.9理论研究表明,在细胞膜上聚集的受体能协同性地影响周围受体的结构从而放大信号.4,10对聚集体结构的研究不仅能丰富目前高度简化的理论模型,也能从分子层次上理解受体构象协同耦合的本质.42003年,Weis等11利用电镜(EM)技术研究了过量表达Tsr然后破碎的大肠杆菌细胞膜提取物.在这些提取物中存在三种类型的细胞膜-受体聚集体形式,即球形胶束状、小型晶体阵列状以及拉链状.分析表明在球形胶束状和小型晶体阵列状聚集体中Tsr都以晶体结构中观察到的TOD为基本结构单元,而拉链状聚集形式则通过Tsr二聚体中复绕的螺旋(coiled-coil helix)尾部相互叠合形成.11,12通过统计多个拉链状聚集体较低分辨率(~2 nm)的图片,得到平均的尾部叠合长度为(6.5±0.6)nm.类似的拉链状聚集形式也存在于Tar、CheA以及CheW的复合物溶液结构中.13不仅如此,冷冻电镜研究结果14也表明,大肠杆菌细胞在过量表达受体Tsr时失去趋化功能且在细胞膜上出现大量的拉链状聚集形式并伴随着细胞膜的内陷变形.有趣的是,同时过量表达Tsr、CheA和CheW的细胞能恢复趋化功能且在细胞膜上恢复正常的聚集形式.考虑到细菌化学趋向性受体胞质区的尾部残基都很保守,11,12这种拉链状聚集被认为在化学趋向性受体聚集中是广泛存在的结构.然而,形成拉链状聚集的分子模型和分子水平的相互作用机制仍然缺乏,不同聚集形式之间的转换过程和机制也完全不清楚.本文通过蛋白质-蛋白质对接方法15研究两个Tsr二聚体可能形成的复合物结构,发现形成倒金字塔形TOD和拉链状聚集的基本作用单元,确定拉链状复合物的结构模型,通过分子动力学(MD)模拟的方法确定其稳定性,并进一步讨论复合物界面相互作用特征.
2 计算方法
2.1 化学趋向性受体二聚体的分子对接
对接计算所用结构来源于大肠杆菌Tsr的胞质区X射线晶体结构(图1,PDB代码:1QU77),该结构具有三重对称性,每一个不对称单位中包含1个二聚体,由两个分别具有227和221残基长度的单体组成.对Tsr二聚体取coiled-coil螺旋尾部N322-T453共264个残基为受体二聚体的结构(图2).这264个残基组成的coiled-coil螺旋长度大约为9.3 nm,比EM实验11,12中观察到的拉链状聚集的尾部叠合长度(6.5 nm)要长,当然也远长于晶体结构中TOD重叠的长度.将该二聚体作为对接的受体和配体,使用ZDOCK 3.0程序16进行对接,计算采用缺省参数,即平动自由度由快速傅里叶变换(FFT)处理,转动欧拉角格子大小为15°,输出程序打分排名前2000的复合物坐标.对所有对接产生的复合物,利用Charmm力场17结合GBSW隐式水模型18在CHARMM C33b119中进行优化,优化后的坐标作为对接结果的研究对象.为研究复合物中两个受体二聚体之间的夹角,我们定义沿着尾部A362-I421(图2中红色线框内部)的最长惯性主轴且指离尾部的方向为受体二聚体的方向(图2中蓝色箭头).倒金字塔式复合物模型的选择主要依赖二聚体之间的角度和晶体结构,而拉链状复合物模型的选择则主要依赖二聚体之间的角度和EM11,12中测量出的平均尾部叠合长度.
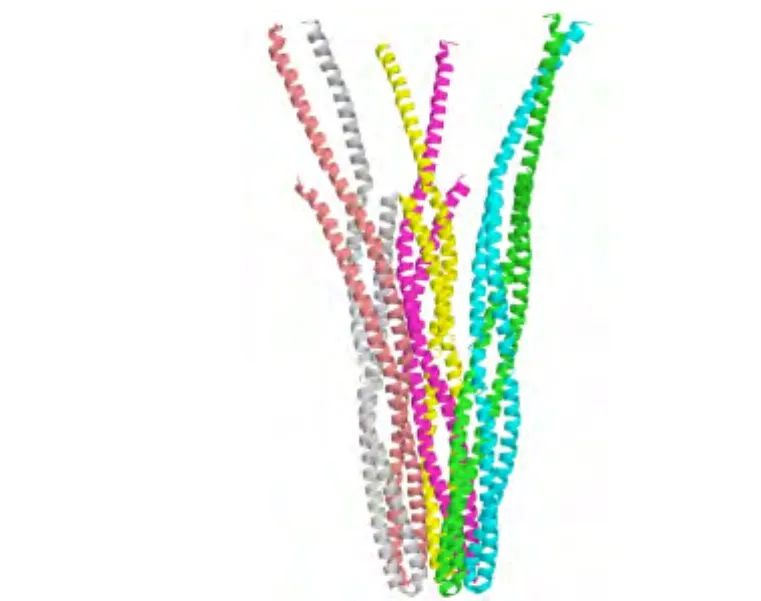
图1 丝氨酸受体Tsr胞质区晶体结构中的TOD结构(PDB代码:1QU77)Fig.1 TOD structure in the crystal structure of serine chemoreceptor Tsr cytoplasmic domain(PDB code:1QU77)TOD:trimer-of-dimers
2.2 分子动力学模拟
对确认的拉链状复合物模型和晶体结构TOD中两个二聚体的复合物利用Gromacs 4.5.4程序20进行了MD模拟.考虑到系统中不存在小分子,为了更好地研究这些复合物在水溶液中的稳定性,模拟使用OPLS-AA力场21描述蛋白质,使用TIP3P模型22描述水分子.为了允许较大的构象变化,在复合物两侧三个方向各加入1.2 nm的水层.整个系统中含有8个单位的负电荷,加入8个Na+以平衡整个系统的电荷,并加入0.1 mol·L-1的NaCl以模拟生理条件.每个最终得到的模拟系统中均含有约80000个原子,具体原子数目随着初始结构的不同有一定的变化.
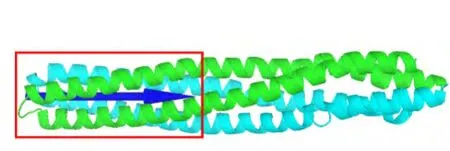
图2 用来对接的受体Tsr二聚体结构及其方向Fig.2 Tsr dimer structure and direction used for docking

图3 具有不同二聚体夹角的对接复合物数量Fig.3 Counts for docking complexes with different dimer angles
3 结果与讨论
3.1 蛋白质-蛋白质对接结果
由ZDOCK计算生成了程序打分排名前2000的二聚体-二聚体复合物结构.对每个结构按照前述对二聚体方向的定义计算二聚体之间的夹角,并统计具有不同夹角的复合物分布情况(图3).注意这里计算的具体分布与输出排名靠前的文件个数有关,但这2000个复合物涵盖了[0°,180°]的范围,使得进一步的研究成为可能.

图4 类倒金字塔形复合物与TOD中的两个二聚体的最小Cα-RMSD值Fig.4 Minimal Cα-RMSD between the inverted pyramid like complexes and any two dimers in TOD RMSD:root mean square deviation

图5 类倒金字塔形复合物与晶体结构TOD的对齐显示Fig.5 Alignments of inverted pyramid like complexes and crystal structures in TOD
首先研究接近晶体结构TOD中夹角的复合物.计算晶体结构TOD中两个二聚体的夹角,结果为17.5°.这一计算结果与前人根据胞质区二聚体长度以及TOD中二聚体在跨膜区之间的距离粗略地算出的20°夹角23,24相近,具体数值根据计算方法的不同可能会有5°以内的差别.这里以20°为中心,定义夹角位于[15°,25°]的复合物为类倒金字塔形复合物,共有82个.分别计算这些复合物与晶体结构TOD中任意两个二聚体之间均方根位移(RMSD)的最小值(图4).这82个复合物中最小的RMSD值为0.16 nm.对应复合物(图5(a))的二聚体之间夹角为17.2°.对齐显示(图5(a))表明通过对接能找到十分接近于晶体结构TOD的倒金字塔复合物.用ZDOCK进一步将这个二聚体-二聚体的复合物和晶体结构TOD中的一个二聚体对接,生成打分排名前2000的二聚体的三聚体,并计算它们与晶体结构TOD的Cα-RMSD值.具有最小Cα-RMSD值 (0.24 nm)的复合物结构(图5(b))与晶体结构TOD很相似,特别在靠近尾部的界面上十分吻合(图5(c)).
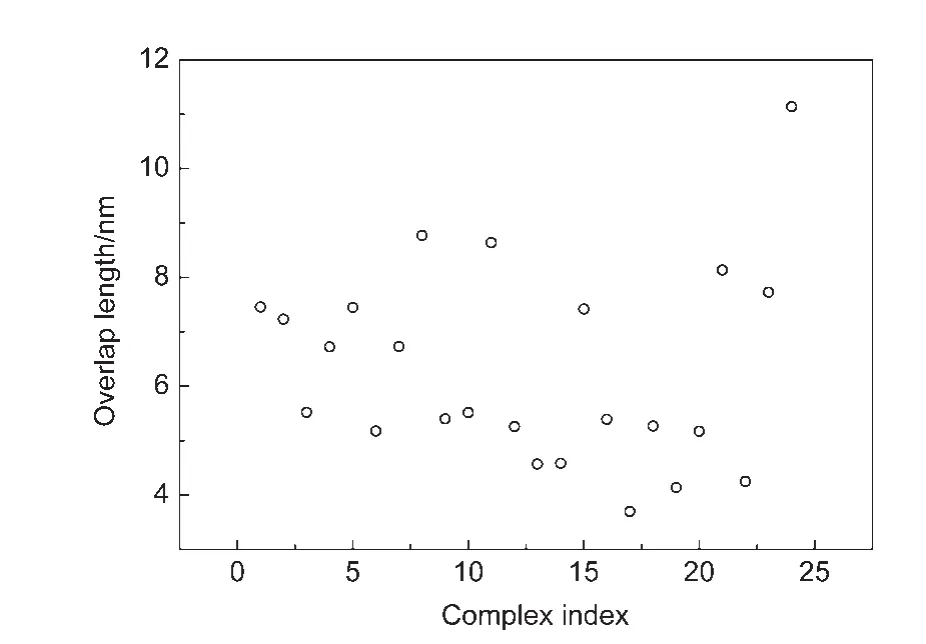
图6 类拉链状复合物二聚体尾部之间的叠合长度Fig.6 Overlap length of dimer tips in zipper like complexes
两个二聚体一般会以反平行方式形成拉链状结构,因此定义二聚体夹角位于[170°,180°]的复合物为类拉链状复合物,共有24个.对每个复合物,定义两个二聚体尾部之间的叠合长度为二聚体最终端部残基E391的平均坐标沿着最长主轴方向的投影距离.计算得到的二聚体尾部叠合长度示于图6.我们取其中尾部叠合长度位于[5.5 nm,7.5 nm]的8个复合物结构(图7(a)),分析表明这8个复合物可以分为两类,分别分布在二聚体的两侧.在大规模的拉链状聚集时,二聚体可以同时利用两侧界面形成扩展的团簇结构.对每一类取最接近EM实验11中拉链状聚集的尾部叠合长度((6.5±0.6)nm)的结构作为最终的拉链状复合物结构模型(图7(b)),它们分别具有7.23和6.72 nm的叠合长度,与EM11中测出的(6.5±0.6)nm接近.观察发现它们主要在尾部N346-L434之间叠合.下文分别称它们为拉链状模型(zipper model)1和2.
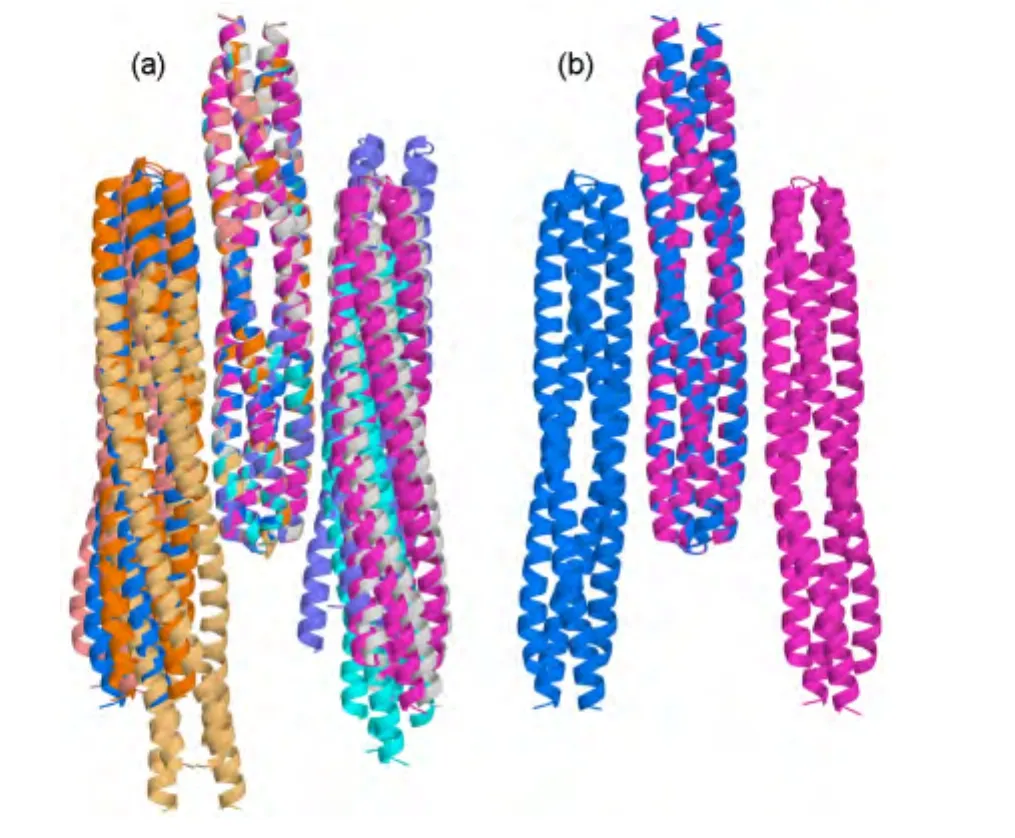
图7 类拉链状复合物结构Fig.7 Structures for zipper like complexes
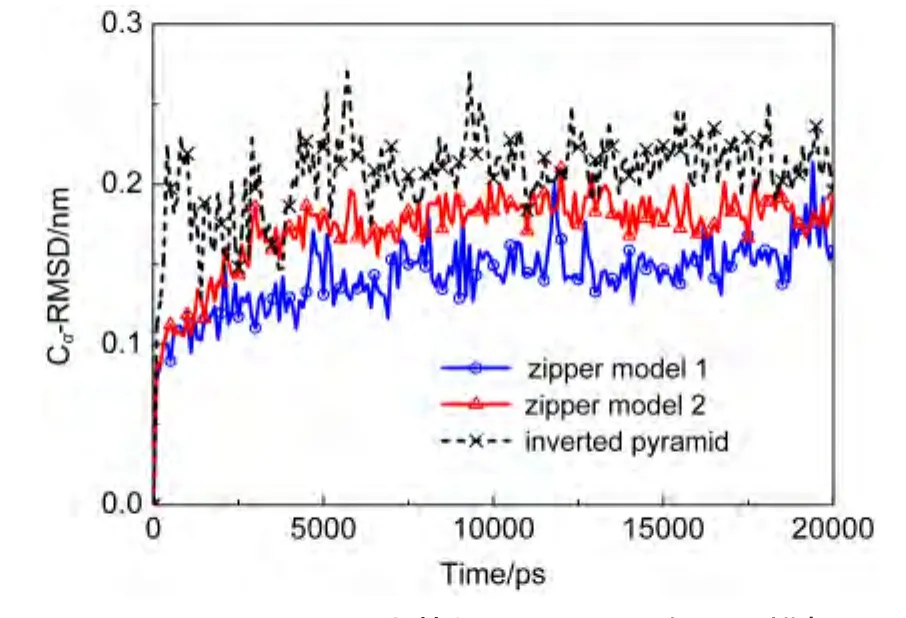
图8 N346-L434残基的Cα-RMSD在MD模拟过程中的变化Fig.8 Cα-RMSD changes for residues within N346-L434 during MD simulations
3.2 分子动力学模拟结果
对拉链状复合物的两个模型以及晶体结构中的倒金字塔复合物结构各进行了20 ns的MD模拟.图8显示的是这三个结构在拉链状复合物的尾部叠合区N346-L434之间89个残基的Cα坐标与起始结构之间的RMSD随模拟时间的变化.三个复合物与初始结构的CαRMSD都不超过0.3 nm,说明两个拉链状复合物模型都比较稳定.
分别对这三个结构模型计算了尾部叠合区Cα原子的均方根涨落(RMSF)(图9).相对倒金字塔复合物结构,拉链状复合物结构在F373-I377之间的RMSF明显较小,可以推测这些区域对结合的贡献是比较大的.
3.3 相互作用分析

图9 N346-L434残基的Cα原子的RMSFFig.9 RMSF for Cαatoms in the residues within N346-L434
对拉链状复合物结构的两个模型,用APBS程序25分别计算四个二聚体单独存在条件下的静电势分布,并用高莹等26,27发展的PP_SITE程序计算界面上的关键疏水和氢键作用残基.将表面势叠合到对应的复合物结构上以分析复合物结合界面的带电性质(图10),并计算复合物受体二聚体周围0.4 nm的配体二聚体的带电及疏水残基(图10(b,c),正电的示为蓝色,负电的示为红色,疏水的示为绿色).可见拉链状模型1(图10(a,b))和拉链状模型2(图10(c,d))只有少量差别,主要集中在复合物界面的边缘.例如,对拉链状模型1,配体二聚体中的E436、D429(靠近coiled-coil螺旋尾部)和R415(靠近界面边缘)与受体二聚体有较强的静电作用;而在拉链状模型2中,配体二聚体中的R409取代了R415,同时E436和D429与受体二聚体之间的距离增加,导致配体二聚体与受体二聚体的作用减弱.重要的是,这两个模型都一致性地显示,静电相互作用形成的盐桥是拉链状复合物形成的最主要的相互作用.另外,界面上的疏水相互作用对界面也有较大贡献,特别是F373和I377的贡献尤为显著.这一结论与图9中通过MD模拟计算出的F373-I377之间的RMSF普遍较小是一致的.图中带电的残基和电势的表面符号一般都比较匹配,特别是R388附近的负电势很强,预示着这种拉链状复合物之间的相互作用很强,这也可以解释为什么化学趋向性受体在细胞膜上能克服让细胞膜变形的弹性能障碍而形成拉链状聚集.值得一提的是,R388也是倒金字塔形TOD形成的关键残基之一,28因此拉链状复合物和倒金字塔复合物相互竞争这些关键作用残基.
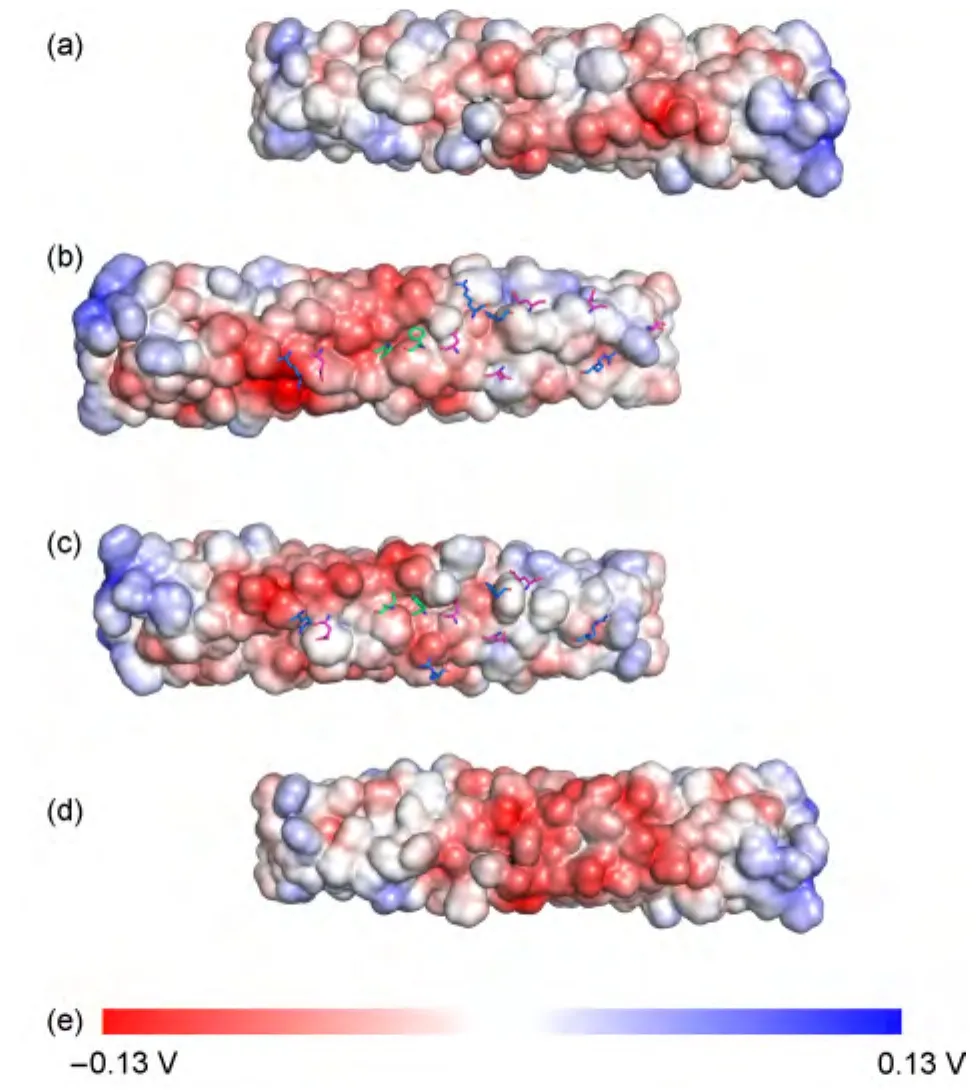
图10 拉链状复合物的表面静电势与关键残基Fig.10 Surface electrostatic potential and key residues of zipper like complexes
4 结论
化学趋向性受体在细胞膜上可以通过TOD为基本单元形成大规模聚集的团簇,也可以通过尾部相互叠合形成拉链状的聚集.为了研究拉链状聚集的分子相互作用机制,我们以晶体结构中的Tsr二聚体为基本单元,通过蛋白质分子对接计算研究了二聚体形成的可能复合物结构.在对接所得到的复合物中,既能找到形成TOD的倒金字塔式复合物,也能找到形成拉链状聚集的拉链状复合物.由于没有EM Map的数据,我们通过二聚体之间的角度和EM中测量出的平均尾部叠合长度数据从对接复合物中选择拉链状复合物模型.结果显示Tsr二聚体的两侧各有一个界面可以通过尾部叠合的方式形成拉链状复合物,并确认两个分别具有7.23和6.72 nm叠合长度的复合物为拉链状复合物模型.对这两个拉链状复合物模型的MD模拟表明其是稳定的.对界面的相互作用分析发现静电和疏水相互作用是拉链状复合物形成的主要原因.R388、F373和I377是最为关键的界面相互作用残基.考虑到对应EM实验的EM Map数据尚未放出,由对接产生拉链状复合物模型的坐标是目前唯一可以得到的分子层次模型.在此基础上,可以进行进一步的全原子或粗粒化模拟.本文中研究的胞质区二聚体片段含有264个残基,对拉链状聚集及二聚体的三聚体的聚集最好含有6个以上的二聚体(即能形成两个以上的TOD),超出了目前的数百残基的全原子模拟范围.29因此,对化学趋向性受体大规模聚集的模拟应该基于本文提供的相互作用机制发展粗粒化模型30并考虑使用耗散粒子动力学31,32等粗粒化模拟中常用的介观模拟技术.本文的研究为化学趋向性受体的拉链状聚集提供了分子相互作用的机制,也为进一步的聚集理论或模拟提供了基础.
(1) Adler,J.Annu.Rev.Biochem.1975,44(1),341.doi:10.1146/annurev.bi.44.070175.002013
(2) Adler,J.;Tso,W.W.Science 1974,184(4143),1292.doi:10.1126/science.184.4143.1292
(3) Hazelbauer,G.L.;Falke,J.J.;Parkinson,J.S.Trends Biochem.Sci.2008,33(1),9.doi:10.1016/j.tibs.2007.09.014
(4) Tu,Y.Annual Review of Biophysics 2013,42,337.doi:10.1146/annurev-biophys-083012-130358
(5)Ottemann,K.M.;Xiao,W.;Shin,Y.K.;Koshland,D.E.,Jr.Science 1999,285(5434),1751.doi:10.1126/science.285.5434.1751
(6) Bi,S.;Yu,D.;Si,G.;Luo,C.;Li,T.;Ouyang,Q.;Jakovljevic,V.;Sourjik,V.;Tu,Y.;Lai,L.Proc.Natl.Acad.Sci.U.S.A.2013,110(42),16814.doi:10.1073/pnas.1306811110
(7)Kim,K.K.;Yokota,H.;Kim,S.H.Nature 1999,400(6746),787.doi:10.1038/23512
(8) Maddock,J.R.;Shapiro,L.Science 1993,259(5102),1717.doi:10.1126/science.8456299
(9) Greenfield,D.;McEvoy,A.L.;Shroff,H.;Crooks,G.E.;Wingreen,N.S.;Betzig,E.;Liphardt,J.PLoS Biol.2009,7(6),e1000137.doi:10.1371/journal.pbio.1000137
(10) Bray,D.;Levin,M.D.;Morton-Firth,C.J.Nature 1998,393(6680),85.doi:10.1038/30018
(11) Weis,R.M.;Hirai,T.;Chalah,A.;Kessel,M.;Peters,P.J.;Subramaniam,S.J.Bacteriol.2003,185(12),3636.doi:10.1128/JB.185.12.3636-3643.2003
(12) Lefman,J.;Zhang,P.;Hirai,T.;Weis,R.M.;Juliani,J.;Bliss,D.;Kessel,M.;Bos,E.;Peters,P.J.;Subramaniam,S.J.Bacteriol.2004,186(15),5052.doi:10.1128/JB.186.15.5052-5061.2004
(13) Francis,N.R.;Levit,M.N.;Shaikh,T.R.;Melanson,L.A.;Stock,J.B.;DeRosier,D.J.J.Biol.Chem.2002,277(39),36755.doi:10.1074/jbc.M204324200
(14) Zhang,P.;Khursigara,C.M.;Hartnell,L.M.;Subramaniam,S.Proc.Natl.Acad.Sci.U.S.A.2007,104(10),3777.doi:10.1073/pnas.0610106104
(15) Zhang,C.S.;Lai,L.H.Acta Phys.-Chim.Sin.2012,28,2363.[张长胜,来鲁华.物理化学学报,2012,28,2363.]doi:10.3866/PKU.WHXB201209172
(16) Mintseris,J.;Pierce,B.;Wiehe,K.;Anderson,R.;Chen,R.;Weng,Z.Proteins 2007,69(3),511.doi:10.1002/prot.21502
(17) Mackerell,A.D.,Jr.;Feig,M.;Brooks,C.L.,III.J.Comput.Chem.2004,25(11),1400.doi:10.1002/jcc.v25:11
(18)Im,W.;Lee,M.S.;Brooks,C.L.,3rd.J.Comput.Chem.2003,24(14),1691.doi:10.1002/jcc.10321
(19) Brooks,B.R.;Brooks,C.L.,3rd.;Mackerell,A.D.,Jr.;Nilsson,L.;Petrella,R.J.;Roux,B.;Won,Y.;Archontis,G.;Bartels,C.;Boresch,S.;Caflisch,A.;Caves,L.;Cui,Q.;Dinner,A.R.;Feig,M.;Fischer,S.;Gao,J.;Hodoscek,M.;Im,W.;Kuczera,K.;Lazaridis,T.;Ma,J.;Ovchinnikov,V.;Paci,E.;Pastor,R.W.;Post,C.B.;Pu,J.Z.;Schaefer,M.;Tidor,B.;Venable,R.M.;Woodcock,H.L.;Wu,X.;Yang,W.;York,D.M.;Karplus,M.J.Comput.Chem.2009,30(10),1545.doi:10.1002/jcc.v30:10
(20) Hess,B.;Kutzner,C.;van der Spoel,D.;Lindahl,E.J.Chem.Theory Comput.2008,4(3),435.doi:10.1021/ct700301q
(21) Kaminski,G.A.;Friesner,R.A.;Tirado-Rives,J.;Jorgensen,W.L.J.Phys.Chem.B 2001,105(28),6474.doi:10.1021/jp003919d
(22) Jorgensen,W.L.;Chandrasekhar,J.;Madura,J.D.;Impey,R.W.;Klein,M.L.J.Chem.Phys.1983,79(2),926.doi:10.1063/1.445869
(23)Vaknin,A.;Berg,H.C.Proc.Natl.Acad.Sci.U.S.A.2006,103(3),592.doi:10.1073/pnas.0510047103
(24) Shimizu,T.S.;Le Novere,N.;Levin,M.D.;Beavil,A.J.;Sutton,B.J.;Bray,D.Nat.Cell Biol.2000,2(11),792.doi:10.1038/35041030
(25) Baker,N.A.;Sept,D.;Joseph,S.;Holst,M.J.;McCammon,J.A.Proc.Natl.Acad.Sci.U.S.A.2001,98(18),10037.doi:10.1073/pnas.181342398
(26) Gao,Y.;Wang,R.;Lai,L.H.J.Mol.Model.2004,10(1),44.doi:10.1007/s00894-003-0168-3
(27) Gao,Y.;Wang,R.X.;Lai,L.H.Acta Phys.-Chim.Sin.2002,18,676.[高 莹,王任小,来鲁华.物理化学学报,2002,18,676.]doi:10.3866/PKU.WHXB20020802
(28) Mowery,P.;Ostler,J.B.;Parkinson,J.S.J.Bacteriol.2008,190(24),8065.doi:10.1128/JB.01121-08
(29)Dror,R.O.;Dirks,R.M.;Grossman,J.P.;Xu,H.;Shaw,D.E.Ann.Rev.Biophy.2012,41,429.doi:10.1146/annurev-biophys-042910-155245
(30) Saunders,M.G.;Voth,G.A.Ann.Rev.Biophy.2013,42,73.doi:10.1146/annurev-biophys-083012-130348
(31)Guigas,G.;Morozova,D.;Weiss,M.Adv.Protein Chem.Struct.Biol.2011,85 143.doi:10.1016/B978-0-12-386485-7.00004-1
(32)Chen,M.L.;Wang,L.X.;Chen,S.S.;Liu,X.Y.Acta Phys.-Chim.Sin.2013,29,1201.[陈美玲,汪理想,陈姗姗,刘晓亚.物理化学学报,2013,29,1201.]doi:10.3866/PKU.WHXB201303202
