利用PCR-DGGE技术分析抗生素对小鼠肠道菌群多样性的影响
2014-09-12王春敏佟丽波韩桂华任继秋
王春敏 佟丽波 韩桂华 任继秋 崔 刚
(佳木斯大学基础医学院微生物教研室,黑龙江 佳木斯 154007)
动物胃肠道内数量和种类多样的微生物与动物的健康和疾病密切相关,已渐渐引起重视并成为研究热点〔1,2〕。传统的肠道菌群分析多采用活菌计数法,迄今为止,肠道中只有极少部分微生物可以被培养。对于用传统方法难以分离培养的细菌或目前尚未了解的细菌来说,传统方法往往难以作出全面的分析。16S rDNA在细菌中普遍存在,其可变区和保守区交错排列,可用于细菌的系统发育分析。基于16S rDNA的变性梯度凝胶电泳(DGGE)技术,理论上可以检测DNA片段中的一个碱基变化,在揭示自然界微生物群落遗传多样性和种群差异方面具有独特的优越性〔3〕。本实验采用PCR-DGGE技术研究不同抗生素对小鼠肠道微生物变化的影响,以探讨不同抗生素对小鼠肠道菌群多样性的影响及机制。
1 材料与方法
1.1主要试剂 QIAamp DNA Stool Mini Kit购于上海东盛创新实验技术有限公司;PCR试剂盒(dNTPs、Buffer、MgCl2、Taq酶)大连宝生物生物有限公司;丙烯酰胺、甲叉双丙烯酰胺尿素、去离子甲酰胺、琼脂糖、溴乙啶购于北京博大泰克生物科技有限公司。16 SrDNA V3可变区扩增通用引物:357f(5′-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACG GGAGGCAGCAG-3′)和518r (5′-ATTACCGCGGCTGCTCC-3′),委托上海生工生物工程公司合成。
1.2实验动物及处理 昆明纯系小鼠30只,雌雄各半,体重(20±2)g购于佳木斯大学实验动物中心。随机分为对照组(A组)、盐酸林可霉素组(B组)和硫酸庆大霉素组(C组)。适应性喂养1 d后,B组灌盐酸林可霉素80 mg·kg-1·d-1,C组灌胃硫酸庆大霉素20 mg·kg-1·d-1,A组灌胃等量生理盐水。2次/d,间隔6 h,连续灌胃5 d。
1.3小鼠粪便肠道菌群总DNA提取及PCR扩增 小鼠灌胃5 d后无菌取各组小鼠粪便样品0.2 g,严格按试剂盒说明操作,提取肠道菌群总DNA,提取的DNA通过1.0%的琼脂糖凝胶电泳检测DNA完整性,并用核酸定量仪测定其DNA含量,置-20℃保存备用。以提取的基因组总DNA为模板,使用16 SrDNA V3可变区通用扩增引物对其进行扩增。PCR的反应体系(50 μl)组成为DNA模板2 μl,引物各1 μl,10倍缓冲液(含Mg2+)5 μl,dNTP 2 μl,Taq酶 0.25 μl,加去离子水补至50 μl。扩增程序为94℃ 5 min,(94℃ 30 s,56℃ 30 s,72℃ 30 s)30个循环,72℃延伸10 min。扩增产物在1.0%琼脂糖凝胶上电泳,溴乙啶染色,紫外光下观察扩增效果。
1.4DGGE分析 采用 Bio-Rad Dcode 进行 DGGE 凝胶电泳。变性剂线性梯度范围为35%~65%,1×TAE缓冲液,先在220 V条件下预电泳10 min,随后在150 V条件下电泳8 h,电泳结束后 ,硝酸银染色〔4〕,用 TANON-2500凝胶成像系统采集图像,观察DNA条带并记录重复3次。
2 结 果
应用DGGE技术分离16S rDNA V3区段PCR产物,三组粪便样品经DGGE后都分离出数目不等的电泳条带,可分辨的条带5~12条,而且不同组标本之间带型存在差异。B、C两组与A相比较,电泳条带减少尤以B组电泳条带最少,且B、C两组之间还存在差异。见图1。
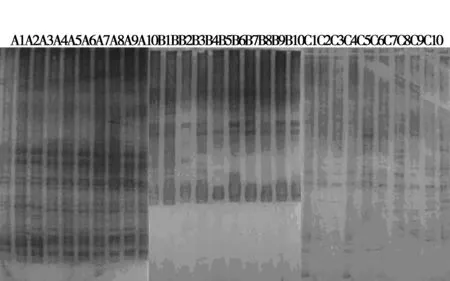
图1 各组细菌16S rDNA V3区段PCR产物
3 讨 论
DGGE技术是由Fischer和Lerman最先提出并应用于检测基因突变的技术〔5〕。Muyzer等〔6〕将其引入微生物分子生态学研究中。该技术打破部分细菌不可培养性的局限,不仅可以比较不同样品中细菌的多样性和相似性,而且还可以通过对目标带的回收、克隆和测序进一步分析特征性的细菌种群。现PCR-DGGE技术已用于多种生态环境微生物菌群研究。其中包括消化道及粪便菌群研究〔7~9〕。DGGE图谱中可区分出很多清晰的条带,每一条带代表某个微生物菌群。每一条带的密度反映细菌的相对构成比例。
本实验采用该技术检测两种抗生素应用前后小鼠肠道细菌16S rDNA V3区段PCR扩增产物,从而了解抗生素应用对小鼠肠道菌群的影响。本实验结果体现出肠道微生态发生较大变化,且不同抗生素对肠道微生态的影响不一致。分析其原因:盐酸林可霉素抗菌谱主要针对革兰阳性菌(包括革兰阳性厌氧菌),庆大霉素抗菌谱主要针对革兰阴性菌,所以两种抗生素分别作用于小鼠肠道后,小鼠肠道内优势菌群的种类和数量必发生不同变化,反映在DGGE图谱中表现为条带出现位置、多少及密度的不同。
综上,采用PCR-DGGE技术可分析肠道菌群多样性,并从分子水平证实抗生素可影响小鼠的肠道菌群结构和多样性。但不同抗生素对哪些优势菌群影响最大还需结合对目标带的回收、克隆和测序等手段明确。
4 参考文献
1吴仲文,李兰鹃,马伟杭,等.肠道菌群正常参考值的检测〔J〕.中国微生态学杂志,2001;13:314-5.
2徐凯进,李兰鹃.肠道正常菌群与肠道免疫〔J〕.国外医学·流行病学传染病学分册,2005;32(3):181-3.
3张宝涛.PCR-DGGE技术及其在微生物生态学中的应用〔J〕.生物信息学,2006;4(3):132-42.
4Sanguninetti CJ,Dias Neto E,Simpson AJ.Rapid silver staining and recovery of PCR products separated on polyacylamide gels〔J〕.Biotechniques,1994;17(5):914-21.
5Fisher SG,Lerman LS.DNA fragments differing by single base-pair substitutions are separated in denaturing gradient gels:correspondence with melting theory〔J〕.Proc Natl Acad Sci USA,1983;80(6):1579-83.
6Muyzer G,de Waal EC,Uitterlinden AG.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA〔J〕.Appl Environ Microbiol,1993;59(3):695-700.
7季天荣,陈 庄.DGGE技术优化及其在肠道微生态研究中的应用〔J〕.广东农业科学,2008;(6):93-6.
8Tannock GW,Munro K,Harmsen HJM,etal.Analysis of the fecal microflora of human subjects consuming a probiotic product containing Lactobacillus rhamnosus DR20〔J〕.Appl Environ Microbriol,2000;66:2578-88.
9于 涛,德力夏提·依米提,买买提·牙森,等.新疆维吾尔族长寿老人肠道菌群多样性分析〔J〕.中国病原生物学杂志,2009;4(4):271-4.
