原核CRISPR-Cas系统的结构功能及应用
2014-08-07冯红,董阁,雍彬,李维
冯 红, 董 阁, 雍 彬, 李 维
(1.四川大学生命科学学院生物资源与生态教育部重点实验室,四川成都610064; 2.四川师范大学生命科学学院,四川成都610066)
CRISPR(Clustered regularly interspaced short palindromic repeats)称为成簇规则间隔的短回文重复序列,最早于1987年在大肠杆菌中发现[1],随后陆续在许多真细菌和古菌基因组中发现了这种重复序列[2-5].2002年R.Jansen等[6-7]详细比较分析原核基因组中的这种重复序列,更重要的是发现在此重复序列的旁侧经常伴随出现保守的系列基因.根据重复序列的排列特征,他们将这种独特的重复序列命名为CRISPR,保守基因编码的蛋白质统称为CRISPR附属蛋白(Cas,CRISPR-association proteins).由于这种序列在细菌和古菌中分布如此之广,因此推测CRISPR-Cas系统应该具有某种细胞功能.
后来,陆续在不同的细菌中检测到CRISPR位点的转录产物 crRNA(CRISPR RNA)[4,8-10],但crRNA通常较编码基因序列短,说明这些转录产物是经过核酸酶修饰加工后产生的.此外,由于Cas蛋白往往包含核酸切割、修饰的功能域,因此有人推测CRISPR-Cas系统可能与DNA复制、修复等DNA代谢有关[11-12].2005年3个独立的研究小组在对CRISPR重复序列的进一步分析比较后,几乎同时发现CRISPR中的间隔序列与噬菌体和质粒的序列完全相同[13-15].所以又提出了新的假说,认为原核CRISPR-Cas系统可能是细菌抵御外来质粒和噬菌体侵入的一种免疫防御机制.2007年R.Barrangou等[16]首次在乳酸菌Streptococcus thermophilus中,用实验证实了CRISPR-Cas系统的生物学功能,它确实是原核细胞抵御噬菌体侵染的一种新的免疫机制.次年,L.A.Marraffini等[17]在Staphylococci中证明CRISPR-Cas系统同样也能够干扰质粒的结合转移.
随后几年,在CRISPR-Cas系统的免疫机制以及Cas蛋白质结构功能等方面的研究取得了重大的进展,而且基于CRISPR-Cas系统发展了一种新的DNA编辑技术,在原核和真核细胞中成功地用于基因组的编辑、改造,显示出巨大的潜力.本文较全面综述了原核CRISPR-Cas系统的分布、组织结构和作用机理以及在基因组编辑等方面应用,以期推动国内在此相关领域的研究.
1 原核基因组广泛存在CRISPR重复序列
CRISPR重复序列具有典型的特征,可以通过简单搜索重复序列在基因组中找到这样的重复序列.但是由于CRISPR重复序列的多样性和整个系统的复杂组成,为了能够更加准确地鉴定出CRISPR-Cas系统,近年来开发了一些专门的计算机软件,用于搜索CRISPR序列及其Cas基因,如CRISPRfinder,PILER-CR,CRT等[18-21].基于CRISPRfinder等计算机软件的搜索比较,构建了2个独立的数据库(http://crispr.u-psud.fr/crispr;http://crispr.genouest.org)[18-19].基于这2个数据库,我们可以看到CRISPR重复序列广泛分布于原核世界,在所有已经分析的基因组中,45.4%(1126/2480)或46.4%(1141/2457)的真细菌、83.3%(125/150)或79.2%(126/159)的古细菌中存在CRISPR重复序列(这2组数据分别来自于以上的两个数据库,截止时间分别为1013/9/18和1013/7/27),而且这些细菌分属于各种不同的类群.迄今为止,Chlamydiae是唯一没有发现CRISPR重复序列的细菌类群,也许这与其专性寄生的生境和较小的基因组有关.此外,在宏基因组测序中也发现了丰富多样的CRISPR重复序列[22-23].

图1 CRISPR-Cas系统的组织构成Fig.1 Organization of the CRISPR-Cas system
典型的CRISPR重复序列由3部分组成(图1):即同向重复序列(directed repeat,R);长度不等的间隔序列(spacer,S);以及位于5′端的前导序列(leader,L).前2种序列依次串联排列,可以拥有2到100个重复.CRISPR位点大多出现在染色体基因组中,也有少数出现在质粒序列中;有的基因组可能包括多个CRISPR位点,有的细菌可能只有一个.
大多数CRISPR包含前导序列,长度在100~500 bp之间,通常富含AT.在不同的CRISPR中,前导序列并不保守,但包含转录起始位点,而且在一些细菌中已经得到了实验验证[24-25],因此前导序列就是启动CRISPR重复序列转录的启动子,在拥有CRISPR系统的细菌细胞内,很容易检测到CRISPR的转录产物[24-26].
同向重复序列通常在23~55 bp之间,中位数集中于24~25,29~30,36~37[18].不同的细菌具有不同的CRISPR重复序列,并且这种重复序列呈现多样性,根据同向重复序列的同源性,大致可以分成12个群以及21个亚群[27].但是在同一个CRISPR排列中,同向重复序列是高度保守的;而且在一些相近的细菌属种间,也具有一定的同源性.由此推测CRISPR-Cas系统可能来自于“偶然”的水平基因转移[28-29].重复序列的另外一个特点是,其序列呈部分回文对称结构,因此成熟的crRNA能够形成一个茎环环结构(图1).
间隔序列位于两个重复序列之间,在同一个CRISPR排列中拥有相似的长度,但序列是独特的.在不同的CRIPSR-Cas系统中,间隔序列的长度大多位于21~72 bp的范围,主要集中在32 bp或35~37 bp之间[27].对于不同的原核种类,间隔序列具有高度的多样性,只有在一个细菌的种内不同菌株之间趋于同源.目前已经明确认为间隔序列来自于噬菌体或质粒,实际上是噬菌体或质粒入侵后留下的“痕迹”,从而赋予细胞获得对相应噬菌体或质粒的免疫防御能力.此外,许多间隔序列在数据库中还不能找到同源序列,这从另一个侧面说明我们对噬菌体等移动元件的多样性仍然缺乏充分的认识.
2 CRISPR-Cas系统的组织结构
CRISPR-Cas系统是由CRISPR重复序列排列与其串联的cas基因组成的一个原核免疫系统(图1).其中,cas基因主要编码核酸酶、DNA解旋酶等切割修饰核酸的相关蛋白质.通过对大量的CRISPRCas系统中编码的蛋白质的鉴定和序列分析,可将Cas蛋白归并为45个蛋白家族[12].除了少数的基因,如cas1和cas2几乎出现在所有的CRISPR-Cas系统中,不同的CRISPR-Cas系统包含各种不同、复杂和异源的编码基因,最多可达到20个以上.主要的Cas蛋白的特征和功能汇总见表1.
根据CRISPR-Cas系统中的Cas蛋白的种类和同源性,可将CRISPR-Cas系统区分成3种类型[48-50],即Type I~III.这3种类型除了包括2个共有基因cas1和cas2之外,还包括特定的“标记”基因,类型I-III的标志基因分别是cas3、cas9和cas10.其次,在每一种类型下再根据cas基因的异同以及cas1的同源性,进一步分成不同的亚型(表2).值得注意的是这些类型和亚型并没有原核种属的特异性,而且同一个细菌菌株也可能包含不同类型的CRISPR-Cas系统.
类型I(Type I)主要出现在真细菌中(~31%),除了标志基因cas3和共有基因cas1和cas2以外,通常还包括cse1、cse2、cas7、cas5、cas5e等基因.该类型下再分成了6个亚型(I-A~E).如Escherichia coliK12中的 CRISPR-Cas系统I-E亚型包括8 个基因(cas1、cas2、cas3、cas5、cas6、cas7、cse1和cse2),14个部分重复的回文序列以及13个间隔序列[48].类型II(Type II)最早在乳酸菌Streptococcus thermophilus中发现,属于比较简单的CRISPR-Cas系统,即除了共有基因cas1和cas2和标志基因cas9之外,包括的其它cas基因比较少,据此可进一步分成2个亚型.

表1 CRIPSR-Cas系统中编码的主要蛋白质家族的特征Table 1 Characterization of the protein families in CRISPR-Cas system
类型III(Type III)约占已知CRISPR-Cas系统的1/4,其中分布有一些重要的病原细菌,如Styphylococcus和Mycobacteria;绝大多数古菌中存在的CRISPR-Cas系统也属于类型III.类型III通常由较多的基因组成,除了标志基因cas10之外,还包括许多编码RAMP蛋白的基因.根据cas1的同源性以及拥有的RAMP蛋白编码基因,可进一步分成2个亚型(III-A和III-B).

表2 CRISPR-Cas系统的分类[47]Table 2 Classification of CRISPR-Cas system
3 CRISPR-Cas系统的免疫机理
自从2007年R.Barrangou等[16]首次在Streptococcus中用实验证明了CRISPR-Cas系统的免疫防御功能后,最近几年在CRISPR-Cas系统作用机理的研究中取得了极大的进展.虽然对整个过程中的某些细节还有待阐明,但大体上可以将CRISPR-Cas系统的免疫作用机理分为3步:即获得间隔序列;crRNA的表达、加工和成熟;以及免疫干扰.其中,crRNA的加工成熟是阐明CRISPR-Cas系统作用机理的核心.
3.1 获得间隔序列间隔序列的获得是一个较为保守的过程,包括吸收外源DNA,并插入CRISPR重复序列中形成一个新的间隔序列,这一过程又称为“适应”(adaption).在分析环境样品的 CRISPR序列时,发现获得间隔序列是非常活跃的[51],说明在自然界中这是一种经常发生的事件.当然,在实验室已经证明细菌确实能够从入侵的外源DNA中捕获间隔序列[16],随后在E.coli,Pseudomonas aeruginosa,Streptococcus agalactiae,Sulfolobus solfataricus等多种不同的细菌中也观察到同样的现象[52-57].一般认为共有基因cas1和cas2编码产物是获得间隔序列所必需的,一是因为这2个基因存在于所有的 CRISPR-Cas类型中;二是在一些CRISPR-Cas系统中敲除cas1或cas2基因后,细菌不能获得新的间隔序列[52,54].但是目前对这一过程的机制并不了解,如外源DNA序列的识别、原间隔序列(protospacer)的切割和整合,以及重复序列的“复制”等.但通过对Cas1和Cas2的生化特性和结构等方面的研究可能提供一些线索.Cas1在体外能够切割核酸,具有内切核酸酶活性,如来自于Pseudomonas的Cas1能够将dsDNA切割成80 bp长的小片段[31],但是80 bp远大于间隔序列的长度,因此推测可能还有其它组分参与间隔序列的获得.来自于S.solfataricus的Cas2能切割富含U的RNA[30],但Desulfovibtio culgaris的 Cas2却不能切割DNA[58],最近发现来自于Bacillus halodurans的Cas2也能将DNA切割成120 bp长的小片段[59].此外,已有实验结果认为其它的Cas蛋白,如Csn2也可能参与间隔序列的获得,一是因为敲除csn2基因后导致不能获得间隔序列[16];二是最新的一个研究表明Csn2蛋白形成一个环状的4聚体,可结合dsDNA末端,并在dsDNA上滑动[60].因此推测Csn2也可能参与识别原间隔序列和整合,并且类似于双链断裂的修复.
在对原间隔序列的分析过程中,发现在原间隔序列旁侧存在一个保守的短序列(2-3 nt),称为PAM(Protospacer-Adjacent Motif),有实验表明PAM序列可能作为一个序列信号用于Cas蛋白对原间隔序列的识别,这些实验证实原间隔序列在插入CRISPR排列的序列中包括PAM的第3个核苷酸,同时成为重复序列的第一个核苷酸[53-54,61],也许这是保证原间隔序列精准插入的一种机制[62].在捕获间隔序列的同时,还需要“复制”产生一个新的重复序列单元,一般认为靠近前导序列的第一个重复序列复制产生新的重复序列[61].最近在S.solfutaricus中发现新的间隔序列可插入在整个CRISPR排列的内部,而不是经常观察到的哪样,即插入在紧邻前导序列的5′端[57].综上所述,CRISPR-Cas系统获得间隔序列的过程大致如图2所示,当噬菌体的染色体注入细胞后,Cas蛋白通过PAM信号识别原间隔序列(protospacer),然后酶切产生前体间隔序列,最后再整合到CRISPR重复序列中,其间包括复制产生一个新的重复序列单元.一般认为在3种类型的CRISPR-Cas系统中,间隔序列的捕获过程是相似的,但在一些细节上存在一定的差异.
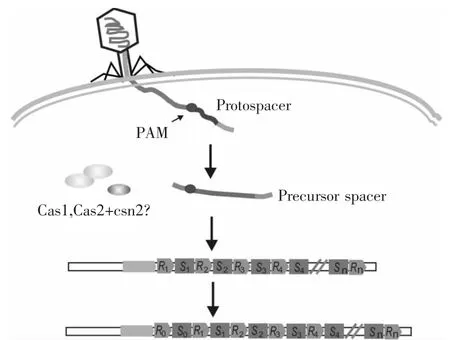
图2 CRISPR-Cas系统的适应示意过程Fig.2 Adaption processing of CRISPR-Cas system
3.2 类型I中crRNA的产生及干扰机理在免疫防御过程中,首先是CRISPR重复序列作为一个遗传位点在前导序列启动子的驱动下,转录产生Pre-crRNA(Precursor crRNA),再通过加工、形成成熟的crRNA,成熟的crRNA包括一个单元的同向重复序列和间隔序列.然后,crRNA结合Cas蛋白质,组成的一个复合体,并作用于同源的外源序列,crRNA以形成R-环(R-loop)的方式侵入靶序列,最后在crRNA的指导下,外源序列被Cas核酸酶降解,即产生所谓的免疫干扰,从而抵御外源DNA(噬菌体、质粒)的侵入.由于CRISPR-Cas系统的复杂和多样性,这一过程的细节和参与的蛋白质均有差异.在类型I中,CRISPR首先转录表达成pre-crRNA,并通过部分回文对称的重复序列形成茎环结构,然后结合Cas6蛋白,并在特定位点切割产生形成一个成熟的crRNA(5′端包括一个完整单元的间隔序列,3′端为茎环单元)[63-64].crRNA生成后,仍然结合在Cas6上,并作为一个核心,招募其它的Cas蛋白,构成一个称为Cascade(CRISPR-associated complex for antiviral defense)的复合体[36].在E.coli(Type I-E)中,Cascade复合体由5个蛋白组成CasABCDE[36].这5个蛋白现在分别称为 Cse1、Cse2、Cse7、Cas5 和 Cas6,它们在 Cascade复合体中的组成为1∶2∶6∶1∶1,其中6个Cse7亚基作为骨架,crRNA负责将Cse7组装成一个“海马”状的核蛋白螺旋,其中Cas6结合在crRNA的3′端[37,65].共表达这5个蛋白质以及加上Cas3和一个人工构建的crRNA(互补噬菌体DNA)就能完全保护E.coli抵御噬菌体λVir的侵染,在平板上不会形成噬菌斑[36].因此,在类型I中,推测crRNA的切割和加工过程大致如图3所示.另外,在Sulfolobus solfataricus(I-A)[64]、Thermoproproteus tenax(I-A)[65],Bacillus halodurans(I-C)[66]和Pseudomonas aeruginosa(I-F)[67]等系统中也分离鉴定出一个类似E.coli(I-E)的Cascade复合体.当然在这些不同的复合体中,蛋白质的组分和比例存在一定的差异,但一般认为在类型I中产生crRNA和形成Cascade复合体的过程和机理是相似的.

图3 CRISPR-Cas系统I型crRNA产生与免疫干扰机理Fig.3 Immune interference mechanism and formation of crRNA in Type I of CRISPR-Cas system
成熟的crRNA指导Cascade复合体识别、并作用于外源DNA(噬菌体)中的原间隔序列,并以形成R-环的方式,入侵靶序列(图3).在此过程中,原间隔序列中的PAM序列决定Cascade复合体与原间隔序列的识别和切割,例如CasA可能参与识别PAM序列,并以此区分自身的DNA[70],而且对PAM的识别并不依赖于互补配对[71].此外,间隔序列中的前8个核苷酸也与最初的识别有关,这个序列被称为“种子”序列(seed sequence)[72].此时,另一个蛋白Cas3被招募到复合体中,负责切割外源DNA[33-34,73-74].有关结构和生化分析证明Cas3本身就是一个核酸酶[33-34].
3.3 类型II中crRNA的产生及干扰机理类型II只有4个cas基因,除了共有基因cas1、cas2外,cas9编码一个较大的蛋白质,序列分析表明Cas9蛋白具有核酸酶和RNase H结构域.2011年利用S.pyogenes(II-A)揭示了该类型产生crRNA的过程[75].在差异 RNA-seq中,首先检测到 PrecrRNA,并用Northern杂交所证实.同时还检测到另外一个RNA,是cas操纵子上游反义链的转录产物,称为 tracrRNA(trans-activating crRNA).同时还发现了这两个 RNA的剪接产物,75 nt的tracRNA和66 nt的crRNA形成的杂交中间产物存在3′端突起,这是RNase III切割后的一个典型末端结构.利用RNase III基因的突变体确实也证明RNaseIII是crRNA加工和成熟必需的组分[75].同时,cas9基因的突变体也证实了cas9参与crRNA的形成[75].因此,在tracrRNA的协同下,RNase III和Cas9组成了一个负责生成crRNA的简单结构(图4).
在类型II中,Cas9与2个部分互补的RNA形成复合体,并在RNA的指导下,识别入侵的外源DNA(这一过程不需要RNase III).这个RNA/蛋白质复合体与类型I相似,也是通过PAM位点识别原间隔序列,有实验表明如果突变PAM,将会导致入侵的外源 DNA逃逸切割降解[76].此外,Cas9/tracRNA/crRNA这样的复合体也能够在体外,特异切割噬菌体或质粒DNA[44,77].因此,这个系统具有成为发展基因组编辑技术的潜力.
3.4 类型III中crRNA的产生及干扰机理类型III包括两个亚型(III-A和III-B),前者主要分布在真细菌中,包括一些重要的病原菌;后者主要分布在古菌中.类型III包括的cas基因比较多,特别是编码的RAMP蛋白质,这些蛋白质与Cas10都是类型III所必需的组分.通过Northern杂交,在正常生长条件下的多种细菌和古菌细胞中检测到crRNA,因此认为这些CRISPR系统是组成型表达的[10,25,78].相反,在类型I-E中,似乎CRISPR的表达受到高度调控[79-80];或者在暴露于噬菌体下,CRISPR被激活表达[81].
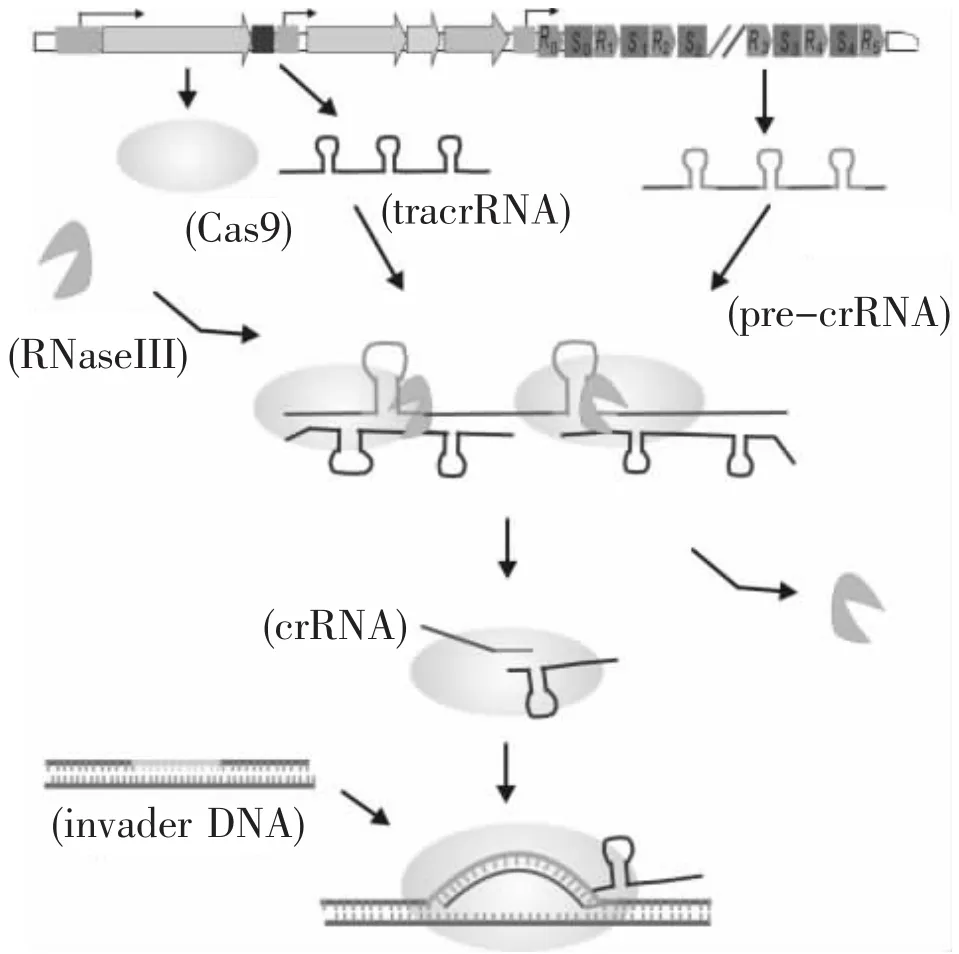
图4 CRISPR-Cas系统II型crRNA产生与免疫干扰机理Fig.4 Immune interference mechanism and formation of crRNA in type II of CRISPR-Cas system
在类型III中,Pre-crRNA的加工需要经历初加工和成熟两个阶段.Pre-RNA首先在同向重复序列中切割,产生一个crRNA中间产物,其两端具有部分重复序列,中间为完整的间隔序列.在成熟阶段,crRNA中间产物的3′端进一步被切割,产生成熟的crRNA.在Styphylococcus epidermidis中(Type III-A),成熟的crRNA长43 bp,少数为37 bp,负责初加工的蛋白质可能包括Cas6、Cas10和Csm4,但具体的作用还不清楚[17,82].在古菌P.furiosus(Type III-B)中,重复序列不是部分回文对称的,很多实验证据认为也是Cas6负责类型III中PrecrRNA的初加工,但是作用机制与类型I不同,Pre-crRNA结合“缠绕”在 Cas6蛋白质的表面(图5),然后在重复序列内部切割pre-crRNA,产生66~72 bp长的中间产物,中间产物将进一步在3′端缩短(负责的核酸酶不清楚),最后产生成熟的crRNA,其中在间隔序列的5′端保留8 nt的保守重复序列[83-85].此外,Cas6的催化特性以及与crRNA的共结构也揭示了Cas6是一个典型的核酸酶,并分别具有独立的DNA结合功能域和催化功能域[86].
类型III-A/B在干扰过程中作用的目标不同.在S.epidermidis(Type III-A)中,能够依赖间隔序列spc-1和spc-2分别抵御质粒的接合转移和噬菌体的入侵,并且入侵DNA中与间隔序列同源的序列(proto-spacer DNA)是类型III-A作用的靶点[17,82],但识别入侵DNA主要通过成熟crRNA与原间隔序列的互补配对[82],而不像类型I/II,这种识别依赖于PAM序列[70-71,76].目前还不清楚哪些Cas蛋白质参与了这一过程.
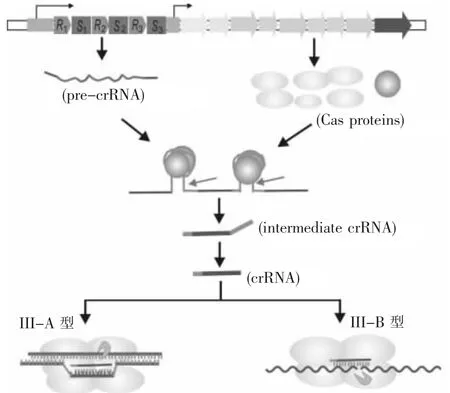
图5 CRISPR-Cas系统I型crRNA产生与免疫干扰机理Fig.5 Immune interference mechanism and formation of crRNA in type III of CRISPR-Cas system
已有的实验证据显示III-B类型在干扰时,作用的目标是RNA分子,这与其它所有的CRISPRCas系统都不相同.在古菌P.furiosus(III-B)中,2009年C.R.Hale等[87]利用质谱技术从分离的复合体中鉴定出Cas10、Cmr5和几个RAMP蛋白质(Cmr1、Cmr3、Cmr4 和 Cmr6),而且纯化的 Cmr复合体只能离体切割crRNA反义的单链的RNA,且位点是特异的;但不能切割任何 dsRNA或DNA[87-88].在另一个属于类型III-B的S.solfataricus中,也鉴定出一个包括7个Cas蛋白的复合体,在crRNA的指导下也只能切割RNA,但这种活性依赖于成熟 crRNA分子5′端 8 nt的重复序列[89].此外,虽然Cas10是类型III的标志蛋白,但根据Cmr复合体的结构组成,认为Cas10不参与靶RNA的切割,只是复合体中的一个结构蛋白质[85].综上所述,在类型III中,crRNA的加工成熟以及免疫干扰作用可归纳见图5.
4 CRISPR-Cas系统的应用
由于CRISPR-Cas系统的生物学功能以及重复序列的多样性和动态特性,CRISPR系统在原核的遗传学研究、抗病毒和生物技术等领域获得了广泛的应用.首先CRISPR-Cas系统的原始功能主要是防御噬菌体等外源入侵的移动元件.鉴于CRISPR-Cas适应功能,理论上可以通过将细菌菌株“暴露”于天然的噬菌体或通过基因工程技术,将小片段的病毒原间隔序列整合到CRIPSR的重复序列中,从而赋予细胞获得相应的免疫功能.这对于一些工业发酵菌株仍然具有一定的实际意义,即基于CRISPR的适应系统构建抗噬菌体的工业菌株,例如有人提出了所谓的“CRISPerization”策略(国际专利WO/2008/108989),以此筛选噬菌体的拮抗菌株.
其次,CRISPR重复序列具有广泛的多样性和动态特征,因此有研究者利用CRISPR排列及重复序列进行细菌的基因分型(genotyping).如1993年利用CRISPR中的同向重复序列对病原菌Mycobacterium tuberculosis进行分型[90],而且数字化后用以监控M.tuberculosis的临床流行学[91].基于同样的原理,测序分析环境样品中的CRISPR重复序列,可以用来监控自然界中原核细菌及其动态变化情况[92].另一方面,由于CRISPR排列中的间隔序列来自于噬菌体,并且借助于DNA二代测序技术,可以获得许多细菌或菌株,甚至环境样品中海量的CRISPR序列信息,可以很方便地用以分析研究原核世界或自然生境中细菌与噬菌体病毒之间的进化、相互作用及其生态适应等[93-94].
最后,由于CRISPR-Cas系统中,一些Cas蛋白具有DNA或RNA特异的切割特性,并能将外源DNA(原间隔序列)插入到基因组的CRISPR重复序列中.特别是类型II编码的Cas9蛋白质,在特定RNA的指导下能够特异切割DNA[44,95].因此,基于CRISPR-Cas系统有望发展新的基因组编辑技术,可用于细胞内基因组的操作,如插入、替换基因组中特定序列[44].正如预期的哪样,在2013年初,几个独立的研究小组分别在《Science》、《Nature Biotechnolgy》等杂志上发表研究成果,利用CRISPRCas系统(Cas9)在哺乳等细胞中成功地实现了基因组的靶向编辑[96-100],预示着这项新技术可能成为“精确”操纵基因组的一种非常强大的工具.
在CRISPR-Cas系统的类型II中,已有的研究显示Cas9蛋白在RNA的指导下,能够特异切割核酸[44,95].根据这样的原理,只需通过简单的构建,即将cas9基因和针对基因组靶序列设计的编码“crRNA”的基因一起共转化受体细胞,然后通过RNA定向靶基因序列,并指导Cas9在特异位点切割靶序列,最后依靠细胞内的DNA修复功能,即可实现在特定靶位点对基因组DNA进行修饰,其原理见图6.例如,2013年哈佛医学院的 P.Mali等[96]依据这样的原理,针对哺乳动物细胞,构建了一个优化的cas9基因表达单元,且在cas9基因的3′端融合了一个核定位序列(NLS);同时设计了一个类似 CRISPR-Cas系统类型 II中的 crRNA/tracrRNA融合基因(称为gRNA,guide RNA),并置于真核U6启动子下;然后转化T293和K562细胞系.结果表明对基因组靶基因的定点修饰率在两种细胞中分别达到了10%~25%和8%~13%.其中,PAM位点是实现定点编辑所必需的,因此在选择靶序列时,需要靶序列至少包括部分PAM序列(图6).随后,这一技术和方法迅速发展,并应用于不同的生物种类,如细菌、酵母菌、植物、线虫、果蝇、斑马鱼和小鼠等[101-106],在基因组的定向编辑方面展现出了巨大的应用潜力.对于果蝇和斑马鱼等动物的胚胎细胞[107-108],甚至可以直接将cas9体外转录的mRNA和gRNA混合物注射胚胎细胞,结果表明在果蝇实验中,有88%的胚胎细胞获得定点修饰,而且这些突变系能够转化为稳定的突变体.这种方法的优点是在细胞内不会残留“污染DNA”.
此外,Cas9/gRNA编辑技术可以很容易进行多点突变或大尺度基因组修饰,理论上可以针对多个靶位点序列设计一组gRNA,将其串联表达,转化细胞后,即可在设计的靶位点指导Cas9切割修饰基因组.如2013年H.Wang等[109]同时用编码Cas9的mRNA和针对5个靶位点的gRNA混合物,注射小鼠胚胎干细胞获得了高效的基因组编辑;进一步将两种gRNA注射胚胎细胞,结果显示获得大多数小鼠(80%)都同时在两个位点实现了基因修饰;另外,在注射gRNA的同时,加入突变寡核苷酸,发现能够产生精确的定点突变.Cas9/gRNA基因编辑技术也能创造缺失突变[98],甚至大片段的缺失[110].如在小鼠中,W.Fujii等[110](2013)在需要缺失的靶序列的两端分别设计2个gRNA,然后注射胚胎细胞,结果表明能够造成这2个靶位点之间的基因组序列丢失,而且缺失的序列超过10 kb.
目前,Cas9/gRNA基因组编辑通过干扰目标基因,还能用于基因表达调控的研究.这项发展主要是利用一个核酸酶失活、但保留了DNA结合特性的Cas9突变体.将突变体基因与gRNA一起转化E.coli,Cas9突变蛋白质在gRNA的指导下结合靶序列,如果是启动子区域则阻止RNA聚合酶对启动子的识别和结合,从而降低靶基因的转录[111-112].基于同样的设计,2013年L.A.Gilbert等[113]在真核中利用同样的失活突变体,进一步与特定的效应蛋白质融合表达,在gRNA的指导下可以通过效应蛋白质调控真核基因的转录表达,而且作用非常专一.这些研究事实上建立了一个基于Cas9/gRNA的DNA结合平台,根据融合的效应蛋白基因,可以在特定的基因位点招募或阻止蛋白质,从而干扰靶基因的表达,作者将此作用称为CRISPR干扰(CRISPRi)[111].也有人利用Cas9/gRNA技术,期望编辑细胞内的HIV基因组、并阻止病毒基因的表达,以此探索HIV等传染性疾病治疗的新途径[114].

图6 基于Cas9/gRNA的基组编辑技术[96]Fig.6 Genomic editing technology based on Cas9
基于Cas9/gRNA的基因组编辑技术正处于发展初期,还需要完善以及进一步拓展应用范围.通常采用图6所示的策略,在gRNA的指导下,利用Cas9特异切割双链靶序列,然后依靠细胞的非同源末端的连接(NHEJ)的机制修复切点,并导致靶序列被修改.由于Cas9能够“忍耐”gRNA与靶序列之间的错配,因此可能会导致脱靶,在非靶位点产生造成不需要的突变.已经在一些实验中观察到这种现象[98,115-116],甚至产生较高的脱靶率[117-118].虽然有研究发现利用较长的gRNA,可以降低脱靶率[110];但也有相反的研究结果,认为较短的但活性较低的 gRNA可能提高靶向性[118].此外,降低Cas9/gRNA的表达量也有利于提高靶向性[110,116,118].因此,在利用Cas9/gRNA进行基因组编辑时,必须在“活性”和特异性之间找到平衡,降低脱靶率成为Cas9编辑技术需要解决的问题.最近的一个研究利用Cas9的切口酶(nickase)突变体,通过设计2个gRNA,分别对应靶序列的互补链,这样可利用Cas9的切口酶活性,分别在靶序列的互补链上产生单链切口,然后通过同源重组的修复,结果发现能够极大地降低脱靶率50~1 500倍[119].脱靶率是所有基因组编辑技术所面临的主要问题之一,但是目前关于Cas9核酸酶的生化性质了解很少;并且Cas9的切割特异性还取决于gRNA以及与靶序列的互补配对,因此寻找更多来源的Cas9[120],或直接采用蛋白质工程改变Cas9的核酸酶活性[119]、或者提高特异性也是将来在优化完善Cas9编辑技术值得尝试的研究课题.
5 结语
CRISPR-Cas系统是一些细菌和古菌进化出来的一种适应型的免疫机制,用以抵御外来病毒、质粒等移动元件的入侵.这是继限制修饰系统、非特异核酸酶等后,在原核细菌中发现的又一种新的机制.除此之外,也有研究证明这个系统在某些细菌中还扮演其它的作用.例如,在条件致病菌Pseudononas aeruginosa中,已有实验证明了CRISPR-Cas系统调控生物膜的形成[121-122].另一个例子来自于粘细菌Myxococcus xanthus,当一个CRISPR位点在转座子插入突变后,细胞不能聚集并发育形成子实体,表明 CRISPR与粘细菌的发育有关[123-124].还有研究发现Cas1参与DNA修复,如2011年M.Babu等[125]发现纯化的双链断裂修复酶RecBCD中包括Cas1,而且敲除Cas1后的E.coli细胞对UV和丝裂霉素(mitomycin)更为敏感.
目前,虽然对CRISPR-Cas系统的功能和作用机理以及参与的蛋白质等诸多方面的研究取得了极大的进展;而且基于CRISPR-Cas系统Cas9发展起来的基因组编辑技术也显示出了巨大的潜力,可能成为“精确”操纵基因组的一种革命性的技术[126].但是也要看到,有关CRISPR-Cas系统免疫功能、组织结构的多样性以及作用机理的了解仍然还非常有限,许多过程中的一些细节依然不太清楚.例如:Cas1/Cas2等蛋白在间隔序列获得的过程中如何发挥作用?CRISPR-Cas怎样区别自身和外来的DNA?免疫作用与其它功能之间的联系和进化有何关系?这些问题必将随着研究的深入和发展逐渐明了.
[1]Ishino Y,Shinagawa H,Makino K,et al.Nucleotide-sequence of theIapgene responsible for alkaline-phosphatase isozyme conversion inEscherichia coliand identification of the gene product[J].J Bacteriol,1987,169:5429-5433.
[2]Nakata A,Amemura M,Makino K.Unusual nucleotide arrangement with repeated sequences in theEscherichia coliK-12 chromosome[J].J Bacteriol,1989,171:3553-3556.
[3]Groenen P M,Bunschoten A E,van Soolingen D,et al.Nature of DNA polymorphism in the direct repeat cluster ofMycobacterium tuberculosis;application for strain differentiation by a novel typing method[J].Mol Microbiol,1993,10:1057-1065.
[4]Mojica F J M,Juez G,Rodríguez-Valera F.Transcription at different salinities ofHaloferax mediterraneisequences adjacent to partially modifiedPstI sites[J].Mol Microbiol,1993,9:613-621.
[5]Mojica F J M,Ferrer C,Juez G,et al.Long stretches of short tandem repeats are present in the largest replicons of the archaeaHaloferax mediterraneiandHaloferax volcaniiand could be involved in replicon partitioning[J].Mol Microbiol,1995,17:85-93.
[6]Jansen R,van Embden J D,Gaastra W,et al.Identification of genes that are associated with DNA repeats in prokaryotes[J].Mol Microbiol,2002,43:1565-1575.
[7]Jansen R,van Embden J D,Gaastra W,et al.Identification of a novel family of sequence repeats among prokaryotes[J].OMICS,2002,6:23-33.
[8]Lillestøl R K,Redder P,Garrett RA,et al.A putative viral defence mechanism in archaeal cells[J].Archaea,2006,2:59-72.
[9]Tang T H,Bachellerie J P,Rozhdestvensky T,et al.Identification of 86 candidates for small non-messenger RNAs from the archaeonArchaeoglobus fulgidus[J].Proc Natl Acad Sci USA,2002,99:7536-7541.
[10]Tang T H,Polacek N,Zywicki M,et al.Identification of novel non-coding RNAs as potential antisense regulators in the archaeonSulfolobus solfataricus[J].Mol Microbiol,2005,55:469-481.
[11]Makarova K S,Aravind L,Grishin N V,et al.A DNA repair system specific for thermophilic Archaea and bacteria predicted by genomic context analysis[J].Nucleic Acids Res,2002,30:482-496.
[12]Haft D H,Selengut J,Mongodin E F,et al.A guild of 45 CRISPR-associated(Cas)protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes[J].PLoS Comput Biol,205,1:474-483.
[13]Mojica F J M,Díez-Villaseñor C,García-Martínez J,et al.Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J].J Mol Evol,2005,60:174-182.
[14]Bolotin A,Quinquis B,Sorokin A,et al.Clustered regularly interspaced short palindrome repeats(CRISPRs)have spacers of extrachromosomal origin[J].Microbiol,2005,151:2551-2561.
[15]Pourcel C,Salvignol G,Vergnaud G.CRISPR elements inYersinia pestisacquire new repeats by preferential uptake of bacteriophage DNA and provide additional tools for evolutionary studies[J].Microbiol,2005,151:653-663.
[16]Barrangou R,Fremaux C,Deveau H,et al.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science,2007,315:1709-1712.
[17]Marraffini L A,Sontheimer E J.CRISPR interference limits horizontal gene transfer inStaphylococciby targeting DNA[J].Science,2008,322:1843-1845.
[18]Grissa I,Vergnaud G,Pourcel C.The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J].BMC Bioinf,2007,8:172.
[19]Rousseau C,Nicolas J,Gonnet M.CRISPI:a CRISPR interactive database[J].Bioinformatics,2009,25:3317-3318.
[20]Bland C,Ramsey T L,Sabree F,et al.CRISPR Recognition Tool(CRT):a tool for automatic detection of clustered regularly interspaced palindromic repeats[J].BMC Bioinformatics,2007,8:209.
[21]Skennerton C T,Imelfort M,Tyson G W.Crass:identification and reconstruction of CRISPR from unassembled metagenomic data[J].Nucleic Acids Res,2013,41:e105.
[22]Berg Miller M E,Yeoman C J,Chia N,et al.Phage-bacteria relationships and CRISPR elements revealed by a metagenomic survey of the rumen microbiome[J].Environ Microbiol,2012,14:207-227.
[23]Willner D,Thurber R V,Rohwer F.Metagenomic signatures of 86 microbial and viral metagenomes[J].Environ Microbiol,2009,11:1752-1766.
[24]Lillestøl R K,Shah S A,Brügger K,et al.CRISPR families of the crenarchaeal genusSulfolobus:bidirectional transcription and dynamic properties[J].Mol Microbiol,2009,72:259-272.
[25]Deng L,Kenchappa C S,Peng X,et al.Modulation of CRISPR locus transcription by the repeat-binding protein Cbp1 inSulfolobus[J].Nucleic Acids Res,2012,40:2470-2480.
[26]Agari Y,Sakamoto K,Tamakoshi M,et al.Transcription profile of Thermus thermophilus CRISPR systems after phage infection[J].J Mol Biol,2009,395:270-281.
[27]Kunin V,Sorek R,Hugenholtz P.Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J].Genome Biol,2007,8:R61.
[28]Godde J S,Bickerton A.The repetitive DNA elements called CRISPRs and their associated genes:evidence of horizontal transfer among prokaryotes[J].J Mol Evol,2006,62:718-729.
[29]Chakraborty S,Snijders A P,Chakravorty R,et al.Comparative network clustering of direct repeats(DRs)and cas genes confirms the possibility of the horizontal transfer of CRISPR locus among bacteria[J].Mol Phylogenet Evol,2010,56:878-887.
[30]Han D,Lehmann K,Krauss G.SSO1450-a CAS1 protein fromSulfolobus solfataricusP2 with high affinity for RNA and DNA[J].FEBS Lett,2009,583:1928-1932.
[31]Wiedenheft B,Zhou K,Jinek M,et al.Structural basis for DNase activity of a conserved protein implicated in CRISPR-mediated genome defense[J].Structure,2009,17:904-912.
[32]Beloglazova N,Brown G,Zimmerman MD,et al.A novel family of sequence-specific endoribonucleases associated with the clustered regularly interspaced short palindromic repeats[J].J Biol Chem,2008,283:20361-20371.
[33]Beloglazova N,Petit P,Flick R,et al.Structure and activity of the Cas3 HD nuclease MJ0384,an effector enzyme of the CRISPR interference[J].EMBO J,2011,30:4616-4627.
[34]Sinkunas T,Gasiunas G,Fremaux C,et al.Cas3 is a single stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system[J].EMBO J,2011,30:1335-1342.
[35]Zhang J,Kasciukovic T,White M F.The CRISPR associated protein Cas4 is a 59 to 39 DNA exonuclease with an iron-sulfur cluster[J].PLoS ONE,2013,7:e4723.
[36]Brouns S J,Jore M M,Lundgren M,et al.Small CRISPR RNAs guide antiviral defense in prokaryotes[J].Science,2008,321:960-964.
[37]Jore M M,Lundgren M,van Duijn E,et al.Structural basis for CRISPR RNA-guided DNA recognition by Cascade[J].Nat Struct Mol Biol,2011,18:529-536.
[38]Hale C R,Zhao P,Olson S,et al.RNAguided RNA cleavage by a CRISPR RNA-Cas protein complex[J].Cell,2009,139:945-956.
[39]Haurwitz R E,Jinek M,Wiedenheft B,et al.Sequence-and structure specific RNA processing by a CRISPR endonuclease[J].Science,2010,329:1355-1358.
[40]Reeks J,Sokolowski R D,Graham S,et al.Structure of a dimeric crenarchaeal Cas6 enzyme with an atypical active site for CRISPR RNA processing[J].Biochem J,2013,452:223-230.
[41]Wang R,Preamplume G,Terns M P,et al.Interaction of the Cas6 riboendonuclease with CRISPR RNAs:recognition and cleavage[J].Structure,2011,19:257-264.
[42]Sashital D G,Wiedenheft B,Doudna J A.Mechanism of foreign DNA selection in a bacterial adaptive immune system[J].Mol Cell,2012,46:606-615.
[43]Sapranauskas R,Gasiunas G,Fremaux C,et al.TheStreptococcus thermophilusCRISPR/Cas system provides immunity inEscherichia coli[J].Nucleic Acids Res,2011,39:9275-9282.
[44]Jinek M,Chylinski K,Fonfara I,et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J].Science,2012,337:816-821.
[45]Garneau J E,Dupuis M E,Villion M,et al.The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J].Nature,2010,468:67-71.
[46]Hale C R,Zhao P,Olson S,et al.RNAguided RNA cleavage by a CRISPR RNA-Cas protein complex[J].Cell,2009,139:945-956.
[47]Han D,Krauss G.Characterization of the endonuclease SSO2001 fromSulfolobus solfataricusP2[J].FEBS Lett,2009,583:771-776.
[48]Makarova K S,Haft D H,Barrangou R,et al.Evolution and classification of the CRISPR-Cas systems[J].Nature Rev Microbiol,2011,9:467-477.
[49]Wiedenheft B,Sternberg S H,Doudna J A.RNA-guided genetic silencing systems in bacteria and archaea[J].Nature,2012,482:331-338.
[50]Bhaya D,Davison M,Barrangou R.CRISPR-Cas systems in bacteria and archaea:versatile small RNAs for adaptive defense and regulation[J].Annu Rev Genet,2011,45:273-297.
[51]Tyson G W,Banfield J F.Rapidly evolving CRISPRs implicated in acquired resistance of microorganisms to viruses[J].Environ Microbiol,2008,10:200-207.
[52]Yosef I,Goren M G,Qimron U.Proteins and DNA elements essential for the CRISPR adaptation process inEscherichia coli[J].Nucleic Acids Res,2012,40:5569-5576.
[53]Swarts D C,Mosterd C,van Passel M W,et al.CRISPR interference directs strand specific spacer acquisition[J].PLoS One,2012,7:e35888.
[54]Datsenko K A.Pougach K,Tikhonov A,et al.Molecular memory of prior infections activates the CRISPR/Cas adaptive bacterial immunity system[J].Nat Commun,2012,3:945.
[55]Cady K C,Bondy-Denomy J,Heussler G E,et al.The CRISPR/Cas adaptive immune system ofPseudomonas aeruginosamediates resistance to naturally occurring and engineered phages[J].J Bacteriol,2012,194:5728-5738.
[56]Lopez-Sanchez M J,Sauvage E,Da Cunha V,et al.The highly dynamic CRISPR1 system ofStreptococcus agalactiaecontrols the diversity of its mobilome[J].Mol Microbiol,2012,85:1057-1071.
[57]Erdmann S,Garrett R A.Selective and hyperactive uptake of foreign DNA by adaptive immune systems of an archaeon via two distinct mechanisms[J].Mol Microbiol,2012,85:1044-1056.
[58]Samai P,Smith P,Shuman S.Structure of a CRISPR-associated protein Cas2 fromDesulfovibrio vulgaris[J].Acta Crystallogr F Struct Biol Cryst Commun,2010,66:1552-1556.
[59]Nam K H,Ding F,Haitjema C,et al.Double-stranded endonuclease activity inBacillus haloduransclustered regularly interspaced short palindromic repeats(CRISPR)-associated Cas2 protein[J].J Biol Chem,2012,287:35943-35952.
[60]Arslan Z,Wurm R,Brener O,et al.Double-strand DNA end-binding and sliding of the toroidal CRISPR-associated protein Csn2[J].Nucleic Acids Res,2013,doi:10.1093/nar/gkt315.
[61]Goren M G,Yosef I,Auster O,et al.Experimental definition of a clustered regularly interspaced short palindromic duplication inEscherichia coli[J].J Mol Biol,2012,423:14-16.
[62]Westra E R,Brouns S J.The rise and fall of CRISPRs-Dynamics of spacer acquisition and loss[J].Mol Microbiol,2012,85:1021-1025.
[63]Wiedenheft B,Lander G C,Zhou K,et al.Structures of the RNA-guided surveillance complex from a bacterial immune system[J].Nature,2011,477:486-489.
[64]Lintner N G,Frankel K A,Tsutakawa S E,et al.The structure of the CRISPR-associated protein Csa3 provides insight into the regulation of the CRISPR/Cas system[J].J Mol Biol,2011,405:939-955.
[65]Plagens A,Tjaden B,Hagemann A,et al.Characterization of the CRISPR/Cas subtype I-A system of the hyperthermophilic crenarchaeonThermoproteus tenax[J].J Bacteriol,2012,194:2491-2500.
[66]Nam K H,Haitjema C,Liu X,et al.Cas5d protein processes pre-crRNA and assembles into a Cascade-like interference complex in subtype I-C/Dvulg CRISPR-Cas System[J].Structure,2012,20:1574-1584.
[67]Haurwitz R E,Jinek M,Wiedenheft B,et al.Sequence-and structure-specific RNA processing by a CRISPR endonuclease[J].Science,2010,329:1355-1358.
[68]Sternberg S H,Haurwitz R E,Doudna J A.Mechanism of substrate selection by a highly specific CRISPR endoribonuclease[J].RNA,2012,18:661-672.
[69]Haurwitz R E,Sternberg S H,Doudna J A.Csy4 relies on an unusual catalytic dyad to position and cleave CRISPR RNA[J].EMBO J,2012,31:2824-2832.
[70]Sashital D G,Wiedenheft B,Doudna J A.Mechanism of foreign DNA selection in a bacterial adaptive immune system[J].Mol Cell,2012,46:606-615.
[71]Westra E R,Semenova E,Datsenko K A,et al.Type I-E CRISPR-Cas systems discriminate target from non-target DNA through base pairing-independent PAM recognition[J].PLoS Genet,2013,9:e1003742.
[72]Semenova E,Jore M M,Datsenko K A,et al.Interference by clustered regularly interspaced short palindromic repeat(CRISPR)RNA is governed by a seed sequence[J].Proc Natl Acad Sci USA,2011,108:10098-10103.
[73]Westra E R,van Erp P B,Kunne T,et al.CRISPR immunity relies on the consecutive binding and degradation of negatively supercoiled invader DNA by Cascade and Cas3[J].Mol Cell,2012,46:595-605.
[74]Howard J A,Delmas S,Ivancic-Bace I,et al.Helicase dissociation and annealing of RNA-DNA hybrids byEscherichia coliCas3 protein[J].Biochem J,2011,439:85-95.
[75]Deltcheva E,Chylinski K,Sharma C M,et al.CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III[J].Nature,2011,471:602-607.
[76]Garneau J E,Dupuis M E,Villion M,et al.The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J].Nature,2010,468:67-71.
[77]Deveau H,Barrangou R,Garneau J E,et al.Phage response to CRISPR-encoded resistance inStreptococcus thermophilus[J].J Bacteriol,2008,190:1390-1400.
[78]Gudbergsdottir S,Deng L,Chen Z,et al.Dynamic properties of theSulfolobusCRISPR/Cas and CRISPR/Cmr systems when challenged with vector-borne viral and plasmid genes and protospacers[J].Mol Microbiol,2011,79:35-49.
[79]Westra E R,Pul Ü,Heidrich N,et al.H-NS-mediated repression of CRISPR-based immunity inEscherichia coliK12 can be relieved by the transcription activator LeuO[J].Mol Microbiol,2010,77:1380-1393.
[80]Mojica F J,Díez-Villaseñor C.The on-off switch of CRISPR immunity against phages inEscherichia coli[J].Mol Microbiol,2010,77:1341-13.
[81]Agari Y,Sakamoto K,Tamakoshi M,et al.Transcription profile of thermos thermophilus CRISPR systems after phage infection[J].J Mol Biol,2010,395:270-281.
[82]Hatoum-Aslan A,Maniv I,Marraffini L A.Mature clustered,regularly interspaced,short palindromic repeats RNA(crRNA)length is measured by q rule mechanism anchored at the precursor processing site[J].Proc Natl Acad Sci USA,2011,108:21218-21222.
[83]Carte J,Wang R,Li H,et al.Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes[J].Genes Dev,2008,22:3489-3496.
[84]Carte J,Pfister N T,Compton M M,et al.Binding and cleavage of CRISPR RNA by Cas6[J].RNA,2010,16:2181-2188.
[85]Shao Y,Li H.Recognition and cleavage of a nonstructured CRISPR RNA by its processing endoribonuclease Cas6[J].Structure,2013,21:385-393.
[86]Wang R,Preamplume G,Terns M P,et al.Interaction of the Cas6 riboendonuclease with CRISPR RNAs:recognition and cleavage[J].Structure,2011,19:257-264.
[87]Hale C R,Zhao P,Olson S,et al.RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex[J].Cell,2009,139:945-956.
[88]Hale C R,Majumdar S,Elmore J,et al.Essential features and rational design of CRISPR RNAs that function with the Cas RAMP module complex to cleave RNAs[J].Mol Cell,2012,45:292-302.
[89]Zhang J,Rouillon C,Kerou M,et al.Structure and mechanism of the CMR complex for CRISPR-mediated antiviral immunity[J].Mol Cell,2012,45:303-313.
[90]Groenen P M,Bunschoten A E,van Soolingen D,et al.Nature of DNA polymorphism in the direct repeat cluster ofMycobacterium tuberculosis;application for strain differentiation by a novel typing method[J].Mol Microbiol,1993,10:1057-1065.
[91]Brudey K,Driscoll J R,Rigouts L,et al.Mycobacterium tuberculosiscomplex genetic diversity:mining the fourth international spoligotyping database(SpolDB4)for classification,population genetics and epidemiology[J].BMC Microbiol,2006,6:23.
[92]Snyder J C,Bateson M M,Lavin M,et al.Use of cellular CRISPR(clusters of regularly interspaced short palindromic repeats)spacer-based microarrays for detection of viruses in environmental samples[J].Appl Environ Microbiol,2010,76:7251-7258.
[93]Garcia-Heredia I,Martin-Cuadrado A B,Mojica FJ,et al.Reconstructing viral genomes from the environment using fosmid clones:the case of haloviruses[J].PLoS One,2012,7:e33802.
[94]Pride D T,Salzman J,Relman D A.Comparisons of clustered regularly interspaced short palindromic repeats and viromes in human saliva reveal bacterial adaptations to salivary viruses[J].Environ Microbiol,2012,14:2564-2576.
[95]Barrangou R.RNA-mediated programmable DNA cleavage[J].Nat Biotechnol,2012,30:836-838.
[96]Mali P,Yang L,Esvelt K M,et al.RNA-guided human genome engineering via Cas9[J].Science,2013,339:823-826.
[97]Cong L,Ran F A,Cox D,et al.Multiplex genome engineering using CRISPR/Cas systems[J].Science,2013,339:819-823.
[98]Jiang W,Bikard D,Cox D,et al.RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J].Nat Biotechnol,2013,31:233-239.
[99]Hwang W Y,Fu Y,Reyon D,et al.Efficient genome editing in zebrafish using a CRISPR-Cas system[J].Nat Biotechnol,2013,31:227-229.
[100]Shen B,Zhang J,Wu H,et al.Generation of gene-modified mice via Cas9/RNA-mediated gene targeting[J].Cell Res,2013,23:720-723.
[101]Cho S W,Kim S,Kim J M,et al.Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease[J].Nat Biotechnol,2013,31:230-232.
[102]Gratz S J,Cummings A M,Nguyen J N,et al.Genome engineering of Drosophila with the CRISPR RNA-guided Cas9 nuclease[J].Genetics,2013,194:1029-1035.
[103]Xiao A,Wang Z,Hu Y,et al.Chromosomal deletions and inversions mediated by TALENs and CRISPR/Cas in zebrafish[J].Nucleic Acids Res,2013,41:e141.
[104]Di Carlo J E,Norville J E,Mali P,et al.Genome engineering inSaccharomyces cerevisiaeusing CRISPR-Cas systems[J].Nucleic Acids Res,2013,41:4336-4343.
[105]Hou Z,Zhang Y,Propson N E,et al.Efficient genome engineering in human pluripotent stem cells using Cas9 fromNeisseria meningitides[J].Proc Natl Acad Sci USA,2013,110:15644-15649.
[106]Nekrasov V,Staskawicz B,Weige D,et al.Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease[J].Nat Biotechnol,2013,31:691-693.
[107]Bassett A R,Tibbit C,Ponting C P,et al.Highly efficient targeted mutagenesis ofDrosophilawith the CRISPR/Cas9 System[J].Cell Rep,2013,4:220-228.
[108]Chang N,Sun C,Gao L,et al.Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos[J].Cell Res,2013,23:465-472.
[109]Wang H,Yang H,Shivalila C S,et al.One-step generation of mice carrying mutations in multiple genes by CRISPR/Casmediated genome engineering[J].Cell,2013,153:910-918.
[110]Fujii W,Kawasaki K,Sugiura K,et al.Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease[J].Nucleic Acids Res,2013,doi:10.1093/nar/gkt772.
[111]Qi L S,Larson M H,Gilbert L A,et al.Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J].Cell,2013,152:1173-1183.
[112]Bikard D,Jiang W,Samai P,et al.Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system[J].Nucleic Acids Res,2013,41:7429-7437.
[113]Gilbert L A,Larson M H,Morsut L,et al.CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J].Cell,2013,154:442-451.
[114]Ebina H,Misawa N,Kanemura Y,et al.Harnessing the CRISPR/Cas9 system to disrupt latent HIV-1 provirus[J].Sci Rep,2013,3:2510.
[115]Mali P,Aach J,Stranges P B,et al.Cas9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering[J].Nat Biotechnol,2013,31:833-838.
[116]Hsu P D,Scott D A,Weinstein J A,et al.DNA targeting specificity of RNA-guided Cas9 nucleases[J].Nat Biotechnol,2013,31:827-832.
[117]Fu Y,Foden J A,Khayter C,et al.High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells[J].Nat Biotech,2013,31:822-826.
[118]Pattanayak V,Lin S,Guilinger J P,et al.High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity[J].Nat Biotechnol,2013,31:839-843.
[119]Ran F A,Hsu P D,Lin C Y,et al.Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J].Cell,2013,154:1380-1389.
[120]Esvelt K M,Mali P,Braff J L,et al.Orthogonal Cas9 proteins for RNA-guided gene regulation and editing[J].Nat Methods,2013,doi:10.1038/NMETH.2681.
[121]Zegans M E,Wagner J C,Cady K C,et al.Interaction between bacteriophage DMS3 and host CRISPR region inhibits group behaviors ofPseudomonas aeruginosa[J].J Bacteriol,2009,191:210-219.
[122]Cady K C,O’Toole G A.Non-identity-mediated CRISPR-bacteriophage interaction mediated via the Csy and Cas3 proteins[J].J Bacteriol,2011,193:3433-3445.
[123]Viswanathan P,Murphy K,Julien B,et al.Regulation of dev,an operon that includes genes essential forMyxococcus xanthusdevelopment and CRISPR-associated genes and repeats[J].J Bacteriol,2007,189:3738-3750.
[124]Thony-Meyer L,Kaiser D.devRS,an autoregulated and essential genetic locus for fruiting body development in Myxococcus xanthus[J].J Bacteriol,1993,175:7450-7462.
[125]Babu M,Beloglazova N,Flick R,et al.A dual function of the CRISPR-Cas system in bacterial antivirus immunity and DNA repair[J].Mol Microbiol,2011,79:484-502.
[126]Mali P,Esvelt K M,Church G M.Cas9 as a versatile tool for engineering biology[J].Nat Methods,2013,10:957-963.
