定量蛋白质组学分析ClpS在分枝杆菌耐药中的功能
2014-06-15古丽莎娜阿地里江冯杉米凯霞邓海腾
古丽莎娜·阿地里江,冯杉,米凯霞,邓海腾
定量蛋白质组学分析ClpS在分枝杆菌耐药中的功能
古丽莎娜·阿地里江1,冯杉1,米凯霞2,邓海腾1
1清华大学生命科学学院教育部生物信息学重点实验室,北京100084 2中国科学院微生物研究所,北京100101
ClpS是原核生物蛋白质降解复合物ClpAPS的重要组成成分,它可以识别某些特定的氨基酸序列并将其呈递给ClpAP以促进其降解。同时,ClpS也抑制了其他蛋白质底物的降解。本研究通过在耻垢分枝杆菌中过度表达ClpS,发现所构建的重组菌株提高了利福平的抗药性。应用定量蛋白质组学技术,我们系统地分析了过度表达ClpS对于细菌蛋白质组的影响,并推测出细菌抗利福平的分子机制:ClpS促进稳态的调整、促进药物沉降以及加速药物代谢。本研究首次通过改变细菌降解复合物的相关蛋白的表达增加细菌的抗药性,并证明蛋白质组学技术是细菌的抗药性研究以及耐药株筛选的重要工具。
ClpAPS蛋白降解系统,ClpS,利福平,定量蛋白质组学
ClpAPS是原核生物存在于胞质中的蛋白质降解系统,其结构和序列与真核生物的26S蛋白酶体同源。ClpAPS是大的蛋白质复合物,由具有蛋白酶功能的ClpP、具有识别及去折叠底物功能的ClpA和具有进一步调控ClpAP降解功能的ClpS组成。ClpP由两个背向的七聚体构成,其结构组成类似于GroEL;ClpA则是中空六聚体,其N端结构域负责识别并去折叠底物,C端结构域则把已经去折叠的底物传递给ClpP进行降解[1-2]。ClpS与ClpA以1∶1比例形成结合的复合物,可进一步对底物的降解过程进行调控[3-4]。
目前关于ClpS在全细胞蛋白质降解过程中的调控机制还不是很清楚,其功能研究显示,对于某些蛋白质底物(例如N端含有FR序列的N-end rule蛋白)ClpS具有促进降解的功能;而对于另一些蛋白质底物(例如C端含有ssrA标签的蛋白)具有抑制降解的功能[1,3-7]。通过控制ClpS的表达水平可以改变细菌内蛋白质的降解途径,从而间接调控其他蛋白在细胞内的水平。由于调控细胞内蛋白水平的蛋白质降解系统具有高度的复杂性,因此研究相关基因过表达的菌株对理解蛋白质降解系统在细菌生长及抗药性等方面都具有重要的价值。
利福平(Rifampicin),化学名称为3-[[(4-甲基-1-哌嗪基)亚氨基]甲基]-利福霉素,是一种广谱性的半合成抗生素。在治疗结核杆菌、麻风病菌等其他细菌感染时,它通常与其他药物联合使用,如异烟肼等[8-9]。利福平通过结合DNA依赖性的RNA聚合酶来抑制细菌RNA的合成,从而达到杀菌的效果[10-11]。目前对利福平有抗性的菌株主要是基于RNA聚合酶的改变以降低与利福平的亲和力[11-12]。
在本项研究中,我们首先在结核分枝杆菌Mycobacterium tuberculosis K.的模式菌株耻垢分枝杆菌Mycobacterium smegmatis L.&N.中建立了过度表达ClpS的重组菌株,分析显示过度表达ClpS的M.smegmatis增加了利福平抗药性。进一步利用TMT标记的定量蛋白质组学技术,在过度表达ClpS的菌中鉴定到了199个上调表达的蛋白和117个下调表达的蛋白。通过功能性分类和代谢途径分析,阐述了过表达ClpS导致细菌利福平抗性增加的分子机制。
1 材料与方法
1.1 质粒、菌株及试剂
质粒pMV261、p0004s和phAE159,菌株M.smegmatis mc2155均由本实验室保存。细菌基因组DNA提取试剂盒、PCR所用DNA聚合酶购自天根生化科技有限公司。分子克隆所用限制性内切酶、T载体购自TaKaRa宝生物公司。噬菌体包装所用的MaxPlax packaging extract购自Epicenter Biotechnologies公司。7H9、利福平、二硫苏糖醇(DTT)、碘乙酰胺(IAM)购自Sigma公司。TMT试剂购自Thermo公司。酶解用胰蛋白酶(Trypsin)购自Promega公司。其他与液质相关的化学试剂均采自国内,色谱纯级别。
1.2 clpS敲除和过度表达菌株的构建
用细菌基因组DNA提取试剂盒提取M.smegmatis mc2155菌株基因组DNA后,使用引物4910-F和4910-R扩增clpS(msmeg_4910)基因片段,并构建到T载体上,经测序验证其序列正确。具体引物序列见表1。将clpS序列亚克隆到pMV261载体上,并电转至M.smegmatis mc2155菌株中,获得过度表达clpS的菌株。
利用分枝杆菌噬菌体的特异转导法(Mycobacteriophages in specialized transduction)敲除clpS[13]。具体步骤如下:利用4910LL和4910LR引物及4910RL和4910RR引物分别扩增clpS基因1.2 kb的上游和下游区域,具体引物序列见表1;利用限制性内切酶DraⅢ酶切PCR扩增产物,克隆到p0004s质粒上,所获得的中间载体与phAE159分别利用PacⅠ酶切和连接,连接产物经过体外包装获得含有clpS上游和下游及筛选标记潮霉素抗性基因的噬菌粒,进一步将其转移到M.smegmatis获得相应的噬菌体;然后将高浓度的重组噬菌体与M.smegmatis mc2155菌株共培养3 h后,在含有潮霉素的7H10平板上筛选。培养5 d后,挑选单菌落,验证获得clpS敲除菌株。
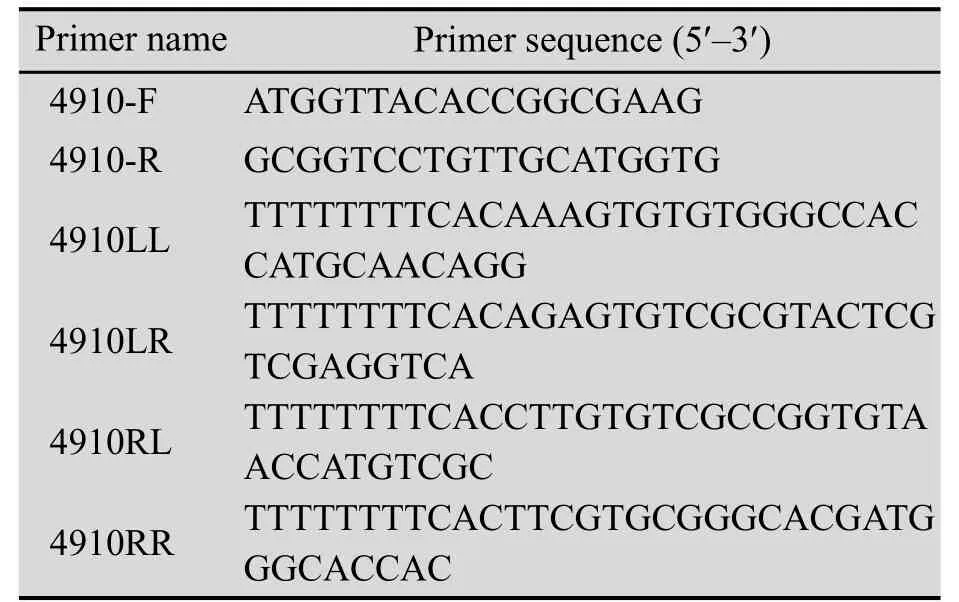
表1clpS基因过表达和敲除所需扩增引物信息Table 1Primers for overexpression and knock-out of gene clpS
1.3 细菌生长曲线的测定
配制含10%ADS、0.5%甘油和0.05%Tween 80的7H9液体培养基,分别在无抗性及加入10 mg/L利福平的7H9液体培养基中测定生长曲线。野生型mc2155、敲除clpS的突变菌株及过度表达clpS菌株均按照1∶100接种量接菌,培养至OD600达到0.8–1.0;将上述菌液分别稀释至OD600为0.01,37℃下继续培养,约每隔4 h取1 mL菌液,测量其在600 nm处的吸光度值(Ultrospec 2 100紫外可见分光光度计GE Healthcare),记录实验数据。
1.4 胶内酶解与TMT标记蛋白质定量
分别培养10 mL野生菌株和10 mL clpS过度表达菌株,用超声破碎仪进行细菌细胞裂解,各取40 μg的裂解蛋白在SDS-PAGE蛋白胶分离,考马斯亮蓝染色后,将胶条切为15块,分别进行脱色、DTT打开二硫键和IAM烷基化处理。在50 mmol/L NH4HCO3溶液中使用胰酶酶解蛋白过夜,之后用含1%TFA和50%ACN的NH4HCO3溶液将酶解后的肽从胶内萃取出来,再用真空浓缩干燥仪将体积缩小。
使用Thermo公司的TMT试剂化学标记酶解后多肽的伯氨基[14],用以对比clpS过度表达菌株与野生型菌株在蛋白水平表达的差异谱。简单来讲,TMT试剂溶解于无水乙腈之中,并加到每个酶解的产物中。在室温下反应1 h之后,用5%的羟氨终止标记反应。TMT标记的肽使用C18的反相柱除盐,并准备上机Q Exactive (Thermo)。我们使用TMT2-126试剂来标记野生型菌株蛋白酶解后所产生的肽,TMT2-127试剂来标记clpS过度表达菌株蛋白酶解后所产生的肽。TMT2-126试剂标记的与TMT2-127试剂标记的肽或蛋白的比例由Proteome Discovery软件(Thermo)所计算。
1.5LC-MS/MS分析及数据处理
对于LC-MS/MS分析,酶解后的产物或TMT标记后的肽在纳升HPLC系统(EASY-nLCⅡTM)上分离,梯度洗脱时间为65 min,流速为0.25 μL/min;之后直接喷射入Thermo Q Exactive质谱仪中。所用分析柱是自制的填充了C18填料(孔径300 Å、粒径5 μm)的石英毛细管柱(直径75 μm,长度150 mm)。流动相A是含0.1%甲酸的水溶液,流动相B是含0.1%甲酸的乙腈溶液。Q Exactive质谱仪由Xcalibur 2.07软件在数据依赖性获取(Data-dependent acquisition)模式下操控,扫描模式为在Orbitrap中(400–1 800 m/z,75 000分辨率)经过一个全谱扫描后,紧接着有10个数据依赖性的二级质谱扫描(碎片能量为30%HCD)。对于每个LC-MS/MS运行的样品,所有得到的一级和二级谱图都被送到Proteome Discovery软件中在M. smegmatis库中进行搜索。
2 结果与分析
2.1 clpS敲除菌株和clpS过度表达菌株的构建及ClpS蛋白表达水平分析
利用分枝杆菌的噬菌体的特异转导法(Mycobacteriophages in specialized transduction)获得clpS敲除菌株。同时,将clpS构建到pMV261获得了clpS过度表达菌株。如图1A插入图片所示,RT-PCR实验结果显示,与野生型(泳道2)相比较,过度表达clpS的菌株(泳道3)其转录水平明显增高;而在敲除clpS的菌株(泳道4)检测不到clpS基因表达。我们进一步利用质谱方法比较了两株菌的ClpS蛋白表达水平。图1展示了一个ClpS蛋白酶解产生的二价肽段LHAAGLATMQQDR的一级质谱,其单同位素峰的质核比在799.40(图1A);其二级质谱b系列离子自b2至b6、y系列离子自y4至y13都能够很好地匹配上(图1B)。在过度表达菌株中ClpS的表达在分值及二级质谱数目上均明显高于野生型;另外,在敲除的突变菌株中,基本找不到匹配程度或得分较好的肽段。我们的实验表明3种菌株中ClpS的表达在转录和翻译水平上存在着差异。
2.2 利福平对于细菌生长的影响
M.smegmatis与M.tuberculosis同属于分枝杆菌科Mycobacteriaceae分子杆菌属Mycobacterium,M.smegmatis是一种条件性致病菌。M.smegmatis全基因组测序分析显示其与M.tuberculosis在毒力基因、双组分条件系统及转录调控因子等方面具有较高同源性,又因为其生长速度优势,M.smegmatis成为较好的M. tuberculosis标准模式菌种[15-17]。M.smegmatis细菌培养在7H9液体培养基中,通过测定600 nm处的吸光度来反映其生长的程度。如图2所示,对于野生型的菌株,大约培养至8–9 h时进入对数期,20 h之后基本进入平台期。而在培养基中加入10 mg/L利福平后,15 h时细菌进入对数期,30 h之后才进入平台期。从整个生长趋势上看,利福平的加入使野生型M.smegmatis细菌的生长滞后了6–7 h。
对于clpS过表达和敲除的菌株来说,其本身生长速率慢于野生型,尤其过表达的菌株还滞后于敲除的菌株约2 h(图2)。加入利福平后,过表达和敲除的菌株的生长速率虽然同样变慢,但其滞后的程度与野生型却不相同:过表达菌株的滞后时间约2–3 h,敲除菌株的滞后时间则为8–9 h。说明利福平对于clpS敲除菌株生长的抑制效果最为明显,野生型次之,clpS过表达菌株则最不敏感。细菌生长曲线经过多次实验验证,有效证明了过表达ClpS蛋白能够提升M.smegmatis对于利福平的抗性。
2.3 过度表达clpS菌株蛋白质组学的分析
关于ClpS的研究显示ClpS参与调节细胞蛋白质的降解,因此推测过度表达ClpS可能在蛋白质水平影响相关蛋白的细胞含量,从而影响了对利福平的抗性。为验证此假设,我们利用TMT标记的定量蛋白质组学技术,系统地分析探究了过度表达ClpS蛋白对细菌蛋白质组变化的影响。
TMT标记技术是目前定量蛋白质组学研究的一项主要手段,通过比较二级质谱图中报告离子的强度来反映对应肽段,以及肽段对应蛋白质的相对含量,图3展示了TMT分子的结构及标记流程。对两个样品中的蛋白进行酶解,所产生的肽段分别用两种不同的TMT试剂进行比较,同一序列的肽段在标记后分子量相同,但在二级质谱图中,产生了两个不同质量的报告基团,如126 Da和127 Da的碎片离子,这两个碎片离子强度的比值代表了产生该肽段的蛋白在两个样品中的相对丰度比值。

图2野生型(方块)、过表达clpS菌株(圆圈)和敲除clpS菌株(三角形)在未加利福平(空心)和10 mg/L利福平(实心)培养基中的生长曲线Fig.2Growth rates measured for the wild-type strain (WT,square),clpS-overexpressionstrain(4910up, circle)and clpS knock-out strain(4910down,triangle) growing in the culture medium treated with(solid)or without(hollow)10 mg/L rifampicin(R).

图3TMT定量标记质谱技术流程示意图(改编自Thermo Scientific TMT使用说明图)(A)和TMT分子同位素标记前(B)和标记后(C)的结构示意图Fig.3Schematic of the quantitation method with TMT Isobaric Mass Tagging.(A)Experimental procedure.(B) Structure of the TMT reagent,which has three parts:the amine-reactive group to modify α-amino groups of N-terminus and the ε-amino groups of lysine residue in tryptic peptides,the mass reporter group that is used in the MS/MS spectrum for quantitation,the spacer arm that balances the mass of reporter group by incorporation of stable isotopic atoms.(C)Structures of isobaric reagents:TMT2-126 and TMT2-127,*indicates the position of13C atom. Peptides with the same sequence generated from two samples have the identical molecular weight after TMT-labeling, but they generate two reporter ions in MS/MS spectra.The intensity ratios of the reporter ions represent the relative protein rations from two samples.
以细菌铁蛋白(Bacterioferritin)为例,图4列出了其被TMT标记的一段二价肽I(TMT)LLLDGLPNYQR的MS/MS二级质谱图,它的单同位素峰的质核比在820.48。碎片后的b、y系列离子用于肽段序列的匹配以确定肽的序列,而TMT试剂断裂后的报告基团则在低质核比的区域被检测到。从图上看,质量为127.13 Da的报告基团的强度是126.13 Da报告基团强度的2.7倍,说明该肽段在clpS过表达菌株中较之野生型提高了2.7倍。通过平均该蛋白所检测到的所有二级质谱的定量信息,即可计算出细菌铁蛋白在过表达菌株中升高的比例(1.64倍)。

图4 细菌铁蛋白一段TMT标记肽I(TMT)LLLDGLPNYQR的二级质谱图Fig.4MS/MS of a TMT-labeled tryptic peptide ILLLDGLPNYQR of bacterioferritin.Samples from the WT strain were labeled with TMT2-126,while those from clpS-overexpression strain were labeled with TMT2-127.
经过3次独立重复实验,我们一共在M.smegmatis中鉴定到了2 000个蛋白,其中有199个上调和117个下调的蛋白。使用David软件对有变化的蛋白进行了功能分类(图5),我们发现变化最多的都是与细胞代谢过程相关的蛋白,分别占上调和下调蛋白比例的32%和29%。其他与氧化还原和转录翻译相关的蛋白也占有相当比例。我们选取了一些具有典型变化的上调和下调蛋白分别列于图6和图7中。
此外,我们对这些变化蛋白进行了细胞位置定位,除去一些未知功能的蛋白外,大部分表达水平变化的蛋白都定位于细胞质中,也有少数定位在细胞膜及周质中,包括上调的23个蛋白,例如细菌铁蛋白Bacterioferritin,以及下调的5个蛋白,例如孔蛋白MspA。

图5 过表达clpS菌株中相比野生型上调的199个蛋白(A)和下调的117个蛋白(B)的蛋白功能分析Fig.5Functional classification of differentially expressed proteins between clpS-overexpression strain and wild-type strain.(A)199 up-regulated proteins.(B)117 down-regulated proteins.
从功能分类上看,ClpS蛋白过度表达的影响涉及到细菌生化的多种途径及功能。首先,很多核酸结合蛋白与转录调节因子的水平发生了变化(图6A和7A),它们的增多或减少注定了ClpS的变化对于细胞内其他蛋白的影响是复杂的,不一定停留在降解的层面,可能影响了一些基因的转录。另外,与蛋白质折叠相关的分子伴侣蛋白和与氧化还原相关的酶也都有一定程度的上调(图6B),这对于保护蛋白并维持细胞稳态具有积极意义。而在与膜相关的蛋白中,蛋白质的表达变化影响了细胞膜的通透性和对药物的敏感性,例如孔蛋白MspA的下调会降低分枝杆菌属细胞对抗生素药物的敏感性,从而产生了一定的耐药性[18-19]。

图6 4组上调蛋白在clpS过表达菌株(灰色)与野生型(白色)的含量对比(TMT标记定量结果)Fig.6Quantitation of selected up-regulated proteins:clpS-overexpression strain(grey)and wild-type strain(white). (A)DNA binding proteins and transcriptional regulators.(B)Chaperone proteins and oxidation-reduction related enzymes.(C)Bacterioferritin and acetyltransferases.(D)Acetyl-CoA synthesis and lipid metabolism related enzymes.
以上这些因素都有可能提升细菌对于利福平的耐药性,然而,细菌铁蛋白(Bacterioferritin)和一系列乙酰基转移酶(Acetyltransferase)表达水平的提高,可能更为直接地导致了细菌耐药性的提升。
2.4过度表达clpS菌株抗利福平机制的推测
首先,如图6 C所示,细菌铁蛋白(Bacterioferritin)在ClpS过表达菌株中相比于野生型提高了1.64倍。细菌铁蛋白主要分布在细胞膜上,由24个同源亚基构成一个中空的高度对称球壳状蛋白,并包含了12个铁卟啉(Heme B),可以摄入、运输和存储大量的铁离子[20-21]。细菌铁蛋白的增多一般暗示细菌对于铁离子摄入的增加,而Fe3+可以和利福平形成微溶于水的复合物[22],铁离子的增多可以有效降低细胞内利福平的浓度。其次,除细菌铁蛋白外,还有3种乙酰基转移酶(Acetyltransferase)有1.5至2倍的上调,这也能有助于加速利福平的代谢。除酚羟基氧化成苯醌以外,利福平的一个主要代谢途径是一个酯官能团水解生成25-去乙酰化利福平;而一些乙酰基转移酶可以参与催化此类反应[23-25]。乙酰基转移酶的上调说明利福平的代谢可能在一定程度上得到了加速,从而起到解毒的作用。
综上,相比于细胞整体稳态的提升,我们认为,铁离子摄入增加和利福平代谢加速,是clpS过表达菌株抗药性增强的主要原因。
此外,实验还发现,受蛋白降解速率变慢影响,多种与氨基酸合成相关的酶也有不同程度的下调,例如图7B列举的乳酸乙酰合成酶(Acetolactate synthase)可以催化支链氨基酸的合成。另外,与脂肪酸合成和氧化相关的一些酶也分别有不同程度的上调和下调(图6D和7A)。我们认为利福平脱乙酰基导致细胞内乙酸浓度的上升,多余的乙酸用来合成乙酰辅酶A,并间接影响到了脂类代谢。如脂肪酸合成酶(Fatty acid synthase),其表达量在过表达的菌株中提升了近3倍(图8),说明脂肪酸的合成可能被活化。

图7 2组下调蛋白在clpS过表达菌株(灰色)与野生型(白色)的含量对比(TMT标记定量结果)Fig.7Quantitation of selected down-regulated proteins:clpS-overexpression strain(grey)and wild-type strain (white).(A)Transcriptional regulators and fatty acid oxidation related enzymes.(B)Amino acid synthesis related enzymes and alcohol dehydrogenases.
3讨论
ClpS是蛋白质降解系统ClpAPS的重要组成部分,它能够引起细菌内诸多蛋白水平的变化,从而改变细菌本身的性质。本文首次报道了过表达ClpS蛋白可以增强细菌对于利福平的耐药性,并使用定量蛋白质组学的方法系统地探讨了过表达菌株相比野生型菌株蛋白质组的改变,并推测ClpS对其他蛋白水平的影响不一定通过降解途径,还会影响到一些转录因子的水平。
ClpS蛋白的过表达能够引起细胞内分子伴侣蛋白和氧化还原相关酶水平的上调,从而增强整个细胞的稳态,以提高细菌对利福平的耐药性。此外,细菌铁蛋白和乙酰基转移酶水平的上升对于利福平的代谢有直接的作用:前者能够增加铁离子的摄入以促进药物沉降,后者则可以加强利福平的去乙酰化以加速药物代谢。因此我们推测过表达ClpS可以促进药物沉降和加速药物代谢以增加对利福平的抗药性。而细胞膜孔蛋白MspA的下调则在一定程度上减少了细菌对利福平的摄入。图9总结了有关ClpS过表达引发的细胞内一系列的变化,无论是通过直接地调控降解途径,还是间接地干扰转录水平,ClpS都会影响这些功能蛋白的水平,从而增强细菌对于利福平的抗性,并在一定程度上抑制了氨基酸的合成并活跃了脂肪酸的合成过程。
综上所述,ClpS过表达影响细菌的生长和耐药性。应用定量蛋白质组学的技术,我们发现ClpS过表达引起316个蛋白表达水平的变化,并推测了ClpS过表达增加耐药性的机理。本研究为工程上研究细菌的耐药性以及筛选新的耐药菌株提供了一个新的思路。
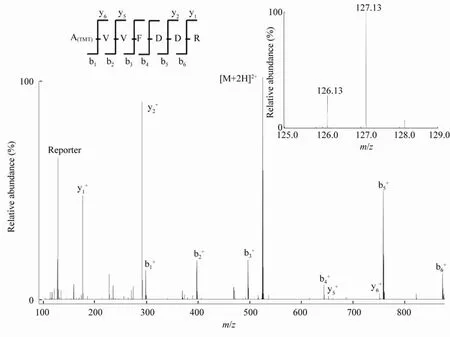
图8 脂肪酸合成酶一段TMT标记肽A(TMT)VVFDDR的二级质谱图Fig.8The MS/MS of a TMT-labeled peptide AVVFDDR of fatty acid synthase.Samples from the WT strain were labeled with TMT2-126,while those from clpS-overexpression strain were labeled with TMT2-127.

图9 过表达ClpS蛋白导致的一些蛋白水平的变化以及可能导致的对利福平抗性增加的机理Fig.9A scheme of proposed mechanisms of drug resistance in rifampicin-treated M.smegmatis.
REFERENCES
[1]KressW,MaglicaZ,Weber-BanE.Clp chaperone-proteases:structure and function.Res Microbiol,2009,160(9):618−628.
[2]Effantin G,Ishikawa T,De Donatis GM,et al. Local and global mobility in the ClpA AAA+ chaperone detected by cryo-electron microscopy functional connotations.Structure,2010,18(5): 553−562.
[3]Dougan DA,Reid BG,Horwich AL,et al.ClpS,a substrate modulator of the ClpAP machine.Mol Cell,2002,9(3):673−683.
[4]Román-Hernández G,Hou JY,Grant RA,et al.The ClpS adaptor mediates staged delivery of N-end rule substrates to the AAA+ClpAP protease.Mol Cell,2011,43(2):217−228.
[5]Erbse A,Schmidt R,Bornemann T,et al.ClpS is an essential component of the N-end rule pathway in Escherichiacoli.Nature,2006,439(7077): 753−756.
[6]Wang KH,Sauer RT,Baker TA.ClpS modulates butisnotessentialforbacterialN-endrule degradation.Genes Dev,2007,21(4):403−408.
[7]Ronny S,Regina Z,Bernd B,et al.ClpS is the recognitioncomponentforEscherichiacoli substrates of the N-end rule degradation pathway. Mol Microbiol,2009,72(2):506−517.
[8]Stephen DL,Alimuddin IZ.Tuberculosis.Lancet, 2011,378(9785):57−72.
[9]Masters SB,Trevor AJ,Katzung BG.Katzung& Trevor's pharmacology.New York:McGraw-Hill, 2005,Medical Pub Division.
[10]Calvori C,Frontali L,Leoni L,et al.Effect of rifamycinonproteinsynthesis.Nature,1965, 207(995):417−418.
[11]Elizabeth AC,NataliyaK,ArkadyM,etal. Structural mechanism for rifampicin inhibition of bacterial RNA polymerase.Cell,2001,104(6): 901−912.
[12]Feklistov A,Mekler V,Jiang Q,et al.Rifamycins do not function by allosteric modulation of binding of Mg2+to the RNA polymerase active center.Proc Natl Acad Sci USA,2008,105(39):14820−14825.
[13]Bardarov S,Bardarov Jr S Jr,Pavelka Jr MS Jr,et al.Specialized transduction:an efficient method for generating marked and unmarked targeted gene disruptionsinMycobacteriumtuberculosis,M. bovis BCG and M.smegmatis.Microbiology,2002, 148(Pt 10):3007−3017.
[14]ByersHL,CampbellJ,vanUlsenP,etal. Candidate verification of iron-regulated Neisseria meningitidis proteins using isotopic versions of tandemmass tags(TMT)andsinglereaction monitoring.J Proteomics,2009,73(2):231−239.
[15]Jean MR,Daniel K.Mycobacterium smegmatis:an absurd model for tuberculosis?Trends Microbiol, 2001,9(10):472−473.
[16]Jaya ST,Deepak S.Mycobacterium smegmatis and tuberculosis.Trends Microbiol,2002,10(2):68−69.
[17]Mudassar A,Christopher HM,David SB,et al. Evaluation of the Mycobacterium smegmatis and BCG models for the discovery of Microbacterium tuberculosis inhibitors.Tuberculosis,2010,90(6): 333−337.
[18]Claudia M,Norbert R,Harald E,et al.The MspA porin promotes growth and increases antibiotic susceptibility of both Mycobacterium bovis BCG andMycobacteriumtuberculosis.Microbiology, 2004,150:853−864.
[19]Michael N,Olga D,Jason H,et al.Mycobacterial outer membranes:in search of proteins.Trends Microbiol,2010,18(3):109−116.
[20]Dautant A,Meyer JB,Yariv J,et al.Structure of a monocliniccrystalformofcytochromeb1 (bacterioferritin)from E.coli.Acta Crystallogr D Biol Crystallogr,1998,54(Pt 1):16−24.
[21]Maria AC.Ferritins,iron uptake and storage from the bacterioferritin viewpoint.EMBO J,2003, 22(9):1959−1968.
[22]SadeghiS,KarimiE.Spectrophotometric determinationofrifampicinthroughchelate formationandchargetransfercomplexationin pharmaceutical preparation and biological fluids. Chem Pharm Bull(Tokyo),2006,54(8):1107−1112.
[23]Strolin Benedetti M,Dostert P.Induction and autoinduction properties of rifamycin derivatives:a review of animal and human studies.Environ Health Perspect,1994,102(Suppl 9):101−105.
[24]Matthew WV,Luiz PS,Michael Y,et al.Structure andfunctionsoftheGNATsuperfamilyof acetyltransferases.Arch Biochem Biophys,2005, 433(1):212−226.
[25]Sousa M,Pozniak A,Boffito M.Pharmacokinetics andpharmacodynamicsofdruginteractions involving rifampicin,rifabutin and antimalarial drugs.JAntimicrobChemother,2008,62(5): 872−878.
(本文责编郝丽芳)
Quantitative proteomics analysis of ClpS-mediated rifampicin resistance in Mycobacterium
GulishanaAdilijiang1,Shan Feng1,Kaixia Mi2,and Haiteng Deng1
1 MOE Key Laboratory of Bioinformatics,School of Life Sciences,Tsinghua University,Beijing 100084,China 2 Institute of Microbiology,Chinese Academy of Sciences,Beijing 100101,China
Adaptor protein ClpS is an essential regulator of prokaryotic ATP-dependent protease ClpAP,which delivers certain protein substrates with specific amino acid sequences to ClpAP for degradation.However,ClpS also functions asthe inhibitor of the ClpAP-mediated protein degradation for other proteins.Here,we constructed the clpS-overexpression Mycobacterium smegmatis strain,and showed for the first time that overexpression of ClpS increased the resistance of M. smegmatis to rifampicin that is one of most widely used antibiotic drugs in treatment of tuberculosis.Using quantitative proteomic technology,we systematically analyzed effects of ClpS overexpression on changes in M.smegmatis proteome, and proposed that the increased rifampicin resistance was caused by ClpS-regulated drug sedimentation and drug metabolism.Our results indicate that the changes in degradation related proteins enhanced drug resistance and quantitative proteomic analysis is an important tool for understanding molecular mechanisms responsible for bacteria drug resistance.
protein degradation system ClpAPS,ClpS,rifampicin,quantitative proteomics
February 25,2014;Accepted:June 10,2014
Haiteng Deng.Tel/Fax:+86-10-62790498;E-mail:dht@mail.tsinghua.edu.cn Kaixia Mi.Tel/Fax:+86-10-57408892;E-mail:mik@im.ac.cn
古丽莎娜·阿地里江,冯杉,米凯霞,等.定量蛋白质组学分析ClpS在分枝杆菌耐药中的功能.生物工程学报,2014, 30(7):1115−1127.
Adilijiang Gulishana,Feng S,Mi KX,et al.Quantitative proteomics analysis of ClpS-mediated rifampicin resistance in Mycobacterium.Chin J Biotech,2014,30(7):1115−1127.
Supported by:Independent Research Funds of Chinese Ministry of Education(No.2012Z02293).
教育部自主科研基金(No.2012Z02293)资助。
Received:December 12,2013;Accepted:March 28,2014
Supported by:National Basic Research Program of China(973 Program)(No.2012CB725204),National High Technology Research and Development Program of Chnia(863 Program)(No.2012AA021505),National Natural Science Foundation of China(Nos.31070039, 31170030,51073081),Tianjin Municipal Key Program(Nos.13JCYBJC24900,13JCZDJC27800).
Corresponding author:Cunjiang Song.Tel/Fax:+86-22-23503866;E-mail:songcj@nankai.edu.cn
国家重点基础研究发展计划(973计划)(No.2012CB725204),国家高技术研究发展计划(863计划)(No.2012AA021505),国家自然科学基金(Nos.31070039,31170030,51073081),天津市重点项目(Nos.13JCYBJC24900,13JCZDJC27800)资助。
时间:2013-11-27网络出版地址:http://www.cnki.net/kcms/detail/11.1998.Q.20131127.1120.003.html
