番茄热激蛋白90的全基因组鉴定及分析
2014-05-25刘云飞万红建杨悦俭韦艳萍李志邈叶青静王荣青阮美颖姚祝平周国治
刘云飞,万红建,杨悦俭,韦艳萍,李志邈,叶青静,王荣青,阮美颖,姚祝平,周国治
1. 浙江省农业科学院蔬菜研究所,杭州 310021;
2. 南京农业大学园艺学院,南京 210095
番茄热激蛋白90的全基因组鉴定及分析
刘云飞1,2,万红建1,杨悦俭1,韦艳萍1,2,李志邈1,叶青静1,王荣青1,阮美颖1,姚祝平1,周国治1
1. 浙江省农业科学院蔬菜研究所,杭州 310021;
2. 南京农业大学园艺学院,南京 210095
热激蛋白90(Heat shock protein 90,Hsp90)是植物应对不良环境胁迫产生的一类特定的抗逆蛋白。文章以番茄(Solanum lycopersicum L.)基因组数据为平台,借助生物信息学方法对Hsp90基因家族进行鉴定与分析。结果表明,番茄至少含有7个Hsp90基因,不均匀分布在6条染色体上,氨基酸序列长度为267~794aa,内含子数目为 2~19;共线性分析发现两对基因(Hsp90-1和 Hsp90-3,Hsp90-5和 Hsp90-7)以片段重复形式存在。MEME(Multiple Em for Motif Elicitation)分析显示,番茄Hsp90基因编码的氨基酸序列具有多个保守基序;聚类分析揭示番茄、水稻(Oryza sativa L.)和拟南芥(Arabidopsis thaliana L.)Hsp90基因可以分为5组,存在3对直系同源基因和4对旁系同源基因;基于RNA-seq数据库表达分析发现,3个基因(Hsp90-5、Hsp90-6和Hsp90-7)在营养器官和生殖器官中表达量较高,4个基因(Hsp90-1、Hsp90-2、Hsp90-3和Hsp90-4)除在番茄转色后10 d的果实中表达量较高外,其余组织中表达量均较低;对Hsp90基因启动子序列进行分析,发现了多个参与植物对逆境胁迫的顺式作用元件,如 HSE、CCAAT-box。此外,qRT-PCR检测结果表明,在叶片热胁迫条件下,番茄Hsp90基因的表达量均存在增强趋势,表明这些基因参与了番茄叶片应对高温胁迫的反应。研究结果为鉴定番茄Hsp90基因的功能和进化起源奠定了基础。
番茄;热激蛋白;基因重复;表达分析
植物生长发育过程中通常受到各种不良环境条件的影响,如非生物胁迫(高温、干旱、盐、冷害等)和生物胁迫(真菌、细菌、病毒及线虫等)。近年来,随着全球气候变暖以及设施栽培的普遍发展,高温胁迫已成为影响植物正常生长发育的主要因素之一[1]。植物在长期进化过程中已形成完善的抵御不良环境条件的调控机制。目前研究发现,高温胁迫条件下植物细胞内热激蛋白(Heat shock protein, Hsp) 的合成显著增加[2~4],并且已证实该蛋白在植物抵御高温过程中发挥着重要作用[2]。热激蛋白广泛存在于动物、植物以及微生物中[5],按分子量大小将 Hsp分为不同的家族,主要包含 Hsp60、Hsp70/Hsp80、Hsp90、Hsp110和小分子量 Hsp(smHsp)家族[6]。Hsp90基因家族结构高度保守,为真核生物细胞质中广泛存在的一类分子伴侣[7,8],参与蛋白的折叠、激活以及成熟过程,同时与信号转导中蛋白构象转化及稳定有关[9]。研究发现,Hsp90基因不仅被非生物胁迫诱导,如盐碱、高温、低温、重金属等[10,11],而且还可参与植物对病原菌的抵抗,Wang等[12]研究发现,Hsp90基因家族成员TaHsp90.2 和TaHsp90.3过表达的小麦植株对条锈病具有显著抗性。
目前,部分植物Hsp90基因家族成员已经鉴定完成。拟南芥(Arabidopsis thaliana L.)中已鉴定出7个 Hsp90基因[13],水稻(Oryza sative L.)中含 9个Hsp90基因[14]。番茄(Solanum lycopersicum L.)为世界上第二大蔬菜作物,同时也是我国蔬菜产业中的主要成员,但其生产常受到高温等非生物胁迫的影响。番茄全基因组测序的完成为在全基因组水平上进行Hsp90基因的挖掘提供了条件[15]。本文对番茄Hsp90基因家族的成员进行了鉴定,并进一步分析Hsp90基因的数目、结构特点、染色体定位、系统发育关系以及表达模式等,以期为番茄Hsp90基因的功能鉴定以及耐热番茄品种的培育奠定基础。
1 材料和方法
1.1 材料
番茄Hsp90基因家族成员相关信息主要来自两个基因组数据库(http://solgenomics.net/和 http://mips. helmholtz-muenchen.de/plant/tomato/searchjsp/index.jsp)。根据Krishna等[13]研究结果在NCBI网站中获得拟南芥Hsp90基因家族成员信息。根据Hu等[14]研究结果,从水稻基因组网站(http://rice.plantbiology.msu. edu/analyses_search_locus.shtml)获得水稻 Hsp90基因家族成员信息。
1.2 方法
1.2.1 番茄Hsp90基因家族的鉴定
本研究采用两种方法对番茄Hsp90基因进行检索:(1)在上述两个数据库中分别输入关键词“heat shock protein 90”进行搜索,获得番茄Hsp90基因相关信息;(2)利用拟南芥Hsp90基因氨基酸序列对番茄数据库进行Blastp搜索。搜索结果中去除重复基因,即获得候选基因,然后通过Pfam网站(http://pfam. janelia.org/)进行鉴定。番茄 Hsp90基因的等电点以及分子量通过网站(http://web.expasy.org/compute _pi/)的pI/Mw计算工具获得。
1.2.2 番茄Hsp90基因内含子/外显子结构分析
将每个番茄 Hsp90基因的核苷酸序列及相应的基因组序列分别以 FASTA格式导入网站(http:// gsds.cbi.pku.edu.cn/),利用在线工具 Gene Structure Display Server(GSDS)[16],绘制Hsp90基因的外显子-内含子结构模式图。
1.2.3 番茄Hsp90基因染色体定位
结合番茄基因组数据库中获得的Hsp90基因位置信息,通过 MapDraw V2.1软件对 7个鉴定的Hsp90基因进行染色体定位。将番茄Hsp90基因名称提交到网站http://chibba.agtec.uga.edu/duplication/ index/locus,分析这些基因的重复特征(串联重复和片段重复)。
1.2.4 番茄Hsp90基因的保守基序分布及序列分析
将番茄Hsp90基因氨基酸序列保存为FASTA格式文件,利用MEME(http://meme.nbcr.net/meme/cgibin/meme.cgi)在线工具对番茄 Hsp90基因进行保守基序分布及序列分析。用DNAMAN6.0软件分析序列之间的相似性。
1.2.5 番茄、拟南芥和水稻Hsp90基因家族比较分析
将获得的拟南芥、水稻和番茄Hsp90基因的氨基酸序列保存为FASTA格式文件,通过软件Clustal X 对 Hsp90基因进行氨基酸序列比对[17],利用MEGA 5.0软件的邻接法(Neighbor-Joining)对拟南芥、水稻和番茄Hsp90基因进行系统发育树的构建[18],Bootstrap值为1000,去除Bootstrap支持率低于60%的节点。
1.2.6 基于RNA-seq数据的表达分析
在番茄功能基因组数据库(http://ted.bti.cornell. edu/cgi-bin/TFGD/digital/home.cgi)中下载D004栽培番茄各组织RNA-Seq测序数据,并搜寻番茄Hsp90基因表达数据,利用软件MeV[19]分析Hsp90基因的表达模式。
1.2.7 番茄Hsp90基因启动子序列鉴定及分析
从番茄基因组网站(http://solgenomics.net)下载所鉴定的7个Hsp90基因上游序列1.5 kb,通过在线软件Plant CARE(http://bioinformatics.psb.ugent.be/ webtools/plantcare/html/)进行分析。
1.2.8 番茄材料处理、Hsp90基因特异性引物设计及qRT-PCR检测体系
对番茄材料 7818D进行热胁迫(42℃)处理,取热激处理0 h、1.5 h、3 h、6 h、12 h和24 h的叶片为材料,3次生物学重复。根据总RNA提取试剂盒(天根生化科技有限公司,北京)的说明书提取RNA,采用琼脂糖凝胶电泳仪检测RNA的纯度,-70℃保存。利用cDNA第1链合成试剂盒(天根生化科技有限公司,北京),取 2 μL总 RNA,按照说明书进行cDNA第1链的合成。
运用BioXM 2.6软件设计番茄Hsp90基因特异引物,由上海英俊生物公司合成。引物信息详见表1。

表1 番茄HSP90基因引物
荧光定量 PCR扩增体系为 20 μL,包括:2× TransStartTMEco Green qPCR SuperMix 10 μL,Passive Reference Dye 0.4 μL,正反向引物(10 μmol/L)各0.4 μL,cDNA 1 μL,ddH2O 7.8 μL。荧光定量PCR扩增条件:95℃ 30 s;95℃ 5 s,55℃ 15 s,72℃ 30 s,40个循环。3次实验重复。运用 2-ΔΔCt方法对数据进行数据分析。
2 结果与分析
2.1 番茄Hsp90基因家族的鉴定
通过对番茄基因组数据库的检索,共鉴定出 7个Hsp90基因,分别命名为Hsp90-1~Hsp90-7(表2)。由表2可以看出,编码氨基酸长度在267~794aa之间,氨基酸数目最多的为 Hsp90-4(Solyc07g0477-90.2.1),含794个氨基酸,其次是Hsp90-2(Solyc05g-010670.2.1),含 787个氨基酸;而编码氨基酸数目最少的为 Hsp90-6(Solyc10g078930.1.1),仅含 267个氨基酸;分子量大小由31.30~90.43 kDa不等,其中Hsp90-4的分子量最大,高达90.43 kDa,最小的为Hsp90-6,仅31.30 kDa;番茄Hsp90基因的等电点变化位于4.94~8.18之间,等电点最高的为Hsp90-6基因,最低的是Hsp90-2基因,除Hsp90-6基因外,其余基因所对应的蛋白质等电点皆在6以下,表明大部分番茄Hsp90蛋白为酸性。
2.2 番茄Hsp90基因聚类及内含子/外显子结构分析
本研究运用MEGA5.0软件对番茄Hsp90基因的氨基酸序列进行系统发育树的构建。结果发现,7个Hsp90基因被明显的分成4个分枝,除Hsp90-6外,Hsp90-1与Hsp90-3、Hsp90-5与Hsp90-7、Hsp90-2与Hsp90-4都分别聚类在同一分枝(图1)。此外,本研究利用番茄基因组数据库信息,通过在线工具GSDS绘制Hsp90基因的结构模式图(图1)。如图1所示: Hsp90基因的内含子数目变化程度较大。其中内含子数目最多的为Hsp90-4,含有19个内含子,其次是Hsp90-2,含18个内含子,且被聚为同一分枝,表明这两个基因的结构较为复杂,而其余 5个番茄 Hsp90基因的内含子数目皆为 2,基因结构相对简单。
2.3 番茄Hsp90基因定位
通过番茄基因组数据库信息,利用 MapDraw V2.1软件将鉴定的番茄7个Hsp90基因定位于6条染色体上(图2), Hsp90-1~ Hsp90-7分别位于3、5、6、7、7、4和12号染色体上,除Hsp90-4和Hsp90-5基因位于同一染色体上,其余 5个基因分别位于不同的番茄染色体,其中Hsp90-1、Hsp90-2和Hsp90-7基因位于染色体的上端,Hsp90-4、Hsp90-5和Hsp90-6则位于染色体下端。通过在线网站(http://chibba.agtec.uga.edu/duplication/index/locus)研究发现,两对基因(Hsp90-1和Hsp90-3,Hsp90-5和 Hsp90-7)所在区域(500 kb范围内)存在共线性关系,前者发生了倒位现象,而后者基因排列顺序相同,因此,这两对基因在进化过程中发生了片段重复。
2.4 番茄Hsp90基因的保守基序分布及序列分析
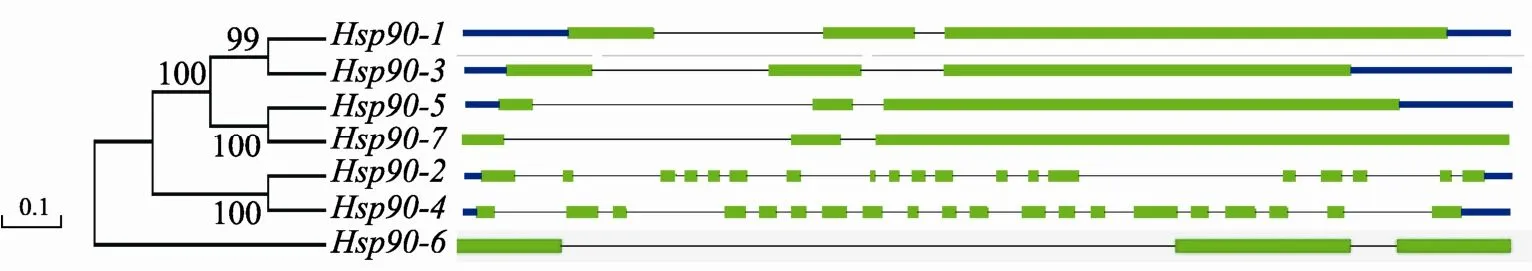
图1 番茄Hsp90基因家族成员系统发育关系及内含子-外显子结构绿色线条为外显子,黑色细线为内含子区,蓝色粗线为非编码区。
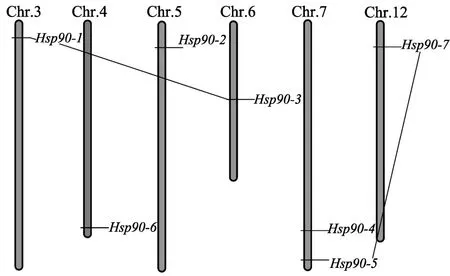
图2 番茄Hsp90基因的染色体定位
利用MEME在线工具对番茄Hsp90基因进行保守基序分析,结果显示:番茄Hsp90基因含有15个保守基序,这些保守基序的长度为15~50aa(表3),其中6个保守基序都包含50个氨基酸,分别是Motif 1、3、4、5、6和8,含有氨基酸数目最少的为Motif 13,仅含有15个氨基酸。通过Pfam在线软件对15个 Motif的结构域进行分析,结果显示 Motif5、7、2和8共同组成了番茄Hsp90蛋白的ATPase结构域,而其余 11个 Motif共同组成了 Hsp保守域。仅Hsp90-1、Hsp90-5和Hsp90-7等3个基因中存在Hsp蛋白C端结构域的MEEVD保守结构,因此,保守基序分析未能显示C端结构。此外,序列相似性分析发现,Hsp90-5和Hsp90-7序列相似性最高,达到96.0%,Hsp90-5和Hsp90-6序列相似性最低,仅为19.0%,剩下的基因之间相似性位于19.4%~95.4%之间,表明番茄Hsp90蛋白之间具有较高的序列多样化。
本文进一步分析了番茄Hsp90蛋白15个基序的保守程度(图 3A)。结果显示:Hsp90-6蛋白仅含 3个保守基序,即Motif9、Motif11和Motif12。这表明此蛋白序列可能存在缺失。除Hsp90-3的Motif 11和12丢失外,其余5个番茄Hsp90蛋白都含有15个保守基序;除Hsp90-2和Hsp90-4中Motif 8的保守性相对较低外,其他基序的保守程度均较高。进一步分析发现15个保守基序中,氨基酸序列保守性较高的为Motif 8(图3B),这一基序除第8、23、24、27、34、35和 37个氨基酸处有一定变化外,其余位置氨基酸均完全保守。而含50个氨基酸的Motif 1的氨基酸序列保守性较低,其中许多位置的氨基酸多样性程度较高,如第3、6、7个氨基酸位置处。

表3 番茄Hsp90蛋白的基序组成
2.5 番茄、拟南芥和水稻Hsp90基因家族比较分析
为深入分析番茄Hsp90基因的进化关系,本研究选取拟南芥、水稻和番茄的Hsp90基因进行系统发育关系分析,参与系统发育树构建的Hsp90基因共23个,其中来自番茄和拟南芥的均为7个,来自水稻的为9个。从图4中可以明显看出,这些基因可分为5个亚枝,分别命名为Class A~Class F。除Class C和Class F亚分枝外,其余进化枝都包含3种植物的Hsp90基因。基因数目最多的为Class A亚枝,含7个Hsp90基因,最少的为Class F亚枝,仅含两个基因。本研究共发现14个Hsp90基因具有同源关系,约占基因总数目的60.86%(14/23)。物种间存在3对直系同源基因(AtHsp90-7和 Os06g50300、AtHsp90-6和Hsp90-4、Hsp90-6和Os09g30438)。而物种内存在 4对旁系同源基因,2对(Hsp90-5和Hsp90-7,Hsp90-1和 Hsp90-3)来自于番茄,2对(Os08g39140和Os09g30418,Os08g38086和Os09g-29840)来自于水稻。育过程中的表达模式,本研究利用番茄功能基因组数据库网站中 RNA-Seq测序数据库(D004),分析Hsp90基因的表达模式(图 5)。结果显示:Hsp90-5基因在根、叶、花、花芽以及果实不同发育阶段表达水平较高;Hsp90-7基因除在叶片中表达量相对较低外,其余组织中均高表达;Hsp90-6基因在参与表达分析的番茄各组织中表达量均较低;此外,本研究还发现在转色期10 d的果实中,Hsp90-4基因表达水平较低,其余5个基因均具有高的表达量;大部分基因在叶片中的表达量较低;基因 Hsp90-2和Hsp90-3在幼果生长过程(1~3 cm)中表达量呈现出先上升后降低的趋势,其中Hsp90-3基因在2 cm幼果和转色10 d的果实中表达水平较高,而在其他组织器官中表达量较低;Hsp90-4基因除在转色 10 d的果实中有少量表达外,在其余组织中表达量均较低。
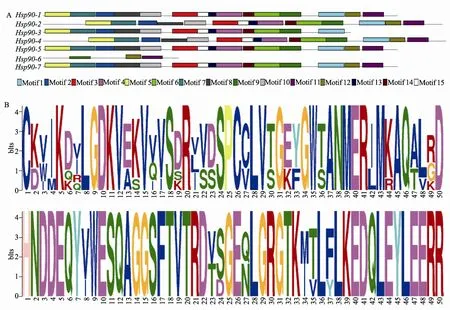
图3 番茄Hsp90蛋白保守基序分布及部分基序保守性分析A:保守基序分布;B:Motif 1和Motif 8 保守性分布。
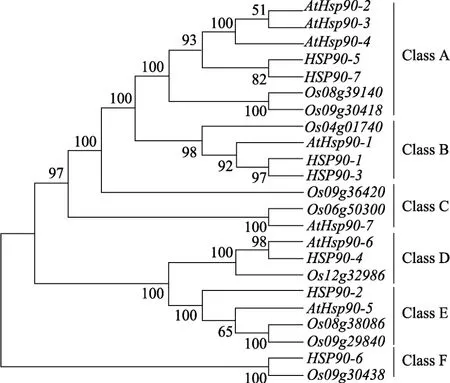
图4 番茄、拟南芥和水稻Hsp90基因的系统发育关系将拟南芥、水稻、番茄的23个Hsp90基因分成6类,分别为Class A~Class F。2.6 番茄Hsp90基因表达分析为揭示番茄Hsp90基因在不同组织以及果实发
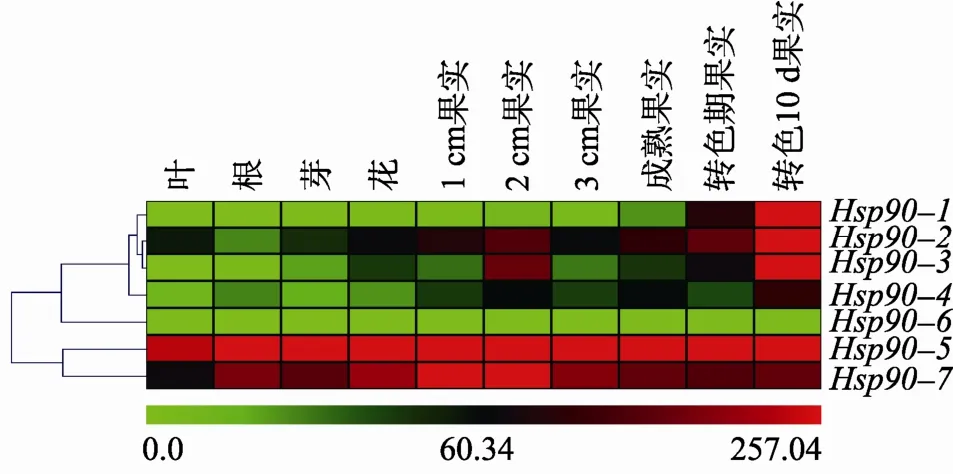
图5 番茄Hsp90基因表达分析红色表示高表达量,绿色表示低表达量。

表4 番茄Hsp90基因启动子区顺式作用元件种类及数目
2.7 番茄Hsp90基因启动子分析
本研究运用Plant CARE在线软件对Hsp90基因启动子序列(上游1.5 kb)进行分析(表4)。结果发现,主要存在3类顺式作用元件,包括非生物胁迫应答反应相关作用元件、激素应答顺式作用元件以及植物抗性胁迫反应相关作用元件。非生物胁迫应答反应相关作用元件主要有:与热胁迫相关的 HSE和CCAAT-box元件,与低温应激反应相关的LTR元件,干旱胁迫相关作用元件MBS。各种激素应答顺式作用元件主要有:乙烯反应作用元件(ERE),脱落酸作用反应元件(ABRE),水杨酸作用反应元件(TCA-element),赤霉素作用反应元件(GARE-motif),茉莉酸甲酯作用元件(CGTCA-motif和TGACG-motif),植物激素反应作用元件(TGA-box)。植物抗性胁迫反应相关作用元件主要有:TC-rich repeats元件。在参与分析的7个基因中,除Hsp90-2外,其余5个基因均含有HSE元件,其中Hsp90-4含有的HSE元件数目多达5个。
2.8 番茄Hsp90基因在高温胁迫下的表达模式
为了进一步明确番茄Hsp90基因是否应答高温胁迫反应,本文运用qRT-PCR分析了番茄叶片在热激处理条件下Hsp90基因的表达情况(图6)。结果表明:所有基因在高温不同处理时间段内均有不同程度的表达,其中 Hsp90-1、Hsp90-4、Hsp90-5和Hsp90-7基因在叶片热激达到12 h时,表达量最高;Hsp90-2和Hsp90-3基因在热激3 h时表达量达到最高,并且它们的表达趋势相近,但基因 Hsp90-2的表达量在处理3 h时呈现为急剧增加趋势,而Hsp90-3基因的表达量增加则更为缓慢。Hsp90-6基因在处理3 h时表达量显著增高,在6 h时有所降低,随后又持续增高。
3 讨 论
高温胁迫通常会引起生物体内相关基因的表达变化,其中热激蛋白(Hsp)为生物体在高温胁迫条件下迅速合成的一类保守蛋白,该蛋白最早发现于果蝇唾液腺染色体[20]。已有研究表明,当植物经过高温胁迫后,植物可对致死高温产生耐受性[21],其中热激蛋白起到了重要作用。另外,低温、干旱、重金属、盐碱、ABA等都可诱导植物产生Hsp[22]。目前,研究者根据Hsp分子量大小,将其分为Hsp60、Hsp70/ Hsp80、Hsp90、Hsp110和小分子量Hsp(smHsp)[6],其中Hsp90是一类重要的热激蛋白,具有高度的保守性,参与调控与维持细胞内多种蛋白质的构象和功能[9]。有研究证实 Hsp90基因参与了胁迫信号的传递和类固醇激酶受体、激酶和转录因子的折叠等生理生化过程[3,23~25]。因此,Hsp90基因具有功能多样化特点。番茄是我国蔬菜产业中的主要作物,且设施栽培面积较广,番茄耐热研究已成为当前研究热点之一。番茄Hsp90基因的鉴定将有利于全面揭示该基因的潜在功能,同时也为植物Hsp90基因的进化分析等研究提供依据。2012年,番茄全基因组测序已经完成[15],这将为人们在全基因组水平上鉴定番茄Hsp90基因家族成员提供条件。
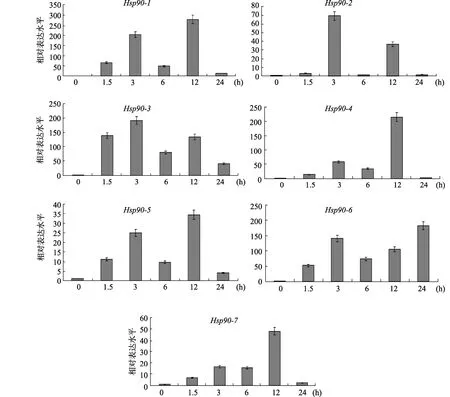
图6 番茄Hsp90基因高温胁迫下的表达模式
本研究共鉴定出7个番茄Hsp90基因(表2)。它们所编码的氨基酸序列长度位于276~794 aa之间,等电点集中在4.00~8.18范围内,除Hsp90-6外,大部分番茄Hsp90蛋白皆为酸性。番茄Hsp90基因不均匀的分布于番茄染色体上,主要集中于染色体的两端(图2),这与水稻Hsp90基因分布情况类似[14]。目前,水稻和拟南芥的比较研究证实基因重复是基因家族进化过程中的重要机制之一[4,26~27]。本研究中,两对番茄Hsp90基因(Hsp90-1和Hsp90-3,Hsp90-5和 Hsp90-7)位于染色体片段重复区段,表明番茄Hsp90基因在进化过程中可能发生了基因复制现象。番茄 Hsp90基因的保守基序分析显示,Hsp90-6仅含3个保守基序,这表明该基因在进化过程中可能发生了部分序列的丢失。除Hsp90-6外,仅Hsp90-3存在两个保守基序的缺失,而其余5个Hsp90基因家族成员所编码的氨基酸序列均高度保守(图3),这与前人对 Hsp90基因的描述相符[9]。Motif5、7、2和 8共同组成了的 ATPase结构域,该结构域为ATP/ADP结合位点,具有 ATPase活性[4],而其余11个 Motif共同组成的 Hsp保守域在维持 ATPase结构域的完整活性中具有关键作用[28]。番茄 Hsp90基因中仅Hsp90-1、Hsp90-5和Hsp90-7等3个基因具有MEEVD保守结构,这表明番茄Hsp蛋白的C端结构域多样性程度较高。
通过拟南芥、水稻和番茄Hsp90基因的系统发育关系分析,发现3对直系同源基因和4对旁系同源基因,其数目约占基因总数目的 60.86%(14/23),表明这些基因在番茄、水稻和拟南芥基因组中,以各自物种特异的方式进行了扩展,这种现象也普遍出现于其他植物基因家族研究中[29,30]。基于 RNA-seq分析发现,Hsp90基因在番茄不同组织中的表达量具有显著性差异,其中 Hsp90-5在根、叶、芽、花和果实不同发育阶段中表达量均较高,这表明该基因与番茄植株正常生理生化活动联系紧密。除Hsp90-4和Hsp90-6基因外,其余5个基因在果实中都具有显著的表达量,这暗示着它们可能参与了番茄果实的生长和发育。Hsp90-2和Hsp90-3基因在幼果生长过程中表达量呈现出先升高后降低的状态,这表明该基因可能参与果实发育的相关调控过程。qRT-PCR研究发现,Hsp90基因在番茄叶片热处理条件下表达量均有所升高,但其变化趋势存在差异,这表明Hsp90蛋白可能参与番茄叶片对高温逆境胁迫的应答。
此外,还发现在每一个番茄Hsp90基因启动子序列中均含有与非生物胁迫相关的顺式作用元件(表 4),其中 HSE已被证实为植物应对热胁迫的作用元件,而CCAAT-box具有增加热诱导启动子的活性[31,32],大部分的番茄 Hsp90基因启动子均含有HSE。此外,Hsp90基因还含有其他作用元件,如激素反应和抗病反应相关作用元件,表明这类基因可能还参与了番茄应对激素胁迫和生物胁迫的应答反应,这些结果将为进一步分析番茄Hsp90基因的作用机理提供理论基础。然而,本文未发现番茄Hsp90基因启动子区顺式作用元件及其数目与基因的实际表达量存在明确的相关性。而引起这一现象的原因可能存在以下 3个方面。首先,基因表达的调控与多种因素相关,如空间、时间调控等模式[33];其次,目前还有许多顺式作用元件还未能鉴定出来,存在被忽略的可能性[34];最后,顺式作用元件还存在多样性,它们所调控基因产生不同的功能,而这通常引起植株的不同表型[35]。
[1] Ahuja I, de Vos RC, Bones AM, Hall RD. Plant molecular stress responses face climate change. Trends Plant Sci, 2010, 15(12): 664-674.
[2] Heckathorn SA, Ryan SL, Baylis JA, Wang D, Hamilton III EW, Cundiff L, Luthe DS. In vivo evidence from an Agrostis stolonifera selection genotype that chloroplast small heat-shock proteins can protect photosystem II during heat stress. Funct Plant Biol, 2002, 29(8): 935-946.
[3] Wegele H, Müller L, Buchner J. Hsp70 and Hsp90-a relay team for protein folding. Rev Physiol Biochem Pharmacol, 2004, 151: 1-44.
[4] Young JC, Moarefi I, Hartl FU. Hsp90: a specialized but essential protein-folding tool. J Cell Biol, 2001, 154(2): 267-274.
[5] Lindquist S. The heat-shock response. Annu Rev Biochem, 1986, 55(1): 1151-1191.
[6] Al-Whaibi MH. Plant heat-shock proteins: a mini review. J King Saud University-Sci, 2011, 23(2): 139-150
[7] Prasinos C, Krampis K, Samakovli D, Hatzopoulos P. Tight regulation of expression of two Arabidopsis cytosolic Hsp90 genes during embryo development. J Exp Bot, 2005, 56(412): 633-644.
[8] 裴丽丽, 徐兆师, 尹丽娟, 李连城, 陈明, 郭玉华, 马有志. 植物热激蛋白 90 的分子作用机理及其利用研究进展. 植物遗传资源学报, 2013, 14(1):109-114.
[9] Picard D. Heat-shock protein 90, a chaperone for folding and regulation. Cell Mol Life Sci, 2002, 59(10): 1640-1648.
[10] Pareek A, Singla SL, Grover A. Immunological evidence for accumulation of two high-molecular-weight (104 and 90 kDa) HSPs in response to different stresses in rice and in response to high temperature stress in diverse plant genera. Plant Mol Biol, 1995, 29(2): 293-301.
[11] Song HM, Fan PX, Li YX. Overexpression of organellar and cytosolic AtHSP90 in Arabidopsis thaliana impairs plant tolerance to oxidative stress. Plant Mol Biol Rep, 2009, 27(3): 342-349.
[12] Wang GF, Wei XN, Fan RC, Zhou HB, Wang XP, Yu CM, Dong LL, Dong ZY, Wang XJ, Kang ZS, Ling HQ, Shen QH, Wang DW, Zhang XQ. Molecular analysis of common wheat genes encoding three types of cytosolic heat shock protein 90 (Hsp90): functional involvement of cytosolic Hsp90s in the control of wheat seedling growth and disease resistance. New Phytol, 2011, 191(2):418-431.
[13] Krishna P, Gloor G. The Hsp90 family of proteins in Arabidopsis thaliana. Cell stress Chaperon, 2001, 6(3): 238-246.
[14] Hu WH, Hu GC, Han B. Genome-wide survey and expression profiling of heat shock proteins and heat shock factors revealed overlapped and stress specific response under abiotic stresses in rice. Plant Sci, 2009, 176(4):583-590.
[15] The Tomato Genome Consortium. The tomato genome sequence provides insights into fleshy fruit evolution, Nature, 2012, 485(7400): 635-641.
[16] 郭安源, 朱其慧, 陈新, 罗静初. GSDS: 基因结构显示系统. 遗传, 2007, 29(8): 1023-1026.
[17] Chenna R, Sugawara H, Koike T, Lopez R, Gibson TJ, Higgins DG, Thompson JD. Multiple sequence alignment with the Clustal series of programs. Nucleic Acids Res, 2003, 31(13): 3497-3500.
[18] Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol, 2011, 28(10): 2731-2739.
[19] Mohr P, Iliadis C. Recommended cross-section of the reaction below 2.5 MeV: A potential tool for quantitative analysis and depth profiling of oxygen. Nucl Instrum Meth A, 2012, 688(1): 62-65.
[20] Tissiéres A, Mitchell HK, Tracy UM. Protein synthesis in salivary glands of Drosophila melanogaster: Relation to chromosome puffs. J Mol Biol, 1974, 84(3): 389-398.
[21] 杜志如. 水稻热激蛋白 Hsp90 基因的克隆及互作蛋白的筛选[学位论文]. 雅安: 四川农业大学, 2008.
[22] Vierling E. The roles of heat shock proteins in plants. Annual Review of Plant Biology, 1991, 42(1): 579-620
[23] Jackson SE, Queitsch C, Toft D. Hsp90: from structure to phenotype. Nat Struct Mol Biol, 2004, 11(12): 1152-1155.
[24] Shinozaki F, Minami M, Chiba T, Suzuki M, Yoshimatsu K, Ichikawa Y, Terasawa K, Emori Y, Matsumoto K, Kurosaki T, Nakai A, Tanaka K, Minami Y. Depletion of Hsp90β induces multiple defects in B cell receptor signaling. J Biol Chem, 2006, 281(24): 16361-16369.
[25] Zuehlke A, Johnson JL. Hsp90 and co-chaperones twist the functions of diverse client proteins. Biopolymers, 2010, 93(3): 211-217.
[26] Yang Z, Wang X, Gu S, Hu Z, Xu H, Xu C. Comparative study of SBP-box gene family in Arabidopsis and rice. Gene, 2008, 407(1-2): 1-11.
[27] Yang ZF, Zhou Y, Wang XF, Gu SL, Yu JM, Liang GH, Yan CJ, Xu CW. Genomewide comparative phylogenetic and molecular evolutionary analysis of tubby-like protein family in Arabidopsis, rice, and popla. Genomics, 2008, 92(4): 246-253.
[28] Meyer P, Prodromou C, Hu B, Vaughan C, Roe SM, Panaretou B, Piper P W, Pearl LH. Structural and functional analysis of the middle segment of hsp90: implications for ATP hydrolysis and client protein and cochaperone interactions. Mol Cell, 2003, 11(3): 647-658.
[29] Jain M, Tyagi AK, Khurana JP. Genome-wide analysis, evolutionary expansion, and expression of early auxinresponsive SAUR gene family in rice (Oryza sativa). Genomics, 2006, 88(3): 360-371.
[30] Zhang SB, Chen C, Li L, Meng L, Singh J, Jiang N, Deng XW, He ZH, Lemaux PG. Evolutionary expansion, gene structure, and expression of the rice wall-associated kinase gene family. Plant Physiol, 2005, 139(3): 1107-1124.
[31] Haralampidis K, Milioni D, Rigas S, Hatzopoulos P. Combinatorial interaction of cis elements specifies the expression of the Arabidopsis AtHsp90-1 Gene. Plant Physiol, 2002, 129(3): 1138-1149.
[32] Czarnecka E, Key J L, Gurley W B. Regulatory domains of the Gmhspl 7.5 heat shock promoter of soybean. Mol Cell Biol, 1989, 9(8): 3457-3463.
[33] Banerjee N, Zhang MQ. Functional genomics as applied to mapping transcription regulatory networks. Curr Opin Microbiol, 2002, 5(3): 313-317.
[34] Higo K, Ugawa Y, Iwamoto M, Korenaga T. Plant cis-acting regulatory DNA elements (PLACE) database: 1999. Nucleic Acids Res, 1999, 27(1): 297-300.
[35] Brown CD, Johnson DS, Sidow A. Functional architecture and evolution of transcriptional elements that drive gene coexpression. Science, 2007, 317(5844): 1557-1560.
(责任编委: 李传友)
Genome-wide identification and analysis of heat shock protein 90 in tomato
Yunfei Liu1,2, Hongjian Wan1, Yuejian Yang1, Yanping Wei1,2, Zhimiao Li1, Qingjing Ye1, Rongqing Wang1, Meiying Ruan1, Zhuping Yao1, Guozhi Zhou1
1. Institute of Vegetables, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, China;
2. College of Horticulture, Nanjing Agricultural University, Nanjing 210095, China
Abstract:Heat shock proteins 90 (Hsp90) are a kind of specific proteins in plant which were produced under environmental stresses. By referring to the tomato genome database, we identified and analyzed Hsp90 gene family members using bioinformatics methods. Results indicated that the tomato genome contained at least 7 Hsp90 genes, which were distributed unevenly on 6 chromosomes. Amino acid sequence length of these proteins ranged from 267 to 794aa. Numbers of intron ranged from 2 to 19. Microsynteny analysis showed that two pairs of Hsp90 genes (Hsp90-1and Hsp90-3, Hsp90-5 and Hsp90-7) were identified by segment duplication. In addition, multiple conservation motifs were found in Hsp90 proteins. Phylogenetic analysis revealed that Hsp90 genes from tomato, rice and Arabidopsis can be divided into 5 groups. Three pair of orthologous genes and four pairs of homologous genes were found. Expression analysis based on RNA-seq showed that the expression of three genes (Hsp90-5, Hsp90-6 and Hsp90-7) was high in vegetable and reproductive organs, while the expression of other four genes (Hsp90-1, Hsp90-2, Hsp90-3 and Hsp90-4) was relatively low except for its expression at the breaking stage of fruit. Analysis of promoter regions of Hsp90 genes showed that multiple cis-elements were involved in plant responses to biotic and abiotic stresses. The expression of 7 genes under heat stress was also detected by qRT-PCR. Expression of all Hsp90 genes in tomato leaf was enhanced. The results indicated that these genes could be participated in tomato leaf response to heat stresses. Together, these results will lay a foundation for analyzing Hsp90 gene function and molecular evolution in the future.
tomato; heat shock protein; gene duplication; expression analysis
2014-02-24;
2014-06-08
国家自然科学基金项目(编号:31301774,31272156),浙江省优先主题农业项目(编号:2011C12004),国家大宗蔬菜产业技术体系(编号:CARS-25-G-16),浙江省农业新品种选育重大科技专项(编号:2012C12903),公益性行业(农业)科研专项经费(编号:201003065),国家科技支撑计划项目(编号:2012BAD02B02)和浙江省蔬菜产业创新团队项目(编号:2009R50026)资助
刘云飞,硕士研究生,专业方向:蔬菜学。Tel: 0571-86404352;E-mail: head_20074152@126.com
万红建,博士,副研究员,研究方向:番茄遗传育种。E-mail: wanhongjian@sina.com
10.3724/SP.J.1005.2014.1043
时间: 2014-9-2 10:35:55
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140911.1047.003.html