利用向日葵重组自交系构建遗传图谱
2014-05-25张永虎于海峰侯建华李素萍吕品于志贤
张永虎,于海峰,侯建华,李素萍,吕品,于志贤
1. 内蒙古农业大学农学院,呼和浩特 010018;
2. 内蒙古自治区农牧业科学院,呼和浩特 010031
利用向日葵重组自交系构建遗传图谱
张永虎1,于海峰2,侯建华1,李素萍2,吕品1,于志贤1
1. 内蒙古农业大学农学院,呼和浩特 010018;
2. 内蒙古自治区农牧业科学院,呼和浩特 010031
以向日葵自选系K55为母本、K58为父本杂交组合,通过单粒传得到的187个F5:6代重组自交系群体为作图材料,联合应用SSR和AFLP标记构建遗传连锁图谱。经过78对SSR引物和48对AFLP引物组合选择性扩增,分别得到341和1119条带,共1460条,分别获得多态性条带184条和393条,共577条多态性条带,占所有条带的39.52%。SSR和AFLP标记各有84个和108个多态性标记偏离孟德尔分离比例(P=0.05),共192个偏分离标记。采用JoinMap4.0软件进行连锁分析,构建了1张总长度为2759.4 cM、包含17个连锁群、连锁495个多态性标记的遗传图谱,其中偏分离标记 170个,标记间的平均图距为 5.57 cM。每个连锁群上分布有 5~72个标记,长68.88~250.17 cM。本图谱为向日葵永久性图谱,为向日葵重要性状QTL定位和基因克隆奠定基础。
向日葵;重组自交系;SSR;AFLP;分子连锁图谱
遗传连锁图谱的构建是遗传学基础研究的重要方面,是复杂性状基因组结构和功能的比较分析、QTLs定位、图位克隆等研究的重要基础,也是当前遗传学研究的热点。伴随着分子生物学的飞速发展,基于不同原理的多种分子标记技术不断被开发出来,常用于遗传作图的分子标记有 RFLP、RAPD、SSR和AFLP标记[1]。在实际研究中RFLP的多态性程度不高,而且大多数 RFLP标记为多个位点;RAPD标记步骤简单,但重复性差且多态性低;SSR多态性高且多数位点单一稳定,常被作为锚定标记;AFLP标记既有RAPD标记方便快捷的特点,又具有RFLP标记的稳定性和可靠性[2,3]。所以,本研究利用SSR标记作为锚定标记,用AFLP标记填补空隙,构建较高密度的分子遗传图谱,既有助于图谱的连锁群或染色体的归并又方便不同连锁群的整合。
向日葵是一种重要的油料作物,其连锁遗传图谱构建研究已有近20年的历史,早期遗传图谱多是利用 RFLP构建,目前向日葵分子遗传图谱的构建落后于其他作物。1995年 Genzbittel等[4]构建了第 1张含有237个RFLP位点、连锁群总长度为1150 cM的向日葵遗传图谱,并于 1999年利用 7个 F2群体的cDNA对这个图谱进行了补充[5]。1996年Peerbolte等[6]利用AFLP标记构建了一个具有523个标记位点的向日葵遗传图谱。2002年 Tang等[7]利用 SSR标记通过RILs群体构建了向日葵遗传连锁图谱,该图谱包含 408个 SSR位点、17条连锁群,总长度为1368.3 cM,标记间距为3.1 cM。2012年黄先群等[8]以重组自交系为材料用35个SSR标记加密了Fabre F构建的包含323个AFLP标记的连锁图谱。至今国内外已经报道了超过10张向日葵分子图谱,构建连锁图谱用到的AFLP标记均为EcoRⅠ和MseⅠ内切酶组合[6,9]。本研究以187个向日葵重组自交系为作图群体,利用PstⅠ和MseⅠ组合的AFLP标记和SSR标记构建高密度的遗传连锁图谱,旨在丰富向日葵遗传连锁图谱的位点,为今后重要性状的QTL定位和基因克隆及分子育种奠定理论基础。
1 材料和方法
1.1 构图群体
以内蒙古农牧业科学院提供的干旱敏感型自选系K55与耐旱型自选系K58配制杂交组合,通过单粒传得到的187个F5:6代重组自交系为作图群体。
1.2 方法
1.2.1 DNA提取
向日葵叶片中多酚的含量比较高,基因组的DNA比较难提纯。故本实验参照陈昆松等[10]改良的CTAB法提取DNA并加以改进,在氯仿:异戊醇(24:1)抽提两次后,再用1.5倍1×CTAB 沉淀一次DNA,提高DNA的质量。
1.2.2 SSR分析
SSR分析的PCR反应条件采用本实验室报道[11]的体系及扩增条件,引物合成按照 NCBI公布的向日葵微卫星序列合成。
1.2.3 AFLP分析
AFLP分析技术参照Vos等[2]的方法,并加以改进,用PstⅠ和MseⅠ内切酶组合同时加入T4连接酶,37℃酶切连接14 h,把接头连接到酶切片段上。然后用不含选择性碱基的引物对酶切连接进行预扩增,将预扩增产物稀释 30倍作为模板,用 PstⅠ3个选择性碱基和 MseⅠ2个选择性碱基的引物进行选择性扩增。扩增产物采用6%的聚丙烯酰胺变性胶电泳,采用银染法检测结果[12]用氢氧化钠显影。
1.3 数据整理和连锁分析
根据SSR和AFLP标记的聚丙烯酰胺检测显影结果,将各株系的带型按亲本类型归类,与母本带型一致的赋值为1,与父本带型一致的赋值为0,由于各种原因带型不清楚的或数据缺失的赋值为 2。图谱构建所采用的软件是JoinMap 4.0,根据软件的要求将获得的标记数据“0”、“1”和“2”原始矩阵转换“b”、“a”和“c”的数据格式,导入软件。在Groupings (tree) 窗口下,用Calculate命令进行标记连锁分组(LOD=2.0-10.0),然后选用LOD≥4.0标记连锁组群,用Create Groups use the Groupings tree命令获得用于作图的组群,用Calculate map命令建立框架图,并采用Kosambi函数[13]进行图谱距离计算。再将能够连锁的偏分离标记逐个加入框架图中得到最终图谱。
2 结果与分析
2.1 亲本间多态性分析
利用双亲组合从 300对 SSR引物中筛选到 78对,从90对AFLP引物组合中筛选到48对多态性好、条带清晰的引物组合。用筛选出的 126对引物组合对 187个 F5:6群体的单株基因组进行多态性分析。由于大部分标记在后代中没有同时出现 AA、Aa和 aa 带型,便于统计所有的标记都按照显性标记统计。这些引物共扩增出1460个标记,其中SSR标记341个,AFLP标记1119个;具有多态性的显性标记577个,184个SSR 标记,393个AFLP标记。经卡方(χ2)测验(P=0.05),有192个标记偏离孟德尔分离比例,占多态性标记的33.28%(表1),其中SSR 84个,AFLP 108个。
2.2 遗传图谱构建
利用JoinMap4.0软件对该群体的577个多态性标记进行连锁分析,得到一张含17个连锁群的遗传图谱(表2,图1),495个标记定位到该图谱上,SSR标记155个,AFLP标记340个,29个SSR标记和53个 AFLP标记没有进入连锁图谱,这些标记来自母本 K55和父本 K58的比例基本为 1:1。图谱全长2759.40 cM,标记间距0~57.88 cM,平均5.57 cM。17个连锁群上的标记数在 5~72个之间,其中 1~8连锁群上分布的标记比较多,连锁群长度在68.88~250.17 cM之间,第1连锁群上分布的标记最多有72个,长250.17 cM,第14连锁群上分布的标记最少只有5个,长度也最短为68.88 cM。

表1 用于组图的不同类型标记
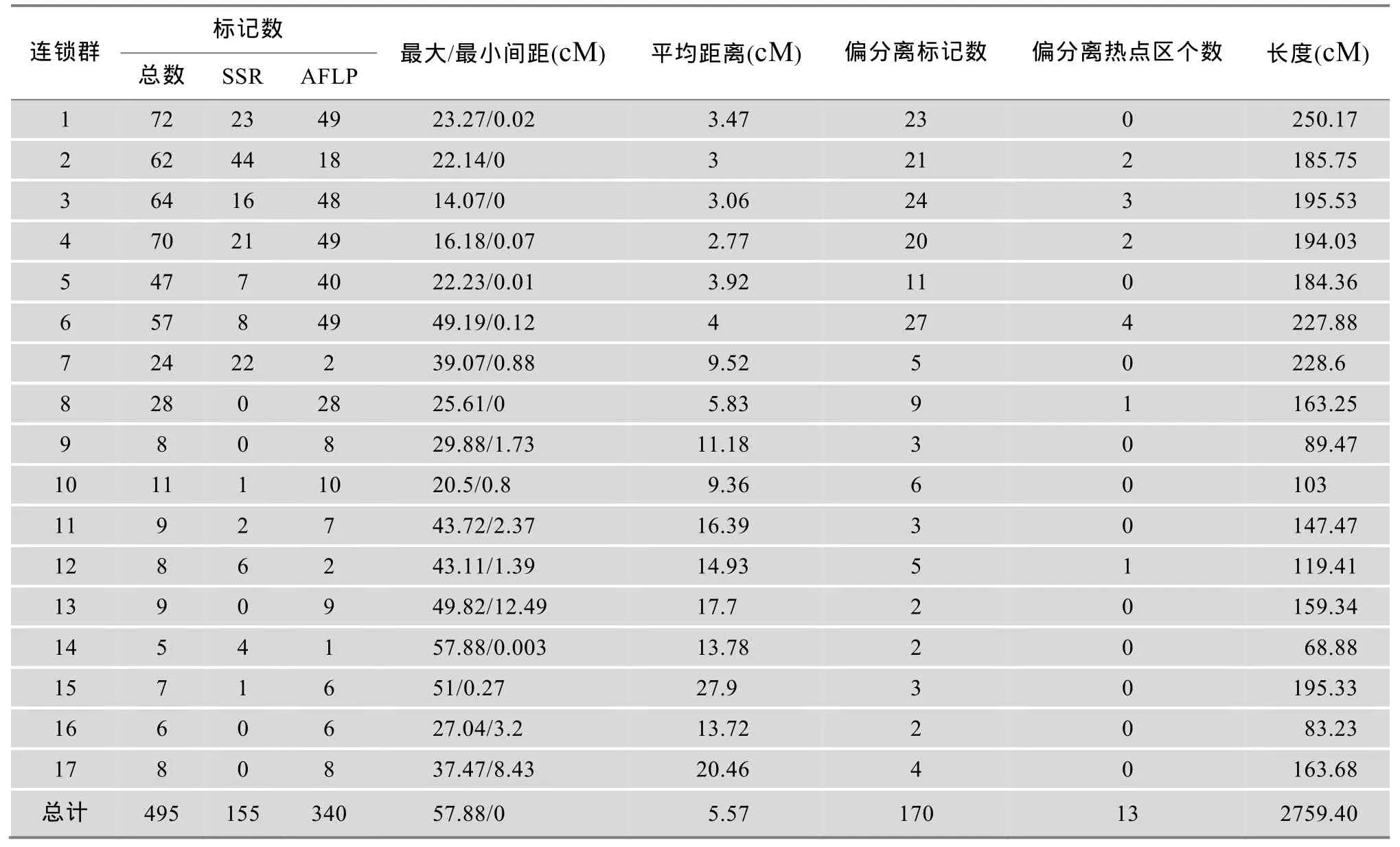
表2 向日葵17个连锁群的特性
经Excel中相关系数函数Pearson计算,本研究中各连锁群的标记数和连锁群长度的相关系数为0.984,达到极显著水平,表明这些标记在图谱上是随机分布的。标记在连锁群上的分布并不均匀,只有第3、第4连锁群上没有出现大于20 cM的间隙,总共出现了30个大于20 cM的间隙,第14连锁群上出现了一个超过50 cM的大间隙。在第1、第3、第4、第6和第8连锁群上出现了以AFLP为主的小范围标记密集区,这些密集区分布于整个连锁群上。SSR和AFLP两种标记在各个连锁群上的比例不一样,只有第2、第7、第12和第14连锁群上的SSR标记多于AFLP标记,第8、第9、第13、第16和第17连锁群上只有AFLP标记。
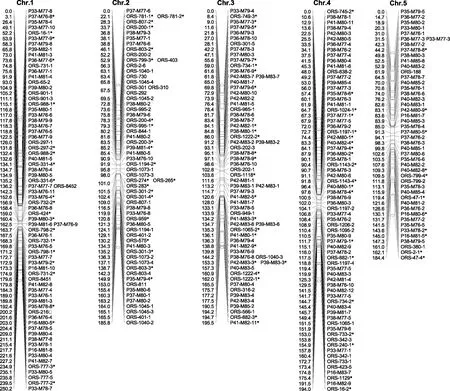
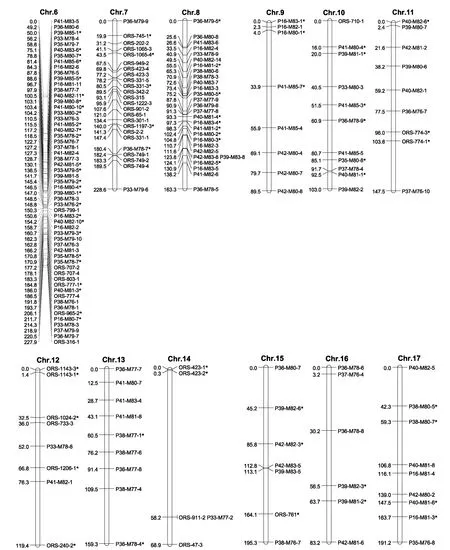
图1 向日葵分子遗传连锁图谱“*”表示偏分离标记。
这张遗传图谱上共分布有170个偏分离标记,每个连锁群上或多或少都有偏分离标记,但大多数的偏分离标记主要分布在少数几个连锁群上,且在连锁群上呈分散状分布,但在第2、第3、第4、第6、第8和第12这6个连锁群上以3~4个偏分离标记聚集形成了13个偏分离热点区。170个偏分离标记中有87个为母本K55的特异位点,83个为父本K58的特异位点,基本符合1:1。
3 讨 论
3.1 构建高密度的分子连锁图谱工作量大、费用高
高密度分子连锁图谱往往是不同研究者共同的成果,有效避免了资源浪费。对不同研究者采用不同群体构建的连锁图谱进行整合,要求有公共的、稳定的锚定标记。SSR标记稳定性好,位点的平均多态信息量最高,AFLP标记多态性检测效率最高[14],因而构建高密度遗传图谱最常用的方法是借助于SSR标记锚定连锁群,利用AFLP标记加密图谱[1]。本研究以 155个 SSR标记锚定连锁群,与 Tang
等[7]2002年构建,并在2003年[15]加密的向日葵SSR分子连锁图谱有很好的吻合性,有78个标记与之相关联。本研究构建的向日葵分子遗传图谱有17个连锁群,总长2759.4 cM,共计495个标记,有340个PstⅠ/MseⅠ组合的AFLP标记,155个SSR标记,标记间平均距离为5.57 cM,是国内最为致密的图谱,达到了QTL定位的要求[16]。目前向日葵遗传图谱构建的缺憾是还没有将向日葵的连锁群与对应的染色体联系起来。相信随着向日葵全基因组测序工作的开展,这17个连锁群与向日葵17条染色体的关系很快就能明确[17]。
3.2 AFLP内切酶选择
AFLP标记检测多态性的效率不仅与供试材料的基因型有关,与所使用的限制性内切酶组合亦相关。Powell等[18]在小麦研究中发现大多数群体使用PstⅠ/MseⅠ比 EcoRⅠ/MseⅠ组合多态性检测效率高,他们认为主要原因在于PstⅠ对碱基甲基化不敏感,所以由甲基化引起 DNA的多态性变化 PstⅠ/MseⅠ也能够检测到。本研究AFLP标记使用PstⅠ/MseⅠ组合多态性为 35.1%,而刘杰等[19]使用的EcoRⅠ/MseⅠ组合多态性是 12.8%,说明向日葵AFLP标记研究中PstⅠ/MseⅠ比EcoRⅠ/MseⅠ的多态性检测率高。大量的研究表明,PstⅠ/MseⅠ组合的背景噪音比较低,扩增出来的条带较少,但是多态性条带比例比较高,容易识别,标记在连锁群上的分布比较均匀,更适合遗传多样性的分析和遗传连锁图的构建。本研究中在第1、第3、第4、第6和第8连锁群上出现了以AFLP为主的小范围标记密集区,这些密集区分布于整个连锁群上,不同于大豆[1]、白菜[20]使用 AFLP构建分子连锁图谱,聚集区分布于着丝粒两端,这可能是大多数分子连锁图谱构建中用到的AFLP标记都是使用EcoRⅠ/MseⅠ双酶切组合,EcoRⅠ对着丝粒附近富含的甲基化胞嘧啶比较敏感,容易在这一区域发生标记聚集现象[1,20]。
3.3 偏分离现象
遗传偏分离是自然界中一种普遍存在的现象[21],是生物进化强有力的动力,也是降低遗传作图精度的因素之一,几乎所有的作图群体都会存在,在远缘杂交组合的分离群体及DH和RIL群体中尤为普遍。Knox等[22]认为造成标记发生偏分离的原因有很多,诸如配子体和孢子体发育阶段基因型的选择、遗传搭车效应、染色体片段的丢失、非同源染色体重组和环境因素等,作图群体的亲本不纯合也会引发偏分离。另外,不同类型的分子标记发生偏分离的几率不同,一般情况下显性标记比共显性标记发生偏分离的比例要高[23]。偏分离标记可能会影响连锁的检验,但偏分离标记和正常分离标记有相同的作图效率,不能直接的把偏分离标记删除,如果将偏分离标记全部筛掉排除,会影响图谱对基因组的覆盖率,还有可能丢掉一些QTL位点[24]。本研究构建出的连锁图谱包含了495个标记,有170个偏离标记,占标记位点的34.34%,本研究所使用的材料为向日葵重组自交系,向日葵属于异花作物,有一定的自交不亲和性,在单粒传的过程中存在株系缺失,这可能是产生较高偏分离的原因之一。
[1] 吴晓雷, 贺超英, 王永军, 张志永, 东方阳, 张劲松, 陈受宜, 盖钧镒. 大豆遗传图谱的构建和分析. 遗传学报, 2001, 28(11): 1051-1061.
[2] Vos P, Hogers R, Bleeker M, Reijans M, van de Lee T, Hornes M, Frijters A, Pot J, Peleman J, Kuiper M, Zabeau M. AFLP: a new technique for DNA fingerprinting. Nucleic Acids Res, 1995, 23(21): 4407-4414.
[3] 张镝, 丁毅. 基于 AFLP标记的中国西藏近缘野生大麦遗传多样性分析. 遗传, 2007, 29(6): 725-730.
[4] Gentzbittel L, Vear F, Zhang YX, Bervillé A, Nicolas P. Development of a consensus linkage RFLP map of cultivated sunflower (Helianthus annuus L.). Theor Appl Genet, 1995, 90(7-8): 1079-1086.
[5] Gentzbittel L, Mestries E, Mouzeyar S, Mouzeyar F, Badaoui S, Vear F, de Labrouhe D T, Nicolas P. A composite map of expressed sequences and phenotypic traits of the sunflower (Helianthus annuus L.) genome. Theor Appl Genet, 99(1-2): 218-234.
[6] Peerbolte RP, Peleman J. The catisol sunflower RFLP map (146 loci) extended with 291 AFLP markers. In: Proceed of the 18th sunflower research workshop. Fargo, North Dakota, USA, , 1996: 174-178.
[7] Tang S, Yu JK, Slabaugh MB, Shintani K, Knapp J. Simple sequence repeat map of the sunflower genome . Theor Appl Genet, 2002(8), 105: 1124-1136.
[8] 黄先群, Genzbitelle L, Fabre F, Saraffi A. SSR分子标记丰富向日葵(Helianthus annuus L.)遗传图谱的研究. 西南农业学报, 2012, 25(3): 1031-1038.
[9] Flores Berrios E, Gentzbittel L, Kayyal H, Alibert G, Sarrafi A. AFLP mapping of QTLs for in vitro organogenesis traits using recombinant inbred lines in sunflower (Helianthus annuus L.). Theor Appl Genet, 2000, 101(8): 1299-1306.
[10] 陈昆松, 李方, 徐昌杰, 张上隆, 傅承新. 改良CTAB法用于多年生植物组织基因组 DNA的大量提取. 遗传, 2004, 26(4): 529-531.
[11] 张永虎, 陈雪, 于海峰, 侯建华, 斯日古楞. 向日葵芽期抗旱相关性状丙二醛含量的 SSR分子标记分析. 内蒙古农业大学学报(自然科学版), 2012, 33(3): 25-29.
[12] Bassam BJ, Caetano-Anolles G, Gresshoff PM. Fast and sensitive silver staining of DNA in polyacrylamide gels. Annal Bio Chem, 1991, 196(1): 80-83.
[13] Kowalski SP, Lan TH, Feldmann KA, Paterson AH. Comparative mapping of Arabidopsis thaliana and Brassica oleracea chromosomes reveals islands of conserved organization. Genetics, 1994, 138(2): 499-510.
[14] 袁力行, 傅骏骅, Warburton M, 李新梅, 张世煌, Khairallah M, 刘新芝, 彭泽斌, 李连城. 利用RFLP、SSR、AFLP和RAPD标记分析玉米自交系遗传多样性的比较研究. 遗传学报, 2000, 27(8): 725-733.
[15] Tang SX, Kishore VK, Knapp SJ. PCR-multiplexes for a genome-wide framework of simple sequence repeat marker loci in cultivated sunflower. Theor Appl Genet, 2003, 107(1): 6-19.
[16] 徐微, 张宗文, 张恩来, 吴斌. 大粒裸燕麦(Avena nuda L.)遗传连锁图谱的构建. 植物遗传资源学报, 2013, 14(4): 673-678
[17] 高猛, 安玉麟, 孙瑞芬, 张艳芳. 向日葵 rRNA基因克隆及其染色体定位研究. 中国细胞生物学学报, 2014, 36(2): 195-199.
[18] Powell W, Thomas WTB, Baird E, Lawrence P, Booth A, Harrower B, Mcnicol JW, Waugh R. Analysis of quantitative traits in barley by the use of Amplified Fragment Length Polymorphisms. Heredity, 1997, 79(1): 48-59.
[19] 刘杰, 刘公社, 齐冬梅, 汪恩华, 李芳芳. 向日葵品种鉴定和纯度分析的AFLP研究. 植物学通报, 2002, 19(4): 444-451
[20] 于拴仓, 王永健, 郑晓鹰. 大白菜分子遗传图谱的构建与分析. 中国农业科学, 2003, 36(2): 190-195.
[21] Konish T, Yano Y, Abe K, Geographic distribution of alleles at the Gal2 locus for segregation distortion in barley. Theor Appl Genet, 1992, 85(4): 419-422.
[22] Knox MR, Ellis TH. Excess heterozygosity contributes to genetic map expansion in pea recombinant inbred populations. Genetics, 2002, 162(2): 861-873.
[23] 刘海燕, 崔金腾, 高用明. 遗传群体偏分离研究进展.植物遗传资源学报, 2009, 10(4): 613-617.
[24] 宋立君. 萝卜遗传连锁图谱构建与主要品质性状 QTLs分析. QTLs分析[学位论文]. 南京: 南京农业大学, 2009: 34-35.
(责任编委: 陈德富)
Construction of a genetic map of sunflower using a population of recombinant inbred lines (RILs)
Yonghu Zhang1, Haifeng Yu2, Jianhua Hou1, Suping Li2, Pin Lv1, Zhixian Yu1
1. Agricultural College, Inner Mongolia Agricultural University, Hohhot 010018, China;
2. Inner Mongolia Academy of Agricultural & Animal Husbandry Sciences, Huhhot 010031, China
A genetic linkage map of sunflower was constructed by combined applying the SSR and AFLP markers using 187 F5:6individuals of recombinant inbred lines (RILs) which derived from the cross between Helianthus annuus K55 and Helianthus annuus K58 through single-seed descent (SSD). Using 78 pairs of SSR primers and 48 pairs of AFLP primer, 341 and 1119 bands were amplified, respectively. Among these 1460 bands, 557 bands (39.52%) were polymorphic, including 184 bands by SSR markers and 393 bands by AFLP markers. In the group of these polymorphic bands, 84 bands from SSR markers and 108 bands from AFLP markers showed the genetic distortion (P = 0.05). A total of 192 segregation distortion markers were obtained in this study. By using the JoinMap 4.0 software to do the linkage analysis, a genetic linkage map was established with length of 2759.4 cM, consisted of 17 linkage groups, and comprised of 495 polymorphic molecular markers including 170 segregation distortion markers. The mean mark-er interval distance is 5.57 cM between markers. In addition, the number of markers in the linkage groups varied from 5 to 72, and the length of linkage groups were from 68.88 cM to 250.17 cM. The genetic map developed in the present study could be used for QTL mapping and gene cloning of sunflower important genes.
sunflower; recombinant inbred lines population; SSR; AFLP; molecular markers linkage map
2014-05-23;
2014-08-18
国家自然科学基金项目(编号:31160288)和内蒙古自然科学基金重点项目(编号:2010Zd06)资助作者简介: 张永虎,博士研究生,研究方向:作物抗性遗传改良。E-mail: zhangyonghu0815@126.com
侯建华,博士,博士生导师,研究方向:作物抗逆性状的遗传改良。E-mail: Houjh68@163.com
10.3724/SP.J.1005.2014.1036
时间: 2014-9-10 10:28:31
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140910.1028.001.html