拟南芥R2R3-MYB家族第22亚族的结构与功能
2014-05-25樊锦涛蒋琛茜邢继红董金皋
樊锦涛,蒋琛茜,邢继红,董金皋
河北农业大学真菌毒素与植物分子病理学实验室,保定 071001
拟南芥R2R3-MYB家族第22亚族的结构与功能
樊锦涛,蒋琛茜,邢继红,董金皋
河北农业大学真菌毒素与植物分子病理学实验室,保定 071001
拟南芥 R2R3-MYB转录因子在拟南芥生长发育、代谢及响应生物和非生物胁迫的调控网络中具有重要作用。根据保守的氨基酸序列,R2R3-MYB转录因子被分为25个亚族,其中第22亚族包含AtMYB44、AtMYB77、AtMYB73和AtMYB70 4个基因,主要响应生物和非生物胁迫。文章从基因功能的相似性、基因表达的一致性和基因结构的保守性3方面综述了第22亚族的4个基因,并综合讨论了其在结构与功能上的冗余性和多样性。
拟南芥;R2R3-MYB转录因子;22亚族;结构与功能
拟南芥MYB转录因子以含有保守的MYB结构域为共同特征,包括经典的 MYB转录因子和MYB-related转录因子,共199个成员。MYB结构域是一段约含 51~52个氨基酸的肽段,一般含有 3个保守的色氨酸残基,间隔18~19个氨基酸规则排列,参与空间结构中疏水核心的形成。根据 MYB蛋白含MYB结构域不完全重复子的个数,可将其分为4类:(1) 单一的MYB结构域蛋白(R1/R2);(2) R2R3蛋白;(3) 3R蛋白;(4) 拟南芥中新发现的4R蛋白。其中,R2R3-MYB转录因子在拟南芥MYB家族中数量最多,其蛋白结构的N端含有非常保守的DNA结合结构域,C端含有不保守的转录激活/抑制结构域[1]。
R2R3-MYB转录因子广泛参与拟南芥的初生和次生代谢、生长发育,响应生物和非生物胁迫等过程。根据R2R3-MYB蛋白的氨基酸序列的保守性不同,将其进一步分成25个亚族(图1)。其中,第22亚族包含AtMYB44、AtMYB77、AtMYB73和AtMYB70 4个基因,主要参与拟南芥抵抗生物和非生物胁迫过程。该亚族基因结构保守、功能相近、表达规律相似,表明该亚族基因在功能上可能存在冗余性,但是该亚族基因在功能和结构上又各具特点。由此我们推测,该亚族基因的相似性和各自的独特性是该亚族基因功能上呈现冗余性和多样性的重要原因。因此,本文从基因功能的相似性、基因表达的一致性和基因结构的保守性等方面对拟南芥R2R3-MYB家族22亚族基因的冗余性和多样性进行分析,为进一步明确R2R3-MYB转录因子的调控机制提供理论依据。
1 R2R3-MYB家族基因功能的多样性
R2R3-MYB家族中第 1、2、11、18、20、22亚族参与调控拟南芥抵抗生物、非生物胁迫反应。拟南芥AtMYB30基因通过正调控HR反应,刺激长链脂肪酸的合成,从而增强拟南芥对病原菌的抗性[2]。AtMYB30可与磷酸酶AtsPLA2-α相互作用,促进磷酸酶 AtsPLA2-α从细胞质转移到细胞核中,使AtMYB30的转录活性受到抑制。AtsPLA2-α突变体增强了对病原菌的抗性,表明AtsPLA2-α是AtMYB30调控拟南芥抗病反应过程的负调节因子[3];AtMYB96基因可以激活植物的脱落酸(ABA)和生长素(Auxin)的信号途径,其超表达突变体通过减少侧根的生长,增强对干旱胁迫的耐受性[4]。进一步研究发现,MYB转录因子具有冷胁迫因子 CBF启动子的结合位点;AtMYB15可与CBF家族的多个基因的启动子结合,AtMYB15的功能缺失导致CBF家族的多个基因的表达水平增强,同时myb15突变体对冷胁迫的耐受性增强,表明 AtMYB15是调控拟南芥冷胁迫相关基因表达的重要成员[5]。
R2R3-MYB家族中第3、4、5、6、7、12亚族参与调控拟南芥的初生和次生代谢过程。拟南芥中AtMYB12参与调控拟南芥根部类黄酮的合成[6],而AtMYB111参与调控子叶中类黄酮的合成[7]。拟南芥AtMYB75基因参与调控植物体内花青素的生物合成,其异位表达能够促进类黄酮类物质生物合成相关基因的表达,使植物的大多数器官表现为紫色[8,10]。部分 MYB转录因子控制植物细胞壁的合成,如第 3亚族的AtMYB58和AtMYB63参与拟南芥苯丙烷和木质素的代谢;AtMYB58和AtMYB63在次生壁加厚的纤维和导管中特异性表达,是木质素合成的特异调控基因;抑制AtMYB58和AtMYB63的表达会导致次生壁缺陷、木质素含量降低;而两者过量表达会激活木质素合成基因的表达,并伴随木质素的异位沉积[11]。
R2R3-MYB家族中第 9、14、15、18、19、21亚族影响拟南芥的生长和发育过程。其中,第 9亚族和第 15亚族均参与调控表皮毛的发育[12];第 14亚族的AtMYB37、AtMYB38、AtMYB68和第21亚族的AtMYB105和AtMYB117参与调控拟南芥腋生分生组织和横向器官的形成[13,16];AtMYB23和AtMYB66参与调控拟南芥根毛的发育[17,18];第 18亚族的AtMYB33和AtMYB65参与调控拟南芥雌蕊和花药的发育[19];第19亚族的AtMYB21和AtMYB24参与调控拟南芥雌蕊发育和纤维的长度[20,21]。
R2R3-MYB家族中第13、16、23、24、25亚族基因的功能尚未完全解析,暂时归为功能待定一类。
例如:第25亚族中包含AtMYB22、AtMYB64、AtMYB100、AtMYB115、AtMYB118和AtMYB119 6个基因,只有AtMYB115和AtMYB118的功能初步确定为参与拟南芥的种子萌发和胚的形成。酵母双杂交实验证实,AtMYB72可以和乙烯信号途径中的抗病因子 EIL3互作,初步确定 AtMYB72是拟南芥抗病的早期信号因子[22,23]。AtMYB26突变体植株能够产生形态和大小均正常的花粉,但是花药的药室内壁次生加厚内壁木质化异常,以至于花药不能正常开裂[24],推测AtMYB26与拟南芥花药的发育相关,但其具体功能还有待进一步的证实。

图1 根据氨基酸序列的保守性对R2R3-MYB转录因子家族的分类[1]
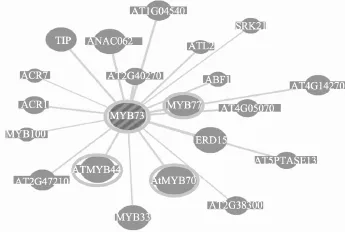
图2 共表达的22亚族基因
2 R2R3-MYB家族22亚族基因功能的相似性
利用GENEMANIA数据库(http://pages.genemania. org/)对R2R3-MYB家族基因进行分析,发现一些亚族基因不仅功能相似,而且存在一些共表达的现象。其中,参与抗逆反应的第22亚族基因的共表达现象最为明显,推测该亚族基因在功能上可能存在一定的冗余性,本文从该亚族 4个基因抗逆功能的相似性、基因表达的一致性和基因结构的保守性等方面进行分析,探讨4个基因在结构和功能上的冗余性和多样性,为明确22亚族各基因间的关系及其调控拟南芥抗逆的机制奠定基础。
2.1 R2R3-MYB家族22亚族基因参与抵抗非生物胁迫反应
R2R3-MYB家族 22亚族基因参与拟南芥响应高盐、干旱、低温等非生物胁迫反应。sos2-1(salt overly sensitive)突变体经250 mmol/L NaCl处理后,AtMYB44、AtMYB77、AtMYB73和AtMYB70的表达水平均出现明显的上调[25];用300 mmol/L NaCl处理拟南芥野生型,AtMYB73的表达水平随着处理时间的延长而增强,处理 6 h达到最大值;同时盐胁迫过程中,AtMYB73突变体中SOS1和SOS3基因的表达水平明显增强,表明AtMYB73可能负调控SOS反应,影响拟南芥抵抗高盐胁迫反应[26]。
ABA处理拟南芥野生型 30 min后,AtMYB44的转录水平明显增强,且在导管和叶片气孔中高效表达,表明AtMYB44可能参与调控拟南芥对干旱胁迫的响应;AtMYB44超表达转基因植株较野生型和敲除突变体对ABA的敏感性增强,并能更为迅速的调控气孔的开合,同时对钠盐胁迫和干旱胁迫的耐受程度明显增强;进一步的研究表明,AtMYB44通过抑制ABA的负调控因子PP2Cs(丝氨酸/苏氨酸蛋白激酶2Cs)对ABA进行正调控[27]。AtMYB73在拟南芥脱水过程中的表达水平没有明显变化,而在复水过程中被显著的诱导,表明AtMYB73也参与调控拟南芥对干旱胁迫的响应[28]。另外,低温处理能够诱导AtMYB73、AtMYB44和AtMYB77的表达,且AtMYB73可以持续长时间上调表达,推测AtMYB73在拟南芥抵抗低温胁迫过程中具有重要功能[29]。
2.2 R2R3-MYB家族22亚族基因参与拟南芥抵抗生物胁迫反应
R2R3-MYB家族 22亚族基因参与调控拟南芥对生物胁迫的响应。AtMYB77、AtMYB73和AtMYB70均能响应沙雷氏菌(Serratia plymuthica)和嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia)的胁迫刺激[30]。AtMYB44和AtMYB73能够响应Pst DC3000和激素SA、MeJA、ACC的刺激[31,32]。例如:接种Pst DC3000后,AtMYB44超表达转基因植株的病斑面积较野生型和敲除突变体明显减小,H2O2、胼胝质和PR1的积累明显增多[31];茉莉酸甲酯(MeJA)处理拟南芥野生型10 min后AtMYB44被诱导表达,但是AtMYB44不影响茉莉酸的信号通路,却影响茉莉酸信号通路下游基因JR2、SP、OXII和AOS的表达[33]。另外,AtMYB44能够响应蚜虫刺激。过敏致病性蛋白(Harpin)可以诱导植物抵抗病原菌的侵染和蚜虫的危害[34],在植物响应 Harpin后乙烯信号调控因子 EIN2受AtMYB44的调控,进而影响植物防御蚜虫的重要元件PP2-A和GSL的表达[35];在研究确定根癌农杆菌毒蛋白的互作蛋白VirE2-interacting protein 1 (VIP1)可以与 AtMYB44的启动子区结合,增强 AtMYB44的表达[36]。亚精胺在植物抗逆和防御反应中具有核心地位,在亚精胺合成酶 SPDS的超表达转基因拟南芥中,AtMYB73表达水平明显高于野生型[37]。
本课题组研究发现,水杨酸、茉莉酸和乙烯均可以不同程度的诱导AtMYB73的表达,同时在水杨酸途径关键基因突变体eds5和sid2、茉莉酸途径关键基因突变体jar1及水杨酸和茉莉酸共同调控的节点基因npr1突变体中,AtMYB73表达均受到明显的抑制,表明AtMYB73响应水杨酸和茉莉酸信号途径;当接种水稻胡麻斑病菌(Bipolaris Oryzae)后,AtMYB73的表达水平明显上调,且 Atmyb73突变体中 H2O2含量明显降低,PR1、NPR1和PDF1.2的表达水平均较野生型明显增强,表明AtMYB73通过水杨酸和茉莉酸途径调控拟南芥抵抗B. Oryzae的侵染[38]。
2.3 R2R3-MYB家族22亚族基因参与调控拟南芥的生长和发育过程
AtMYB77敲除突变体中,生长素相关基因的表达水平明显减弱;低浓度的IAA处理或营养缺乏时,AtMYB77敲除突变体的侧根密度明显减少;AtMYB77的 C端可以和生长素响应因子 ARFs互作,表明AtMYB77可以响应生长素的刺激[39];在雌蕊衰老的不同阶段,AtMYB73的表达水平发生明显的变化,推测AtMYB73参与调控胚珠的发育[40];在茎形成过程中,AtMYB73、AtMYB77和AtMYB44同时上调表达,表明AtMYB73、AtMYB77和AtMYB44可能参与次生生长过程[41]。此外,AtMYB73和AtMYB77还能够响应光的刺激,当黑暗处理 30 min后,AtMYB73和AtMYB77的表达水平均发生明显的变化[42]。
3 R2R3-MYB家族22亚族基因表达的一致性
3.1 22亚族基因的共表达现象
在拟南芥防御反应过程中,R2R3-MYB家族22亚族基因AtMYB44、AtMYB77、AtMYB73和AtMYB70表现出明显的共表达现象[30,43,44]。早在1998年,研究报道了在胚发育的晚期AtMYB44和AtMYB77表达水平均明显增强[45];在fus3、lec1和abi3突变体中,AtMYB44和AtMYB77的表达均受到明显的抑制。AtMYB44、AtMYB77和AtMYB73在拟南芥机械损伤后均有不同程度的上调表达[46]。2004年,Guan等[47]发现AtMYB77和AtMYB73均可以响应损伤刺激。在sos2-1(salt overly sensitive)突变体中,AtMYB44、AtMYB77、AtMYB73和AtMYB70在NaCl处理后均明显的上调[25]。在编码碳酸酐酶基因 βCA1的敲除突变体中,AtMYB73和AtMYB77的表达水平均明显下调[48]。2010年,Liu等[44]发现,AtMYB44和AtMYB73均能响应Harpin蛋白的刺激;同时在研究油菜素内酯时,发现bri1(brassinosteroid-insensitive 1)突变体中AtMYB44、AtMYB77和AtMYB73均明显下调[43]。2012年,Wenke等[30]发现AtMYB77、AtMYB73和 AtMYB70均能响应根际细菌沙雷氏菌(Serratia plymuthica)和嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia)的胁迫。2013年,Han等[49]发现了AtMYB44和AtMYB73共同响应茉莉酸诱导的抗性反应。
3.2 22亚族基因的时空表达规律的一致性
利用AtGenExpress Visualization Tool(http://www. weigelworld.org/resources/microarray/AtGenExpress/)[50],本文获得了22亚族4个基因在不同时期、不同部位(根、茎、叶、整个植株、分生部位、花、雄蕊、雌蕊和种子)的表达数据(图 3)。通过对比分析 4个基因在不同的组织部位、不同时间的表达水平,发现AtMYB44、AtMYB77、AtMYB73和AtMYB70的时空表达规律表现出明显的一致性。尤其是在17 d的根部、7 d的子叶、15 d的叶片、21 d的幼苗和8周龄的长角果等部位,4个基因的表达均处于较高的水平;在1 cm长的叶片上、茎的第2个节间处、幼苗第7 d的幼苗绿色部位、6周龄成熟的花粉中和早期心形胚中,4个基因的表达均处于较低的水平。
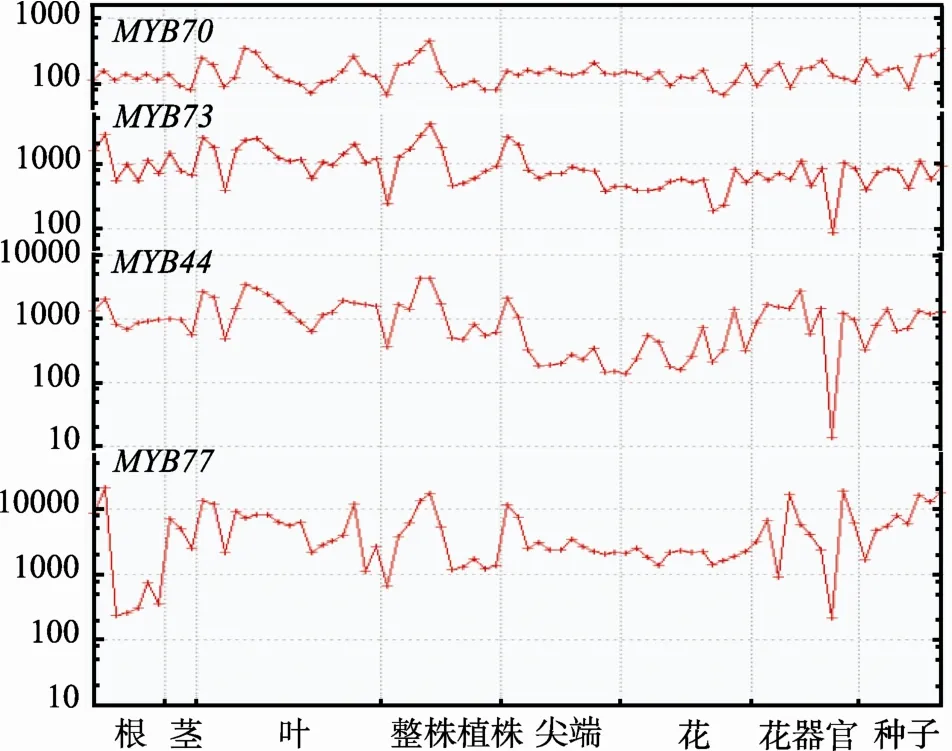
图3 22亚族基因的表达规律
4 R2R3-MYB家族22亚族基因结构的保守性
4.1 22亚族基因启动子元件的相似性
22亚族基因存在明显的共表达现象,本文推测AtMYB44、AtMYB77、AtMYB73和AtMYB70的启动子区可能存在着相似的顺式作用元件。利用plantCARE (http://bioinformatics.psb.ugent.be/webtools/ plantcare/html/)软件[51],本文对22亚族4个基因的启动子区(ATG前 1500 bp区域)进行分析,发现 4个基因共同含有的元件有11个,除了包括转录必备元件CAAT-box和TATA-box之外,还含有能够响应ABA的元件ABRE,响应热胁迫的元件HSE,MYB转录因子结合的区域MBS,参与防御反应的TC-rich repeats,响应生长素的TGA-element,4个光响应元件Box4、G-BOX、G-box和GAG-motif (表1 A)。这些结果说明,22亚族4个基因均能够响应植物激素(脱落酸、生长素),逆境胁迫(干旱、热和防御反应)和光信号(表1 A)。
AtMYB73、AtMYB44和AtMYB77均含有赤霉素响应元件 P-box和参与胚乳早期形成的元件 Skn-1_motif。而AtMYB44、AtMYB77和AtMYB70均含有响应光的MYB转录因子结合区域MRE(表1 B)。此外,AtMYB44和AtMYB70均含有2个响应参与抵抗坏死型病原菌的茉莉酸元件 TGACG-motif和CGTCA-motif,AtMYB44和AtMYB77含有响应水杨酸的元件 TCA-element和响应真菌激发子的Box-W1元件,而AtMYB73和AtMYB77含有2个响应赤霉素的元件GARE-motif和TATC-box,AtMYB73和AtMYB70均含有响应低温胁迫的LTR元件。
另外,通过分析每个顺式作用元件在不同基因启动子中数量和位置,结果显示这些与抗逆有关的顺式作用元件的数量和位置呈现多样性,并没有表现出我们预期的相似性。这表明每个基因通过转录调控被开启的模式是需要多种转录因子的进行不同形式的组合来完成的。
4.2 22亚族蛋白质结构的保守性
利用SOPMA网站[52],进一步对22亚族4个基因编码蛋白质的保守结构域进行分析。结果发现,22亚族的4个蛋白质一级结构域在1~100个氨基酸内均含有2个典型的SANT结构域,构成了经典的R2R3-MYB结构(图4),在具有转录激活或抑制的C端均含有若干个短的重复序列。利用 Jpred3和SOPMA软件,对AtMYB44、AtMYB77、AtMYB73和 AtMYB70蛋白质的二级结构进行分析。结果发现,4个蛋白中α-螺旋、β转角、无规则卷曲所占比例也表现出明显的一致性;其中,AtMYB77和AtMYB44相似程度较高,AtMYB73和AtMYB70相似度较高(表2)。该结果与亚族分类的结果相一致(图1)。
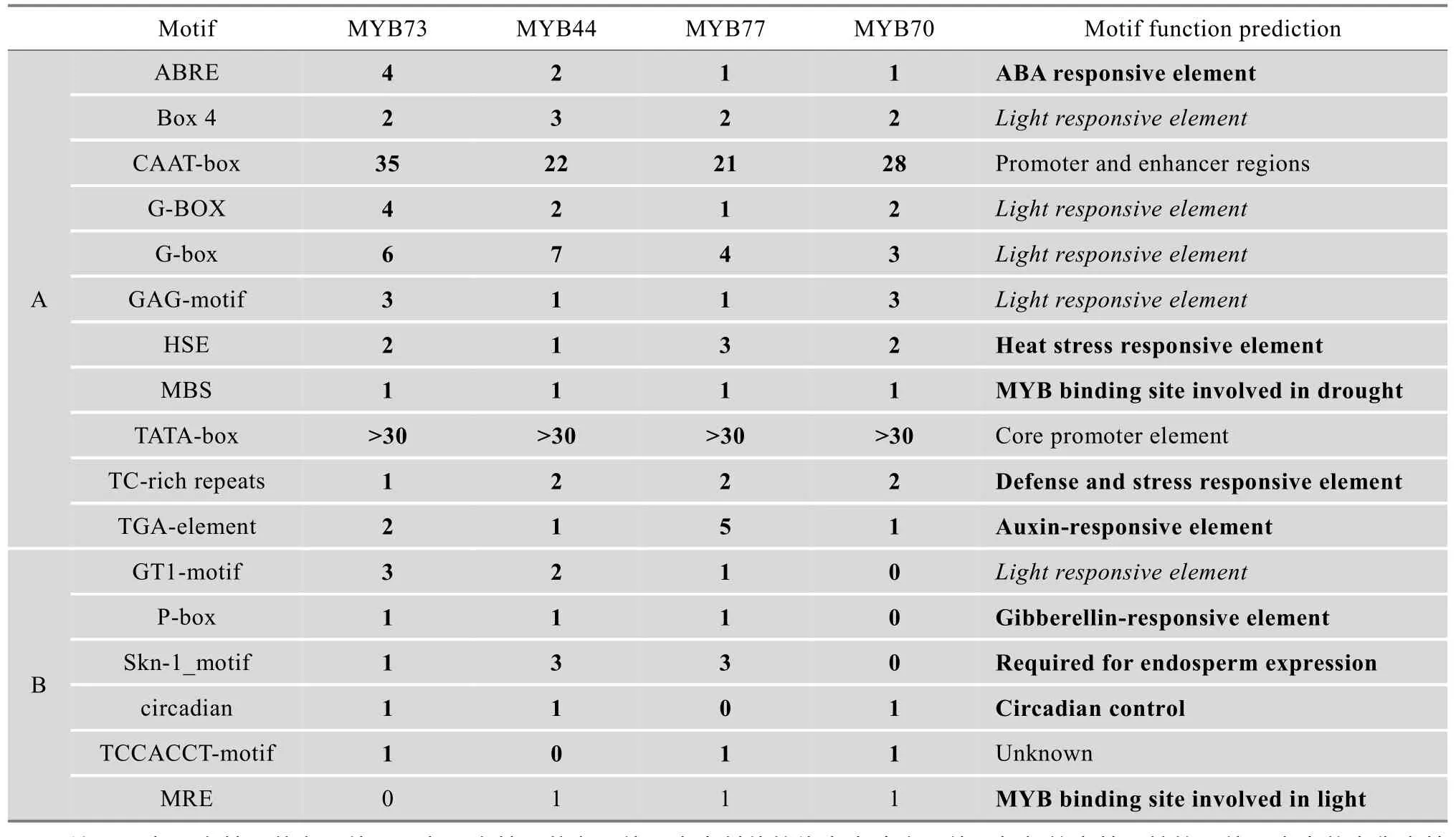
表1 22亚族4个基因的启动子元件分析

图4 22亚族4个蛋白的SANT保守结构域
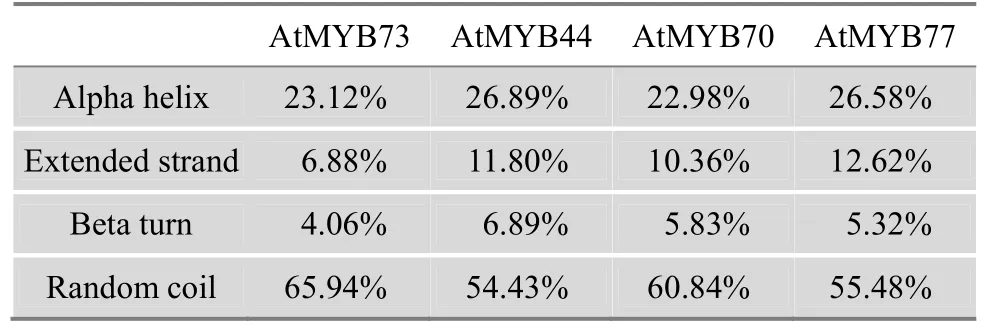
表2 22亚族4个蛋白的二级结构分析
5 展 望
拟南芥R2R3-MYB家族22亚族主要参与拟南芥抵抗生物和非生物胁迫过程,但是该亚族中 4个基因AtMYB44、AtMYB73、AtMYB77和AtMYB70功能并未完全明确。研究较为深入的是AtMYB44,目前已确定AtMYB44位于表达调控的上游区域,影响拟南芥的多种代谢过程。AtMYB44参与调控拟南芥对生物和非生物胁迫的响应及其生长、发育过程。在R2R3-MYB家族22亚族中,AtMYB77和AtMYB44相似程度最高(图 1),分别被称为 AtMYBR2和AtMYBR1,表明它们可能存在相同的功能。但是,目前仅发现AtMYB77在生长素刺激的根部发育过程中起调控作用。而AtMYB73的功能与AtMYB44较为相似,AtMYB73不仅参与拟南芥抵抗生物和非生物胁迫过程,在拟南芥的发育过程中也起着重要作用。AtMYB70的相关研究相对较少,目前仅知AtMYB70可能参与Ca2+信号的调控。事实上,22亚族4个基因在其介导的植物响应逆境胁迫过程中的具体功能及其相互关系尚不清楚,比如22亚族4个基因的上、下游结合因子仍未确定,4个基因在各种抗逆信号转导途径中的交叉调节的机制(冗余性和多样性)还有待进一步深入研究。随着各种基因功能研究方法的不断发展及酵母双杂交、染色质免疫沉淀等技术体系的应用,22亚族及R2R3-MYB转录因子所调控的植物重要生理生化活动的机制将会进一步得到阐明。
植物在生长发育过程中,经常受到各种环境(低温、干旱、土壤盐渍化及病原物侵染等)的胁迫。在长期的进化过程中,植物形成了抵抗逆境胁迫的多种防御机制。当受到逆境胁迫时,植物体内产生一系列的信号传递,激发植物自身的防御体系,提高抵抗胁迫的能力。植物对逆境胁迫的响应是一个涉及多基因、多信号途径、多基因产物的复杂过程。转录因子作为一种具有特殊结构的蛋白分子在植物抗逆过程中发挥重要的调控功能,它直接或间接的调控多个抗逆相关的功能基因的表达。阐明抗逆相关各转录因子的功能以及调控机制是目前植物胁迫生物学研究的热点领域,在提高作物对逆境胁迫的分子育种中,增加或改良某个关键转录因子的调控功能,可以激活或抑制多个功能基因的表达,从而提高植物的综合抗逆性(抗冻、抗旱、抗盐及抗病虫害等),为培育具有广谱、持久抗性的作物新品种具有重大意义,将在植物抗逆性改良方面拥有广阔的应用前景。
[1] Dubos C, Stracke R, Grotewold E, Weisshaar B, Martin C, Lepiniec L. MYB transcription factors in Arabidopsis. Trends Plant Sci, 2010, 15(10): 573-581.
[2] Bolton MD, Thomma BP, Nelson BD. Sclerotinia sclerotiorum (Lib.) de Bary: biology and molecular traits of a cosmopolitan pathogen. Mo Plant Pathol, 2006, 7(1): 1-16.
[3] Froidure S, Canonne J, Daniel X, Jauneau A, Brière C, Roby D, Rivas S. AtsPLA2-α nuclear relocalization by the Arabidopsis transcription factor AtMYB30 leads to repression of the plant defense response. Proc Natl Acad SciUSA, 2010, 107(34): 15281-15286.
[4] Seo PJ, Xiang F, Qiao M, Park JY, Lee YN, Kim SG, Lee YH, Park WJ, Park CM. The MYB96 transcription factor mediates abscisic acid signaling during drought stress response in Arabidopsis. Plant Physiol, 2009, 151(1): 275-289.
[5] Agarwal M, Hao Y, Kapoor A, Dong CH, Fujii H, Zheng X, Zhu JK. A R2R3 type MYB transcription factor is involved in the cold regulation of CBF genes and in acquired freezing tolerance. J Biol Chem, 2006, 281(49): 37636-37645.
[6] Mehrtens F, Kranz H, Bednarek P, Weisshaar B. The Arabidopsis transcription factor MYB12 is a flavonol-specific regulator of phenylpropanoid biosynthesis. Plant Physiol, 2005, 138(2): 1083-1096.
[7] Stracke R, Jahns O, Keck M, Tohge T, Niehaus K, Fernie AR, Weisshaar B. Analysis of PRODUCTION OF FLAVONOL GLYCOSIDES-dependent flavonol glycoside accumulation in Arabidopsis thaliana plants reveals MYB11-, MYB12-and MYB111-independent flavonol glycoside accumulation. New Phytol, 2010, 188(4): 985-1000.
[8] Bhargava A, Mansfield SD, Hall HC, Douglas CJ, Ellis BE. MYB75 functions in regulation of secondary cell wall formation in the Arabidopsis inflorescence stem. Plant Physiol, 2010, 154(3): 1428-1438.
[9] Böhner S, Gatz C. Characterisation of novel target promoters for the dexamethasone-inducible/tetracycline-repressible regulator TGV using luciferase and isopentenyl transferase as sensitive reporter genes. Mol Gen Genet, 2001, 264(6): 860-870.
[10] Zuluaga DL, Gonzali S, Loreti E, Pucciariello C, Degl'-Innocenti E, Guidi L, Alpi A, Perata P. Arabidopsis thaliana MYB75/PAP1 transcription factor induces anthocyanin production in transgenic tomato plants. Funct Plant Biol, 2008, 35(7): 606-618.
[11] Zhou JL, Lee C, Zhong RQ, Ye ZH. MYB58 and MYB63 are transcriptional activators of the lignin biosynthetic pathway during secondary cell wall formation in Arabidopsis. Plant Cell, 2009, 21(1): 248-266.
[12] Baumann K, Perez-Rodriguez M, Bradley D, Venail J, Bailey P, Jin H, Koes R, Roberts K, Martin C. Control of cell and petal morphogenesis by R2R3 MYB transcription factors. Development, 2007, 134(9): 1691-1701.
[13] Keller T, Abbott J, Moritz T, Doerner P. Arabidopsis REGULATOR OF AXILLARY MERISTEMS1 controls a leaf axil stem cell niche and modulates vegetative development. Plant Cell, 2006, 18(3): 598-611.
[14] Müller D, Schmitz G, Theres K. Blind homologous R2R3 Myb genes control the pattern of lateral meristem initiation in Arabidopsis. Plant Cell, 2006, 18(3): 586-597.
[15] Hong SH, Kim HJ, Ryu JS, Choi H, Jeong S, Shin J, Choi G, Nam HG. CRY1 inhibits COP1-mediated degradation of BIT1, a MYB transcription factor, to activate blue light-dependent gene expression in Arabidopsis. Plant J, 2008, 55(3): 361-371.
[16] Kirik V, Lee MM, Wester K, Herrmann U, Zheng ZG, Oppenheimer D, Schiefelbein J, Hulskamp M. Functional diversification of MYB23 and GL1 genes in trichome morphogenesis and initiation. Development, 2005, 132(7): 1477-1485.
[17] Kang YH, Kirik V, Hulskamp M, Nam KH, Hagely K, Lee MM, Schiefelbein J. The MYB23 gene provides a positive feedback loop for cell fate specification in the Arabidopsis root epidermis. Plant Cell, 2009, 21(4): 1080-1094.
[18] Lee MM, Schiefelbein J. WEREWOLF, a MYB-related protein in Arabidopsis, is a position-dependent regulator of epidermal cell patterning. Cell, 1999, 99(5): 473-483.
[19] Millar AA, Gubler F. The Arabidopsis GAMYB-like genes, MYB33 and MYB65, are microRNA-regulated genes that redundantly facilitate anther development. Plant Cell, 2005, 17(3): 705-721.
[20] Mandaokar A, Browse J. MYB108 acts together with MYB24 to regulate jasmonate-mediated stamen maturation in Arabidopsis. Plant Physiol, 2009, 149(2): 851-862.
[21] Mandaokar A, Thines B, Shin B, Markus Lange B, Choi G, Koo YJ, Yoo YJ, Choi YD, Choi G, Browse J. Transcriptional regulators of stamen development in Arabidopsis identified by transcriptional profiling. Plant J, 2006, 46(6): 984-1008.
[22] Van der Ent S, Verhagen BW, Van Doorn R, Bakker D, Verlaan MG, Pel MJ, Joosten RG, Proveniers MC, Van Loon L, Ton J, Pieterse CMJ. MYB72 is required in early signaling steps of rhizobacteria-induced systemic resistance in Arabidopsis. Plant Physiol, 2008, 146(3): 1293-1304.
[23] Segarra G, Van der Ent S, Trillas I, Pieterse CMJ. MYB72, a node of convergence in induced systemic resistance triggered by a fungal and a bacterial beneficial microbe. Plant Biol, 2009, 11(1): 90-96.
[24] Steiner-Lange S, Unte US, Eckstein L, Yang C, Wilson ZA, Schmelzer E, Dekker K, Saedler H. Disruption of Arabidopsis thaliana MYB26 results in male sterility due to non-dehiscent anthers. Plant J, 2003, 34(4): 519-528.
[25] Kamei A, Seki M, Umezawa T, Ishida J, Satou M, Akiyama K, ZHU JK, Shinozaki K. Analysis of gene expression profiles in Arabidopsis salt overly sensitive mutants sos2-1 and sos3-1. Plant Cell Environ, 2005, 28(10): 1267-1275.
[26] Kim JH, Nguyen NH, Jeong CY, Nguyen NT, Hong S-W, Lee H. Loss of the R2R3 MYB, AtMyb73, causes hyper-induction of the SOS1 and SOS3 genes in response to high salinity in Arabidopsis. J Plant Physiol, 2013, 170(16): 1461-1465.
[27] Jung C, Seo JS, Han SW, Koo YJ, Kim CH, Song SI, Nahm BH, Do Choi Y, Cheong JJ. Overexpression of At-MYB44 enhances stomatal closure to confer abiotic stress tolerance in transgenic Arabidopsis. Plant Physiol, 2008,146(2): 623-635.
[28] Oono Y, Seki M, Nanjo T, Narusaka M, Fujita M, Satoh R, Satou M, Sakurai T, Ishida J, Akiyama K, Iida K, Maruyama K, Satoh S, Yamaguchi-Shinozaki K, Shinozaki K. Monitoring expression profiles of Arabidopsis gene expression during rehydration process after dehydration using ca 7000 full-length cDNA microarray. Plant J, 2003, 34(6): 868-887.
[29] Fowler S, Thomashow MF. Arabidopsis transcriptome profiling indicates that multiple regulatory pathways are activated during cold acclimation in addition to the CBF cold response pathway. Plant Cell, 2002, 14(8): 1675-1690.
[30] Wenke K, Wanke D, Kilian J, Berendzen K, Harter K, Piechulla B. Volatiles of two growth-inhibiting rhizobacteria commonly engage AtWRKY18 function. Plant J, 2012, 70(3): 445-459.
[31] Zou BH, Jia ZH, Tian SM, Wang XM, Gou ZH, Lü BB, Dong HS. AtMYB44 positively modulates disease resistance to Pseudomonas syringae through the salicylic acid signalling pathway in Arabidopsis. Funct Plant Biol, 2013, 40(3): 304-313.
[32] Shim JS, Jung C, Lee S, Min K, Lee YW, Choi Y, Lee JS, Song JT, Kim JK, Choi YD. AtMYB44 regulates WRKY70 expression and modulates antagonistic interaction between salicylic acid and jasmonic acid signaling. Plant J, 2013, 73(3): 483-495.
[33] Jung C, Shim JS, Seo JS, Lee HY, Kim CH, Do Choi Y, Cheong JJ. Non-specific phytohormonal induction of AtMYB44 and suppression of jasmonate-responsive gene activation in Arabidopsis thaliana. Mol Cells, 2010, 29(1): 71-76.
[34] Liu RX, Chen L, Jia ZH, Lü BB, Shi HJ, Shao WL, Dong HS. Transcription factor AtMYB44 regulates induced expression of the ETHYLENE INSENSITIVE2 gene in Arabidopsis responding to a harpin protein. Mol Plant Microbe Interact, 2011, 24(3): 377-389.
[35] Lü BB, Li XJ, Sun WW, Li L, Gao R, Zhu Q, Tian SM, Fu MQ, Yu HL, Tang XM, Zhang CL, Dong HS. AtMYB44 regulates resistance to the green peach aphid and diamondback moth by activating EIN2-affected defences in Arabidopsis. Plant Biol, 2013, 15(5): 841-850.
[36] Pitzschke A, Djamei A, Teige M, Hirt H. VIP1 response elements mediate mitogen-activated protein kinase 3-induced stress gene expression. Proc Natl Acad Sci USA, 2009, 106(43): 18414-18419.
[37] Kasukabe Y, He LX, Nada K, Misawa SH, Ihara I, Tachibana S. Overexpression of spermidine synthase enhances tolerance to multiple environmental stresses and up-regulates the expression of various stress-regulated genes in transgenic Arabidopsis thaliana. Plant Cell Physiol, 2004, 45(6): 712-722.
[38] Jia J, Xing JH, Dong JG, Han JM, Liu JS. Functional analysis of MYB73 of Arabidopsis thaliana against Bipolaris oryzae. Agric Sci China, 2011, 10(5): 721-727.
[39] Shin R, Burch AY, Huppert KA, Tiwari SB, Murphy AS, Guilfoyle TJ, Schachtman DP. The Arabidopsis transcription factor MYB77 modulates auxin signal transduction. Plant Cell, 2007, 19(8): 2440-2453.
[40] Skinner DJ, Gasser CS. Expression-based discovery of candidate ovule development regulators through transcriptional profiling of ovule mutants. BMC Plant Biol, 2009, 9: 29.
[41] Ko JH, Han KH, Park S, Yang J. Plant body weight-induced secondary growth in Arabidopsis and its transcription phenotype revealed by whole-transcriptome profiling. Plant Physiol, 2004, 135(2): 1069-1083.
[42] Lee D, Polisensky DH, Braam J. Genome-wide identification of touch-and darkness-regulated Arabidopsis genes: a focus on calmodulin-like and XTH genes. New Phytol, 2005, 165(2): 429-444.
[43] Kim SY, Kim BH, Lim CJ, Lim CO, Nam KH. Constitutive activation of stress-inducible genes in a brassinosteroid-insensitive 1 (bri1) mutant results in higher tolerance to cold. Physiol Plant, 2010, 138(2): 191-204.
[44] Liu RX, Lü BB, Wang XM, Zhang CL, Zhang SP, Qian J, Chen L, Shi HJ, Dong HS. Thirty-seven transcription factor genes differentially respond to a harpin protein and affect resistance to the green peach aphid in Arabidopsis. J Biosciences, 2010, 35(3): 435-450.
[45] Kirik V, Kölle K, Miséra S, Bäumlein H. Two novel MYB homologues with changed expression in late embryogenesis-defective Arabidopsis mutants. Plant Mol Biol, 1998, 37(5): 819-827.
[46] Cheong YH, Chang HS, Gupta R, Wang X, Zhu T, Luan S. Transcriptional profiling reveals novel interactions between wounding, pathogen, abiotic stress, and hormonal responses in Arabidopsis. Plant Physiol, 2002, 129(2): 661-677.
[47] Guan Y, Nothnagel EA. Binding of arabinogalactan proteins by Yariv phenylglycoside triggers wound-like responses in Arabidopsis cell cultures. Plant Physiol, 2004, 135(3): 1346-1366.
[48] Ferreira FJ, Guo C, Coleman JR. Reduction of plastid-localized carbonic anhydrase activity results in reduced Arabidopsis seedling survivorship. Plant Physiol, 2008, 147(2): 585-594.
[49] Han Y, Mhamdi A, Chaouch S, Noctor G. Regulation of basal and oxidative stress-triggered jasmonic acid-related gene expression by glutathione. Plant Cell Environ, 2013, 36(6): 1135-1146.
[50] Schmid M, Davison TS, Henz SR, Pape UJ, Demar M, Vingron M, Schölkopf B, Weigel D, Lohmann JU. A gene expression map of Arabidopsis thaliana development. NatGenet, 2005, 37(5): 501-506.
[51] Lescot M, Déhais P, Thijs G, Marchal K, Moreau Y, Van de Peer Y, Rouzé P, Rombauts S. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences. Nucleic Acids Res, 2002, 30(1): 325-327.
[52] Geourjon C, Deleage G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments. Comput Appl Biosci, 1995, 11(6): 681-684.
(责任编委: 张宪省)
Structure and function of the 22nd subfamily in Arabidopsis R2R3-MYB family
Jintao Fan, Chenxi Jiang, Jihong Xing, Jingao Dong
Mycotoxin and Molecular Plant Pathology Laboratory, Agricultural University of Hebei, Baoding 071001, China
R2R3-MYB transcription factors of Arabidopsis play important roles in regulatory networks controlling development, metabolism and responses to biotic and abiotic stresses. R2R3-MYB transcription factors can be divided into 25 subfamilies based on the conserved amino acid sequences. In these subfamilies, the 22nd subfamily that responses to biotic and abiotic stresses includ AtMYB44, AtMYB77, AtMYB73 and AtMYB70. In this review, we summarize these 4 genes of the 22nd subfamily from three aspects, including the similarity of gene function, consistency of gene expression and conservation of the genetic structure. Then we discuss the redundancy and diversity about gene structure and function of these 4 genes.
Arabidopsis thaliana; R2R3-MYB transcription factors; 22 subfamily; structure and function
2014-05-16;
2014-08-04
国家自然科学基金项目(编号:31200203),河北省自然科学基金项目(编号:C2012204032)和高等学校博士学科点专项科研基金项目(编号:20121302120007)资助
樊锦涛,硕士研究生,专业方向:分子生物学。E-mail: afanjintao@126.com
邢继红,教授,研究方向:分子生物学。Tel: 0312-7528142;E-mail: xingjihong2000@126.com;
董金皋,教授,博士生导师,研究方向:植物分子病理学。Tel: 0312-7528266;E-mail: dongjingao@126.com
10.3724/SP.J.1005.2014.0985
时间: 2014-9-24 10:17:09
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140924.1017.001.html