产L-苹果酸重组大肠杆菌的构建
2014-05-04吴亚斌石贵阳
吴亚斌,张 梁,石贵阳
(江南大学 工业生物技术教育部重点实验室 粮食发酵工艺与技术国家工程实验室,无锡 214122)
L-苹果酸是生物体内三羧酸循环的重要一员,在生物体中具有重要的生理功能[1];同时也是天然果酸的重要组成部分,其酸味持久柔和,是优良的酸味剂。因为上述特性,L-苹果酸在医药、食品等多个行业中都有广泛用途[2]。合成单一手性的L-苹果酸主要方法有细胞/酶转化法和微生物发酵法[3],胡永红等[4]以富马酸钙为底物,采用游离延胡索酸酶,直接转化生成苹果酸钙,在40℃、pH 7.0~7.5的条件下,每升延胡索酸酶液能在20~28 h可将3.2 kg的富马酸钙转化生成苹果酸钙,转化率高达99.9%;周小燕等[5]使用L-苹果酸高产突变株曲霉N1-14'在5 L罐上发酵120 h,L-苹果酸产量达105.88 g/L,平均产生速率0.883 g/(L·h),糖酸转化率78.43%。
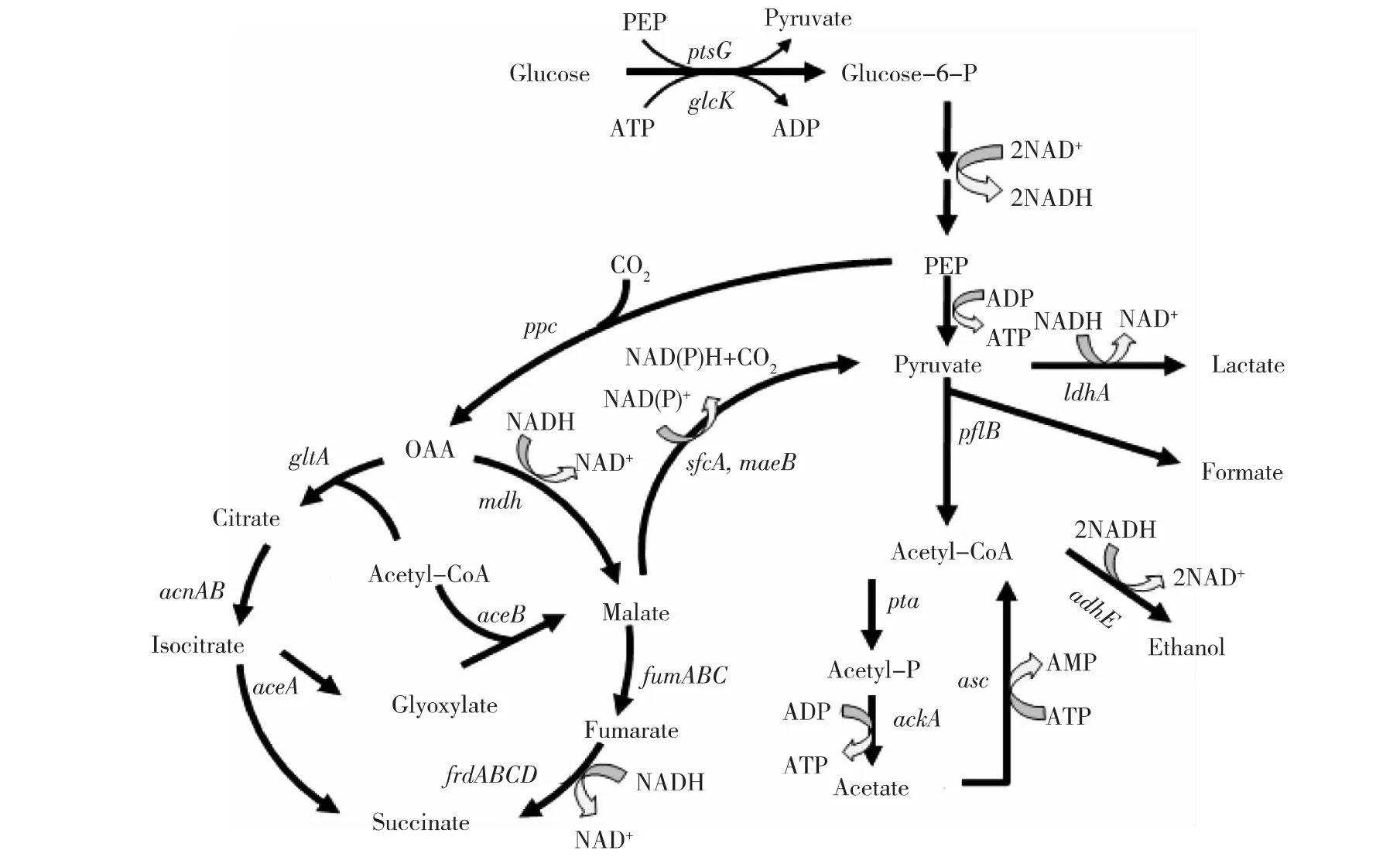
图1 野生大肠杆菌厌氧混合酸发酵途径Fig.1 Matabolic pathway of anaerobic mixed acid fermentation in wide-type E.coli
大肠杆菌遗传背景清楚、易于进行代谢调控,操作简便,在厌氧条件下进行混合酸发酵(图1),可发酵糖生成甲酸、乙酸、乳酸、琥珀酸和乙醇等产物。通过代谢工程和基因敲除技术对大肠杆菌代谢途径进行改造,可消除副产物的积累,提高目的产物的产量。如 Zhou等[6]和 Jantama等[7]利用基于FLP/FRT重组酶系统的基因敲除技术对大肠杆菌的代谢途径进行改造,获得生产不同物质的重组大肠杆菌,其产物包括乙醇、丙酮酸、乳酸和琥珀酸等。基于此研究思路,在大肠杆菌中构建L-苹果酸的合成途径,同时消除杂酸的影响,有可能获得较高的产量与转化率。
笔者以所在实验室前期构建的产琥珀酸重组大肠杆菌 E.coli B0013-1050[8](敲除乙酸激酶、磷酸乙酰转移酶基因ackA-pta、乳酸脱氢酶基因ldhA、丙酮酸甲酸裂解酶基因pflB、乙醇脱氢酶基因adhE和酶IICBGlc编码基因ptsG)为出发菌株,利用Red同源重组结合Xer/dif重组技术[9-10]继续敲除富马酸酶基因和苹果酸酶基因,构建1条葡萄糖-PEP-草酰乙酸-L-苹果酸的合成途径,以实现L-苹果酸在大肠杆菌中的积累。在此基础上,通过克隆表达来源于黄曲霉的苹果酸脱氢酶基因,强化合成途径,进一步提高L-苹果酸的转化率。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和质粒
重组菌株 E.coli 2010、E.coli 2020、E.coli 2030、E.coli 2040和重组菌E.coli B0013-1050由笔者所在实验室研究人员构建。
质粒:质粒pSK-difGm和同源重组的协助质粒pKD46、质粒pNpheA保藏于笔者所在实验室;重组 质粒 pMD18-T-fumB'::difGm、pMD18-T-fumC'::difGm、 pMD18-T-maeB'::difGm、pUC19-pNmdh、pBR322-pNmdh 和 pTH18-pNmdh、pMD18-T-maeB'::difGm-pNmdh均为笔者所在研究课题组研究人员构建。
1.1.2 引物
本研究课题设计的引物(表1)均交由上海生工生物工程公司代为合成。
1.1.3 工具酶和试剂
所用 DNA限制性内切酶 EcoRI、BamHI、SmaI、HindIII和 KpnI,Taq DNA 聚合酶,反转录试剂盒均购自Fermentas公司;T4 DNA连接酶、pMD18-T simple vector、pfu高保真DNA聚合酶均为宝生生物工程有限公司产品;小量质粒快速提取试剂盒、小量DNA片段纯化回收试剂盒和小量DNA片段胶回收试剂盒为北京博大泰克生物基因技术有限责任公司产品;L-阿拉伯糖购自上海生工生物工程公司;酵母提取物和胰蛋白胨为Oxoid公司产品;其他试剂药品均为国产分析纯。

表1 本研究中所用引物Table 1 Primers used in this research
1.1.4 培养基
LB培养基(g/L):胰蛋白胨10,酵母浸出物5,NaCl 10。配制固体培养基时加入15 g/L琼脂粉。
M9 培养基(g/L):Na2HPO4·12H2O 15.12,KH2PO43.0,NaCl 0.5,NH4Cl 1.0。使用时需按体积分数0.1%的量补加Mg2+(1 mol/L)和微量元素,并添加适量葡萄糖溶液作为C源。
微量元素(mmol/L):FeCl3·6H2O 8.88,CoCl2·6H2O 1.26,CuCl2·2H2O 0.88,ZnCl22.20,Na2MoO4·2H2O 1.24,H3BO31.21,MnCl2·4H2O 2.5。
抗生素:氨苄青霉素,终质量浓度100 μg/mL;庆大霉素,终质量浓度30 μg/mL;卡那霉素,终质量浓度 30 μg/mL。
1.2 方法
1.2.1 目标基因的敲除
利用Red同源重组结合Xer/dif重组技术依次敲除富马酸酶基因fumB和 fumC,苹果酸酶基因sfcA和maeB。参照文献[9]的基因敲除方法,依次构建制备在dif-Gm-dif片段左右带有目标基因上下游各200 bp左右同源臂的突变盒。将已经导入质粒pKD46的大肠杆菌目标菌株在含氨苄青霉素的LB培养基中培养,加入L-阿拉伯糖诱导使其产生Red重组酶,制备感受态细胞,将同源重组片段与感受态细胞按适当比例充分混合,电击转化并后培养1 h,涂布氨苄青霉素和庆大霉素的双抗平板,30℃培养,挑选阳性转化子。以验证引物对其进行验证。将验证结果正确的菌株30℃下进行传代,使细胞内Xer重组酶系统发挥作用自动识别dif位点,切除dif位点间的抗性基因,完成目的基因的敲除。
1.2.2 发酵及产物检测
摇瓶发酵:重组菌经平板活化,挑取单菌落接入LB培养基中,37℃、200 r/min培养10~12 h,离心收集菌体,加入适量M9重悬,转接M9培养基,葡萄糖初始质量浓度5 g/L,37℃、200 r/min培养12 h。补加葡萄糖溶液至糖质量浓度到30 g/L,以50 g/L的量加入CaCO3,塞上硅胶塞,转入厌氧发酵,37℃、200 r/min发酵36 h。
15 L罐发酵:重组菌的活化和转接与摇瓶发酵一致,M9中培养12 h后,作为发酵罐的种子液。发酵罐装液量为6 L,接种量为5%,发酵温度37℃,初始葡萄糖质量浓度5 g/L,通气量3 L/min,搅拌转速200 r/min,好氧培养使菌体生长,适时调节搅拌转速(200~500 r/min)使得溶解氧体积分数大于30%,以NH3H2O控制pH为7。有氧培养至菌体浓度达到最大,停止通气,搅拌速率降为100 r/min,进入厌氧转化阶段。按6 g/L加入NaHCO3,并以2.4 mol/L K2CO3和1.2 mol/L KOH溶液代替NH3H2O控制pH为7,37℃恒温厌氧发酵30 h。
产物检测:葡萄糖浓度使用生物传感分析仪(山东省科学院生物研究所)检测;有机酸产量由高效液相色谱测定仪,泵为Dionex P680,紫外检测器为Dionex UVD170U,色谱柱为反向 C18色谱柱Nucleosil 100-5 C18,液相检测条件参照文献[11]。
1.2.3 黄曲霉总RNA的提取及cDNA的制备
黄曲霉总RNA的提取方法参照文献[12],处理过后的总RNA按照反转录试剂盒的说明进行反转录PCR,得到cDNA,用于苹果酸脱氢酶基因mdh的PCR扩增。
1.2.4 黄曲霉苹果酸脱氢酶基因mdh在重组菌中的表达
以质粒pNpheA(含有组成型启动子PN25)和黄曲霉cDNA为模版,通过重叠PCR扩增得到带有启动子PN25的mdh基因,连接不同拷贝数载体质粒(pUC19、pBR322和pTH18),构建重组质粒pUC19-pNmdh、pBR322-pNmdh 和 pTH18-pNmdh,并导入重组菌中进行表达。
2 结果与讨论
2.1 基因敲除重组菌的构建结果
图2为基因sfcA、fumB、fumC和maeB敲除验证结果。扩增sfcA基因时,先后使用2对不同的上下游引物进行PCR,但未得到任何扩增产物,推测sfcA基因已被敲除,再设计引物sfcA1和sfcA2,将目的基因包括其上下游各300 bp左右一起扩增,但PCR结果只有约200 bp,之后以野生菌E.coli B0013染色体为模板,PCR结果与预期(2379 bp)相符合,验证结果见图2(a)。由图2(a)可知:sfcA基因已被敲除,原因可能是在前期菌株改造过程中,某个突变盒的同源臂与该基因有较高同源性,导致基因同时被敲除。
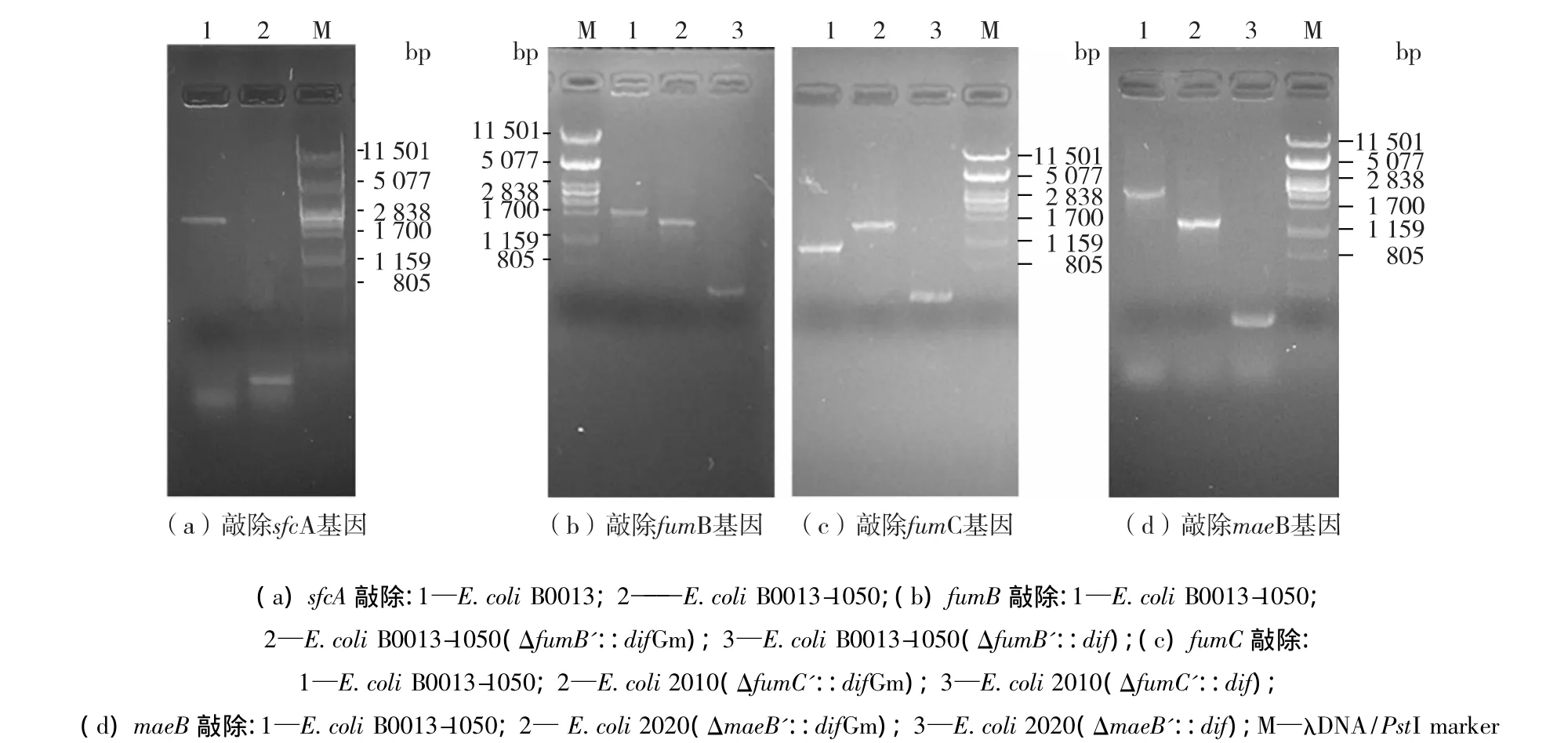
图2 基因敲除验证Fig.2 Identification of gene knockout
基因fumB、fumC和maeB的敲除验证见图2(c)~图2(d),使用相同引物对出发菌株和基因敲除菌株进行PCR,扩增产物的大小有明显差异,证明目标基因片段已从染色体上删除。对3株基因敲除重组菌分别命名为 E.coli 2010、E.coli 2020和E.coli 2030。对于fumA基因,考虑到其主要在有氧条件下作用[13],为保证菌体在有氧条件下的正常生长,本研究未对其进行敲除。
2.2 重组菌的生理特性与发酵实验结果
利用Red同源重组系统结合Xer/dif重组酶系统,成功构建1株富马酸酶基因(fumB和fumC)和苹果酸酶基因(sfcA和maeB)重叠缺失突变株E.coli 2030,将该重组菌分别于LB和M9培养基中培养,测定600 nm处吸光值A600,以E.coli B0013-1050为对照菌株,绘制生长曲线,结果见图3。由图3可知:所构建的重组菌的生长状况对比于出发菌株没有太大差别,表明敲除fumB、fumC和maeB基因并不会对菌体的好氧生长产生影响。
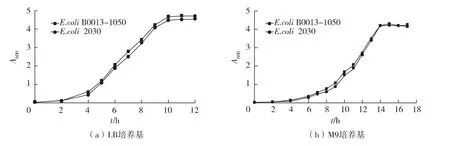
图3 E.coli B0013-1050及其突变株E.coli 2030的生长特性比较Fig.3 Aerobic growth comparison bewteen E.coli B0013-1050 and its mutant 2030 in LB medium and M9 medium supplemented with 5 g/L glucose
对重组菌E.coli 2010、2020和2030进行摇瓶发酵试验,以出发菌E.coli B0013-1050作为对照,发酵结果见表2。由表2可知:敲除基因fumB的重组菌E.coli 2010厌氧发酵的主要产物是丙酮酸和琥珀酸,和出发菌相比较,琥珀酸的产量下降,葡萄糖-琥珀酸转化率由出发菌的38.9%下降为26%。而继续敲除fumC基因的重组菌E.coli 2020的琥珀酸产量却只有微弱的下降,并未到达预期中完全消除琥珀酸积累的目的。
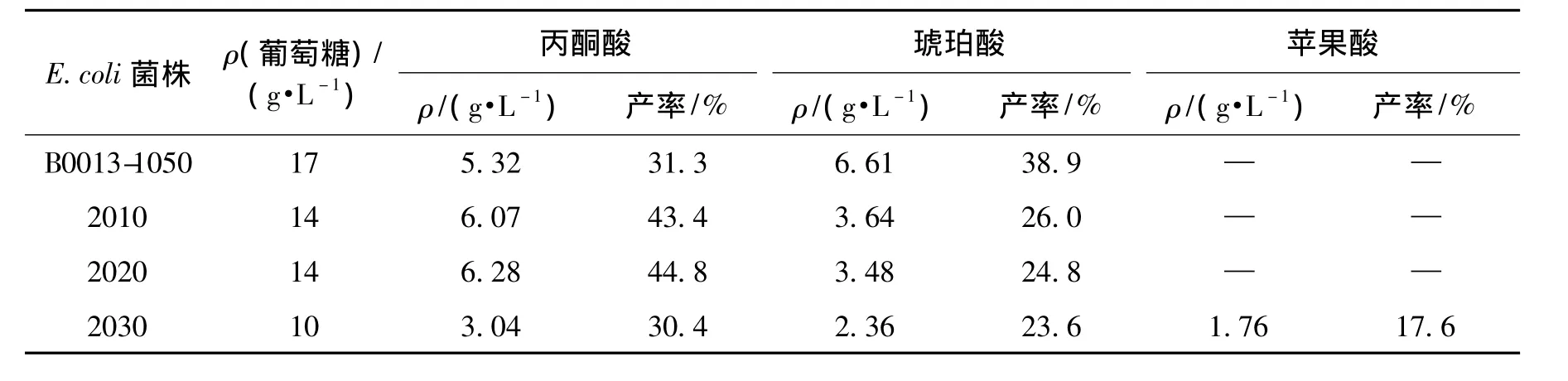
表2 基因敲除重组菌摇瓶发酵结果Table 2 Fermentation results ofrecombinant strains with gene knockout in shake flask
与之前2株重组菌相比,继续敲除苹果酸酶基因的重组菌E.coli 2030发酵产物中,琥珀酸的转化率变化不大,而丙酮酸的转化率下降,最终生成苹果酸1.76 g/L,葡萄糖-苹果酸转化率为17.6%。
重组菌2030在15 L罐中发酵,考察其产酸能力,发酵结果如图4所示。由图4可知:厌氧发酵30 h,耗糖24 g/L,最终产 L-苹果酸 12.5 g/L,葡萄糖-苹果酸转化率为 52.1%,生产强度 0.42 g/(L·h)。丙酮酸转化率下降到19.3%。发酵液中还检测到琥珀酸和少量的乳酸。
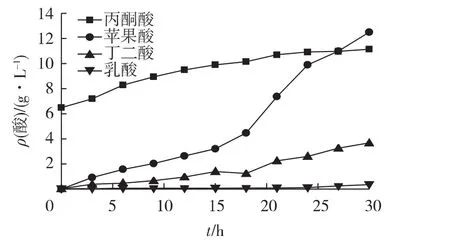
图4 重组大肠杆菌E.coli 2030在15 L发酵罐中发酵产酸情况Fig.4 Acid production of recombinantE.coli 2030 in a 15-L bioreactor
2.3 苹果酸脱氢酶基因mdh表达量对L-苹果酸产率的影响
苹果酸脱氢酶(malate dehydrogenase,MDH,EC 1.1.1.37)是笔者所构建的L-苹果酸合成途径中的一个关键酶,通过强化MDH的表达,可引导代谢流更多地走向L-苹果酸合成途径,提高目的产物的转化率。Peleg等[14]发现在黄曲霉中L-苹果酸的合成主要来自于草酰乙酸,苹果酸脱氢酶在L-苹果酸生产中起到了重要作用。本文此前构建的合成途径也是由草酰乙酸生成L-苹果酸,因此克隆了来源于黄曲霉的苹果酸脱氢酶基因mdh,连接至不同拷贝数载体,并在重组菌E.coli 2030中进行表达,考察其表达量对L-苹果酸产率的影响。
绘制含不同拷贝数质粒的重组菌在M9培养基中的生长曲线,结果见图5。由图5可知:在相同的培养条件下,携带高拷贝质粒的重组菌所能达到的菌体浓度明显小于携带中低拷贝数质粒的重组菌,这是因为更多的能量被用于蛋白质合成而影响了菌体的生长。其余2个重组菌的生长状况相似,对比于重组菌E.coli 2030,最终的菌体浓度也没有太大差别。这表明苹果酸脱氢酶的表达量应控制在中低水平,过高则会影响菌体的生长。
以菌株E.coli 2030作为对照,对含不同拷贝数质粒的重组菌进行摇瓶发酵试验,发酵结果见表3。由表3可知:厌氧发酵36 h后,相较于对照菌,L-苹果酸的产量和转化率都有不同程度的提高,并且随着拷贝数的降低,L-苹果酸的转化率也逐渐升高。上述结果表明,想要获得较好的强化作用,需要将苹果酸脱氢酶的表达量控制在较低的水平,而不是越高越好。

图5 携带不同拷贝数质粒的重组菌生长曲线Fig.5 Growth curves of recombinant strains harboring recombinant plasmids with different copy numbers

表3 含有不同拷贝数质粒的重组菌产L-苹果酸能力的比较Table 3 L-malic acid production comparison bewteen recombinant strains harboring recombinant plasmids with different copy numbers
2.4 重组菌E.coli 2040的构建及其发酵实验结果
为了实现苹果酸脱氢酶基因的稳定遗传并控制其表达量在较低水平,将mdh基因整合表达于重组菌中。根据之前的基因敲除方法,将同源重组突变盒maeB'::difGm连接至pMD18-T-maeB'::pNmdh,构建突变盒 maeB'::difGm-pNmdh,使用该突变盒在敲除maeB基因的同时,将基因mdh整合于重组大肠杆菌染色体上。基因整合的验证结果见图6。由图6可知,mdh已整合于重组菌染色体上,将该菌命名为E.coli 2040。
图7为E.coli 2040在15 L罐中厌氧发酵结果。由图7可知:厌氧发酵30 h,耗糖23.2 g/L,最终产L-苹果酸14 g/L,葡萄糖-苹果酸转化率为60.3%,生产强度0.47 g/(L·h)。
对于利用大肠杆菌直接发酵生产L-苹果酸,国内外学者已有一定的研究,Zhang等[15]以产琥珀酸重组大肠杆菌KJ073为出发菌,继续敲除富马酸酶fumAC和fumB,延胡索酸还原酶frdBC,苹果酸酶sfcA和maeB,得到菌株XZ658,在3 L罐中,经两段式发酵72 h,耗糖32.8 g/L,最终产苹果酸34 g/L,葡萄糖-苹果酸转化率为103.8%,生产强度0.47 g/(L·h),其他的杂酸只有少量的琥珀酸、乳酸和乙酸(1 g/L左右),是目前产L-苹果酸大肠杆菌中转化率最高的1株。但由于fumA基因的缺失,重组菌的生长受到影响,导致发酵周期的延长(有氧培养16 h,发酵周期72 h)。本文菌株拥有较好的生长性能,可为后期的厌氧转化提供较高的初始菌体浓度,并缩短整个发酵周期,在此基础上继续对菌株进行改造,提高苹果酸的产量及转化率,则可获得性能更为优良的菌株。
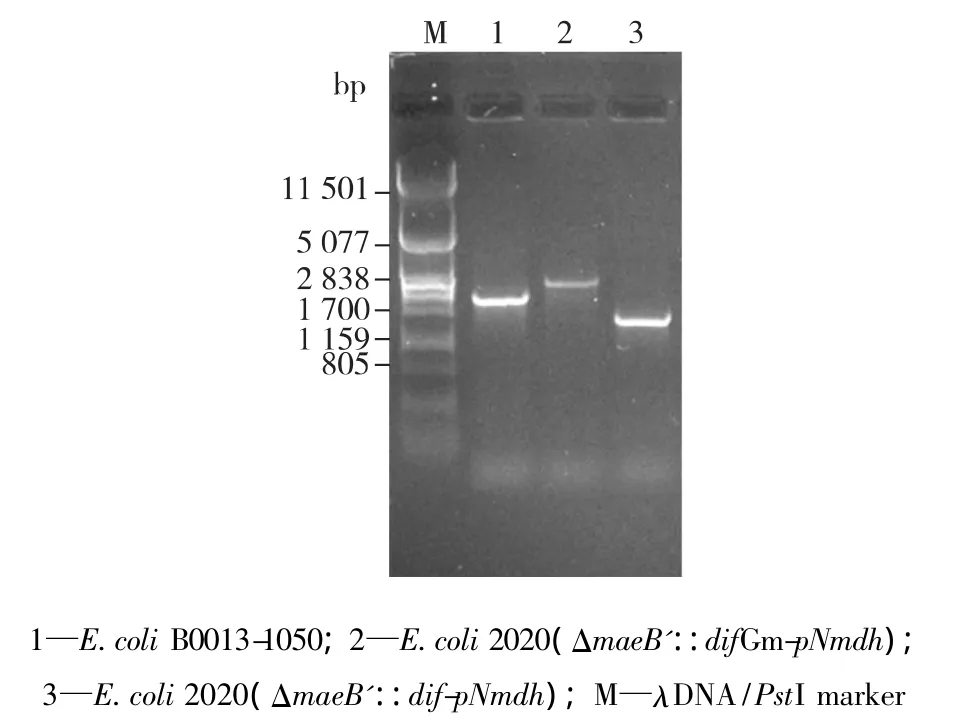
图6 mdh基因整合表达的验证Fig.6 Identification of genemadh integrant expression
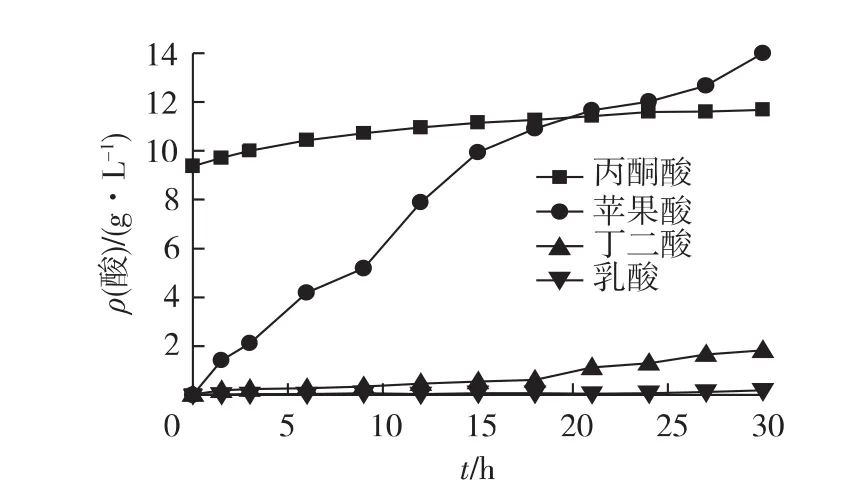
图7 重组大肠杆菌E.coli 2040在15 L发酵罐中发酵产酸情况Fig.7 Acid production of recombinantE.coli 2040 in a 15-L bioreactor
3 结论
基于重组大肠杆菌E.coli B0013-1050的琥珀酸生成途径,利用Red同源重组结合Xer/dif重组系统敲除富马酸酶基因fumB和fumC,苹果酸酶基因sfcA和maeB,成功构建L-苹果酸合成途径,经15 L罐发酵产L-苹果酸12.5 g/L,葡萄糖-苹果酸转化率为52.1%,主要杂酸为丙酮酸及琥珀酸;之后引入来源于黄曲霉的苹果酸脱氢酶,强化该途径,使L-苹果酸产量提高到14 g/L,葡萄糖-苹果酸转化率提高到60.3%,同时降低了丙酮酸及琥珀酸的转化率。
[1] 金其荣,张继民,徐勤.有机酸发酵工艺学[M].北京:中国轻工业出版社,1989.
[2] 汪多仁,陈体庆.L-苹果酸的开发与应用[J].精细化工原料及中间体,2003(9):22-25.
[3] 胡永红,欧阳平凯.苹果酸工艺学[M].北京:化学工业出版社,2009.
[4] 胡永红,欧阳平凯,沈树宝,等.反应分离耦合技术生产L-苹果酸工艺过程的优化研究[J].生物工程学报,2001,17(5):503-505.
[5] 周小燕,吴清平,蔡芷荷,等.曲霉N1-14'胞质酶活性与产L-苹果酸能力的关系[J].微生物学报,2000,40(5):500-505.
[6] Zhou S,Yomano L P,Shanmugam K T,et al.Fermentation of 10%(w/v)sugar to D(-)-lactate by engineered Escherichia coli B[J].Biotechnol Lett,2005,27(23/24):1891-1896.
[7] Jantama K,Zhang Xueli,Moore J C,et al.Eliminating side products and increasing succinate yields in engineered strains of EscherichiacoliC[J].BiotechnolBioeng,2008,101(5):881-893.
[8] 曹剑磊,周丽,张梁,等.产琥珀酸重组大肠杆菌的构建及发酵性能[J].应用与环境生物学报,2010,16(6):851-857.
[9] 周丽,牛丹丹,李宁,等.基于Red重组系统和Xer重组系统的大肠杆菌多基因删除方法[J].微生物学通报,2010,37(6):923-928.
[10] Bloor A E,Cranenburgh R M.An efficient method of selectable marker gene excision by Xer recombination for gene replacement in bacterial chromosomes[J].Appl Environ Microbiol,2006,72(4):2520-2525.
[11] 杨毅,李崎,陈蕴,等.反相高效液相色谱法(RP-HPLC)测定啤酒中有机酸[J].食品与发酵工业,2003,29(8):6-12.
[12] 淡明,黄海波,郭安平,等.一种简单高效的真菌总RNA提取方法[J].福建热作科技,2006,31(4):19-20.
[13] Woods S A,Schwartzbach S D,Guest J R.Two biochemically distinct classes of fumarase in Escherichia coli[J].Biochim Biophys Acta,1988,954:14-26.
[14] Peleg Y,Barak A,Scrutton M C,et al.Malic acid accumulation by Aspergillus flavus[J].Appl Microbiol Biotechnol,1989,30(2):176-183.
[15] Zhang X,Wang X,Shanmugam K T,et al.L-malate production by metabolically engineered Escherichia coli[J].Appl Environ Microbiol,2011,77(2):427-434.
